基因组发掘策略指导铁载体类化合物的发现
江志波,李星星,任卫聪,侍媛媛,高荣梅,李玉环,武临专,洪斌
基因组发掘策略指导铁载体类化合物的发现
江志波*,李星星*,任卫聪,侍媛媛,高荣梅,李玉环,武临专,洪斌
100050 北京,中国医学科学院北京协和医学院医药生物技术研究所/卫健委抗生素生物工程重点实验室
利用基因组发掘策略指导铁载体类化合物的发现。
首先,在获得灰产色链霉菌()CPCC 200274 全基因组序列的基础上,利用 antiSMASH 对其编码的潜在次级代谢产物生物合成基因簇进行生物信息学分析,通过 Blastp 比对,判断该菌株中是否存在铁阻遏蛋白(DmdR1)同源基因,并借助于 MEME、FIMO 等软件寻找包含 DmdR1 蛋白结合位点(iron box)的基因簇,则该基因簇可能为铁载体类化合物的生物合成基因簇;其次,摸索合适的发酵条件,利用实时定量 RT-PCR(qRT-PCR)技术检测相关生物合成基因的表达水平;最后分离纯化目标铁载体类化合物,验证此基因组发掘策略的可行性。
利用 antiSMASH 从灰产色链霉菌 CPCC 200274 基因组中发现了 5 个含有基因、标注为铁载体的生物合成基因簇。Blastp 比对提示该菌株基因组中存在 DmdR1 的同源蛋白 SGC5642,可能参与铁载体类化合物生物合成的调控。结合 MEME、FIMO 软件分析结果,5 个含的基因簇中只有 cluster 29 含有 iron box 序列,推测 cluster 29 可能编码铁载体类化合物。根据以上信息,利用 qRT-PCR 方法确定了该基因簇表达的发酵条件,分离纯化获得了 3 个铁载体类化合物,经结构鉴定分别为去铁敏 B,去铁敏 E和 N1-羟基-N1-琥珀酰基尸胺。
以基因组发掘策略为指导,从微生物基因组中发现铁载体类化合物生物合成基因簇和铁阻遏蛋白结合位点,并通过 qRT-PCR 方法在转录水平确定目标基因簇的方法,成功获得 3 个铁载体类化合物。该策略为解决目前 antiSMASH 对于铁载体类化合物生物合成基因簇预测的局限性和该类化合物基因簇的快速定位、新生物合成元件的发掘提供了新思路,为新结构铁载体类化合物的寻找及合成生物学改造奠定了基础。
基因组发掘; 铁载体; 灰产色链霉菌; qRT-PCR
铁是维持微生物生命活动的重要元素,作为辅因子参与催化多种生化反应,如氧化作用和电子转移过程等,另外还参与 DNA 和 RNA 的生物合成[1]。由于自然环境中可溶性铁离子浓度低,微生物通过合成一类对铁离子具有强螯合作用(aff> 1030)的有机小分子化合物(分子量通常为 500 ~ 2000 D)来摄取环境中的低浓度铁离子,这类物质被称为铁载体[2]。在细菌细胞膜上,存在铁载体转运蛋白,如 TonB 等[3]。研究表明,铁载体可以作为抗生素分子进入胞内的载体,在抗菌药物研发中起到了重要作用[4]。将抗生素分子偶联到铁载体上,形成的一类新型分子称为“木马分子”,能够克服细胞的外膜屏障[5]。根据木马策略设计的化合物 Bal30072 对碳青霉烯耐药的肠杆菌等多种革兰阴性菌具有明显的生长抑制活性,已经完成I期临床研究[6]。铁载体化合物去铁敏 B(deferoxamine B,DFOB)在临床还被用于治疗因代谢紊乱等因素引起的铁离子中毒[7]。鉴于铁载体的多种生物学功能和潜在应用价值,从自然界寻找新结构铁载体成为目前天然产物发现的重要方向之一。
随着基因组技术和天然产物生物合成机制研究的不断深入,大量抗生素等次级代谢产物的生物合成基因簇被报道。根据已报道生物合成基因(簇)来预测潜在的次级代谢产物结构和性质的生物信息学分析技术得到快速发展,这一技术被称为基因组发掘技术[8]。AntiSMASH(https:// antismash.secondarymetabolites.org/)是目前综合性最强的一个分析工具,整合了许多算法和软件,能分析识别微生物基因组中的次级代谢产物生物合成基因簇,被广泛应用于新天然产物的发现。放线菌(特别是链霉菌)是已知抗生素如链霉素、红霉素、万古霉素等的主要来源。自 2001 年第一个放线菌(天蓝色链霉菌)的基因组序列被报道以来,研究者发现放线菌基因组中往往存在十几个甚至几十个结构多样的次级代谢产物生物合成基因簇,数量远远大于从该菌株中已经分离得到的天然产物。这些次级代谢产物大多数在实验室条件下难以表达,被称为“沉默”或“隐性”基因簇[9],其中包括铁载体类化合物,因此需要结合生物信息学分析,采用有效的激活方法,针对性地指导铁载体类化合物的分离。
目前已发现几百个铁载体类化合物,对其生物合成机制也已进行了深入研究[10]。一类是由非核糖体肽合成酶(nonribosomal peptide synthetases,NRPSs)家族的多酶复合体负责合成的铁载体(NRPS-dependent siderophore),但 antiSMASH对于此类型生物合成基因簇不能直接给出铁载体的预测,需要研究者根据可能产物的结构特点来推测。另一类是以不依赖于 NRPS 的方式(NRPS-independent siderophore,NIS)来合成,由一类特殊合成酶家族(IucA_C)参与,在铁载体 aerobactin 生物合成过程中负责将6-羟基-6-乙酰基赖氨酸与柠檬酸脱水缩合[11],antiSMASH 对于含有同源基因的基因簇都预测为铁载体的生物合成基因簇,但这些预测的基因簇并不一定都能编码铁载体类化合物。
铁载体生物合成基因的转录表达受到一类阻遏蛋白(天蓝色链霉菌中为 DmdR1)的调控,且该调控作用与铁离子浓度密切相关[12]。当细胞内铁离子浓度足够高时,DmdR1 蛋白识别铁载体生物合成基因簇中的结合位点(iron box)序列并与之结合,抑制其生物合成基因的表达,而在贫铁条件下阻遏解除,细胞合成铁载体以摄取环境中的铁,这是细胞维持内稳态的一种调控机制[13]。由于 antiSMASH 对于铁载体生物合成基因簇预测的局限性,本文依据铁载体合成的调控机制,提出了一个新的发掘策略。本文以灰产色链霉菌()CPCC 200274 为研究对象,生物信息学分析结果表明,cluster 29 与去铁敏的生物合成基因具有同源性,且在其基因簇上含有阻遏蛋白的“iron box”。利用 qRT-PCR 方法检测相关基因的表达量确定了该基因簇表达的发酵条件,借助于色谱联用技术(HPLC-MS 等)和“闪式”分离技术从发酵液中获得了 3 个铁载体类化合物,说明此基因组发掘策略适用于铁载体类化合物生物合成基因簇的快速定位和分离。
1 材料与方法
1.1 材料
1.1.1 菌株 灰产色链霉菌()CPCC 200274 由中国药用微生物菌种保藏管理中心提供。
1.1.2 试剂和耗材 Trizol 试剂购自美国 Life Tech 公司;TransScript First-Strand cDNA Synthesis SuperMix 试剂盒购自北京全式金生物技术有限公司;Fast Start Universal SYBR Green Master Rox试剂盒购自瑞士 Roche 公司;色谱甲醇、乙腈购自上海泰坦科技股份有限公司;纯水为屈臣氏蒸馏水经 Millipore 纯化仪制备;丙酮、甲酸、三氯化铁等为分析级,购自北京化工厂。分析和半制备色谱柱[CAPCELL PAK C18 AQ,4.6(或 10.0)× 250 mm,5.0 μm]购自日本资生堂公司。
1.1.3 仪器 CFX 96TM实时荧光定量检测系统为美国 Bio-Red 公司产品;Sepacore®flash systems X10 中压制备液相色谱系统为瑞士BuChi 公司产品;LC-20AD 高压液相分析半制备系统为日本岛津公司产品;1290-1956 单四级杆质谱仪为美国 Agilent 公司产品;600 MHz 核磁共振仪为美国Varian 公司产品。
1.2 方法
1.2.1 链霉菌基因组 DNA 提取及序列测定 将CPCC 200274 接种于 5 ml TSB(BD Cat.211825)培养基中,28 ℃、220 r/min 振荡培养 48 h,5000 r/min 离心 10 min,收集10 ml 菌体,并用 STE 缓冲液(0.3 mol/L 蔗糖、25 mmol/L Tris-HCl、10 mmol/L EDTA,pH 8.0)洗涤菌体一次。用 5 ml STE 缓冲液重悬菌体,加入终浓度为 5 mg/ml 的溶菌酶,在 37 ℃恒温水浴中孵育 30 min。然后加入 2.5 ml 2% SDS,轻轻混匀后,加入等体积的酚:氯仿:异戊醇(25:24:1)混合物,在室温下轻轻摇匀,12 000 r/min,4 ℃离心 10 min,将上清液转移到另一离心管中。重复抽提 1 ~ 2 次,直至界面没有白色变性蛋白的存在。加入等体积的氯仿抽提一次以除去残留的酚,5000 r/min,4 ℃离心 10 min。再次将上清液转移到另一离心管中,加 RNA 酶(15 μg/ml)消化 1 h后加入等体积的酚:氯仿:异戊醇抽提一次,用等体积的氯仿抽提一次以除去残留的酚,加入1/20 体积的 5 mol/L NaCl 和等体积异丙醇,充分混匀至 DNA 沉淀。用洁净无菌的玻璃棒缠绕 DNA,将其移至新的离心管中,70% 的乙醇洗涤1 ~ 2 次,室温晾干后溶于 500 μl 纯水,保存于4 ℃ 备用。经电泳检测合格后,送深圳华大基因股份有限公司进行全基因组序列测定。
1.2.2 生物信息学分析 采用 AntiSMASH (Version 4.2.0,https://antismash.secondarymetabolites. org/)预测菌株次级代谢产物生物合成基因簇;Blastp(https://blast.ncbi.nlm.nih.gov/Blast.cgi)比对 DmdR1 的同源蛋白;Clustal Omega(https://www. ebi.ac.uk/Tools/msa/clustalo/)进行 DmdR1 同源蛋白的多序列比对。MEME(Version 4.12.0,http:// meme-suite.org/tools/meme)根据文献报道 DmdR1 的结合位点,生成“iron box”的一致性序列。根据 MEME 生成的一致性序列,以 FIMO(Version 4.11.1,http://meme-suite.org/tools/ fimo)寻找目标基因组中“iron box”的相似序列。
1.2.3 菌株活化、培养与发酵条件 菌株在 YMS 固体培养基(0.4% 酵母抽提物、1% 麦芽膏、0.4% 可溶性淀粉、0.0005% CoCl2•6H2O、2% 琼脂)中28 ℃培养 7 d 后,取约 2.0 cm2大小的带菌琼脂块转接到 100 ml 种子培养基 TSB(BD Cat.211825)中,于 28 ℃、200 r/min 振荡培养48 h 后,按 5%(V/V)的接种量转接到发酵培养基(贫铁:2.0% 可溶性淀粉、0.05% NaCl、0.1% KNO3、0.05% K2HPO4、0.05% MgSO4,pH 7.4 ~ 7.6;富铁:在贫铁发酵培养基中加入终浓度 0.01 mol/L FeCl3)中,相同条件继续培养 7 d。
1.2.4 RNA 提取及实时定量qRT-PCR 在菌株发酵 48 h 时,收取 2 ml 发酵液,12 000 r/min 离心 5 min,弃上清,菌体立即冻于液氮中。提取 RNA 时,将菌体于冰上解冻,重悬于 250 μl TE 溶液 中(含 10 mg/ml溶菌酶),37 ℃孵育 30 min。加1 ml Trizol 试剂并按说明书操作步骤分离并纯化 RNA。然后采用 TransScript First-Strand cDNA Synthesis SuperMix 按说明书操作步骤将 mRNA 逆转录合成第一链 cDNA。采用Fast Start Universal SYBR Green Master Rox 进行实时定量 qRT-PCR,qPCR 反应体系包括逆转录合成的 cDNA 模板 2.5 μl、待考察基因引物 400 nmol/L、SYBR Green Rox Mix 12.5 μl 和 DEPC 水5 μl。反应条件如下:94 ℃ 10 min;94 ℃30 s;60 ℃30 s;72 ℃30 s(40 个循环数)。扩增作为内参。采用 2-ddCt法计算各基因表达变化倍数。引物序列见表1。

表 1 qRT-PCR 所用引物
1.2.5 HPLC-MS 分析方法 发酵液经 3000 r/min离心 30 min,除去菌体和不溶物,取 10 ml 上清液进行固相萃取,其中 60% 甲醇水洗脱液经0.22 μm 滤膜过滤后,用于 HPLC-MS 检测。
液相色谱采用两相梯度洗脱的方法,其中 A 相为甲醇,B 相为 0.1%(V/V)甲酸水溶液。A 相在 40 min 内由 10%(V/V)逐渐递增至 70%(V/V),流速为 1.0 ml/min,洗脱液经过色谱柱后,使用三通等分至二极管阵列检测器和质谱检测器中。检测波长为 435 nm,柱温箱设定为 25 ℃,进样体积为 20 μl。
质谱检测条件:喷雾电压 4.5 V;载气为氮气,流速为 10 L/min;离子源温度 325 ℃;全扫描模式,范围为300 ~ 1200;数据使用仪器自带软件(Data Analysis, version 3.3)进行处理。
1.2.6 化合物分离纯化和制备过程 贫铁发酵上清液(6.0 L)经大孔树脂(XAD-2,6.0 L)吸附后,依次以 3%、10%、30%、50% 丙酮水溶液洗脱。HPLC-MS 检测铁载体集中在 30% 丙酮水溶液部分,减压浓缩 30% 丙酮水溶液,得粗品 1003.5 mg。经过中压快速分离系统(ODS 反相硅胶为载体)10% ~ 90% 甲醇水溶液(70 min)梯度洗脱,收集 20% ~ 30% 甲醇水溶液,减压浓缩后得混合物 100.5 mg。混合物经过半制备高压液相系统分离获得化合物1(R= 15.5 min,25.5 mg),2(R=17.5 min,5.3 mg)和3(R= 5.5 min,5.2 mg)。
取等量化合物1(DFOB,5.0 mg)于两支洁净试管中,分别加入 FeCl3(0.1 mol/L,10 μl)或Ga2(SO4)3(0.1 mol/L,10 μl)水溶液,室温下振荡反应 0.5 h 后,冷冻干燥除去溶剂,得 DFOB-Fe(5.1 mg)和 DFOB-Ga(4.9 mg)。
1.2.7 化合物抗病毒活性检测方法 MDCK 细胞以 2.5 × 104/孔的密度接种于 96 孔培养板,置 5% CO2细胞培养箱,37 ℃培养。24 h 后加入不同浓度待测化合物,同时设置细胞对照组。继续培养 48 h,观察细胞病变,以 Reed & Muench 法计算半数细胞毒浓度(TC50)。同样密度接种细胞,24 h 后用 100 倍半数组织细胞感染量(TCID50)的病毒(流感病毒 A/汉防/359/95,H3N2)感染2 h 后,移去未吸附的病毒。加入不同稀释度样品及阳性对照药(利巴韦林和磷酸奥司他韦),同时设细胞对照组和病毒对照组。待病毒对照组病变达 4+(75% ~ 100%)时观察各给药组细胞病变,用 Reed & Muench 法计算样品半数抑制浓度(IC50),并根据 TC50/IC50比值计算选择指数(SI)。
2 结果
2.1 生物信息学分析
对灰产色链霉菌()CPCC 200274进行了基因组 DNA 提取及高通量测序,获得了基因组精细图,其基因组为一个 10 397 305 bp的线性染色体,GC 含量为 70.82%,含有 9790 个基因,与 GenBank 中ATCC 14511 的完整基因组大小、基因数量相当(10 764 674 bp,9840 个基因),核苷酸序列一致性为 99%。AntiSMASH(Version 4.2.0)分析显示CPCC 200274 基因组中至少含有 47 个不同类型的次级代谢产物生物合成基因簇,包括非核糖体肽类(NRPS)、聚酮类(polyketides,PKS)、萜类(terpene)等,其中有5 个基因簇含有基因_,为铁载体类化合物生物合成基因簇(图 1),而ATCC 14511 的基因组中同样预测有 5 个基因簇含有基因。
文献报道天蓝色链霉菌中铁载体生物合成基因的转录表达受到阻遏蛋白 DmdR1 的调控[12],我们首先分析了 DmdR1 在放线菌中的保守性,发现在随机选择的 15 株放线菌基因组中(链霉菌属 11 株、游动放线菌属 2 株、拟无枝酸菌属 1 株、糖多孢菌属 1 株)都存在至少一个铁阻遏蛋白 DmdR1 的同源蛋白(E 值 < e-80),提示该蛋白在放线菌中保守性较高(代表性菌株比对结果如图 2 所示)。在CPCC 200274中,SGC5642 与 DmdR1 的氨基酸序列在全长范围内一致性为 97%(224/230)(图 2),提示 SGC5642 可能参与铁载体表达的调控。DmdR1 结合位点(iron box)保守性较高,在天蓝色链霉菌中目前已知的 iron box 位点包括以下 7 个[12, 14-15]:desAp-SCO2782(ttaggttaggctcacctaa)、desEp-SCO2780(tgaggttaggctaacctac)、cchAp-SCO0499(ttagcttagg ctcacctaa)、cchEp-SCO0495(ctcgattaggctcgcctta)、cchFp-SCO0494(tgagggtaggcttacctca)、cchJp-SCO0490(ttaggttaggctcgcctaa)和 cdtCp-SCO7400(ttaggtta ggctaaccttc),我们利用 MEME 软件将已知的 iron box 生成一致性序列,结果如图 3 所示,是包含G的回文序列。利用 FIMO 软件分析灰产色链霉菌基因组,结果显示在预测到的所有 47 个次级代谢产物生物合成基因簇中只有 cluster 29 基因簇中含有两个 iron box 位点(图 4),分别位于 cluster-29-C 和 cluster-29-E 的启动子区,说明此基因簇可能为铁载体的生物合成基因簇,其表达水平可能受铁离子浓度的调控。
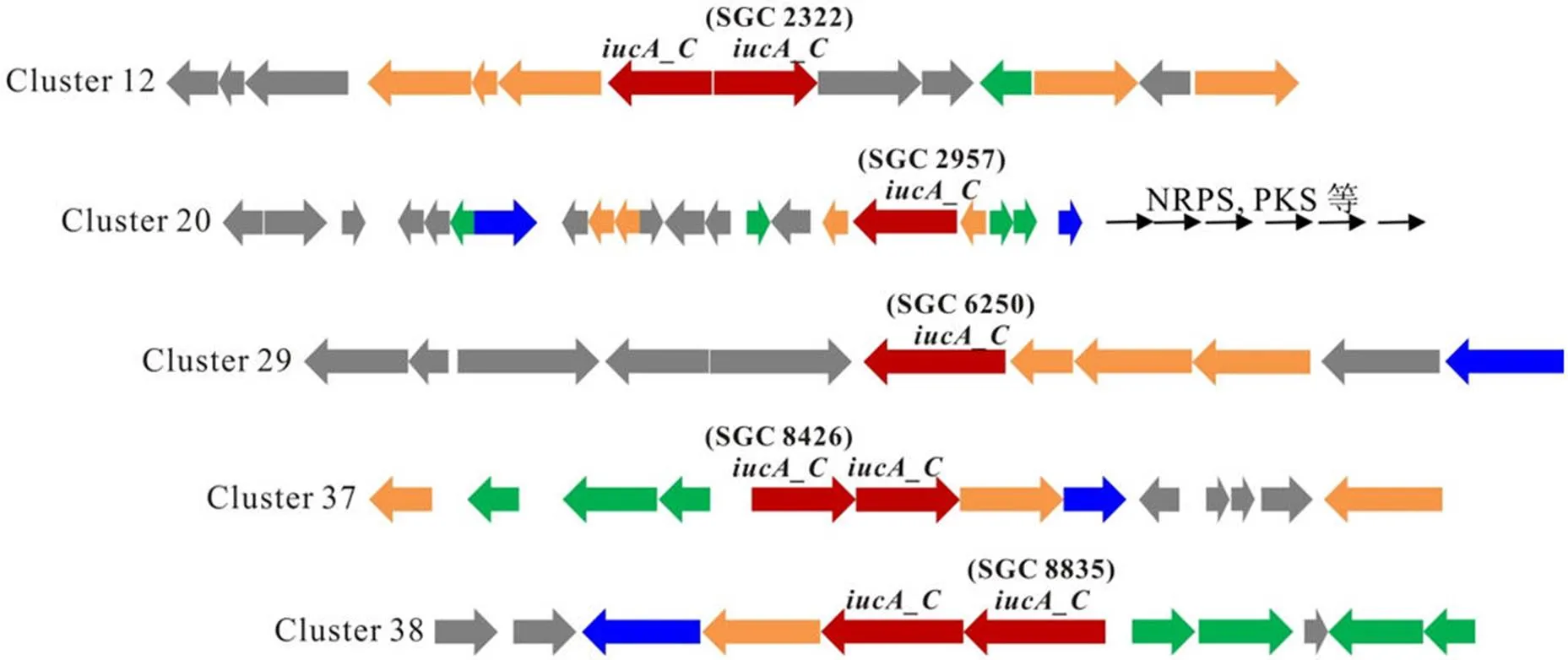
图 1 灰产色链霉菌 CPCC 200274 中预测的铁载体类化合物生物合成基因簇[红色代表iucA_C基因,黄色代表其他生物合成基因,绿色代表调控基因,蓝色代表转运基因,灰色代表其他未知功能基因。由于cluster 20 超过 180 kb,用黑色箭头(未按比例)表示相邻的 NRPS 和 PKS 等基因]
Figure 1 Clusters related to the biosynthesis of siderophores in the genome ofCPCC 200274 (Red arrows stand for_, yellow arrows for other synthetic genes, green arrows for regulatory genes, blue arrows for transporter genes, gray arrows for other genes. As the size of cluster 20 is over 180 kb, genes for adjacent NRPS and PKS are represented by unscaled black arrows)

图 2 8 株放线菌中铁阻遏蛋白 DmdR1 的同源比对
Figure 2 Homology comparison of iron dependent repressor DmdR1 in 8 strains of actinomycetes
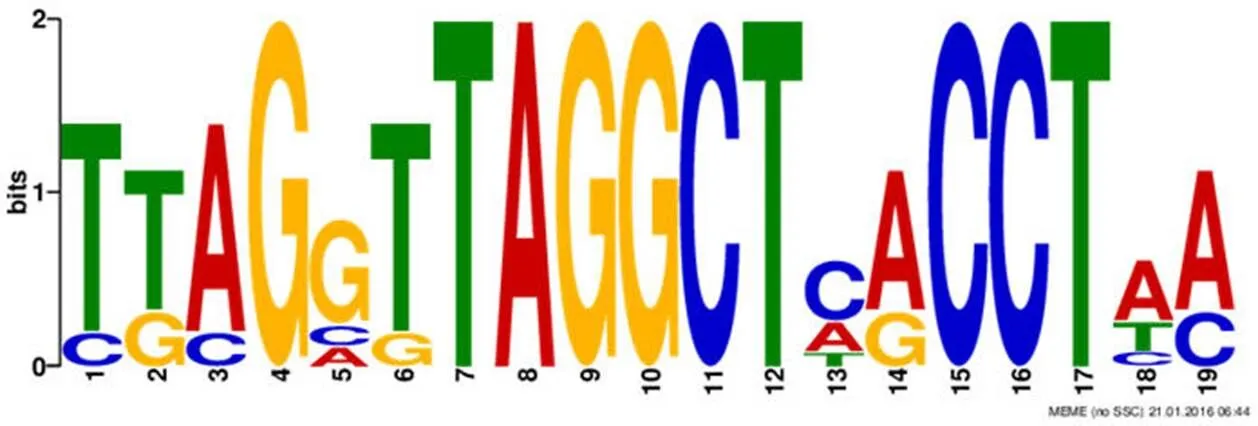
图 3 MEME 软件分析得到 iron box 的一致性序列
Figure 3 MEME analysis for the consensus sequence of iron box
将 cluster 29 与天蓝色链霉菌(M145)中负责去铁敏生物合成的基因簇[16]进行比较,发现该基因簇含有与去铁敏类铁载体关键合成基因~高度同源的基因(图 4),可能编 码去铁敏类的铁载体。Cluster 29 中基因的大小、预测的功能、同源基因以及氨基酸序列相似性见 表 2。
2.2 qRT-PCR 考察预测的基因簇转录水平
根据以上生物信息学分析的结果提示,我们设计了贫铁(< 10-3mol/L)和富铁(0.01 mol/L)两种条件对实验菌株分别进行发酵,提取 RNA,采用 qRT-PCR 对 antiSMASH 预测的 5 个可能的铁载体类基因簇中关键合成基因_的转录水平进行考察,结果如图 5 所示,只有 cluster 29在贫铁条件下被激活,而其余 4 个基因簇的表达不受铁离子浓度的调控,与生物信息学预测的结果一致。
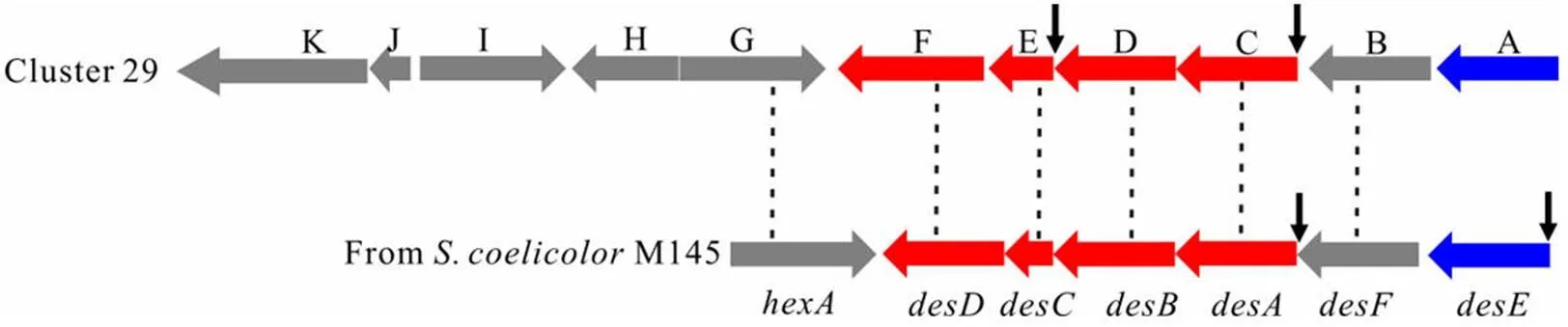
图 4 Cluster 29 与去铁敏生物合成基因簇的比较[黑色短箭头表示 DmdR1 结合序列(iron box)的位置]
Figure 4 Organization of the genes cluster 29 comparing togene cluster inr M145 [The vertical arrows indicate the location of DmdR1 binding sites (iron box)]
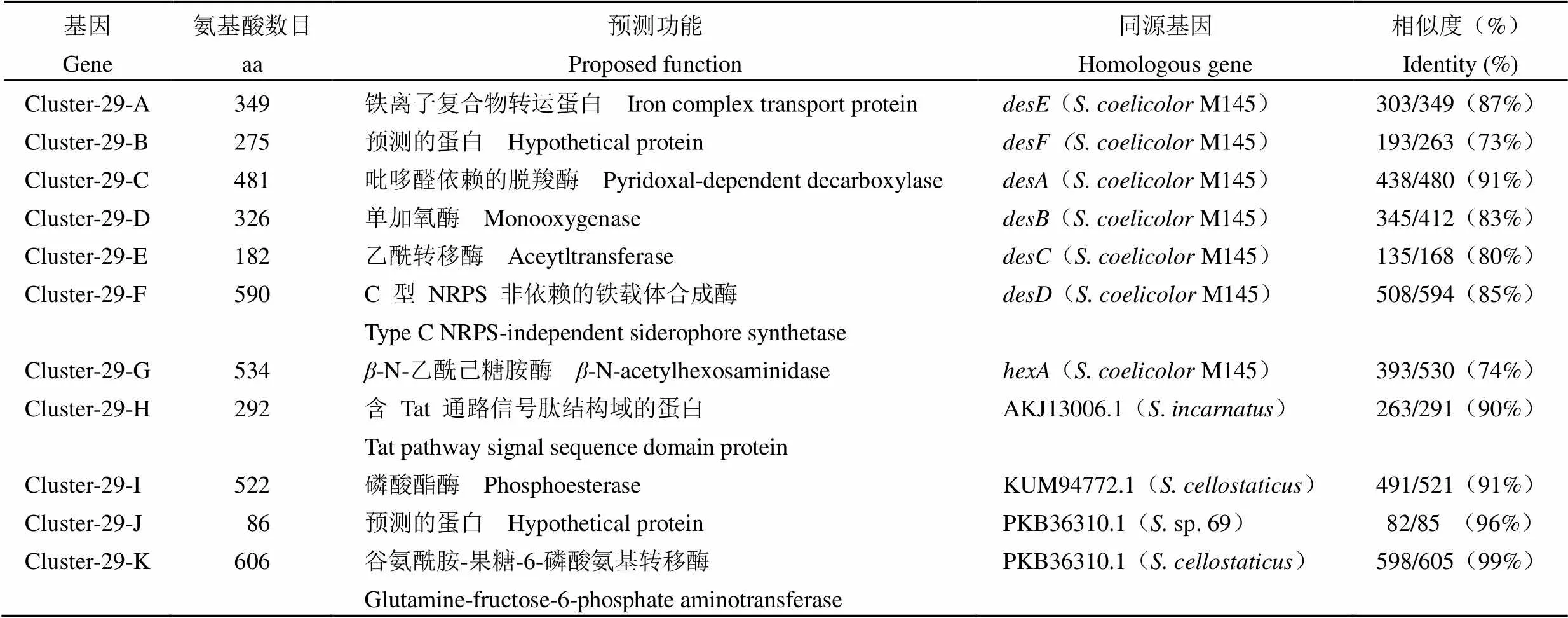
表 2 Cluster 29中基因的功能及与同源基因的相似度
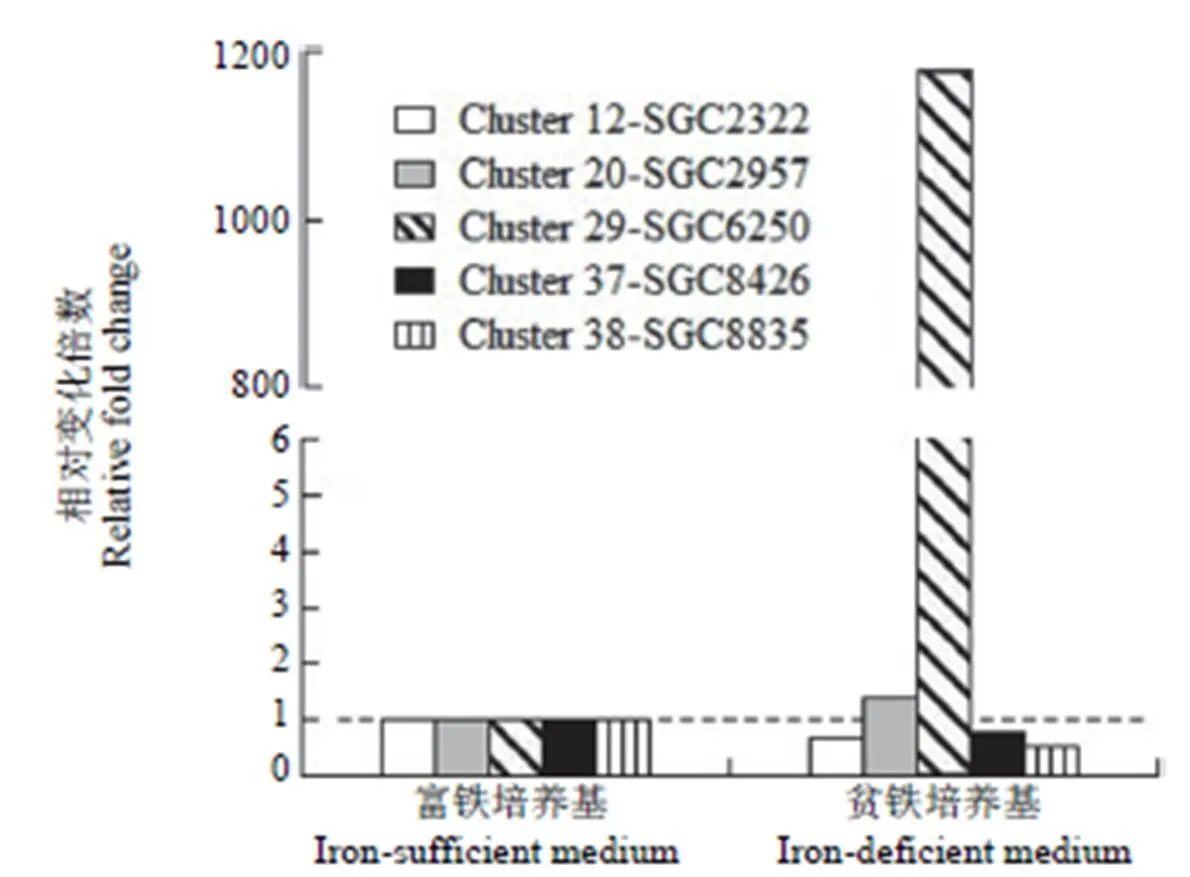
图 5 基因簇 cluster 12、20、29、37 和 38 中的关键合成基因在富铁和贫铁两种培养条件下的转录水平
Figure 5 The transcriptional levels of the key synthetic genes in cluster 12, 20, 29, 37 and 38 under iron-sufficient and iron-deficient conditions
2.3 铁载体类化合物的分离纯化和结构鉴定
根据铁载体与 Fe3+络合后复合物在波长435 nm下存在特定吸收的特点,定性检测发酵产物中铁载体类化合物,结果如图 6 所示。在富铁培养基发酵产物中,由于基因簇被沉默,未检测到特定的铁载体类化合物产生(图 6A);贫铁的发酵液中,由于缺乏Fe3+,激活基因簇表达的游离铁载体在 435 nm 下吸收较弱(保留时间R= 12 min 处的峰不是铁载体类化合物)(图 6B);在贫铁发酵液中加入一定量 FeCl3(0.01 mol/L),使游离的铁载体与 Fe3+形成络合物,分别在 19.7 min(峰 1)、21.5 min(峰2)和 22.1 min(峰3)增加了 3 个具有特征吸收的峰(图 6C)。说明在贫铁条件下表达 3 个铁载体类化合物,能与铁离子特异性结合。借助于中压制备和 HPLC 纯化获得了峰 1、2 和 3 对应的化合物1、2和3,结构如图 7 所示。
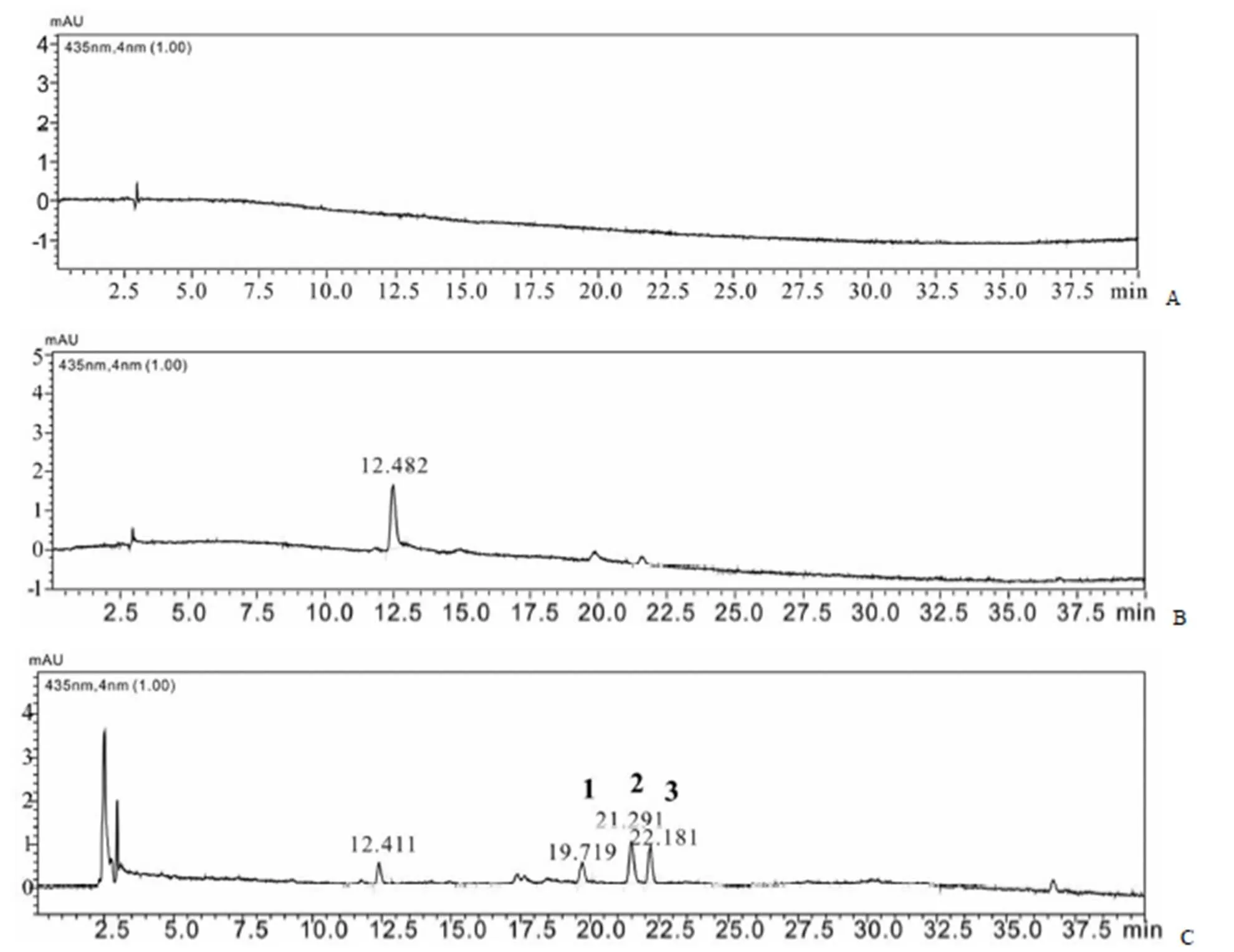
图 6 HPLC 方法检测富铁和贫铁发酵条件下铁载体含量(A:富铁培养基发酵液,含 0.01 mol/L FeCl3;B:贫铁培养基发酵液;C:在 B 中加入 FeCl3,使铁离子浓度达到 0.01 mol/L)
Figure 6 The HPLC for analyzing theabundance of siderophores under fermentation condition of sufficient and deficient iron(A: The iron-sufficient fermentation broth, containing 0.01 mol/L FeCl3; B: The iron-deficient fermentation broth; C: Adding 0.01 mol/L FeCl3to the iron-deficient fermentation broth of B)
化合物1,白色无定型粉末,易溶于水,难溶于氯仿、丙酮等低极性有机溶剂。在低分辨质谱测试样品中分别加入等物质量的Ga2(SO4)3和 FeCl3水溶液后得到准分子离子627.2 [M + Ga-2H]+和 614.2 [M + Fe-2H]+,推测1的分子量(M)为 560 D,与文献报道的去铁敏 B(DFOB)一致。1的镓(III)络合物的1H NMR 谱(以 D2O 为溶剂)中给出了 DFOB-Ga 所有的质子信号,包括两组互相偶合的亚甲基信号ppm2.35(4H,t,= 7.2 Hz,H2-9,20),2.45,1.96(each 2H,m,H2-10,21)归属为琥珀酰基;与羟氨相连的亚甲基ppm4.64 ~ 4.84(6H,m,H2-2,13,24);与氨基相连的亚甲基ppm2.88,3.00,3.05(each 2H,t,= 7.2 Hz,H2-6,17,28);饱和脂肪族质子ppm2.18(18H,m,H2-3,4,5,14,15,16,25,26,27),以及一个属于乙酰基甲基质子信号ppm1.26(3H,s)。这些数据与文献报道的 DFOB-Ga 相吻合[17]。确定1 为去铁敏 B。
化合物2,白色无定型粉末,易溶于水、甲醇等极性溶剂。LC-MS 测试样品中分别加入等物质量的Ga2(SO4)3和 FeCl3水溶液得到准分子离子分别为667.2 [M + Ga-2H]+和 654.2 [M + Fe-2H]+,推测2的分子量(M)为 600 D,比1大 40 D,与文献报道的去铁敏 E(DFOE)一致。2 的1H NMR 谱与1相比明显少了一个甲基信号,而在属于琥珀酰基的亚甲基区域(2.0 ~ 2.5 ppm)处增加了一组互相偶合的亚甲基(积分总数多出了4 个质子),这些信息与去铁敏E 结构相符合[18]。推测2为去铁敏 E。
化合物3,无色油状物,易溶于甲醇、丙酮等有机溶剂。LC-MS 给出3的准分子离子峰219.3 [M + H]+和 241.3 [M + Na]+,提示其分子量为 218 D,结合其 NMR 数据推测其分子式应为 C9H18N2O4。3的1H NMR 谱(DMSO-6为溶剂)给出了羟肟酰胺类化合物的质子信号:ppm2.29,2.41(each 2H,t,= 7.2 Hz,H2-10,11),3.07(2H,t,= 7.2 Hz,H2-6),3.02(2H,t,= 7.2 Hz,H2-2)和 1.28,1.37,1.58(each 2H,m,H2-3,4,5)。3的波谱数据与文献[18]报道的1-hydroxy-1-succinyl cadaverine(HSC)一致,提示3应为1-羟基戊二胺与琥珀酸单脱水缩合产物,化学结构如图 7所示。
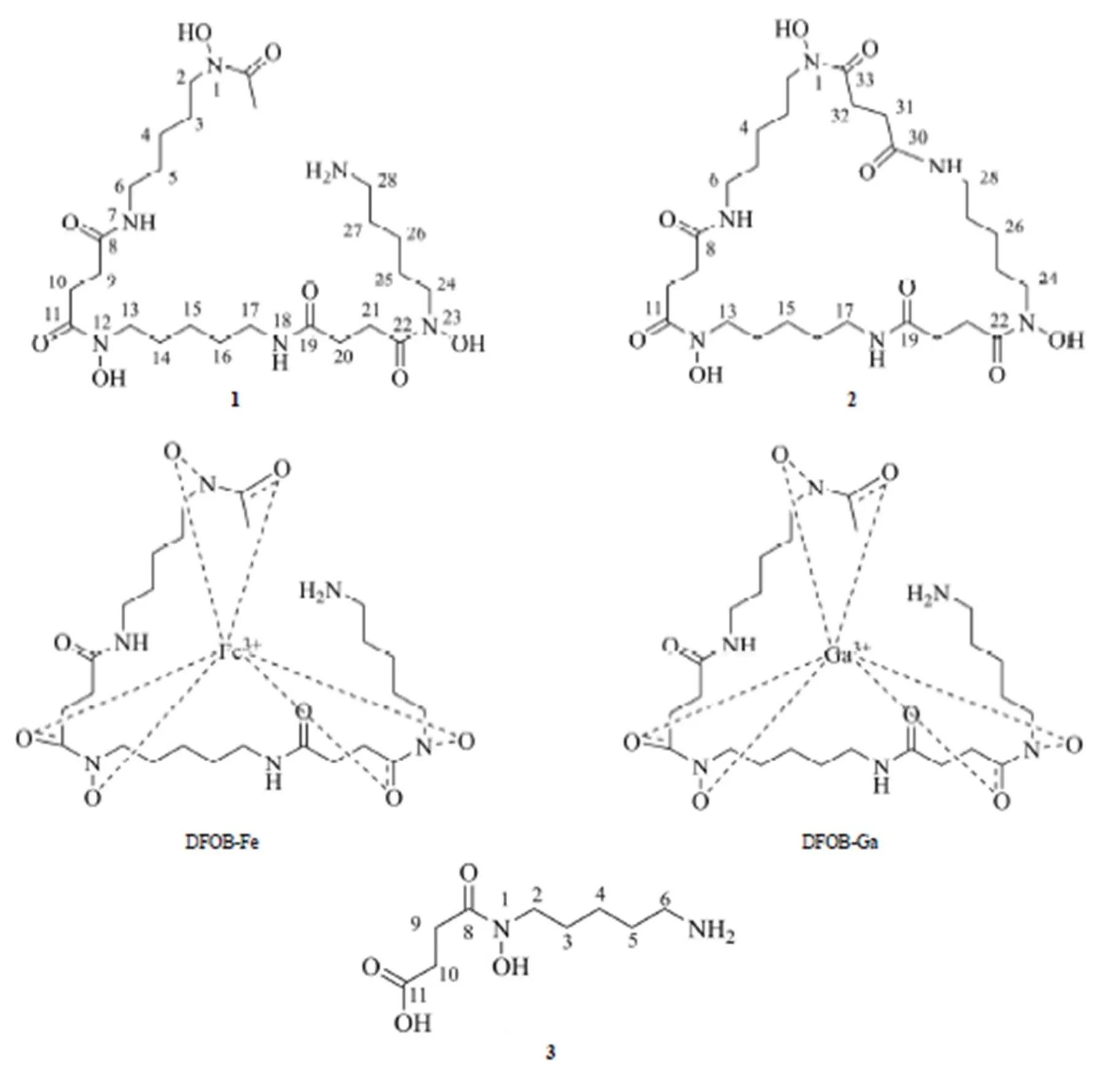
图 7 化合物1、2、3、DFOB-Fe 和DFOB-Ga 结构
Figure 7 Chemical structures of 1, 2, 3, DFOB-Fe and DFOB-Ga
化合物1~ 3的化学结构中均含有体现铁载体功能的羟肟酰胺片段,均属于铁载体类化合物。化合物1和2来源于同一个生物合成前体[16],具有相似的化学结构,差异在化合物1(去铁敏 B)末端氨基发生了乙酰化,分子为链状结构;而化合物2(去铁敏 E)的末端氨基与分子内羧基缩合形成闭环结构。化合物3是1和2的生物合成中间体之一。
2.4 化合物的活性评价
化合物1(DFOB)及其金属离子络合物 DFOB-Fe 和 DFOB-Ga 在流感病毒 A/汉防/ 359/95 病毒株上进行了活性测试。以磷酸奥司他韦和利巴韦林为阳性对照,结果表明三者具有一定抗病毒活性(表 3),IC50分别为 > 0.07 μg/ml(DFOB)、0.74 μg/ml(DFOB-Fe)和 0.01 μg/ml(DFOB-Ga),但选择指数较低。所有化合物对枯草芽孢杆菌、大肠杆菌和绿脓假单胞菌等无明显生长抑制作用。

表 3 DFOB、DFOB-Fe 和 DFOB-Ga 抗病毒活性结果
3 讨论
灰产色链霉菌()CPCC 200274 为杀稻瘟菌素的产生菌[19]。借助于 antiSMASH 网站整合的多种算法对该菌株基因组进行生物信息学分析后,发现其富含多种类型化合物生物合成基因簇,其中有 5 个基因簇预测为铁载体生物合成基因簇,包含 NIS 类型的铁载体类生物合成基因()。对预测到的 NRPS 基因簇进行了逐一分析,未找到与已知 NRPS 类铁载体生物合成基因簇同源的基因簇。经进一步生物信息学分析,这 5 个预测为铁载体生物合成基因簇以及所有的 NRPS 基因簇中只有 cluster 29 含有 DmdR1 蛋白结合位点 iron box,说明除了 cluster 29 之外,其余基因簇的表达可能均不受铁离子浓度影响。进一步分析显示这 4 个基因簇中同源基因与其他生物合成基因(如 NRPS、PKS 等)相邻,提示这些基因簇可能编码其他类型的化合物,有待进一步研究。
去铁敏化学结构中通常含有三组羟肟酰胺类官能团,在溶液状态下能够与三价金属离子(如 Fe3+和Al3+等)生成稳定的无毒络合物,因此去铁敏在临床主要用于治疗铁离子或者铝离子浓度过高引起的金属离子中毒。在中东等地区,去铁敏还被用于治疗因球蛋白生成障碍性贫血引起的听力异常[20]。除此之外,去铁敏与抗生素偶联的天然“木马分子”ferrimycin A1 和 salmycins 对抑制革兰阳性菌生长有效,它们结构中的抗生素均为氨基糖苷类。基于去铁敏与 β-内酰胺(罗拉碳头孢)或氟喹酮类(环丙沙星)抗生素偶联合成的“木马分子”对多重耐药细菌(ESKAPE)有效,最低抑菌浓度(MIC)达到 64 ~ 1 μmol/L[21]。铁载体化学合成比较困难,除了选择性差,反应路线长,总收率低等缺点之外,还体现在保护基团的脱除比较困难[22]。酰胺氮原子的直接氧化(羟基化)产物复杂,氧化剂的量和氧化条件难以控制,因此,目前市售的该类铁载体绝大多数来源于微生物发酵。从微生物来源发现新型铁载体类化合物具有潜在的应用价值。
基于微生物生物信息学分析的基因组发掘技术已经成为寻找新结构天然产物的重要手段之一[8]。研究表明,微生物尤其是放线菌的基因组中存在大量的隐性生物合成基因簇,这些基因簇在通常实验室培养条件下表达量低或者不表达,借助于传统的分离技术无法富集足够量进行结构解析。因此,科学家开发出多种有效手段用来激活这些沉默基因簇,如改变发酵条件的OSMAC 方法[23],过表达基因簇中正调控基因,敲除负调控基因和异源表达等已经被成功应用,并获得了大量新结构天然产物[24]。我们发现在链霉菌属和游动放线菌属、拟无枝酸菌属等稀有放线菌中均存在 DmdR1 的同源蛋白,且 iron box 序列保守,在此基础上本文利用铁载体的生物合成调控特性,从 antiSMASH 预测的 5 个铁载体生物合成基因簇中排除了 4 个不含 iron box 序列且不受铁离子浓度调控的基因簇,用贫铁发酵条件成功激活并分离到了 cluster 29 对应的 3 个铁载体类化合物。该策略将可用于放线菌中新结构铁载体类化合物的高效发现。自然界中还存在能与其他金属离子(如 Zn2+、Cu2+、Mn2+等)螯合的天然产物,具有不同的生物活性,随着其调控蛋白的发现和调控机制的解析(例如 Zn2+摄取调控蛋白 Zur[25-26]),本文报道的基因组发掘策略还可推广至更多的具有不同金属离子螯合功能的天然产物的发现。
本文通过生物信息学分析发现灰产色链霉菌()CPCC 200274 基因组上存在多种天然产物生物合成基因簇,其中有 5 个为可能的铁载体生物合成基因簇。借助 antiSMASH 网站次级代谢产物预测、MEME、FIMO 等生物信息学分析的结果,发现其中一个基因簇与去铁敏生物合成基因簇(~)具有高度同源性,且包含调控蛋白 DmdR1 的结合位点,由此借助于改变培养基中的铁离子浓度结合 qRT-PCR 的方法确定了该基因簇表达条件,并成功从该菌株的发酵液中分离得到去铁敏 B、E和1-羟基-1-琥珀酰基尸胺,通过 LC-MS 和 NMR 等方法确定了三者的结构。本工作为铁载体类化合物生物合成基因簇的快速定位、新生物合成元件的积累及定向化学分离提供了新思路,为新结构铁载体类化合物的寻找及合成生物学改造奠定了基础。
[1] Abbaspour N, Hurrell R, Kelishadi R. Review on iron and its importance for human health. J Res Med Sci, 2014, 19(2):164-174.
[2] Khan A, Singh P, Srivastava A. Synthesis, nature and utility of universal iron chelator - Siderophore: a review. Microbiol Res, 2018, 212-213:103-111.
[3] Dias PJ, Sá-Correia I. The drug: H+ antiporters of family 2 (DHA2), siderophore transporters (ARN) and glutathione: H+ antiporters (GEX) have a common evolutionary origin in hemiascomycete yeasts. BMC Genomics, 2013, 14:901-933.
[4] Dhusia K, Bajpai A, Ramteke PW. Overcoming antibiotic resistance: Is siderophore Trojan horse conjugation an answer to evolving resistance in microbial pathogens? J Control Release, 2018, 269:63-87.
[5] Möllmann U, Heinisch L, Bauernfeind A, et al. Siderophores as drug delivery agents: application of the “Trojan Horse” strategy. Biometals, 2009, 22(4):615-624.
[6] Straubinger M, Blenk H, Naber KG, et al. Urinary concentrations and antibacterial activity of BAL30072, a novel siderophore monosulfactam, against uropathogens after intravenous administration in healthy subjects. Antimicrob Agents Chemother, 2016, 60(6):3309-3315.
[7] Jacobs A. Iron chelation therapy for iron loaded patients. Br J Haematol, 1979, 43(1):1-5.
[8] Choi SS, Katsuyama Y, Bai L, et al. Genome engineering for microbial natural product discovery. Curr Opin Microbiol, 2018, 45:53-60.
[9] Chiang YM, Chang SL, Oakley BR, et al. Recent advances in awakening silent biosynthetic gene clusters and linking orphan clusters to natural products in microorganisms. Curr Opin Chem Biol, 2011, 15(1):137-143.
[10] Huang TT, Lin SJ, Deng ZX. Recent advances in mechanism of siderophore biosynthesis in actinomycetes. Microbiol China, 2011, 38(5):765-773. (in Chinese)
黄婷婷, 林双君, 邓子新. 放线菌中铁载体生物合成机制研究进展. 微生物学通报, 2011, 38(5):765-773.
[11] de Lorenzo V, Neilands JB. Characterization of iucA and iucC genes of the aerobactin system of plasmid ColV-K30 in Escherichia coli. J Bacteriol, 1986, 167(1):350-355.
[12] Flores FJ, Martín JF. Iron-regulatory proteins DmdR1 and DmdR2 of Streptomyces coelicolor form two different DNA-protein complexes with iron boxes. Biochem J, 2004, 380(Pt 2):497-503.
[13] Escolar L, Pérez-Martín J, de Lorenzo V. Opening the iron box: transcriptional metalloregulation by the Fur protein. J Bacteriol, 1999, 181(20):6223-6229.
[14] Barona-Gómez F, Lautru S, Francou FX, et al. Multiple biosynthetic and uptake systems mediate siderophore-dependent iron acquisition in Streptomyces coelicolor A3(2) and Streptomyces ambofaciens ATCC 23877. Microbiology, 2006, 152(Pt 11):3355-3366.
[15] Bunet R, Brock A, Rexer HU, et al. Identification of genes involved in siderophore transport in Streptomyces coelicolor A3(2). FEMS Microbiol Lett, 2006, 262(1):57-64.
[16] Barona-Gómez F, Wong U, Giannakopulos AE, et al. Identification of a cluster of genes that directs desferrioxamine biosynthesis in Streptomyces coelicolor M145. J Am Chem Soc, 2004, 126(50): 16282-16283.
[17] Bergeron RJ, Pegram JJ. An efficient total synthesis of desferrioxamine B. J Org Chem, 1988, 53(14):3131-3134.
[18] Fujita MJ, Sakai R. Heterologous production of desferrioxamines with a fusion biosynthetic gene cluster. Biosci Biotechnol Biochem, 2013, 77(12):2467-2472.
[19] Cone MC, Yin X, Grochowski LL, et al. The blasticidin S biosynthesis gene cluster from Streptomyces griseochromogenes: sequence analysis, organization, and initial characterization. Chembiochem, 2003, 4(9):821-828.
[20] Badfar G, Mansouri A, Shohani M, et al. Hearing loss in Iranian thalassemia major patients treated with deferoxamine: a systematic review and meta-analysis. Caspian J Intern Med, 2017, 8(4):239-249.
[21] Wencewicz TA, Long TE, Möllmann U, et al. Trihydroxamate siderophore-fluoroquinolone conjugates are selective sideromycin antibiotics that target Staphylococcus aureus. Bioconjug Chem, 2013, 24(3):473-486.
[22] Lin YM, Miller MJ. Practical synthesis of hydroxamate-derived siderophore components by an indirect oxidation method and syntheses of a DIG-siderophore conjugate and a biotin-siderophore conjugate. J Org Chem, 1999, 64(20):7451-7458.
[23] Wei HJ, Lin ZJ, Li DH, et al. OSMAC (one strain many compounds) approach in the research of microbial metabolites - a review. Acta Microbiol Sinica, 2010, 50(6):701-709. (in Chinese)
韦洪娟, 林贞建, 李德海, 等. 单菌多次级代谢产物方法及其在微生物代谢产物研究中的应用. 微生物学报, 2010, 50(6):701-709.
[24] Challis GL. Genome mining for novel natural product discovery. J Med Chem, 2008, 51(9):2618-2628.
[25] Gaballa A, Helmann JD. Identification of a zinc-specific metalloregulatory protein, Zur, controlling zinc transport operons in Bacillus subtilis. J Bacteriol, 1998, 180(22):5815-5821.
[26] Patzer SI, Hantke K. The ZnuABC high-affinity zinc uptake system and its regulator Zur in Escherichia coli. Mol Microbiol, 1998, 28(6):1199-1210.
Discovery of siderophore compounds using genome mining strategy
JIANG Zhi-bo, LI Xing-xing, REN Wei-cong, SHI Yuan-yuan, GAO Rong-mei, LI Yu-huan, WU Lin-zhuan, HONG Bin
NHC Key Laboratory of Biotechnology of Antibiotics, Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100050, China
To discover siderophores using genome mining strategy.
Firstly, the antiSMASH was used to analyze the whole genome sequence ofCPCC 200274 for identifying the biosynthetic gene cluster of potential secondary metabolites. Blastp was used to determine the homologues of iron dependent regulatory protein DmdR1 from. Using MEME and FIMO analysis, the biosynthetic gene clusters of siderophore were identified which contain DmdR1 protein binding site(s) (iron box). Secondly, to explore suitable fermentation conditions, real-time RT-PCR (qRT-PCR) was used to detect the expression level of related biosynthetic genes. Finally, the target siderophores were isolated and purified to verify the feasibility of this strategy.
Five clusters containinggene were identified from the genome ofCPCC 200274 by antiSMASH. By Blastp analysis, SGC5642 was indicated to be homologous with DmdR1, which is involved in the regulation of siderophore’s biosynthesis. Cluster 29 might encode siderophores, as it was the only iron box containing cluster among the five predicted siderophore clusters by MEME and FIMO analysis. Based on the above information, the fermentation conditions for the siderophore production were determined by qRT-PCR. Three compounds were isolated and purified and their structures were identified as deferoxamine B, deferoxamine E and1-hydroxy-1-succinyl cadaver amine.
Under the guidance of our siderophore mining strategy, the biosynthetic gene clusters of siderophores are predicted in the microbial genome and the transcriptional level are determined. Three siderophore compounds are successfully obtained fromCPCC 200274. This strategy provides a new idea for the rapid identification of siderophore biosynthetic gene clusters and new biosynthetic elements, and lays a new foundation for the search and synthesis of new structural siderophores through biosynthetic biology.
Genome mining; Siderophores;; qRT-PCR
HONG Bin, Email: binhong69@hotmail.com
10.3969/j.issn.1673-713X.2019.02.001
国家自然科学基金(81603006、81630089、81703398、81872780);“重大新药创制”国家科技重大专项(2018ZX09711001- 006-011、2018ZX09711001-007-001);中国医学科学院医学与健康科技创新工程(2016-I2M-2-002、2016-I2M-3-012)
洪斌,Email:binhong69@hotmail.com
2018-11-05
*同为第一作者

