基于高光谱图像及深度特征的大米蛋白质含量预测模型
孙 俊,靳海涛,芦 兵,武小红,沈继锋,戴春霞
基于高光谱图像及深度特征的大米蛋白质含量预测模型
孙 俊,靳海涛,芦 兵,武小红,沈继锋,戴春霞
(江苏大学电气信息工程学院,镇江 212013)
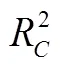
无损检测;光谱分析;模型;高光谱图像;堆叠自动编码器;深度特征;大米;蛋白质含量
0 引 言
中国的大米生产量居世界首位,大米的消费数量也居世界前列,有超过半数的人口将大米作为主食[1],大米的主要营养成分是蛋白质,它富含人体所必需的氨基酸、赖氨酸[2],蛋白质含量决定了大米的营养品质和食味品质[3]。因此,蛋白质含量是评价大米品质的重要指标。
目前,大米蛋白质含量检测的主要措施是化学试剂测定法[4],该方法虽然准确性高,但对样本具有破坏性,且消耗化学试剂量大,成本高。因此,如何快速无损精确地检测大米蛋白质含量是当前大米生产及储存中亟需解决的问题。
在众多新兴技术中,光谱技术可以检测目标内部的特征[5],文献[6-7]采用近红外光谱技术实现大米蛋白质含量检测,效果良好,但光谱技术缺乏目标的空间信息,建模效果有待进一步提升;计算机视觉图像技术可以对样本外部表面信息进行分析,但对于其内部的特征无法反演[8]。高光谱成像技术可以提取光谱信息和图像信息,获取更全面的信息,并可借助于数学手段构建模型,实现对被测对象的无损检测[9],由于其快速、精确、非破坏的特点,高光谱无损检测技术已经被广泛应用于农作物品种分类[10]和营养元素检测[11]等研究中。然而,高光谱图像数据量庞大,波段间相关性较强,冗余度高,若直接对高光谱图像数据进行数据分析,耗时较长,且最终效果不理想,会造成Huges现象[12]。因此,对高光谱图像数据进行降维处理是十分有必要的。
目前,对高光谱图像光谱数据的降维方法从数据相关性角度分为线性方法和非线性方法。主成分分析法[1]、线性判别方法[13]等作为传统的线性方法,都是采用线性映射的手段处理高光谱图像数据的,但是高光谱波段间较强的相关性导致了这些方法在最终处理效果上的不足,而深度学习作为非线性方法通过引入神经网络先天的非线性,弥补了这些不足[14-16]。堆叠自动编码器(stacked auto-encoder,SAE)[17-18]是一种无监督深度学习技术,分别对每层深度网络进行训练,从而更加高效地进行高光谱图像数据降维处理。
另一方面,对于高光谱图像的图像信息,目前的处理方法是提取其颜色或纹理等特征,赵娟等[19]通过提取高光谱图像的纹理特征,建立牛肉的嫩度判别模型,最终精度为94.44%;董高等[20]提取小麦的形态特征,研究发现基于形态特征进行建模的效果并不理想。深度学习的方法通过对整幅图像进行特征提取,可以获得该高光谱图像的图像深度特征,包含了更加全面的信息,目前,还未有相关文献对其报道。
本文采用SAE对高光谱图像的光谱信息和图像信息及其融合信息分别进行特征提取,获得其深度特征,利用支持向量机回归(support vector regression,SVR)建立预测模型并采用网格搜索法对惩罚因子和核参数进行寻优,以提高模型的预测性能,实现大米蛋白质含量的高效无损检测。
1 材料与方法
1.1 样本制备与处理
文献[21]表明,大米在存放过程中,由于大米蛋白质中的氨基酸分解,总蛋白质含量呈逐渐减少的趋势。为了加速大米内部氨基酸反应,获取不同蛋白质含量梯度的大米样本,试验前于镇江某大型超市购买良记金轮膳选泰香米1 kg,共称取6组样本,每组100 g,将6组样本置于BPS-100CL型液晶恒温恒湿箱内(上海一恒科学仪器有限公司),在45 ℃、95%相对湿度的条件下分别放置0,24,48,72,96和120 h,取出后在各组样本中选取30粒大小相近、颗粒饱满、颜色透明的米粒作为一个样本,每组选取70个样本,共制备420个样本,立即放入密封的试剂瓶中送往高光谱实验室进行高光谱图像采集。
1.2 高光谱图像采集及校正
本试验所采用的高光谱成像系统主要包括光纤、卤素灯光源(3900-ER型,Illumination Technologies, USA)、高光谱图像摄影仪(VNIP-HIS-sMOS,五铃光学股份有限公司,中国台湾)、移动平台控制器和处理器等部分。高光谱图像摄影仪中的摄像机为CCD相机,光谱仪为可见光/近红外光谱仪,光谱范围为400~1 000 nm,光谱分辨率为2.8 nm,图像分辨率为1 024像素×473像素。
将各组样本均匀平铺于移动平台上进行图像采集,移动平台移动速度设置为1.99 mm/s。由于光源强度分布不均匀及暗电流噪声的存在,为了提高获取图像的信噪比,需要在扫描前对每个样本进行黑白标定[22]。首先放置白板(反射率约为100%)于移动平台上,曝光时间设置为7 ms,获得全白标定图像,保持曝光时间不变,再盖上CCD相机镜头,获得反射率约为0的全黑标定图像,随后取下CCD相机镜头片,放入大米样本,调节曝光时间为17 ms,获得未标定图像raw。黑白标定后的图像的反射光强度可表示为
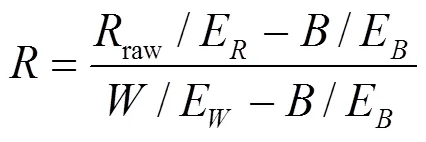
式中、、raw和均为相应图像的反射光强度;E为样本的曝光时间,ms;E为全黑标定的曝光时间,ms;E为白板标定的曝光时间,ms。
1.3 蛋白质含量测定
采集高光谱图像后,采用凯氏定氮法[4]测定样本蛋白质含量。将各样本称质量并记录,置于研钵中研磨至粉末状,然后移入100 mL定氮瓶,置于消化仪做消化处理至内容物全部碳化,随后进行盐酸标准滴定液滴定。通过最终消耗的盐酸标准滴定液体积与氮含量的关系计算每个样本蛋白质含量,蛋白质含量计算公式为
式中为样本中蛋白质含量,g/100g;1为消耗盐酸标准滴定液体积,mL;2为试剂空白消耗盐酸标准滴定液体积,mL;为盐酸标准滴定液浓度,mol/L;为样本质量,g;3为吸收消化液体积,mL;为氮换算蛋白质系数,大米为5.95;100为换算系数。
高温高湿条件下不同放置时间的大米样本蛋白质含量变化趋势如图1所示,通过试验测得大米样本初始蛋白质含量为7.78 g/(100 g),在存放24,48,72,96和120 h后,蛋白质含量分别为7.73,7.70,7.68,7.65和7.62 g/(100 g)。可见,大米样本在经高温高湿条件下放置处理后,蛋白质含量的变化很小,在放置120 h后蛋白质含量减少0.16 g/(100 g)。大米在高温高湿条件下,呼吸作用等各项生理活动加快,消耗了部分占总蛋白量很少的生理蛋白(清蛋白、球蛋白),与文献[21]结果一致。

注:放置温度为45℃,相对湿度为95%,下同。
1.4 样本感兴趣区域(region of interest,ROI)选取
ROI的提取方式与大小会影响到最终高光谱图像数据的获取质量[23]。本文ROI提取流程如图2所示,原始样本高光谱图像如图2a所示,为加强样本与背景的差异性,使用波段比算法处理高光谱图像,根据多个像素点光谱曲线选取一个较大反射率(702.36 nm)波长与一个较小反射率(502.31 nm)波长做商运算,结果如图2b所示。然后采用阈值分割法进行处理,经多次测试,阈值选择0.94时分割效果最佳,分割得到二值图如图2c所示,将该二值图应用掩膜于原始高光谱图像,选取ROI,如图2d所示,为方便后续处理,将ROI图像尺寸统一为100像素×100像素。

图2 大米样本ROI提取流程
1.5 堆叠自动编码器
自动编码器(auto-encoder, AE)主要由编码器和解码器两部分构成,在对输入样本进行学习时,其目的是通过编码和解码将输入数据在输出中重构出来[24]。从某种程度而言,AE是一个小型的深度学习模型,该模型主要包括输入层、隐含层和输出层3层。
假设有无标签的训练样本,AE的编码任务是将输入训练样本通过非线性函数映射到隐含层,设为隐含层的神经单元激活值,其数学表达式为
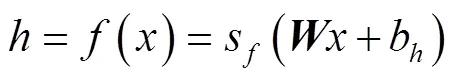
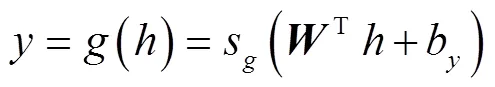
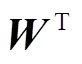
定义重构误差为,表示为
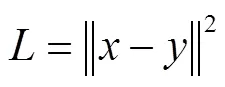
通过不断修改参数来最小化L,此时的y保留了x的大部分信息[25]。在实际应用中,用到的往往并不是单独的一个AE,而是将多个AE通过级联的方式堆叠构成的多层AE,这个多层AE被命名为SAE[26]。SAE模型如图3所示,该SAE模型由2个AE堆叠而成,隐含层2作为SAE最终的降维结果输出的深度特征。
这种结构使得SAE的隐含层是非线性的,从而具有更准确的捕获复杂数据的能力[27]。
1.6 光谱信息与图像信息融合及深度特征提取
通过阈值分割法从原始高光谱图像中提取整个大米样本区域作为ROI,一方面,提取ROI内平均光谱信息,每个高光谱图像包含478个波段,故每个样本高光谱数据维度是478。另一方面,大米中蛋白质含量的变化会导致其颜色、纹理及透明度等变化[28],为了获取更加全面的图像信息,提取ROI中每个波段的图像,将ROI提取结果保存为478幅普通的100像素×100像素RGB图像,并转换为灰度图像,图像尺寸统一为28像素´28像素[29]。为了获取更加精确的图像信息,减少噪声的影响,以光谱曲线的3个波峰760、930和980 nm为中心,获取(740~770 nm)、(910~950 nm)、(960~1 000 nm)3个波长范围内的图像,取每个像素点对应的灰度均值,获得该样本的特征图像,将每幅图像扁平归一化为一维向量,共包含784个像素点,每个图像对应的数据维度是784。高光谱图像数据融合方式包括数据层融合和特征层融合,由于光谱特征和图像特征在性质上有较大的差异,如果对两者进行特征层融合,各特征所占比重的不同会影响最终建模的精度,为避免该问题的出现,本文将光谱信息和图像信息进行数据层融合,数据总维度为1 262,输入到SAE提取融合信息的深度特征。
1.7 建模方法及评价指标
大米样本高光谱图像的光谱信息与图像信息融合流程如图4所示。

图4 光谱信息与图像信息融合流程
在SAE提取的光谱深度特征、图像深度特征及二者融合信息的深度特征基础上,采用支持向量机回归(support vector regression,SVR)建立大米蛋白质含量预测模型,并引用网格搜索法对SVR中核参数和惩罚因子寻优。该方法采用5折交叉验证法在[-10,10]选择最优和,步长设置为0.5,对每个和建立SVR模型并计算验证集均方根误差,和最佳值由最小验证集均方根误差决定[30]。
2 结果与分析
2.1 基于光谱深度特征的建模结果与分析
为了获取更加全面的光谱信息,本文选取整个大米样本区域作为ROI,提取ROI的平均光谱信息作为该样本的高光谱数据。全样本的原始高光谱曲线如图5a所示。高光谱数据在获取过程中,往往会受到仪器噪声等因素的影响,存在吸光度的非线性、基线变动和附加散射变动等问题,不可避免产生误差。为了减少噪声对模型的干扰,本文采用基于时域局部最小二乘法拟合的多项式平滑(savitzky-golay,SG)滤波方法[32]对原始高光谱曲线进行预处理,经过SG滤波后的高光谱曲线如图5b所示,与图5a原始样本高光谱曲线相比,毛刺减少,曲线平滑度得到有效提升。
大米样本的高光谱曲线可以有效地表征大米中蛋白质等主要成分的化学信息,蛋白质等化学信息可以通过C-H官能团、O-H官能团和N-H官能团在特定波长下的伸缩变动体现。由图5可知,虽然整体的反射光谱曲线波动不明显,没有特别明显的波峰波谷,但在760、930和980 nm仍有小幅度的波峰出现。结合图1分析可知,这些特征波段和蛋白质中C-H键的振动和断裂有直接关系,这些特征波段处的波动正是由于高温高湿条件下不同放置时间的大米内部蛋白质含量不同而引起的,随着放置时间的增加,蛋白质含量减少,反射率逐渐增加。因此,高光谱曲线可以直观地反映高温高湿条件下不同放置时间大米蛋白质含量的差异。
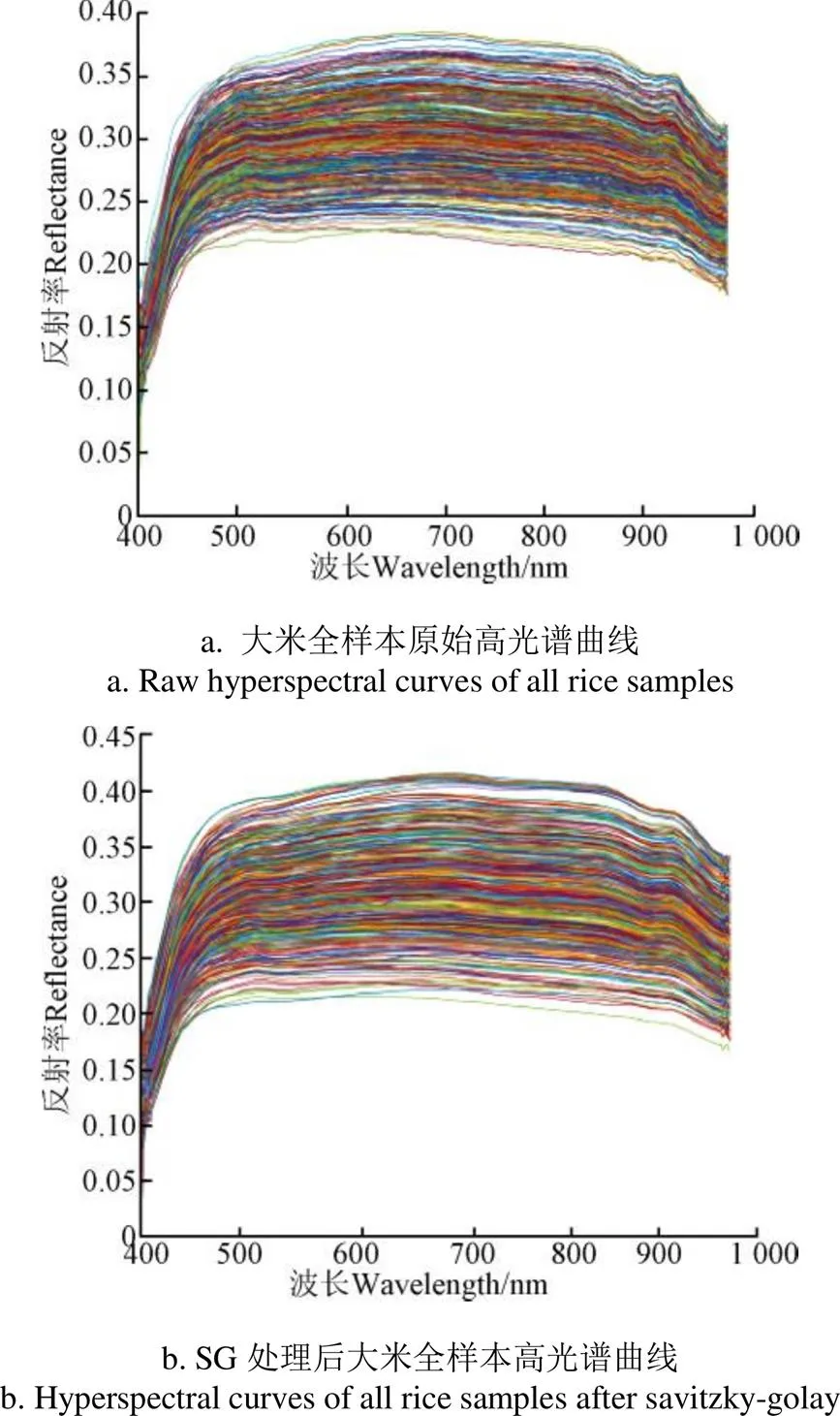
注:图中反射率曲线对应的样本放置时间从下到上依次为0,24,48,72,96和120 h,全样本量为420个。
针对高温高湿条件下6组不同放置时间的大米样本(每组包含70个样本,训练集46个,预测集24个),分别采用3层隐含层SAE寻找最优规模提取光谱深度特征,并利用SVR建立回归模型,结果如表1所示,其中预测值表示每组样本中预测集样本的预测值均值。
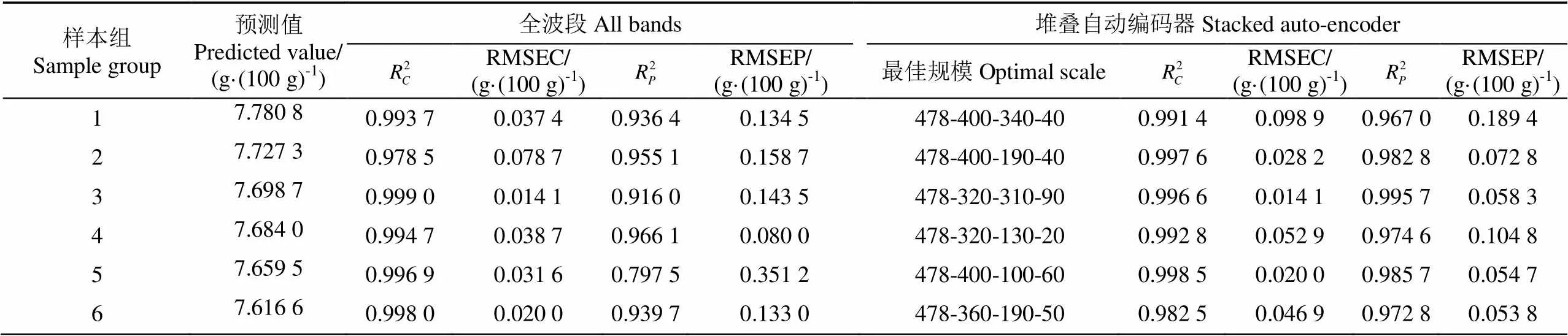
表1 基于光谱深度特征信息的模型预测结果
注:最佳规模表示SAE数据维度和各层神经元最佳个数。如478-400-290-70表示数据维度是478,神经元个数分别是400、290、70,下同。
Note: Optimal scale means the data dimension and the optimal number of cell of each layer of SAE. E.g. 478-400-290-70 means data dimension is 478, the number of neurons is 400, 290 and 70 respectively, the same below.
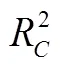
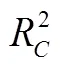
对于SAE隐含层个数的设置,文献[16]表明,对于小样本而言,SAE隐含层个数并非越多越好。经过试验,通过SAE参数规模寻优,发现不同组样本的最佳SAE规模不同,其中3层隐含层的建模效果普遍较好,而且将最初的478维降低到了较低维度20~90,减小了建模成本,提高了建模效率,与文献[16]报道一致。
2.2 基于图像深度特征的建模结果与分析
图像深度特征提取结果如图6所示,对于图像信息,由于原始图像尺寸均为100像素´100像素,所含冗余信息较多,故在特征提取前,将图像尺寸统一为28像素´28像素。以第4组中某样本为例,其高光谱图像如图6a所示,将高光谱图像扁平化,转换为784维的列向量,为了提高运算效率,将该向量归一化,结果如图6b所示,并作为SAE的输入提取图像深度特征,结果如图6c所示。图6d是经过SAE解码重构后的图像信息,经对比发现,重构信息与原始信息大致趋势相同。使用与2.1节相同方法划分数据集,采用SVR建立回归模型,结果如表2所示。
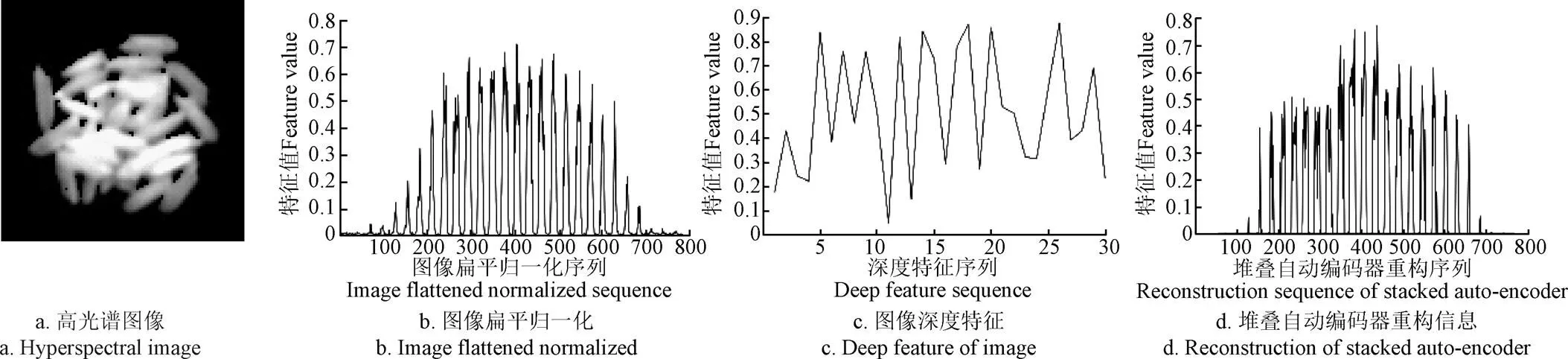
图6 图像深度特征提取结果

表2 基于图像深度特征的模型预测结果
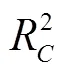
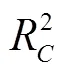
2.3 基于光谱与图像融合信息深度特征的建模结果与分析
为获取更加全面的高光谱图像数据,将478维光谱数据和784维图像数据进行融合,得到1 262维融合信息,输入到SAE提取深度特征,使用与2.1节相同方法划分各组样本集,采用SVR建立回归模型。经过试验,建模结果如表3所示。
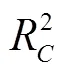
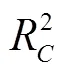
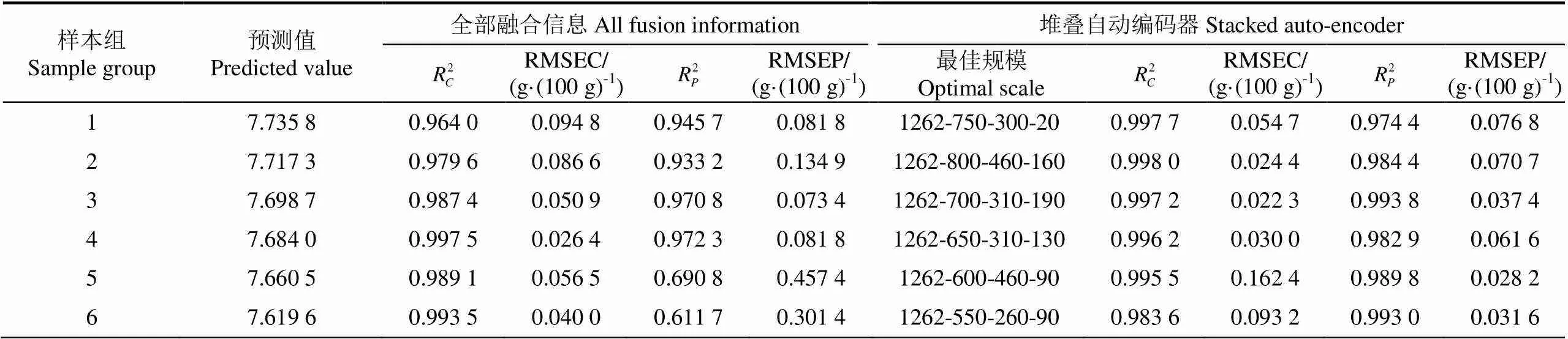
表3 基于光谱-图像融合信息建模结果

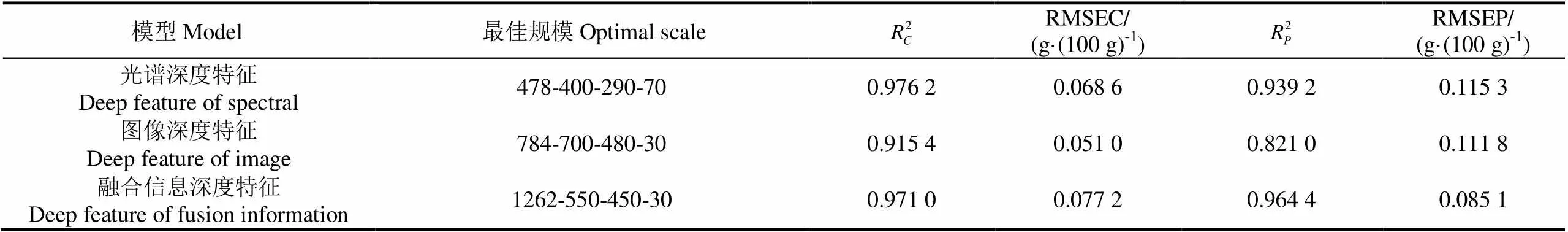
表4 基于全样本的不同模型预测结果对比
3 讨 论
在已有的研究中,大米的蛋白质含量无损检测大都基于近红外光谱,刘建学等[33]以二阶导数谱建立大米蛋白质含量预测模型,预测值和测定值间的相关系数达到0.94;Bagchi等[34]建立SNV-mPLS模型,预测糙米和米糠蛋白质含量,模型的决定系数分别为0.918、0.567;Lin等[35]建立了多元线性回归、偏最小二成回归和人工神经网络模型对大米蛋白质含量进行数据分析,最终多元线性回归的模型效果最佳,预测集决定系数为0.806,预测集标准误差为0.266%。本文采用SAE提取大米样本的高光谱图像光谱深度特征,结合支持向量机建立蛋白质含量预测模型,对于全样本的预测集决定系数为0.939 2,均方根误差为0.115 3 g/(100 g)。模型的效果优于上述文献方法,相较于文献[34],本文模型预测集决定系数提升了14.17%,进一步验证了本文算法对于高光谱数据特征提取的高效性。
此外,传统高光谱图像的图像信息处理方法主要通过提取颜色、纹理等特征进行建模,但这些特征往往是片面的,最终的建模效果有一定的提升空间。本文首先将高光谱图像按波段分离,通过获取图像的灰度值均值作为该样本的特征图像,最终得到784维的列向量,采用SAE提取其深度特征,结合支持向量机回归建立蛋白质含量预测模型,最终建模的训练集决定系数和均方根误差为0.915 4和0.051 0 g/(100 g),预测集决定系数和均方根误差为0.821 0和0.111 8 g/(100 g),建模效果良好,验证了图像深度特征提取方法的可行性。
4 结 论
本研究采用SAE学习获取大米高光谱图像的光谱、图像以及二者融合信息的深度特征,并结合支持向量机回归建立大米蛋白质含量预测模型,得出以下主要结论:
1)SAE算法提取特征功能强大。经由SAE提取深度特征后,基于全部样本的光谱信息、图像信息以及二者融合信息的模型预测结果显示,预测集决定系数分别为0.939 2、0.821 0、0.964 4,且均方根误差较低,表明SAE适于高光谱图像数据的降维。
2)对于高光谱图像的图像信息,与传统的提取颜色、纹理等片面信息不同,本文采用SAE提取图像信息的深度特征,结果表明,模型的预测集决定系数和均方根误差分别是0.821 0和0.111 8 g/(100 g),说明该深度特征包含了更加全面的图像信息,为高光谱图像的图像处理方法提供了新思路。

[1] 孙俊,金夏明,毛罕平,等. 高光谱图像技术在掺假大米检测中的应用[J]. 农业工程学报,2014,30(21):301-307.
Sun Jun, Jin Xiaming, Mao Hanping, et al. Application of hyperspectral imaging technology for detecting adulterate rice [J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2014, 30(21): 301-307. (in Chinese with English abstract)
[2] 贾潇,赵谋明,贾春晓,等. 大米蛋白与阿魏酸酶法交联物的乳化特性和抗氧化稳定性[J]. 食品科学,2017,38(13):131-137.
Jia Xiao, Zhao Mouming, Jia Chunxiao, et al. Emulsifying properties and antioxidant stability of enzymatically cross-linked products between rice protein and ferulic acid[J]. Food Science, 2017, 38(13): 131-137. (in Chinese with English abstract)
[3] 陈媛,张志国. 大米蛋白研究进展[J]. 粮食与油脂,2017,30(7):13-16.
Chen Yuan, Zhang Zhiguo. Research progress in rice protein [J]. Cereals and oils, 2017, 30(7): 13-16. (in Chinese with English abstract)
[4] 国家卫生和计划生育委员会.食品安全国家标准食品中蛋白质的测定:GB5009.5-2016[S]. 北京: 中国标准出版社,2016.
[5] 彭彦昆,赵芳,李龙,等. 利用近红外光谱与PCA-SVM识别热损伤番茄种子[J]. 农业工程学报,2018,34(5):159-165.
Peng Yankun, Zhao Fang, Li Long, et al. Discrimination of heat-damaged tomato seeds based on near infrared spectroscopy and PCA-SVM method [J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2018, 34(5): 159-165. (in Chinese with English abstract)
[6] 刘明博,李玉鹏,唐延林,等. 大米蛋白质含量近红外光谱检测模型研究[J]. 中国农学通报,2013(12):212-216.
Liu Mingbo, Li Yupeng, Tang Yanlin, et al. Study on NIR spectral detection model of rice protein content[J]. Chinese Agricultural Science Bulletin, 2013(12): 212-216. (in Chinese with English abstract)
[7] Miryeong S, Franklineii B, Annam M C, et al. Near-infrared spectroscopy for determination of protein and amylose in rice flour through use of derivatives[J]. Cereal Chemistry, 2004, 81(3): 341-344.
[8] 贾桂锋,陈伟,冯耀泽. 基于生物散斑图像和惯性矩谱分析的牛肉掺腐检测[J]. 农业工程学报,2018,34(16):281-286.
Jia Guifeng, Chen Wei, Feng Yaoze. Quantification detection of beef adulteration with spoiled beef based on biospeckle imaging and inertia moment spectrum [J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2018, 34(16): 281-286. (in Chinese with English abstract)
[9] Camps-Valls G, Tuia D, Bruzzone L, et al. Advances in hyperspectral image classification[J]. IEEE Signal Processing Magazine, 2014, 31(1): 45-54.
[10] 刘小丹,冯旭萍,刘飞,等. 基于近红外高光谱成像技术鉴别杂交稻品系[J]. 农业工程学报,2017,33(22):189-194.
Liu Xiaodan, Feng Xuping, Liu Fei, et al. Identification of hybrid rice strain based on near-infrared hyperspectral imaging technology[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2017, 33(22): 189-194. (in Chinese with English abstract)
[11] 孙红,郑涛,刘宁,等. 高光谱图像检测马铃薯植株叶绿素含量垂直分布[J]. 农业工程学报,2018,34(1):149-156.
Sun Hong, Zheng Tao, Liu Ning, et al. Vertical distribution of chlorophyll in potato plants based on hyperspectral imaging[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2018, 34(1): 149-156. (in Chinese with English abstract)
[12] 孙俊,金夏明,毛罕平,等. 基于高光谱图像光谱与纹理信息的生菜氮素含量检测[J]. 农业工程学报,2014,30(10):167-173.
Sun Jun, Jin Xiaming, Mao Hanping, et al. Detection of nitrogen content in lettuce leaves based on spectroscopy and texture using hyperspectral imaging technology[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2014, 30(10): 167-173. (in Chinese with English abstract)
[13] 臧卓,林辉,杨敏华. ICA与PCA在高光谱数据降维分类中的对比研究[J]. 中南林业科技大学学报,2011,31(11):18-22.
Zang Zhuo, Lin Hui, Yang Minhua. Comparative study on descending dimension classification of hyperspectral data between ICA algorithm and PCA algorithm[J]. Journal of Central South University of Forestry & Technology, 2011, 31(11): 18-22. (in Chinese with English abstract)
[14] Liu Z, He Y, Cen H, et al. Deep feature representation with stacked sparse auto-encoder and convolutional neural network for hyperspectral imaging-based detection of cucumber defects[J]. Transactions of the ASABE, 2018, 61(2): 425-436.
[15] Yu J, Huang D, Wei Z. Unsupervised image segmentation via Stacked Denoising Auto-encoder and hierarchical patch indexing[J]. Signal Processing, 2017: 143, 346-353.
[16] 孙俊,靳海涛,武小红,等. 基于低秩自动编码器及高光谱图像的茶叶品种鉴别[J]. 农业机械学报,2018,49(8):316-323.
Sun Jun, Jin Haitao, Wu Xiaohong, et al. Tea variety identification based on low-rank stacked auto-encoder and hyperspectral image[J]. Transactions of the Chinese Society for Agricultural Machinery, 2018, 49(8): 316-323. (in Chinese with English abstract)
[17] Zhou F F. Improving prediction of self-interacting proteins using stacked sparse auto-encoder with PSSM profiles[J]. International Journal of Biological Sciences, 2018, 14(8): 983-991.
[18] Zhang F, Du B, Zhang L. Saliency-guided unsupervised feature learning for scene classification[J]. IEEE Transactions on Geoscience & Remote Sensing, 2014, 53(4): 2175-2184.
[19] 赵娟,彭彦昆. 基于高光谱图像纹理特征的牛肉嫩度分布评价[J]. 农业工程学报,2015,31(7):279-286.
Zhao Juan, Peng Yankun. Distribution of beef tenderness grading based on texture features by hyperspectral image analysis[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2015, 31(7): 279-286. (in Chinese with English abstract)
[20] 董高,郭建,王成,等. 基于近红外高光谱成像及信息融合的小麦品种分类研究[J]. 光谱学与光谱分析,2015,35(12):3369-3374.
Dong Gao, Guo Jian, Wang Cheng, et al. The classification of wheat varieties based on near infrared hyperspectral imaging and information fusion[J]. Spectroscopy and Spectral Analysis, 2015, 35(12): 3369-3374. (in Chinese with English abstract)
[21] 孙威,刘英. 高气温高湿环境对大米氨基酸的影响[J]. 武汉轻工大学学报,2013(1):24-27.
Sun Wei, Liu Ying. Effects of treatment of high temperature and high relative humidity on rice amino acids[J].Journal of Wuhan Polytechnic University, 2013(1): 24-27. (in Chinese with English abstract)
[22] 朱文静,毛罕平,周莹,等. 基于高光谱图像技术的番茄叶片氮素营养诊断[J]. 江苏大学学报:自然科学版,2014,35(3):290-294.
Zhu Wenjing, Mao Hanping, Zhou Ying, et al. Hyperspectral imaging technology of nitrogen status diagnose for tomato leaves[J]. Journal of Jiangsu University: Natural Science Edition, 2014, 35(3): 290-294. (in Chinese with English abstract)
[23] 赵凡,董金磊,郭文川. 高光谱图像光谱提取区域对猕猴桃糖度检测精度的影响[J]. 现代食品科技,2016(4):223-228.
Zhao Fan, Dong Jinlei, Guo Wenchuan. Effect of spectral extraction regions in hyperspectral images on the precision in determining the sugar content of kiwifruits[J]. Modern Food Science & Technology, 2016(4): 223-228. (in Chinese with English abstract)
[24] Bengio Y, Lamblin P, Dan P, et al. Greedy layer-wise training of deep networks[J]. Advances in Neural Information Processing Systems, 2007, 19: 153-160.
[25] Baldi P. Autoencoders, unsupervised learning and deep architectures[C]// International Conference on Unsupervised and Transfer Learning Workshop, 2011: 37-50.
[26] Vincent P, Larochelle H, Bengio Y, et al. Extracting and composing robust features with denoising autoencoders[C]// International Conference on Machine Learning, ACM, 2008: 1096-1103.
[27] Jaitly N, Hinton G E. Using an auto-encoder with deformable templates to discover features for automated speech recognition[C]// The 14th Annual Conference of the International Speech Communication Association, Lyon, 2013: 25-29.
[28] 郭玉宝. 大米储藏陈化中蛋白质对其糊化特性的影响及其相关陈化机制研究[D]. 南京: 南京农业大学,2012.
Guo Yubao. The Effects of Protein on Pasting Properties of Rice During Storage Ageing and its Related Ageing Mechanism[D]. Najing: Nanjing Agricultural University, 2012. (in Chinese with English abstract)
[29] Chen Y, Lin Z, Zhao X, et al. Deep learning-based classification of hyperspectral data[J]. IEEE Journal of Selected Topics in Applied Earth Observations & Remote Sensing, 2014, 7(6): 2094-2107.
[30] Cheng P, Fan W, Xu Y. Quality grade discrimination of Chinese strong aroma type liquors using mass spectrometry and multivariate analysis[J]. Food Research International, 2013, 54(2): 1753-1760.
[31] Guo W, Shang L, Zhu X, et al. Nondestructive detection of soluble solids content of apples from dielectric spectra with ANN and chemometric methods[J]. Food and Bioprocess Technology, 2015, 8(5): 1126-1138.
[32] 孙俊,丛孙丽,毛罕平,等. 基于高光谱的油麦菜叶片水分CARS-ABC-SVR预测模型[J]. 农业工程学报,2017,33(5):178-184.
Sun Jun, Cong Sunli, Mao Hanping, et al. CARS-ABC-SVR model for predicting leaf moisture of leaf-used lettuce based on hyperspectral[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2017, 33(5): 178-184. (in Chinese with English abstract)
[33] 刘建学,吴守一,方如明. 近红外光谱法快速检测大米蛋白质含量[J]. 农业机械学报,2001,32(3):68-70.
Liu Jianxue, Wu Shouyi, Fang Ruming. Rapid measurement of rice protein content by near infrared spectroscopy[J]. Transactions of the Chinese Society for Agricultural Machinery, 2001, 32(3): 68-70. (in Chinese with English abstract)
[34] Bagchi T B, Sharma S, Chattopadhyay K. Development of NIRS models to predict protein and amylose content of brown rice and proximate compositions of rice bran[J]. Food Chemistry, 2016, 191: 21-27.
[35] Lin L, Lu F, Chang Y. Prediction of protein content in rice using a near-infrared imaging system as diagnostic technique[J]. International Journal of Agricultural and Biological Engineering, 2019, 12(2): 195-200.
Prediction model of rice protein content based on hyperspectral image and deep feature
Sun Jun, Jin Haitao, Lu Bing, Wu Xiaohong, Shen Jifeng, Dai Chunxia
(212013,)
nondestructive detection; spectrum analysis; models; hyperspectral imaging; stacked auto-encoder; deep feature; rice; protein content
10.11975/j.issn.1002-6819.2019.15.036
S126
A
1002-6819(2019)-15-0295-09
2018-12-11
2019-06-06
国家自然科学基金资助项目(31471413);江苏高校优势学科建设工程资助项目PAPD(苏政办发2011 6号);江苏省六大人才高峰资助项目(ZBZZ-019);常州市科技支撑(社会发展)项目(CE20185029)
孙 俊,教授,博士,博士生导师。研究方向为计算机技术在农业工程中的应用。Email:sun2000jun@ujs.edu.cn
孙 俊,靳海涛,芦兵,武小红,沈继锋,戴春霞. 基于高光谱图像及深度特征的大米蛋白质含量预测模型[J]. 农业工程学报,2019,35(15):295-303. doi:10.11975/j.issn.1002-6819.2019.15.036 http://www.tcsae.org
Sun Jun, Jin Haitao, Lu Bing, Wu Xiaohong, Shen Jifeng, Dai Chunxia. Prediction model of rice protein content based on hyperspectral image and deep feature[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2019, 35(15): 295-303. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2019.15.036 http://www.tcsae.org

