印度南瓜高通量转录组测序与数据分析
刘建汀,温文旭,朱海生*,王 彬,白昌辉,张前荣,温庆放*
(1. 福建省蔬菜遗传育种重点实验室,福建 福州 350013;2. 福建省农业科学院作物研究所,福建 福州 350013;3. 福建省蔬菜工程技术研究中心,福建 福州 350013)
【研究意义】印度南瓜(CucurbitamaximaDuch.)作为葫芦科(Cueurbitaceae)南瓜属(Cucurbita)重要的蔬菜作物,在我国各地普遍栽培,种植面积和产量均居世界前列[1]。印度南瓜肉质致密,富含维生素C、胡萝卜素和多糖多酚等,栽培面积逐年扩大,具有良好的市场效益[2]。目前印度南瓜的研究多集中于营养成分[3-4]、遗传育种[5-9]、栽培技术[10]和采后生理[11-13]等方面。了解印度南瓜农艺性状的基因定位及其控制次生代谢途径基因的类型和功能[14-16],有助于推进印度南瓜营养价值的开发及利用和优良品种的选育研究。【本研究切入点】近年来,关于印度南瓜在医疗保健方面研究已受到广泛关注,并逐渐成为研究印度南瓜的一个热点,这对于印度南瓜优良品种选育、各类生物代谢合成途径以及基因资源丰富程度提出了更高要求。高通量测序技术极大地推动了非模式物种的全基因组测序工作,能够快速、精准地获得所需丰富分子标记信息、目的基因资源和代谢通路类型。【前人研究进展】利用Illumina HiSeq 2000高通量测序平台获得的转录组信息可以为后续开展印度南瓜功能基因功能鉴定、分子标记以及相关代谢途径的鉴定提供参考[17-18]。林艳玲[19]利用Illumina Hi SeqTM2000系统进行转录组测序,共获得73 434条Unigenes,平均长度为877 nt,分别有51 407、37 640、17 735、29 197条Unigenes比对到Nr、GO、COG功能分类以及KEGG经典代谢通路当中。Wyatt等[20]报道了美洲南瓜果实和种子的转录组,通过Illumina HiSeqTM2000测序得到141 838 600个读序,组装获得55 949个Unigene,在公共数据库(Swiss-prot,TrEMBL,TAIR10,Nr)注释到的功能基因超过62 %。近两年,印度南瓜相关的转录组测序已陆续开展,这对于印度南瓜基因资源挖掘、基因功能鉴定以及基因组学相关的研究分析提供了可靠的科学基础[21-22]。【拟解决的关键问题】本研究通过Illumina HiSeq 2000高通量测序平台对印度南瓜转录组进行测序,利用公共数据库对测序结果进行生物信息学分析,为印度南瓜多糖多酚、类黄酮、莨菪烷、哌啶和吡啶生物碱等重要的生物代谢途径及其相关功能基因和分子标记信息的开发奠定基础。
1 材料与方法
1.1 试验材料
测序材料印度南瓜取自福建省农业科学院作物所蔬菜中心福清东张基地蔬菜育苗大棚。试验于2017年3月1号选取50粒饱满的印度南瓜种子,清洗干净后用次氯酸钙溶液(100~200 mL/L)消毒10 min,再次用清水洗净残留的次氯酸钙后播种到育苗盘中。于2017年3月15日上午9时从20株生长正常、大小相似的印度南瓜幼苗中随机选取3株作为生物学重复,活体包装后送至北京百迈客生物科技有限公司(Biomarker Technologies Co,LTD,Beijing)测序。研究采用Trizol法分别提取3株印度南瓜幼苗叶片总RNA,各样本分别取等量混合组成3个RNA池,并依次采用Qubit 2.0、Nanodrop和Aglient 2100分析检测印度南瓜RNA样品的浓度、纯度和完整性。通过以下3个步骤构建印度南瓜cDNA文库:①用磁珠和磁分离器分离并纯化出mRNA,将纯化后的mRNA进行随机打断并作为模板,利用random hexamers作为引物反转录合成第一条cDNA链;②加入反应液(由缓冲液、dNTPs、RNA聚合酶H和DNA聚合酶I)合成第二条cDNA链;③利用AMPure XP beads核酸试剂纯化cDNA,再加EB缓冲液洗脱之后进行末端修复、C端加A尾并连接测序接头、利用琼脂糖凝胶电泳选择片段大小,最后通过PCR扩增得到印度南瓜转录组cDNA文库。
1.2 印度南瓜转录组测序
通过Illumina HiSeq 2000测序平台和PE125测序方法对1.1中构建好的印度南瓜cDNA文库进行高通量测序,测序得到的原始图像数据经过滤得到纯化后到高质量的读序(clean reads),再利用Trinity软件进行转录组de dovo分析组装。Trinity通过序列之间的overlap信息组装得到Transcripts,最后用TGICL聚类和Phrap拼接软件分别对Transcripts进行同源聚类和拼接获得不含N的组装片段(Unigene)。印度南瓜转录组分析项目包括测序组装结果分析、Unigene基因功能注释及分类、基因表达量(FPKM,Fragments Per Kilobase of transcript per Million mapped reads)统计分析以及SSRs特征分析等。
1.3 印度南瓜基因功能注释
通过Blastx(https://blast.ncbi.nlm.nih.gov/Blast.cgi)下线比对工具,将印度南瓜Unigene与蛋白数据库进行比对(设定E值≤1E-5),匹配相似性>30 %的基因功能注释信息,选取与其Unigene序列编码具备最高相似性的蛋白作为该Unigene的蛋白功能注释信息。蛋白比对数据库包括非冗余蛋白数据库Nr(Non-redundant protein database,ftp://ftp.ncbi.nih.gov/blast/db/)、蛋白质序列数据库SwissProt(http://www.uniprot.org/,SwissProt protein database)、蛋白质直系同源和功能注释数据库eggNOG(v4.5)(A database of orthologous groups and functional annotation,http://eggnogdb.embl.de/)、真核生物蛋白质同源数据库KOG(euKaryotic Orthologous Groups,http://www.ncbi.nlm.nih.gov/KOG/)、蛋白质直系同源数据库COG(Cluster of Orthologous Groups,http://www.ncbi.nlm.nih.gov/COG/)、蛋白质家族域数据库Pfam(Protein families database,http://pfam.xfam.org/)、基因本体论数据库GO(Gene Ontology,http://www.geneontology.org/)、东京基因与基金组百科全书KEGG(Kyoto Encyclopedia of Genes and Genomes,http://www.genome.jp/kegg/)。
2 结果与分析
2.1 印度南瓜转录组测序结果分析与组装
通对印度南瓜嫩叶进行转录组测序,共获得26 083 711个片段(reads),其中包含7789 098 902(7.79 Gb)个核苷酸序列信息;各样品Q30碱基百分比均不小于93.11 %,GC含量为46.64 %。该结果表明,印度南瓜转录组测序数据量和质量都很高,因此,可用于为后续的数据组装提供原始数据。
随后利用Trinity软件进行序列组装,组装共获得179 524条Transcript。总序列信息达222 022 708 bp(0.22 Gb),平均长度1236.73 nt,Transcript的N50为1947 bp。其中各长度占比如图1所示,长度200~300、300~500、500~1000、1000~2000 bp、≥2000 bp分别占17.81 %、15.74 %、18.49 %、27.25 %、20.72 %。所得Transcript序列再次组装后得到68 073条Unigene,Unigene总序列信息达44 206 542 bp(44.2 Mb),平均长度649.40 bp,长度200~300、300~500、500~1000、1000~2000 bp、≥2000 bp分别占38.47 %、27.60 %、16.48 %、11.19 %、6.25 %。本研究中Unigene的N50为1070 bp,表明印度南瓜转录组测序组装完整性较高。

图1 Transcript和Unigene长度分布Fig.1 Transcript and Unigene length distribution
将建库后的各印度南瓜样品Clean Data与组装得到的Transcript或Unigene库进行序列比对,结果表明,测序得到的26 083 711个reads中有19 901 135个reads具有表达量,占总量的76.3 %,其中仅比对到1个位置的reads有9521 986个(占47.85 %),而比对到2个或2个以上位置的reads(多基因家族序列)为10 379 149个(占52.15 %)。所有印度南瓜68 073个Unigene的FPKM平均值为14.03,其中最大值为59 317.29(ID:c27343.graph_c0),最小值(除0外)为0.10(ID:c48851.graph_c0),FPKM值为0的Unigene有10 699个,其中有11 659个Unigene的FPKM值大于10,另有18 038个Unigene基因的FPKM值小于1。
2.2 印度南瓜Unigene功能注释
使用BLAST[23]软件将西葫芦转录组测序获得的Unigene序列分别与六大数据库Nr、Swiss-Prot、GO、COG、KOG、eggNOG 4.5和KEGG进行比对。利用KOBAS 2.0和HMMER软件分别与KEGG、Pfam数据库比对获取Unigene响应的注释信息。在各数据库获得的功能注释Unigene统计数如图2所示,总共有38 177条Unigene在上述8个数据库得到注释,其中,Nr数据库注释的Unigene最多,为37 542条(占总Unigenes的98.34 %),其次为Pfam数据库,为33 927条,在COG数据库中注释的结果最少,为9938条(仅占总Unigenes的26.03 %)。

图2 Unigene注释统计Fig.2 Annotation statistics of Unigene
2.3 印度南瓜Unigene的Nr及SwissProt数据库比对分析
通过BLAST程序对印度南瓜测序后获得的68 073个Unigene进行Nr和SwissPort数据库比对分析(E≤1E-5),由图3-A可知,37 542个(55.15 %)Unigene在Nr数据库中能找到相似序列。其中E值小于1E-150的Unigene有7986个(21.27 %),E值介于1E-100~1E-150的Unigene有3622个(9.65 %),E值介于1E-50~1E-100的Unigene有8389个(22.35 %),E值介于1E-5~1E-50的Unigene有17 545个(46.73 %)。Nr功能注释匹配的物种如图3-B所示,其中香瓜13 737个Unigene(36.62 %)、黄瓜13 347个Unigene(35.58 %)、可可2346个Unigene(6.25 %)、拟南芥720个Unigene(1.92 %)、葡萄662个Unigene(1.76 %)、木棉595个Unigene(1.59 %)、香橙(287个Unigene(0.77 %)、毛果杨215个Unigene(0.57 %)、巴旦木210个Unigene(0.56 %)、芝麻210个Unigene(0.56 %)等。匹配序列相似度(identity)如图3-C所示,相似度80 %以上的Unigene有25 848个(68.85 %),相似度40 %~80 %的Unigene有11 532个(30.72 %),相似度低于40 %的Unigene有162个(0.43 %)。
与Nr相比SwissProt数据来源较少,因此可找到相似度高(E<1e-150,相似度>80 %)序列的Unigene大幅减少。本研究中印度南瓜Unigene在SwissPort数据库共找到23 946个(35.18 %)相似序列。如图3-D所示,其中E值小于1E-150的Unigene有3257个(13.60 %),E值介于1E-100~1E-150的Unigene有2125个(8.87 %),E值介于1E-50~1E-100的Unigene有4891个(20.43 %),E值介于1E-5到1E-50的Unigene有13 673个(57.10 %);SwissPort功能注释匹配的物种图3-E,其中拟南芥18 422个(76.93 %)、水稻1109个(4.63 %)、番茄253个(1.06 %)、大豆250个(1.04 %)、豌豆230个(0.96 %)、马铃薯226个(0.94 %)、玉米182个(0.76 %)、黄瓜136个(0.57 %)、菠菜123个(0.51 %)等。由图3-F可知,相似度80 %以上的Unigene有6417个(26.80 %),相似度40 %~80 %的Unigene有15 485个(64.67 %),相似度低于40 %的Unigene有2044个(8.54 %)。
2.4 印度南瓜Unigene的Go数据库分类
GO(Gene Ontology)数据库是基因功能国际标准化分类体系,包含生物学过程(Biological Process)、细胞组分(Cellular Component)和分子功能(Molecular Function)3个部分,分别描述了基因产物可能参与的生物学过程、所处的细胞环境和行使的分子功能。由图4可知,21 414个Unigene被分为3个本体51个功能组,46 420个GO条目被分类到细胞组分的17个功能组中,其中细胞部分(1036 422.33 %)、细胞(1036 422.33 %)、细胞器(775 316.70 %)以及膜(589 812.71 %)功能组中涉及的Unigene较多;26 165个GO条目分类到16个分子功能组,其中催化活性(1133 843.33 %)和结合活性(1053 340.26 %)功能组中涉及的Unigene较多;65 232个GO条目被分类到20个生物学过程功能组,代谢进程(1476 122.63 %)、细胞进程(1303 119.98 %)以及单一生物进程(1124 717.24 %)功能组中涉及的Unigene较多。
由图4可知,21 414个Unigene被分为3个本体51个功能组,46 420个GO条目被分类到细胞组分的17个功能组中,其中细胞部分(10 364,22.33 %)、细胞(10 364,22.33 %)、细胞器(7753,16.70 %)以及膜(5898,12.71 %)功能组中涉及的Unigene较多;26 165个GO条目分类到16个分子功能组,其中催化活性(11 338,43.33 %)和结合活性(10 533,40.26 %)功能组中涉及的Unigene较多;65 232个GO条目被分类到20个生物学过程功能组,代谢进程(14 761,22.63 %)、细胞进程(13 031,19.98 %)以及单一生物进程(11 247,17.24 %)功能组中涉及的Unigene较多。
2.5 印度南瓜Unigene的KOG、COG和eggNOG数据库分类
通过KOG、COG和eggNOG数据库比对Unigene,可对获得注释的Unigene进行蛋白功能描述和功能分类。本研究将印度南瓜Unigene与KOG、COG和eggNOG数据库进行比对,并对其结果进行功能分类统计。结果表明,KOG、COG和eggNOG数据库分别注释到20 011个、9938个和33 927个Unigene,根据其功能划分为25个类(表1)。其中KOG数据库中一般功能预测(3456个)注释到的Unigene最多,其次是翻译后修饰、蛋白折叠和分子伴侣(2017个),而细胞运动(7个)注释到的Unigene最少;COG数据库中同样是一般功能预测(2551个)注释到的Unigene最多,而胞外结构未注释到Unigene,核结构中仅注释到1个Unigene;eggNOG数据库中功能未知的Unigene有7665个,其次是一般功能预测6165个,而细胞运动仅注释到3个Unigene。由表1可知,印度南瓜转录组测序结果极为丰富,获得的Unigenes涉及到了植物生长发育过程中的所有生命活动。

表1 印度南瓜Unigene的KOG、COG和eggNOG数据库分类
2.6 印度南瓜Unigene的Pfam数据库分析
Pfam(Protein family)数据库建立了每个蛋白质家族的氨基酸序列的HMM(Hidden Markov Model)统计模型,是目前最全面的蛋白质结构域注释的分类系统,可用于识别蛋白的结构域序列,从而初步预测蛋白质的功能。将印度南瓜的68 073个Unigene进行Pfam数据库编码蛋白结构域功能分析,Pfam数据库注释到21 823个Unigene,共分为7311类。Pfam数据库注释到最多的蛋白结构域为蛋白激酶结构域(Protein kinase domain)共计906个Unigene,其次为蛋白酪氨酸激酶(Protein tyrosine kinase)872个Unigene和PPR重复家族(PPR repeat family)632个Unigene;其他注释到的数量较多的蛋白功能区域分别为WD结构域(WD domain)243个、G-beta重复(G-beta repeat)228个、细胞色素P450(Cytochrome P450)214个、反转录酶(Reverse transcriptase)47个、线粒体载体蛋白(Mitochondrial carrier protein)93个、AP2结构域(AP2 domain)99个、转移酶家族(Transferase family)271个、RNA识别基序(RNA recognition motif)259个、WRKY转录因子结构域(WRKY DNA-binding domain)56个、螺旋-环-螺旋结构域(Helix-loop-helix DNA-binding domain)80个、NB-ARC结构域(NB-ARC domain)25个、GRAS家族(GRAS domain family)59个、类GDSL脂肪酶/酰基水解酶(GDSL-like Lipase/Acylhydrolase)60个。
2.7 印度南瓜Unigene的KEGG数据库功能注释
KEGG是系统分析基因产物功能及其在细胞中参与代谢途径的数据库,通过KEGG分析能够把基因及其表达信息形成一个整体的研究网络。本研究中印度南瓜转录组测序获得68 073个Unigene序列,其中有15 074个Unigene在KEGG数据库中得到注释,并共涉及到的127个代谢途径(表2)。其中注释较多Unigene的有:核糖体代谢途径(676个,ID:ko03010)、碳代谢代谢途径(665个,ID:ko01200)、氨基酸的生物合成代谢途径(595个,ID:ko01230)、植物激素信号传导代谢途径(529个,ID:ko04075)、内质网蛋白质处理代谢途径(429个,ID:ko04141)等。注释较少的Unigene有:花色素苷生物合成代谢途径(ID:ko00942)和芥子油苷的生物合成代谢途径(ID:ko00966)仅识别到1个Unigene。另外,类胡萝卜素生物合成代谢途径(86个,ID:00906)、N-多糖生物合成代谢途径(82个,ID:ko00510)以及类黄酮生物合成代谢途径(52个,ID:ko00941),这些代谢途径中的Unigene为之后开展印度南瓜次生代谢产物合成途径及其分子调控奠定了基础。

表2 印度南瓜Unigene的KEGG代谢途径分析

续表2 Continued table 2

续表2 Continued table 2

续表2 Continued table 2
2.8 印度南瓜转录组SSRs特征分析
从印度南瓜68 073个Unigene中筛选出大于1 Kb以上的Unigene共11 871个,借助MISA(A MIcroSAtellite identification tool,http://pgrc.ipk-gatersleben.de/misa/misa.html)软件,分别按单碱基类型(Mono-nucleotide type)、双碱基类型(Di-nucleotide type)、三碱基类型(Tri-nucleotide type)、四碱基类型(Tetra-nucleotide type)、五碱基类型(Penta-nucleotide type)、六碱基类型(Hexa-nucleotide type)重复和混合类型(Compound type,位点≥2)SSR等进行搜索,搜索结果标记为p1、p2、p3、p4、p5、p6和c(表3~4)。印度南瓜含有SSR位点的Unigene共5391个,其中含有单碱基重复(p1)类型的Unigene最多,共2906个占总数的53.90 %;双碱基重复(p2)类型的Unigene共942个,三碱基重复(p3)类型的Unigene有1096个,四碱基重复(p4)类型的Unigene共89个,五碱基重复(p5)类型的Unigene共10个,六碱基重复(p6)类型的Unigene共18个。含有至少2个SSR位点的Unigene共319个,含有至少2个位点且存在共用碱基的类型有11个。通过对印度南瓜SSR位点进行分析,了解其组成和类型分布特征,可为进一步开展印度南瓜及其近缘种遗传图谱构建、基因组差异表达分析及其通用性引物设计等奠定科学基础。
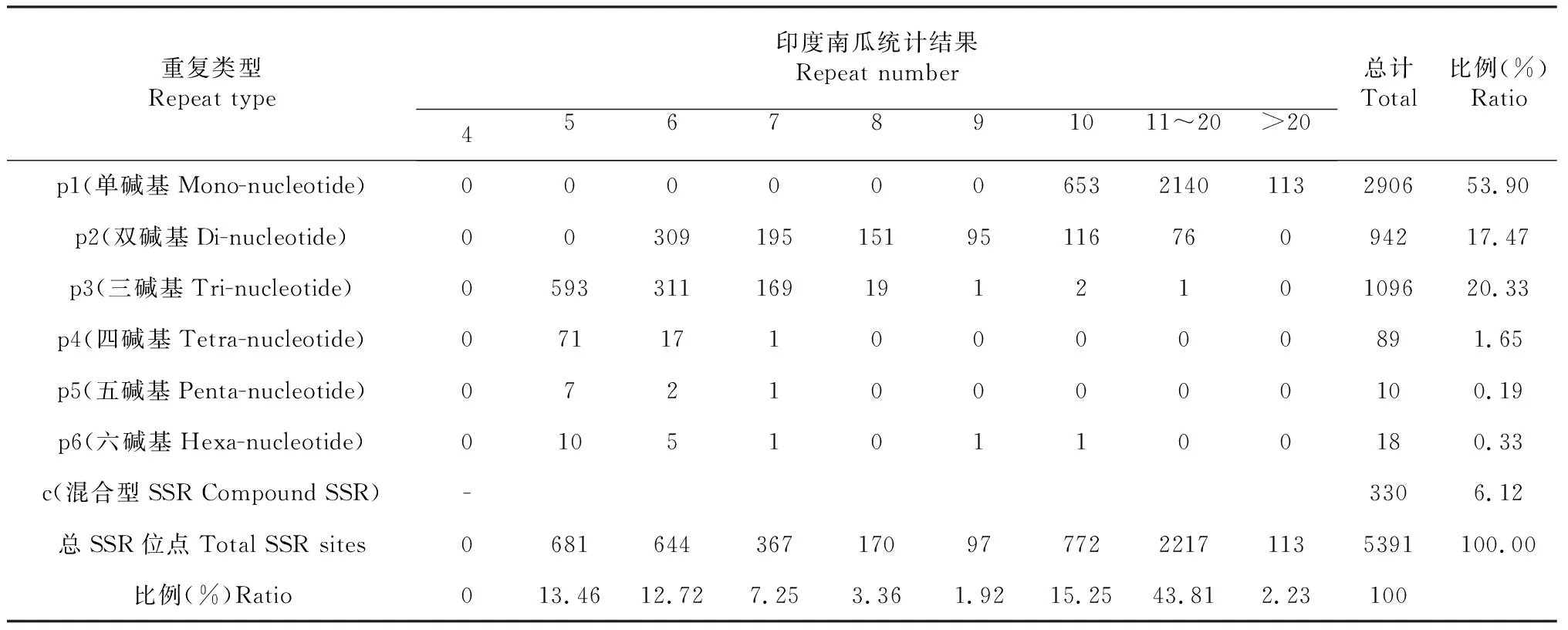
表3 印度南瓜SSR优势碱基组成
3 讨 论
近年来,转录组测序技术在多种植物基因组及合成生物学等研究方面应用广泛,并取得重大进展。Zhu等[24]利用转录组测序(RNA-seq)技术对普通丝瓜品种“福丝3号”进行鲜切褐变差异分析,获得58 073条有效序列,从中筛选出27 301条Unigene在丝瓜鲜切褐变不同时间段差异表达基因(DEGs),并获得了15条差异表达全长基因序列。在南瓜属作物中,Wu等[25]使用Illumina HiSeqTM2000对中国南瓜进行了转录组测序,得到52 849 316个读序组装后得到62 480个Unigene,在Nr、Swiss Port和COG中分别注释到了47 596、34 368和16 700个功能基因,通过筛选获得了4794对SSR引物。本研究对印度南瓜进行转录组测序,从3个RNA池中共获得26 083 711个reads,包含了7 789 098 902(7.79 Gb)个核苷酸序列信息,Q30小于93.11 %,GC含量为46.64 %。组装获得179 524条Transcript,Transcript序列再次组装后得到68 073个Unigene,平均长度649.40 bp,N50为1070 bp,表明印度南瓜测序测序质量较好,能为后续数据分析提供很好的原始数据。

表4 印度南瓜SSR重复基序分布情况
前人研究表明,在不同植物的转录组数据结果分析中,皆存在大量Unigene未获得匹配的情况,如:玉米[26]、亚麻芥[27]、龙眼[28]、喀西茄[29]等,未注释到的Unigene与其长度和数据库信息缺乏有关。本研究通过Illumina HiSeqTM2000测序平台对印度南瓜转录组进行测序,通过拼接获得了68 073个印度南瓜Unigene。研究通过进一步对组装获得的Unigene在Nr、Swiss-Prot、GO、COG、KOG、eggNOG4.5、KEGG、Pfam等公共数据库进行了比对,获得功能注释的Unigene共计38 177个,占Unigene总数56.08 %。进一步分析发现,在Nr数据库中找到37 542个相似序列,其中36.62 %(13 737个)的Unigene注释匹配到甜瓜;35.58 %(13 347个)Unigene注释匹配到黄瓜;在GO数据库中21 414个Unigene被分为3个本体51个功能组,印度南瓜的Unigene几乎涵盖了植物所有功能,但是仍然存在较多的Unigene未被注释需要利用其他数据库进一步补充。研究表明,印度南瓜提取物中含有抗癌、抗糖尿病和抗肥胖等重要功效[30-33]。本研究获得注释到的Unigene广泛涉及各类生命代谢活动,KEGG数据库中共注释到15 074个Unigene分布在127个代谢途径中,其中涉及到了类黄酮、莨菪烷、哌啶、吡啶生物碱、花色素苷和芥子油苷等次生代谢产物合成途径。印度南瓜转录组测序KEGG代谢通路分析表明共有52条Unigene参与到类黄酮代谢通路中,该代谢途径能够有效合成许多具有抗癌、抗氧化、抗病毒、增强免疫力等多种功能的黄酮类化合物,这为今后从印度南瓜成熟果实入手挖掘控制黄酮类化合物的生物合成关键基因提供了重要的基因资源[34]。
目前已开发的南瓜属SSR标记主要来自于美洲南瓜的基因组以及转录组[35-36]、印度南瓜基因组以及转录组[37-38]和中国南瓜转录组[39-40]数据,这些SSR标记具有一定通用性,由于同一个属内不同种之间具有一定保守性,且在印度南瓜上表现为多态性较低,限制了其在印度南瓜上的应用。基于转录组的SSR标记较一般的分子标记具有信息量大和通用性好的优势。朱海生等[41]对美洲南瓜转录组测序获得的SSR种类较为丰富,共检测出7478个SSR位点,各种重复类型的出现频率有较大差异,含6种SSR重复类型,其中单核苷酸占总SSR的47.90 %。本研究从印度南瓜68 073个Unigene中筛选出5391个SSR位点,其中含有单碱基重复类型的Unigene最多,共2906个占总数的53.90 %,表明在印度南瓜中和美洲南瓜测序获得的结果相似,均以单碱基型重复所占比例居多。本研究为印度南瓜种质资源遗传多样性分析、遗传图谱构建、基因定位与克隆及分子标记辅助育种等奠定了基础。现有的印度南瓜SSR分子标记数量远远不能满足印度南瓜分子生物学研究的需求,因此,大量开发SSR标记仍是目前印度南瓜研究的重要工作之一,今后需增加南瓜品种测试包括中国南瓜和美洲南瓜,以进一步获得较高频率的SSR位点和丰富的SSR类型。
4 结 论
研究获得印度南瓜获得质量较好的转录组测序,并对组装获得的Unigene进行基因功能注释以及KEGG代谢通路和SSR序列基本特征分析,为后续基因资源挖掘、基因功能鉴定以及遗传多样性分析和遗传图谱构建提共依据。

