TaNAC14转基因小麦外源目标基因拷贝数及遗传稳定性分析
张立坚,池 青,杜琳颖,郭利建,李玉莲,刘香利,3,马 猛,3,赵惠贤,3
(1.西北农林科技大学生命科学学院,陕西杨凌 712100; 2.山东省农业科学院作物研究所,山东济南 250100;3.西北农林科技大学旱区作物逆境生物学国家重点实验室,陕西杨凌 712100)
小麦(Triticumaestivum)作为世界三大主要粮食作物之一,其产量直接关系着世界粮食安全。1992年Vasil等首次获得转基因小麦[1],此后许多小麦研究工作者尝试通过转基因技术进行小麦遗传育种改良[2-3]。目前小麦遗传转化主要采用基因枪法和农杆菌介导法[3-4],基因枪法转化效率不高,通常因为外源基因会出现多位点插入整合,导致外源基因沉默[5];然而农杆菌介导的遗传转化,转基因植株外源基因插入整合位点和转基因拷贝数较少[2]。外源基因以单拷贝插入整合到受体植物中时,其表达水平较高且遗传稳定性较好;而外源基因以多拷贝插入整合到受体植物时,其表达水平很低甚至会导致基因沉默,而且目标基因的遗传稳定性也较差[6-7]。因此,在小麦转基因研究中,进行T0代转基因再生植株外源基因拷贝数检测,筛选获得单拷贝转基因植株是非常关键的环节,该步骤为后续筛选获得遗传稳定且高效表达的转基因株系奠定了基础[8]。
检测转基因植物外源基因拷贝数最经典的方法是Southern印迹杂交(Southern blot)[9],该方法准确度较高,但也存在DNA样品用量大、操作步骤繁琐、部分检测步骤还需要放射性同位素等缺陷[10]。针对这些不足,近年来研究者建立了样品用量少、简单、快速、灵敏度高和高通量的实时定量PCR(qRCR)方法来检测转基因植物中外源基因的拷贝数[11]。Weng等[12]同时用Southern blot和TaqMan qPCR两种方法对转基因油菜(Brassicanapus)进行外源基因拷贝数检测,结果表明用TaqMan qPCR方法测定的拷贝数和用Southern blot测定得到的拷贝数是一致的,从而证明用TaqMan qPCR检测转基因植物外源基因拷贝数的方法体系是准确可靠的。刘振华等[13]以外源脂氧合酶(LOX)抑制基因(Loxi)的转基因小麦株系为材料,用TaqMan qPCR方法快速准确地测定了其外源基因拷贝数,同时分析了转基因小麦株系和非转基因对照的种子LOX活性,结果发现外源Loxi的拷贝数与种子LOX活性呈负相关。
转录因子是真核生物细胞内一类具有转录调控功能的蛋白质,它可以通过与特定基因启动子区特定的顺式作用元件结合,调控靶标基因在生物体内的时空表达[14]。NAC转录因子是一类植物特异的转录因子家族,在植物生长发育过程中起着重要的作用[15]。Mohammed等[16]研究发现拟南芥(Arabidopsisthaliana)和水稻(Oryzasativa)分别有154个和117个NAC转录因子成员,这些NAC转录因子广泛参与调控植物生长发育和响应逆境胁迫。在拟南芥NAC转录因子家族成员中,CUC1、CUC2和CUC3等基因均参与分生组织和器官边界的形成[17];根尖和侧根中高表达的NAC1基因受吲哚乙酸(IAA)的诱导,能促进侧根的形成[18];拟南芥ANAC019、ANAC055和ANAC072基因的诱导表达,可以显著提高植株的抗旱能力[19-21]。Jiang等[22]发现在水稻中过表达OsNAC2,可以显著影响水稻的株型和产量。
基于全基因组序列信息预测,Guerin等[23]发现小麦NAC转录因子家族共包含488个成员,在小麦响应逆境胁迫等生物学过程中发挥着重要的作用。Xia等[24]证明TaNAC8对小麦条锈病病原感染和非生物胁迫都有响应。Uauy等[25]发现小麦中调控衰老的NAC转录因子可以改善籽粒中蛋白质、锌和铁的含量。Chen等[26]发现转基因过表达TaRNAC1能促使小麦根长和生物量增加,以及抗旱性提高。本课题组克隆得到TaNAC14基因,将TaNAC14与TaRNAC1这两个基因进行序列比对,并在最新小麦基因组序列IWGSC Ref.v1.0[27]进行搜索,结果发现TaNAC14(TraesCS2B02G343600)与TaRNAC1(TraesCS2D02G324700)高度相似,达97.65%,TaNAC14位于小麦B基因组上,TaRNAC1位于小麦D基因组上,二者是来自不同亚基因组的旁系同源基因。小麦miRNA及降解组研究发现TaNAC14是miRNA164的调控靶标之一,表达谱分析显示TaNAC14/TaRNAC1在小麦籽粒发育时期表达水平较高(文章尚未发表)。但是,目前TaNAC14/TaRNAC1在小麦籽粒生长发育中的生物学功能尚不清楚。
为了揭示TaNAC14基因在小麦籽粒生长发育中的生物学功能,本课题组采用农杆菌介导法获得了转TaNAC14基因小麦。本研究主要对转基因小麦中插入整合外源基因TaNAC14的拷贝数进行测定,同时对转基因小麦中外源基因的遗传稳定性和表达水平进行分析,旨在筛选出单拷贝、能够稳定遗传并且能高水平表达的TaNAC14转基因小麦株系,为后续TaNAC14的功能研究奠定基础,为小麦育种提供新的遗传稳定的种质材料。
1 材料与方法
1.1 材料与载体
植物材料为春性小麦JW1(野生型,转基因受体材料)和14个T0代转基因小麦再生植株(通过农杆菌介导的pCAMBIA3301-P35S-TaNAC14遗传转化而来),由济南邦迪生物科技有限公司提供。植物表达载体pCAMBIA3301-P35S-TaNAC14由本实验室构建,其T-DNA区结构如图1所示。
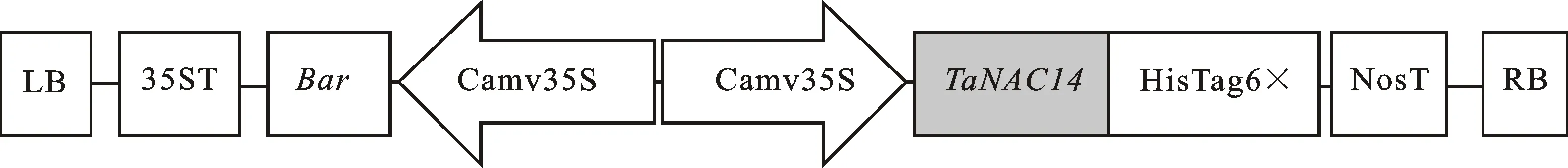
LB:左边界;RB:右边界;35S T:35S终止子;Bar:筛选标记基因,通过编码膦丝菌素乙酰转移酶来增强植株对BASTA的耐受性;Camv35S:花椰菜花叶病毒35S启动子;HisTag6×:6×His标签;Nos T:Nos基因终止子。
1.2 仪器与试剂
定量仪器为CFX96实时定量PCR Detection System(BIO-RAD);2 × Es Taq MasterMix购自康维世纪生物有限公司;Premix Ex TaqTM(Probe qPCR)(RR390A)、TB GreenTMPremix Ex TaqTMII(Tli RNaseH Plus)(RR820Q)和反转录试剂盒PrimeScriptTMRT Master Mix(Perfect Real Time)(RR036A)均购自大连宝生物有限公司;RNA提取试剂盒(RP3202)购自北京百泰科生物技术有限公司。
1.3 方法
1.3.1 小麦基因组DNA的提取
采用CTAB法提取三叶期小麦叶片基因组DNA[28]。DNA样品的浓度利用电泳检测和分光光度计确定,通过比较波长230 nm吸光值(OD230)、260 nm吸光值(OD260)和280 nm吸光值(OD280)的比值以判断DNA样品的纯度。
1.3.2 小麦总RNA的提取及cDNA 的合成
取9个T1代阳性植株和野生型JW1的三叶期植株叶片,用RNA提取试剂盒提取总RNA,用反转录试剂盒合成第一链cDNA。反转录条件为:25 ℃退火5 min,42 ℃延伸30 min,70 ℃反转录酶失活15 min,获得 cDNA。
1.3.3 T0代转基因小麦再生植株的鉴定
本研究根据前期研究的方法[29],用100 mg·L-1的BASTA溶液均匀涂抹于三叶一心期的野生型小麦JW1和14株T0代转基因小麦的第一片完全展开叶片上,涂抹面积为整个叶片的1/3,于温室18 ℃培养7 d后观察植株叶片表型。
因为转基因小麦植株中插入序列含有TaNAC14+Nos(终止子)序列,而TaNAC14来自于小麦本身,所以本研究以目标基因TaNAC14之后的Nos序列为PCR扩增的目标序列,对T0代转基因植株进行PCR鉴定。用Primer 5.0设计序列特异引物,进行序列特异PCR扩增检测转基因小麦再生植株中的阳性植株,引物由西安擎科泽西生物科技有限责任公司合成(表1)。目的片段长度为192 bp。PCR反应体系为:2×Es Taq MasterMix 0.2 μL,上游引物Nos-F(10 μmol·L-1)0.5 μL,下游引物Nos-R(10 μmol·L-1)0.5 μL,模板(50 ng·μL-1)2 μL,ddH2O补至20 μL。PCR反应条件为:94 ℃预变性4 min;94 ℃变性1 min,58 ℃退火30 s, 72 ℃延伸15 s,35个循环;72 ℃延伸10 min。PCR产物用1%的琼脂糖凝胶电泳分离,核酸染料染色,用凝胶成像仪观察。比较BASTA涂抹和PCR结果,统计T0代转基因小麦阳性植株和阴性植株的数量。

表1 TaNAC14基因的探针引物和定量引物信息
1.3.4 TaqMan qPCR扩增
以小麦内源控制籽粒硬度的主效单拷贝基因Pinb(Puroindolineb,GenBank登录号:LOC543301)为内参基因,以转基因小麦植株中目标基因TaNAC14之后的Nos序列为外源目标序列进行TaqMan qPCR扩增。内参基因和外源目标序列的探针引物设计通过IDT(Integrated DNA Technologies)网站中Primer Quest Tool设计(http://sg.idtdna.com/Primerquest/Home/Index),由Invitrogen公司(上海)合成(表1);普通引物采用Primer 5.0进行设计分析,由西安擎科泽西生物科技有限责任公司合成(表1)。PCR反应体系为:2 × Premix Ex Taq 12.5 μL,模板(50 ng·μL-1)1 μL,上下游引物(10 μmol·L-1)各0.5 μL,TaqMan探针(5 μmol·L-1)1.5 μL,ddH2O补至25 μL。PCR反应条件为:95 ℃预变性30 s;95 ℃变性5 s, 60 ℃延伸20 s,40个循环。每个样品设置3次生物学重复,每个重复样品进行3次技术重复。
1.3.5 内参基因Pinb和外源序列Nos扩增标准曲线的建立
以野生型小麦JW1为阴性对照,ddH2O为空白对照,以5倍梯度稀释的转基因植株TaNAC14-3的基因组DNA为模板进行qPCR,设置3个重复,获得不同浓度模板的Ct值,根据不同浓度模板对应的Ct值与其浓度对数值LogC0(C0为qPCR的起始浓度) 之间的关系分别建立Pinb和Nos的扩增标准曲线。
1.3.6 T0代转基因小麦再生植株中外源序列Nos拷贝数的检测
转基因小麦外源目标基因拷贝数的测定参考Weng等[12]的方法。
拷贝数计算公式:
X0/R0=10[(Ctx-Ix)/Sx-(CtR-IR)/SR],其中X0和R0分别为外源序列Nos和内参基因Pinb的拷贝数,CtX和CtR分别为外源序列Nos和内参基因Pinb的PCR扩增荧光量达到阈值的循环数,IX与IR分别为外源序列Nos和Pinb标准曲线的截距,SX和SR分别为外源序列Nos和内参基因Pinb标准曲线的斜率。本研究采用的内参基因Pinb为小麦中的单拷贝纯合基因,因此公式中的R0为2。
1.3.7 单拷贝转基因小麦再生植株T0:1代株系转基因的遗传稳定性分析
将上述鉴定的T0代单拷贝转基因小麦植株收获的种子在温室中加代种植,获得T1代转基因小麦株系。将100 mg·L-1的BASTA溶液均匀涂抹于三叶一心期的野生型小麦和T1代转基因小麦植株完全展开叶片,鉴定和统计T1代转基因阳性和阴性植株数目,分析转基因小麦T0:1代株系转基因的遗传情况。
1.3.8 T1代转基因小麦株系目的基因表达水平测定
以9株T1代阳性植株和野生型JW1的cDNA为模板,通过qPCR来检测目标基因的表达水平,以小麦β-actin(GenBank登录号:MF405765.1)基因作为内参进行归一化处理。qPCR以野生型小麦JW1为阴性对照,ddH2O作为空白对照,每个样品设置3个独立的生物学重复。检测引物NAC14-F、NAC14-R、ACTIN-F和ACTIN-R采用Primer 5.0进行设计,由西安擎科泽西生物科技有限责任公司合成(表1)。PCR反应体系:2×TB GreenTMPremix Ex TaqTMII 10 μL,上下游引物(10 μmol·L-1)各0.5 μL,cDNA模板1 μL,ddH2O补至20 μL。PCR反应条件为:95 ℃预变性3 min;95 ℃变性15 s,60 ℃延伸1 min,45个循环。每管PCR扩增反应的特异性由仪器记录的溶解曲线证实。采集分析各样品的Ct值,各样品中目标基因相对表达水平按照改良的2-△△Ct方法[30]计算。用EXCEL软件进行数据统计和分析,用SPSS软件进行差异显著性分析[31]。
2 结果与分析
2.1 T0代转基因小麦再生植株鉴定结果
用100 mg·L-1的BASTA溶液均匀涂抹于三叶一心期的野生型小麦JW1和14个转基因小麦T0代再生植株的第一片完全展开叶片,7 d后观察叶片表型。野生型小麦JW1和部分转基因再生植株的表型如图2所示,野生型小麦JW1涂抹BASTA溶液后叶片发黄枯萎,转基因再生植株中有5株(TaNAC14-2、TaNAC14-6、TaNAC14-7、TaNAC14-8和TaNAC14-11)表型与野生型JW1相同,即涂抹叶片发黄枯萎,表明它们为转基因阴性植株;其他9株再生植株涂抹BASTA溶液后叶片无明显变化,表明这些植株为转基因阳性植株。

JW1:野生型;TaNAC14-2:转基因阴性; TaNAC14-3:转基因阳性;JW1和TaNAC14-2叶片变黄枯萎;TaNAC14-3叶片无明显变化。
进一步以野生型JW1和14株T0代转基因再生植株的基因组DNA为模板,进行目标序列特异PCR扩增,扩增产物用琼脂糖凝胶电泳检测。结果显示阳性对照pCAMBIA3301质粒和上述9株转基因阳性植株均能扩增出大小为192 bp的目的条带,而空白对照(水)、阴性对照野生型小麦JW1和5株转基因阴性植株均没有相应的扩增条带(图3)。

M:Marker DL2000;H2O:ddH2O;N:阴性对照JW1;P:阳性对照pCAMBIA3301质粒;1~14:转基因植株TaNAC14-1~TaNAC14-14。
总之,BASTA溶液涂抹和目标序列特异PCR扩增鉴定结果完全一致,14个转TaNAC14基因小麦的T0代再生植株中有9个为转基因阳性株系。
2.2 T0代转基因小麦再生植株外源序列拷贝数检测结果
2.2.1 TaqMan qPCR扩增及标准曲线的建立
以转基因植株TaNAC14-3基因组DNA的5倍系列稀释液作为模板,分别用内参基因Pihb和目标序列Nos的特异引物和探针引物进行qPCR扩增。
以LogC0为X轴,C0相对应的Ct值为Y轴,分别建立Pinb和Nos的标准曲线(图4A和4B)。由图4可以看出,内参基因Pinb扩增标准曲线的方程为y=-3.139x+35.142,R2= 0.998,纵轴截距IR=35.142,斜率SR= -3.139,PCR扩增效率为107.9%(图4A)。外源序列Nos扩增标准曲线方程为y=-3.109x+31.705,R2= 0.999,纵轴截距IX=31.705,斜率SX= -3.109,PCR扩增效率为108.9%(图4B)。这两个扩增标准曲线的相关系数分别为 0.998和0.999,都接近于1,表明得到的标准曲线能够用于检测T0代转基因小麦植株目标基因拷贝数。

图4 内参基因Pinb(A)和外源序列Nos(B)的标准曲线
2.2.2 T0代转基因小麦植株外源序列的拷贝数
以9株T0代转基因小麦阳性植株基因组DNA为模板,分别用内参基因Pinb和外源序列Nos的特异引物和探针进行TaqMan qPCR扩增,得到的外源序列和内参基因扩增的Ct值见表2。各转基因小麦植株的外源序列拷贝数X0计算结果如表2所示。由表2可以看出,9个T0代转基因小麦阳性株系中有5个为转基因单拷贝,4个为双拷贝。

表2 T0代转基因小麦植株外源序列拷贝数
2.3 外源基因单拷贝插入整合的小麦T0:1株系转基因的遗传稳定性
由于转基因植株的种子萌发率低且植株生长状况不佳,仅获得2个T0:1株系TaNAC14-3(7株)和TaNAC14-9(4株)。采用BASTA涂抹方法鉴定这2个T0:1代小麦株系所有T1代植株转基因阳性与阴性植株的数量(表3),结果表明,TaNAC14-3的7株T1代转基因植株中有5株(TaNAC14-3-1、TaNAC14-3-2、TaNAC14-3-3、TaNAC14-3-5、TaNAC14-3-6)为阳性,TaNAC14-9的4株T1代转基因植株中有2株(TaNAC14-9-1、TaNAC14-9-2)为阳性。这表明本研究得到的外源目标基因TaNAC14是可遗传的。

表3 T0:1转基因小麦株系目标基因的遗传稳定性分析
2.4 T1代转基因小麦植株目标基因的表达量
用RT-qPCR方法,分别对上述得到的7个T1代转基因阳性植株和野生型JW1叶片目标基因的表达水平进行测定。结果(表4)表明,与对照JW1相比,各转基因植株的目标基因TaNAC14表达量均极显著增加,并且2个株系的每个植株间的表达水平存在一定的差异。

表4 转基因小麦植株目标基因 TaNAC14的表达水平
3 讨 论
前人研究报道转基因表达载体上Nos终止子序列的检测结果可被用作判断转基因作物的主要依据[32-33]。因此,通过检测Nos终止子序列的拷贝数来确定T0代转基因小麦植株中插入整合的外源基因拷贝数是可信的。在小麦遗传改良过程中,农杆菌介导的遗传转化法获得的转基因植株,外源目标基因常以低拷贝随机插入整合到受体植株中,这有利于外源目标基因的稳定遗传和表达,为转基因植株的功能研究和育种应用提供了条件[34]。本研究利用TaNAC14基因通过农杆菌介导法对小麦进行遗传转化,通过筛选标记基因和目标序列特异PCR方法鉴定获得了9株T0代转基因阳性植株,其中5株为单拷贝转基因植株,4株为双拷贝,单拷贝插入整合的比率接近 55.6%。
转基因功能研究或转基因作物育种的前提是外源基因能够插入整合至受体基因组中,而且能高水平表达和稳定遗传。本研究对获得的单拷贝插入整合的T0代小麦转基因过表达植株在温室进行加代繁殖,获得了T1代转基因小麦过表达株系。在此基础上,本研究对T0:1代TaNAC14转基因小麦株系的目标基因遗传稳定性进行分析,结果显示T0代转基因植株TaNAC14-3和TaNAC14-9的目标基因均遗传至T1代;进一步用RT-qPCR对T1代转基因小麦植株中目标基因TaNAC14的表达水平进行检测,结果显示,转基因植株TaNAC14的表达量均极显著高于野生型。这说明本研究获得了目标基因TaNAC14能够稳定遗传而且高水平表达的转基因小麦株系,这为后续TaNAC14基因的生物功能研究提供了基础材料。本研究计划后期对TaNAC14的转基因小麦株系继续加代繁殖和筛选鉴定,以期获得纯合的转基因小麦株系,进而研究TaNAC14基因对小麦生长发育和产量性状的影响。
4 结 论
本研究采用BASTA涂抹和PCR检测相结合的方法,从14株T0代TaNAC14转基因再生植株中鉴定出9株阳性植株;用TaqMan qPCR方法检测了9株T0代转基因阳性小麦植株外源基因的拷贝数,得到5株单拷贝转基因植株。对这些单拷贝转基因植株的T0:1株系目标基因的遗传情况进行分析,表明TaNAC14是可遗传的。对T1代转基因小麦株系目标基因TaNAC14表达水平进行测定,结果表明,转基因小麦中TaNAC14的表达水平与野生型相比,显著增加。本研究获得的可遗传且高水平表达的TaNAC14转基因小麦株系为后续该目标基因的功能研究奠定了重要基础。

