金属离子对肿瘤抑制蛋白p53影响的分子模拟
王卫京,詹世平,丁仕强
(大连大学 环境与化学工程学院,辽宁 大连 116622)
0 引言
蛋白p53是一种肿瘤抑制蛋白,具有调控细胞周期,进行DNA修复等功能,还能够启动不能够修复细胞的凋亡过程,使细胞进入程序性死亡过程,阻止癌变细胞的产生。而p53突变导致肿瘤的产生,约一半的肿瘤中存在p53的突变。
人体内存在多种金属离子,这些金属离子对p53有不同的影响。薛永来等[1]用光谱法研究了9种金属离子与p53DNA结合结构域的相互作用,发现Ba2+,Ca2+,Co2+,Mn2+,Ni2+没有引起蛋白质二级结构变化,Zn2+,Mg2+和Fe3+会导致蛋白质结构细微调整,Cu2+的作用会导致蛋白螺旋结构大量丢失。陈洋[2]用原子力显微镜研究了不同浓度的镁离子对p53-DNA结合的影响,认为高浓度的镁离子可以促进p53蛋白的聚集和p53-DNA的自组装。王帅[3]研究了镁离子对p53PDB与靶基因相互作用的影响,发现镁离子是一个影响和调节p53转录激活活性的因子。丁超[4]利用分子对接技术,设计了以p53/MDM2PP3为靶点的联芳基乙烯骨架的新型分子,这些分子表现出了较好的抗癌活性。许先进[5]采用分子模拟的方法,用SMD技术在分子水平上揭示了抗癌多肽p28和癌症抑制因子p53复合物解离过程。本文利用分子模拟研究了Na+,K+,Ca2+,Mg2+,Zn2+离子对p53蛋白的作用。
1 分子模拟过程
本次模拟采用分子模拟软件NAMD2.12进行分子动力学计算,可视化软件VMD1.9.3进行建模和辅助显示,计算力场选用CHARMM力场。从蛋白质数据库(RCSB PDB)下载p53晶体结构文件(PDB序号:2OCJ),经过VMD提取得到该分子的单独结构,如图1所示。通过VMD软件将该蛋白质分子放入球状水体中,如图2所示,再分别加入浓度为 0.2 mM 的 NaCl,KCl,CaCl2,MgCl2,ZnCl2。构建好体系后用NAMD2.12进行动力学计算,计算温度为310 K。先对体系进行2 ps的能量最小化,再对体系进行100 ps的动力学计算。
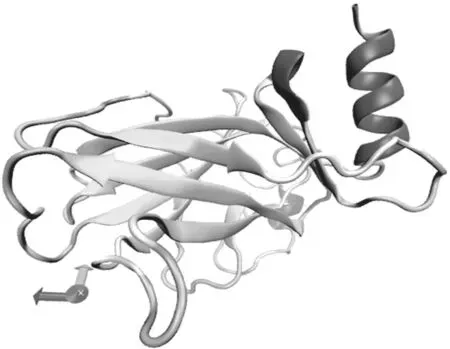
图1 p53蛋白质结构

图2 放入蛋白质分子的球状水体
2 结果与讨论
通过分子动力学模拟,计算蛋白质分子的RMSD值,即均方根偏差。RMSD值是衡量分子在模拟过程中各原子运动幅度的一个值。RMSD值越大,表示分子在模拟过程中各原子的位置变化越剧烈,否则,各原子位置变化较小。
图3是分子模拟的计算结果。从图3中可以看出,加入Mg2+后,p53蛋白质的RMSD值要明显比加入Na+,K+,Ca2+,Zn2+离子的值要高。这表明在Mg2+的作用下,p53蛋白质中各原子的运动幅度要高于在其他离子作用下的运动幅度。也就是说,Mg2+引起蛋白质构象变化较大,而这种大的变化有可能导致p53蛋白的突变。
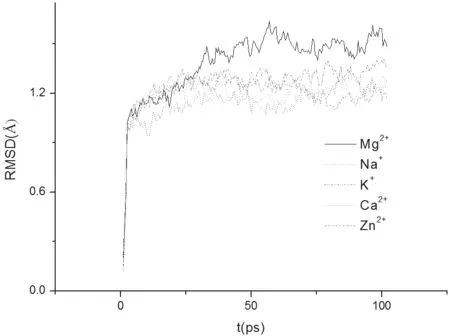
图3 不同离子作用下p53的RSMD值
图4、图5从不同角度显示了分子模拟过程中,Mg2+作用下p53蛋白质分子中各氨基酸残基的RMSD值分布情况,其中红色表示残基活动剧烈,蓝色表示运动幅度较小,而白色则介于二者之间。从图中可以看出,在Mg2+影响下,蛋白质分子有几处氨基酸残基运动幅度较大,特别是图4右方α螺旋附近区域,有氨基酸残基运动剧烈。该区域是p53蛋白的核心区域,负责与DNA的结合,其氨基酸残基运动幅度较大有可能增加p53蛋白质与DNA结合概率,但是过大的运动幅度则该区域构象有可能发生突变,使p53蛋白质失去与DNA结合的能力,丧失DNA修复等功能。
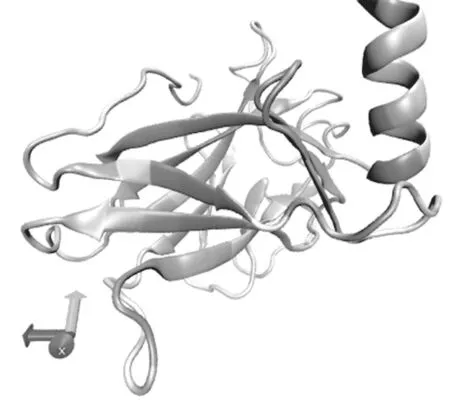
图4 氨基酸残基RMSD分布图
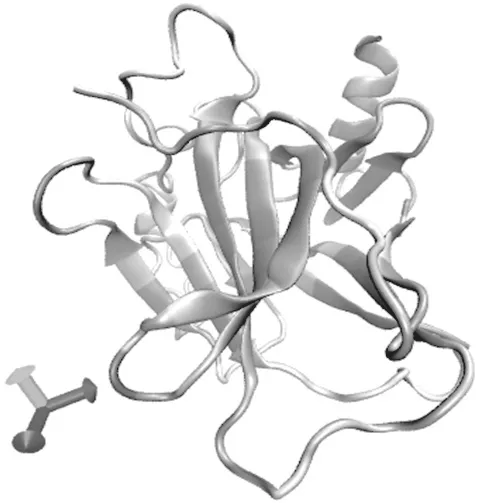
图5 氨基酸残基RMSD分布图
3 结论
本文研究了 Na+,K+,Ca2+,Mg2+,Zn2+离子对p53蛋白的影响。分子模拟结果显示,Mg2+离子较其他离子对p53有较大的影响,引起蛋白质构象变化较大。在p53蛋白的核心区域,残基的RMSD较大,表明Mg2+对该蛋白质核心区域影响较大,影响了其与DNA的结合能力,从而最终对p53的功能产生影响。

