菠萝蜜茎叶全长转录组分析
潘敏 于旭东 蔡泽坪 李佳佳 罗佳佳 周丹 楚文清 瞿倩
摘 要:菠蘿蜜(Artocarpus heterophyllus)是热带地区重要的木本植物,具有较高的经济价值。试验以菠萝蜜正常及叶绿素合成缺失突变体幼苗为研究材料,在PacBio Sequel平台上进行茎叶全长转录组测序,最终得到95 701个Isforms,在对转录本进行聚类纠错后利用NR、NT、GO、KOG、KEGG、Swiss-Prot、InterPro 7个数据库进行功能注释,共有93 367个转录本被注释。七大数据库中分别有92 187(NR)、90 326(NT)、74 548(Swiss-Prot)、75 396(KEGG)、77 022(KOG)、81 906(InterPro)和65 500(GO)个转录本获得功能注释。共检测到85 091个CDS和55 608个SSR序列,并预测到了4 753个转录因子(Transcription Factors,TFs),1 667个转录调控子(Transcriptional Regulators,TRs)以及4 710个受体激酶(Receptor-like Kinases,RLKs)。
关键词:菠萝蜜;全长转录组;功能注释中图分类号:S718.43 文献标识码:A
Transcriptome Data Analysis of Artocarpus heterophyllus Stems and Leaves
PAN Min1, YU Xudong1, CAI Zeping1*, LI Jiajia1, LUO Jiajia2,3, ZHOU Dan1, CHU Wenqing1, QU Qian1
1. College of Forestry, Hainan University / Key Laboratory of Genetics and Germplasm Innovation of Tropical Special Forest Trees and Ornamental Plants, Ministry of Education, Haikou, Hainan 570228, China; 2. College of Tropical Crops, Hainan University, Haikou, Hainan 570228, China; 3. Tropical Crops Genetic Resources Institute, Chinese Academy of Tropical Agricultural Sciences / Key Laboratory of Crop Gene Resources and Germplasm Enhancement in Southern China, Ministry of Agriculture and Rural Affatrs, Danzhou, Hainan 571737, China
Abstract: Artocarpus heterophyllusis an important woody plant in tropical regions withhigh economic value. This test used the normal and chlorophyll deficient mutant ofA. heterophyllusseedlings as the research materials. The transcriptome of the stems and leaves was sequenced by PacBio Sequel, 95 701 isoformswerefinally found. After cluster and polish, using NR, NT, GO, KOG, KEGG, Swiss-Prot and InterPro 7 databases for functional annotation, a total of 93 367 transcripts were annotated. There were 92 187 (NR), 90 326 (NT), 74 548 (Swiss-Prot), 75 396 (KEGG), 77 022 (KOG), 81 906 (InterPro) and 65 500 (GO) transcripts annotated in the seven databases respectively. In addition, a total of 85 091 CDS and 55 608 SSR sequences were detected, 4 753 transcription factors, 1 667 transcriptional regulators and 4 710 receptor-like kinases were predicted.
Keywords: Artocarpus heterophyllus; transcriptome; functional annotation
DOI: 10.3969/j.issn.1000-2561.2020.07.002
菠萝蜜(Artocarpus heterophyllus)为桑科(Moraceae)木波罗属(Artocarpus),在食用、药用、园林、经济等方面都具有较高价值[1]。当前,关于菠萝蜜的研究大多集中在生长发育、果实开发利用、病害防治等方面[2-4]。对于分子生物学方面研究较少。目前已有菠萝蜜花被转录组分析报道,但有关茎叶的报道还未出现[5]。由于缺乏基因组序列,所以分子生物学方面的研讨受到限制。
在果树植物的研究领域中,葡萄(Vitis vinifera)是第一个完成全基因组测序的物种[6]。至今已有数十种果树的基因组序列相继问世,其中包含热带水果香蕉(Musa nana)和菠萝(Ananas comosus)[7-8]。基因组测序的完成能够加深对物种的了解并加快分子育种的速度[9]。目前桑科植物中只有川桑(Morus notabilis)完成了基因组测序[10]。因此,研究菠萝蜜转录组测序可丰富其分子信息,为研究其他桑科植物提供参考[11]。
第三代测序技术具有读长长的优点,可快速简便地获得大量转录本序列,实现更多新基因的挖掘[12]。近年来,三代测序是开发基因组资源以及分子标记的一种重要途径,此技术已经在无参考基因组的条件下为多种植物建立了转录组数据库[13]。本研究利用PacBio Sequel平台对菠萝蜜进行全长转录组测序,不仅为二代测序数据拼接提供模板,还为进一步探索茎叶基因表达奠定基础。
1 材料与方法
1.1材料
组织样品采自生长40 d的白化和正常菠萝蜜幼苗[14],取其成熟叶、嫩茎和老茎节间进行混样后液氮中速冻和保存,用于RNA提取。
1.2方法
1.2.1 RNA提取和检测 将样品放入含有液氮的研钵中充分研磨成粉末,转入放有裂解液的EP管中,采用CTAB法提取RNA[15]。取适量样品进行检测,于?80 ℃保存。RNA提取流程与检测均委托华大基因公司进行。
1.2.2 文库构建和测序 在PacBio Sequel平台上测序菠萝蜜样本并建立PacBio ISO-Seq文库,得到原始聚合酶读取序列。读取序列去接头,用SMRT analysis套件[16]进行插入片段识别(reads of insert, ROI)、分类、聚类和校正,最终获得高质量的全长一致性序列。将各个文库的高质量全长序列合并到一起,在无参考基因组的情况下,使用cd-hit对聚类和纠错后的转录本去冗余。转录组测序委托华大基因公司进行。
1.2.3 CDS预测和SSR检测 使用TransDecoder. LongOrfs(https://transdecoder.github.io)对Unige ne.fa进行开放阅读框搜索,提取长读码框序列。基于序列相似性用Diamond Blastp将Unigene.fa比对到Swiss-Prot数据库中,利用Hmmscan对BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)结果在Pfam数据库筛选。利用TransDecoder.Predict预测Unigene.fa的编码序列。使用MISA[17] (http://pgrc. ipk-gatersleben.de/misa)对转录本进行SSR检测。
1.2.4 功能注释 利用BLAST[18]对转录本进行NR、NT、KOG、KEGG和Swiss-Prot注释。使用Blast2GO[19]以及NR注釋结果进行GO注释,使用InterProScan5[20]进行InterPro注释。使用iTAK在线工具(http://itak.feilab.cn)鉴定转录因子(transcription factors,TFs)、转录调控子(transcriptional regulators,TRs)以及受体激酶(receptor-like kinases,RLKs)。
2 结果与分析
2.1 数据质控
在PacBio Sequel平台上,将菠萝蜜正常及白化病的茎、叶混合样品进行全长转录组测序,共建立一个PacBio ISO-Seq文库,原始数据聚合酶读取(polymerase reads)序列中,总读取共有490 303个(13.58 GB)。其中,最大聚合酶读取(polymerase reads)序列长度为210 091 bp,N50为49 592 bp。最大聚合酶读取(polymerase reads)经过处理后得到的Subreads共有8 661 811个(12.80 GB),Subreads最大长度为132 829 bp,N50为1835 bp。
通过插入片段的识别,将来自同一环状分子的Subreads聚类在一起,在总共1个SMRT cell的测序中平均检测13次后得到893 030 085 bp的CCS(circular consensus sequencing)数据量。reads测序平均长度和平均质量值分别为1882 bp和0.94,可充分体现试验数据的可靠性,插入片段(reads of insert,ROI)共有474 542个。
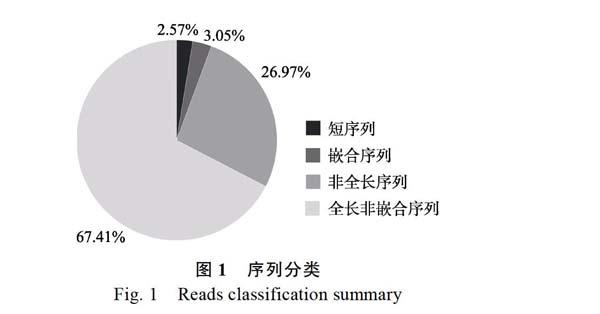
在所有的ROI中,全长非嵌合序列(full-length non-chimeric reads)共有319 878个,占67.41%,其次是非全长序列(non-full-length reads)占26.97%;嵌合体序列(chimeric)以及短序列(short reads)只占极少数,分别为3.05%和2.57%,可以看出只有极少数reads为嵌合reads(图1)。
全长非嵌合序列被聚类成一致性序列共有189 629个,后续分析只采用了high QV的一致性序列125 429个。每个文库聚类和纠错后得到的高质量序列最终合并到一起去冗余,得到转录本95 701个(表1、图2)。转录组测序数据已经上传至NCBI的SRA数据库,登录号PRJNA579273。
2.2 CDS预测
对转录组进行CDS(coding sequence)预测,共获得序列85 091个。片段大小297~6240 nt,总长度为93 949 176 nt。其中,长度在100~500 nt的片段有12 833个,占15.0%;500~1000 nt的有29 699个,占34.9%;1000~1500 nt的有23 147个,占27.2%;1500~2000 nt的有12 381个,占14.6%;长度>2000 nt的有7031个,占8.3%。序列片段大小大多数处于1000~2000 nt,说明菠萝蜜茎叶转录组的质量处于良好状态(图3A、表2)。
2.3 SSR检测
对转录本进行SSR检测,结果表明总共有55 608个序列。其中,最占优势的是单核苷酸重复(27 746),占比49.9%,其次是二核苷酸(13 734)占24.7%,三核苷酸(11 120)占20.0%,四核苷酸、五核苷酸以及六核苷酸占极少数,分别为1.38%、1.71%和2.31%。
在检测到的结果中,二核苷酸里AG/CT最多(9300),最少的是CG/CG(19)。在三核苷酸里AAG/CTT最多(3860),最少的是ACT/AGT(109)(图3B)。
2.4功能注释
将转录本与各大功能数据库比对,分别有92 187(NR)、90 326(NT)、74 548(Swiss-Prot)、75 396(KEGG)、77 022(KOG)、81 906(InterPro)和65 500(GO)个基因获得功能注释(表3)。
注:Intersection:被七大数据库中所有数据库注释上的转录本总数及比例;Overrall:被七大数据库中任意一个数据库注释上的转录本总数及比例。
Note: Intersection: Total number and proportion of transcripts annotated by all databases in seven databases; Overrall: Total number and proportion of transcripts annotated by any one of the seven databases.
2.4.1 NR注释分类 NR注释中相同转录本能比对上的较多相似物种有川桑(Morus notabilis)、枣树(Ziziphus jujuba)、核桃(Juglans regia)和樱桃(Prunus avium),且所占比例分别为71.26%、8.25%、1.53%和1.44%,另外比对到其他已知物种比例为17.52%(图4A)。
转录组在与NR、InterPro、Swiss-Prot、KOG和KEGG 5个数据库比对之后分别特有的转录本数为1998、30、9、9和15个。NR与InterPro的相同转录组最多有81 866个,关系最为紧密。其中InterPro与KOG、InterPro与KEGG、KOG与KEGG共有的转录本为0(图4B)。
2.4.2 KOG功能分布统计 KOG数据库共有 77 022个转录本被注释,根据功能大致可分为25类。功能种类比较全面,参与了较多的生命活动。一般功能预测(general function prediction)占最多(17 987),其次是信号转导机制(signal transdu ction mechanisms)(13 308)和翻译后修饰蛋白质翻转折叠(posttranslational modification, protein turnover,chaperones)(9769),细胞运动(cell motility)数量最少(189)(图5A)。
图A中1:翻译核糖体结构和生物组成;2:转录;3:信号传导机制;4:次级代谢产物的合成运输、分解;5:RNA加工与修饰;6:复制、重组与修复;7:翻译后修饰蛋白质翻转折叠;8:核算运输和代謝;9:核结构;10:脂质运输和代谢;11:细胞外分泌物的运输;12:无机离子运输和代谢;13:大致预测一般功能;14:未知功能;15:胞外结构;16:能量的产生与转换;17:防御机制;18:细胞骨架;19:辅酶的运输和代谢;20:染色质结构与运动;21:细胞膜/包膜生物合成;22:细胞运动;23:细胞周期调控;24:碳水化合物运输与代谢;25:氨基酸运输与代谢。图B中,1:运输和代谢;2:信号传导;3:膜转运;4:转运;5:折叠、分选和降解;6:翻译;7:复制折叠;8:全局和概览图;9:碳水化合物代谢;10:氨基酸代谢;11:类脂化合物代谢;12:能量代谢;13:辅助因子和维生素代谢;14:其他次级生物代谢;15:核苷酸代谢;16:其他氨基酸代谢;17:聚糖生物合成与代谢;18:萜类和酮类化合物代谢;19:环境适应;20:消化系统。图C中,1:细胞过程;2:代谢进程:3:生物调节;4:生物进程调控;5:应激反应;6:定位;7:组织或生物起源细胞组件;8:信号传导;9:多细胞生物过程;10:发育进程;11:繁殖;12:繁殖进程;13:生物过程的负调控;14:多生物过程;15:生物进程的正调控;16:生长;17:免疫系统过程;18:节律进程;19:细胞增殖;20:碳利用;21:脱毒;22:生物粘附;23:氧素利用;24:移动;25:细胞;26:细胞部分;27:细胞膜;28:膜部分;29:细胞器;30:高分子配合物;31:细胞器的部分;32:膜包围内腔;33:胞外区;34:病毒;35:病毒组分;36:超分子络合物;37:细胞连接;38:共质体;39:胞外区域部分;40:拟核;41:连接;42:催化活性;43:载体活性;44:转录调节活性;45:结构分子活性;46:分子功能调节器;47:信号传导活性;48:抗氧化活性;49:分子传感器活性;50:养分贮液囊活性;51:分子载体活性;52:蛋白质标记物;53:翻译调节活性。
A: Functional distribution of KOG annotation; B: Functional distribution of KEGG annotation; C: Functional distribution of GO annotation.
In figure A, 1: Translation, ribosomal structure and biogenesis; 2: Transcription; 3: Signal transduction mechanisms; 4: Secondary metabolites biosynthesis, transport and catabolism; 5: RNA processing and modification; 6: Replication, recombination and repair; 7: Posttranslational modification, protein turnover, chaperones; 8: Nucleotide transport and metabolism; 9: Nuclear structureLipid transport and metabolism; 10: Lipid transport and metabolism; 11: Intracellular trafficking, secretion, and vesicular transport; 12: Inorganic ion transport and metabolism; 13: General function prediction only; 14: Function unknown; 15Extracellular structures; 16: Energy production and conversion; 17 Defense mechanisms; 18 Cytoskeleton; 19: Coenzyme transport and metabolism; 20: Chromatin structure and dynamics; 21: Cell wall/membrane/ envelope biogenesis; 22: Cell motility; 23 Cell cycle control, cell division, chromosome partitioning; 24: Carbohydrate transport and metabolism; 25: Amino acid transport and metabolism. In figure B, 1: Transport and catabolism; 2: Signal transduction; 3: Membrane transport; 4: Translation; 5: Folding, sorting and degradation; 6: Transcription; 7: Replication and repair; 8: Global and overview maps; 9: Carbohydrate metabolism; 10: Amino acid metabolism; 11: Lipid metabolism; 12: Energy metabolism; 13: Metabolism of cofactors and vitamins; 14: Biosynthesis of other secondary metabolites; 15: Nucleotide metabolism; 16: Metabolism of other amino acids; 17: Glycan biosynthesis and metabolism; 18: Metabolism of terpenoids and polyketides; 19: Environmental adaptation; 20: Digestive system. In figure C, 1: Cellular process; 2: Metabolic process; 3: Biological regulation; 4:Regulation of biological process; 5: Response to stimulus; 6: Localization; 7: Cellular component organization or biogenesis; 8: Signaling; 9: Multicellular organismal process; 10: Developmental process; 11: Reproduction; 12:Reproductive process; 13: Negative regulation of biological process; 14: Multi- organism process; 15: Positive regulation of biological process; 16: Growth; 17: Immune system process; 18: Rrhythmic process; 19: Cell proliferation; 20: Carbon utilization; 21: Detoxification; 22: Biological adhesion; 23: Nitrogen utilization; 24: Locomotion; 25: Cell; 26: Cell part; 27: Membrane; 28: Membrane part; 29: Organelle; 30: Macromolecular complex; 31: Organelle part; 32: Membrane-enclosed lumen; 33: Extracellular region; 34: Virion; 35: Virion part; 36: Supramolecular complex; 37: Cell junction; 38: Symplast; 39: Extracellular region part; 40: Nucleoid; 41: Binding; 42: Catalytic activity; 43: Transporter activity; 44: Transcription regulator activity; 45: Structural molecule activity; 46: Molecular function regulator; 47: Signal transducer activity; 48: Antioxidant activity; 49: Molecular transducer activity; 50: Nutrient reservoir activity; 51: Molecular carrier activity; 52: Protein tag; 53: Translation regulator activity.
续图5 功能注释
Fig. 5 Function notes (continued)
2.4.3 KEGG功能分布统计 KEGG数据库共注释到75 396个转录本。根据代谢途径可分为5个分支,细胞加工(cell processing)(3215)、环境信息处理(environmental information processing)(4592)、遗传信息处理(genetic information processing)(16 305)、代谢(metabolism)(41 632)和有机体系统(organist system)(2928)。参与代谢途径中数量最多的通路是全局和概览图(global and overview maps)(16 331),其次为碳水化合物代谢(carbohydrate metabolism)(6758)(图5B)。
2.4.4 GO功能分布统计 GO数据库中共有65 500个转录本被注释。可分为生物过程(biological processes)(85 290)、细胞组成(cell composition)(117 187)和分子功能(molecular function)(75 861)3大类53分支。细胞组成中细胞(Cell)占最多(22 816),拟核(nucleoid)最少(30);生物过程中最多的是细胞过程(cellular process)(24 569),其次是代谢进程(metabolic process)(23 323),移动(locomotion)最少(8);执行分子功能这一大类中连接(binding)(34 271)和催化活性(catalytic activity)(32 416)数量最多,翻译调节活性(translation regulator activity)最少(3)(图5C)。
2.5 预测TFs、TRs以及RLKs
95 701个序列共预测出TFs 4753个,66个TF家族,其中GRAS家族最多(327),其次是bHLH(321);最少的家族是ULT(2)(图6、表4)。
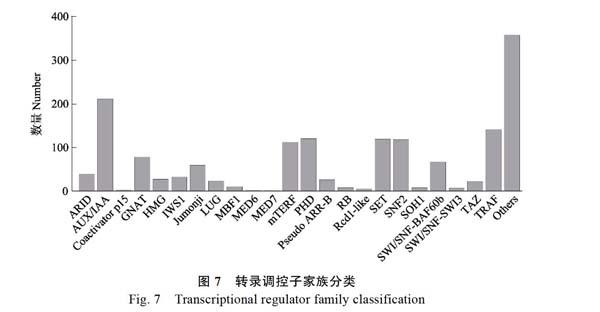
共预测出TRs 1667个,共有23个TR家族,其他家族有360个。数量最多的是AUX/IAA家族(214),其次是家族TRAF(144),最少的家族是MED7(4)(图7、表4)。
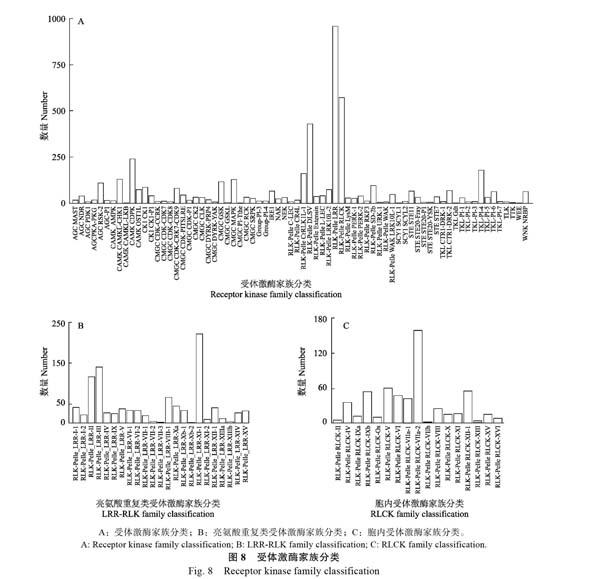
RLKs位于细胞膜上在植物信号传导过程起重要作用。通过数据检测到4710个RLKs,共分为72个家族。其中亮氨酸重复类受体激酶(leuc ine repeat receptor-like kinases,LRR-RLKs)和胞内受体激酶(receptor-like cytoplasmic kinases,RLCKs)分别有961和572个,分别含有23和17个亚家族(图8、表4)。

3 讨论
本研究采取第三代高通量测序技术结合生物信息学分析,得到菠萝蜜全长转录组数据。通过聚类纠错去冗余得到最终高质量序列9501个和N50为1949 bp,可看出三代测序读长长且连续性较高[21]。对转录组序列进行编码区域预测,根据结果显示,N50大于1000 bp且GC含量稳定说明序列组装完整性较高,可以为后续SSR检测及功能注释分析提供保障。在本试验检测到的SSR序列中,属单核苷酸重复最多,接近总SSR的一半。另外,二核苷酸以及三核苷酸重复数量均在一万以上,三者都可以为未来SSR分子标记开发奠定基础。
通过公共数据库对转录组比对分析,共有93 367个序列成功注释,占比97.56%,充分表明三代测序结果的较高精确性。推测剩余的2334条序列未被注释的原因可能是由于本身是非编码序列或者不完整序列[22]。根据NR注释情况来看,注释到川桑基因最多,体现了菠萝蜜与其亲缘性较高,也为将来二者基因表达详细比较提供充分的数据基础。比较KOG、KEGG、GO 3个数据库功能统计情况,发现注释到KOG的转录本数最多,其中以一般功能、信号转导机制、核酸运输和代谢的相关基因最为丰富。
由于菠萝蜜基因组还未被测序,限制其在分子生物学方面的研究。Hu等[5]对菠萝蜜花被进行转录组测序,得到32 495个高质量序列。本试验所获得的高质量序列比花被多的原因也许是由于测序的组织混合样品较多。花被高质量序列在七大数据库中获得功能注释的基因占比40%~80%,相对而言茎叶略比花被注释到的基因多,出现此结果差异也许是因为器官组织不同。
TFs调控众多基因表达,掌握其家族分类可对其功能有更深入的了解[23]。在TFs中,bHLH家族共有321个,目前已有关于桑树bHLH家族分析的报道[24]。本研究预测菠萝蜜体内的TFs、TRs、RLKs數量以及详细的家族分类,为未来研究抗性基因表达以及详细的家族分析提供数据。
本研究的转录组数据和各项结果丰富了菠萝蜜的分子信息,可为未来二代测序差异表达基因提供模板,进一步讨论其关于分子生物学方面的研究。
参考文献
[1] Manjeshwar S B, Arnadi R S, Raghavendra H,et al. Nutritional and pharmacological properties ofArtocarpus heterophyllusLam. (jackfruit): A review[J]. Food Research International, 2011, 44(7): 1800-1811.
- 苏兰茜, 白亭玉, 鱼 欢, 等. 盐胁迫对2种菠萝蜜属植物幼苗生长及光合荧光特性的影响[J]. 中国农业科学, 2019, 52(12): 2140-2150.
- 张 玲, 赖梓昊, 李春海, 等. 菠萝蜜风味果冻的研制[J]. 食品研究与开发, 2019, 40(1): 158-164.
- 黄纬君, 胡福初, 沈文涛, 等. 菠萝蜜锈病分级标准及田间病害调查[J]. 分子植物育种, 2019, 17(7): 2379-2385.
- Hu L, Wu G, Hao C, et al. Transcriptome and selected metabolite analyses reveal points of sugar metabolism in jackfruit (Artocarpus heterophyllus Lam.)[J]. Plant Science, 2016, 248: 45-56.
[6] Jaillon O, Aury J M, Noel B,et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla[J]. Nature, 2007, 449(7161): 463-467.
[7] D'Hont A, Denoeud F, Aury J M,et al. The banana (Musa cuminata) genome and the evolution of monocotyledonous plants[J]. Nature, 2012, 488(7410): 213-217.
[8] Ming R, VanBuren R, Wai C M,et al. The pineapple genome and the evolution of CAM photosynthesis[J]. Nature Genetics, 2015, 47(12): 1435-1442.
[9] Barthelson R, McFarlin A J, Rounsley S D,et al. Plantagora: modeling whole genome sequencing and assembly of plant genomes[J]. PLoS One, 2011, 6(12): e28436.
[10] 桑树基因组研究团队. 桑树(川桑Morus notabilis)全基因组测序[J]. 蚕学通讯, 2013, 33(4): 1-11.
[11] 张文秀, 张 丽, 寇一翾, 等. 中国濒危植物金钱松转录组测序及生物信息学分析[J]. 江西农业大学学报, 2019, 41(4): 761-772.
[12] 张得芳, 马秋月, 尹佟明, 等. 第三代测序技术及其应用[J]. 中国生物工程杂志, 2013, 33(5): 125-131.
[13] Franssen S U, Shrestha R P, Br?utigam A,et al. Comprehensive transcriptome analysis of the highly complexPisum sativumgenome using next generation sequencing[J]. BMC Genomics, 2011, 12(1): 227.
[14] 付 影, 于旭东, 蔡泽坪, 等. 菠萝蜜白化突变体的性状研究[J]. 热带作物学报, 2018, 39(6): 1081-1086.
- 刘春晓, 黄小庆, 刘自广, 等. 十字花科植物种子低分子RNA提取方法比较[J]. 基因组学与应用生物学, 2019, 38(3): 1236-1241.
- 朱兴正, 夏丽飞, 陈林波, 等. 保护品种云茶1号茶树全长转录组测序分析[J]. 茶叶科学, 2018, 38(2): 193-201.
- Thiel T, Michalek W, Varshney R K, et al. Exploiting EST databases for the development and characterization of gene- derived SSR-markers in barley (Hordeum vulgare L.)[J]. Theoretical and Applied Genetics, 2003, 106(3): 411-422.
- Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410.
- Conesa A, Gotz S, Garcia-Gomez J M, et al. Blast2GO: a un i versal tool for annotation, visualization and analysis in fu n c tional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676.
- Quevillon E, Silventoinen V, Pillai S, et al. InterProScan: protein domains identifier[J]. Nucleic Acids Research, 2005, 33(Web Server issue): w116-120.
- 李 慧, 涂 斌, 于英威, 等. 印度谷螟转录组测序及功能注释分析[J]. 河南工业大学学报(自然科学版), 2019, 40(4): 95-101, 113.
- Rui H, Zhenmin B, Shan W, et al. Transcriptome Sequencing and De Novo Analysis for Yesso Scallop (Patinopecten yessoensis) Using 454 GS FLX[J]. PLoS One, 2011, 6(6): e21560.
- Gu Z Y, Zhu J, Zhang L, et al. Transcription factors participate in response to powdery mildew infection in Paeonia lacti ?ora[J]. Scientia Horticulturae, 2019, 257: 108535.
- 惠 甜, 沈兵琪, 王连春, 等. 桑树bHLH转录因子家族全基因组鉴定与分析[J]. 分子植物育种, 2019, 17(17): 5624-5637.

