不同植物FERONIA基因密码子使用偏好性分析
赵森 邓力华 陈芬
摘要:为了了解植物FERONIA(FER)基因在长期进化过程中所形成的密码子使用模式及它们之间的亲缘关系,采用Codon W、CUSP在线程序和SPSS等分析软件对不同植物的FER基因密码子进行碱基组成分析、对应性分析、PR2-Plot分析、中性绘图分析和ENC-Plot分析并进行聚类分析。结果表明,单子叶植物FER基因密码子的平均G+C含量高于双子叶植物,且单子叶植物偏好使用以G/C结尾的密码子,而双子叶植物偏好使用以A/T结尾的密码子。对应性分析结果显示,在单子叶植物、双子叶植物中,向量轴1(Axis 1)可解释的变异分别占总变异的42.25%、28.56%。在单子叶植物中,Axis 1与总G+C含量、GC3含量、G3含量、C3含量呈极显著正相关(P<0.01),而在双子叶植物中则呈极显著负相关(P<0.01)。不同的碱基组成是单子叶植物和双子叶植物形成密码子使用偏好性的重要原因。PR2-Plot分析结果、中性绘图结果和ENC-Plot分析结果表明,自然选择是形成植物FER基因密码子使用偏好性的主要驱动力。基于编码序列(Coding sequence,CDS)构建的系统进化树比基于同义密码子相对使用度(RSCU)得出的聚类分析结果更接近传统的分类结果。本研究结果可为进一步开展植物分子进化和基因工程研究奠定基础。
关键词:FERONIA基因;密码子使用偏好性;分子进化
中图分类号:Q754;Q755文献标识码:A文章编号:1000-4440(2020)05-1073-09
Abstract:To understand the usage pattern of codons formed in the long-term evolutionary process of FERONIA (FER) genes in plants and their genetic relationships, base composition analysis, correspondence analysis, PR2-Plot analysis, neutral plotting analysis and ENC-Plot analysis on FER gene codons of different plants were conducted by using Codon W, on line program of CUSP and analysis software like SPSS, and the cluster analysis was conducted. The results showed that the average content of G and C in FER gene codons of monocots was higher than that of dicots, and the monocots preferred G/C ending codons while dicots preferred A/T ending codons. The results of correspondence analysis showed that, Axis 1 accounted for 42.25% and 28.56% of the total variation in monocots and dicots respectively. Axis 1 presented significantly positive correlation (P<0.01) with contents of total G+C, GC3, G3 and C3 in monocots, while in dicots the correlation between Axis 1 and contents of total G+C, GC3, G3, C3 was significantly negative respectively. Different nucleotide composition is the important reason in the formation of codon usage preference in monocots and dicots. Results of PR2-Plot analysis, neutral plotting analysis as well as ENC-Plot analysis showed that, natural selection was the main driving force in the forming of codon usage preference of FER genes in plants. Phylogenetic tree constructed by coding sequence (CDS) was more similar with traditional classification results than the phylogenetic tree constructed by relative synonymous codon usage (RSCU). The results can lay foundation for further research on molecular evolution and gene engineering.
Key words:FERONIA gene;codon usage preference;molecular evolution
根據中心法则,遗传信息流的方向是从DNA经RNA到蛋白质,密码子则是生物体内核酸信息和蛋白质信息相互沟通的桥梁。构成生物体的氨基酸约有20种,而密码子却有64种,这就存在一个氨基酸由多个不同密码子编码的现象(甲硫氨酸、色氨酸除外)。编码同一氨基酸的不同密码子称为同义密码子,而同义密码子并不是被随机使用的,这种同义密码子不能被均等使用的现象称为密码子使用偏好性[1]。密码子使用偏好性在自然界普遍存在,可以通过影响RNA加工、蛋白质翻译和蛋白质折叠等生命过程来调节基因表达,进而改变细胞功能[2]。在长期的进化过程中,每个物种都形成了其独特的密码子使用模式。一般来说,同一物种对基因密码子的使用偏好性较为一致,并且物种的亲缘关系越近,密码子的使用偏好性越一致,而功能越相似的基因,其密码子的使用偏好性越趋于一致[3]。关于密码子使用偏好性假说主要有中性理论和选择-突变-漂变理论[4-7]。有研究结果显示,密码子使用偏好性受碱基组成、碱基突变、自然选择、tRNA丰度、基因表达水平、基因长度等因素影响[8-12]。通过对特定物种或基因进行密码子使用偏好性分析,有望为基因结构、基因表达、环境适应和分子进化分析等研究提供帮助。
FERONIA(FER)基因是植物类受体蛋白激酶基因(RLK)家族的一员,在庞大的RLK家族中,FER基因又归属于植物特有的crRLK基因家族。FER蛋白编码区只在5′-UTR(非翻译区)有1个短的内含子,在其他位置没有内含子。FER蛋白是一种膜受体蛋白,具有明显的4个结构域,即胞外N端的信号肽、2个重复的Malectin-like结构域、中间的跨膜结构域和胞内的丝氨酸/苏氨酸激酶结构域[13]。FER蛋白最早是在擬南芥花粉管与助细胞的识别过程中被人们认识的,FER蛋白在胚珠助细胞的丝状器上极性表达,当花粉管进入FER基因突变的胚珠后,花粉管顶端并不破裂而是在胚珠内部继续生长,从而无法完成受精过程[13-15]。FER蛋白作为正调节因子,可直接激活ABI2的磷酸酶活性,从而负调节植物对ABA的响应[16]。有研究发现,FER蛋白能促进拟南芥根和叶片细胞的伸长[17-18],而在种子发育过程中则通过抑制细胞伸长来控制种子大小[19]。此外,FER蛋白还参与了Ca2+的信号转导及植物抗病免疫[20-21]。越来越多的研究结果表明,FER蛋白在植物生长发育、应对环境胁迫及抗病免疫等过程中发挥着重要作用。目前,除了拟南芥中的FER基因结构和功能已经得到深入研究外,关于花生[22]、橡胶[23]等物种的相关研究正在开展。鉴于FER基因对植物的重要作用,本研究拟通过分析植物FER基因密码子的碱基组成和使用偏好性,为深入研究FER基因的功能奠定基础,同时使人们更好地理解基因的水平转移和家族分化情况[24]。
1材料与方法
1.1数据来源
本研究选用的47种植物的FER基因序列全部来自GenBank数据库(https://www.ncbi.nlm.nih.gov/nucleotide/),其中9种来自单子叶植物,38种来自双子叶植物。所有FER基因的编码序列(CDS)长度均大于300 bp,均以ATG为起始密码子,以TAA、TAG、TGA为终止密码子。
1.2分析方法
从GenBank数据库中下载47种植物FER基因的CDS,参照Mazumdar等[7]的方法,使用Codon W1.4.2软件和CUSP在线程序(http://www.bioinformatics.nl/emboss-explorer/)计算CDS密码子第3位碱基(A3、T3、C3和G3)含量、密码子第1位的G+C(GC1)含量、第2位的G+C(GC2)含量和第3位的G+C(GC3)含量、有效密码子数(ENC)、同义密码子相对使用度(RSCU)和密码子适应指数(CAI)等参数。用Excel 2016对数据进行整理和作图,用SPSS 22对各相关参数进行相关性分析,用Codon W 1.4.2进行对应性分析。采用邻接法(Neighbor-joining, NJ),通过MEGA 5对所有CDS构建系统进化树,并通过自检举法(Bootstrap method=1 000)进行校正。用SPSS 22对所有FER基因序列的RSCU进行系统聚类分析。
2结果与分析
2.1植物中FER基因密码子使用偏好性相关参数比较
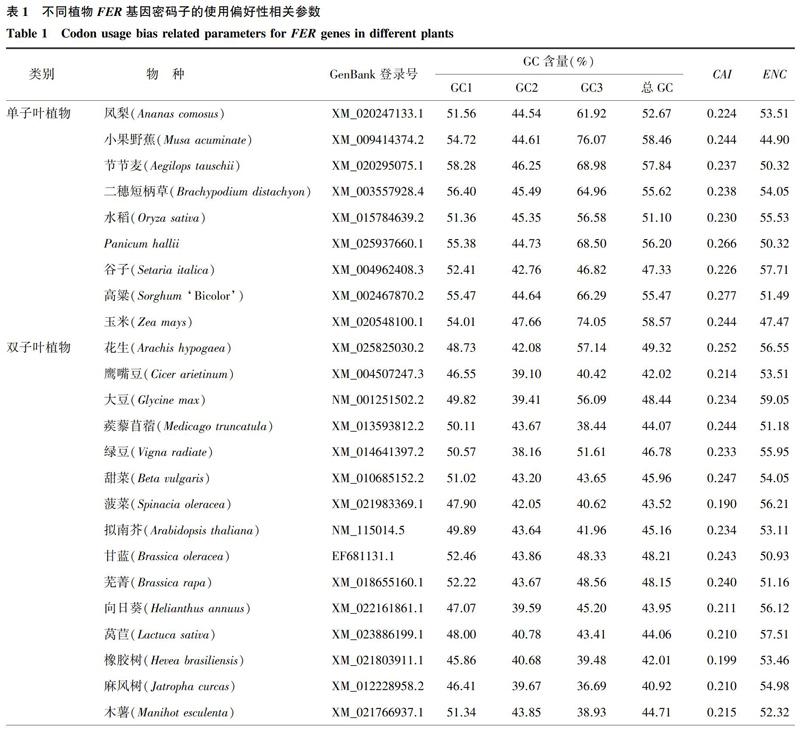
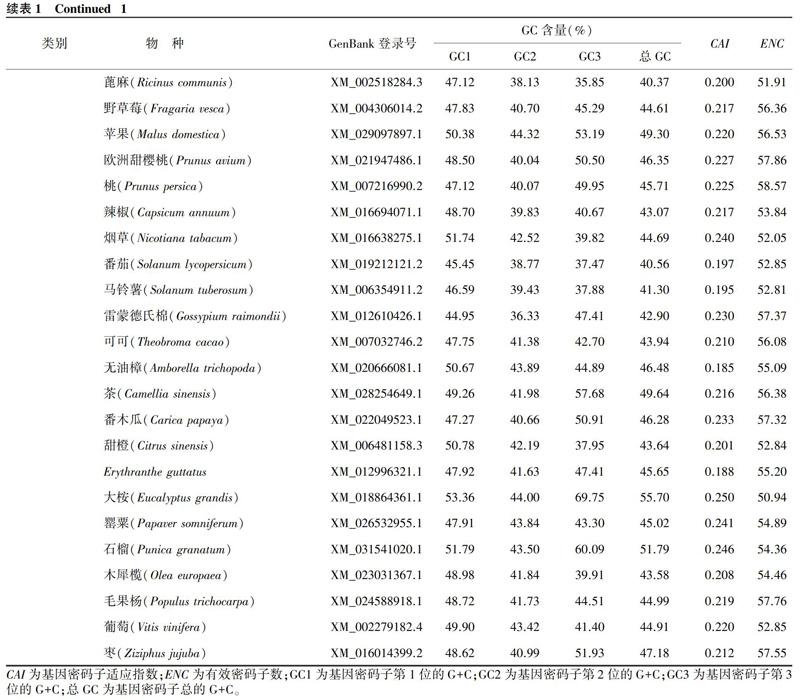
从表1可以看出,在单子叶植物中,FER基因的ENC均值为51.70,有7种单子叶植物FER基因的ENC大于50.00,说明这些单子叶植物的密码子使用偏好性较弱;在双子叶植物中,FER基因的ENC为50.93~59.05,ENC均值大于单子叶植物,为54.79,说明其对密码子的使用偏好性比单子叶植物弱。此外,单子叶植物、双子叶植物FER基因的CAI均值都较低,分别为0.243、0.220,这也进一步说明,在2种类型的植物中,FER基因对密码子的使用偏好性均较弱。
从表1还可以看出,在单子叶植物中,FER基因密码子的总G+C(总GC)含量为47.33%~58.57%,平均为54.81%,而在双子叶植物中,FER基因密码子的总GC含量为40.37%~55.70%,平均为45.39%。由此可见,单子叶植物中FER基因密码子的GC含量高于双子叶植物中FER基因密码子的GC含量,且二者间差异极显著(t=7.753, P<0.01)。t检验结果显示,单子叶植物FER基因密码子的GC3含量均值(64.91%)与双子叶植物FER基因密码子的GC3含量均值(45.82%)间差异极显著(t=6.598, P<0.01)。值得注意的是,除谷子外,其他单子叶植物FER基因密码子的总GC含量、GC3含量均高于50.00%,而在双子叶植物中,FER基因密码子的总GC含量高于50.00%的只有大桉、石榴。可以看出,单子叶植物中FER基因密码子的总GC含量、GC3含量整体上显著高于双子叶植物,单子叶植物FER基因偏好使用以G/C结尾的密码子,而双子叶植物FER基因偏好使用以A/T结尾的密码子。
2.2植物FER基因密码子使用偏好性的对应性分析
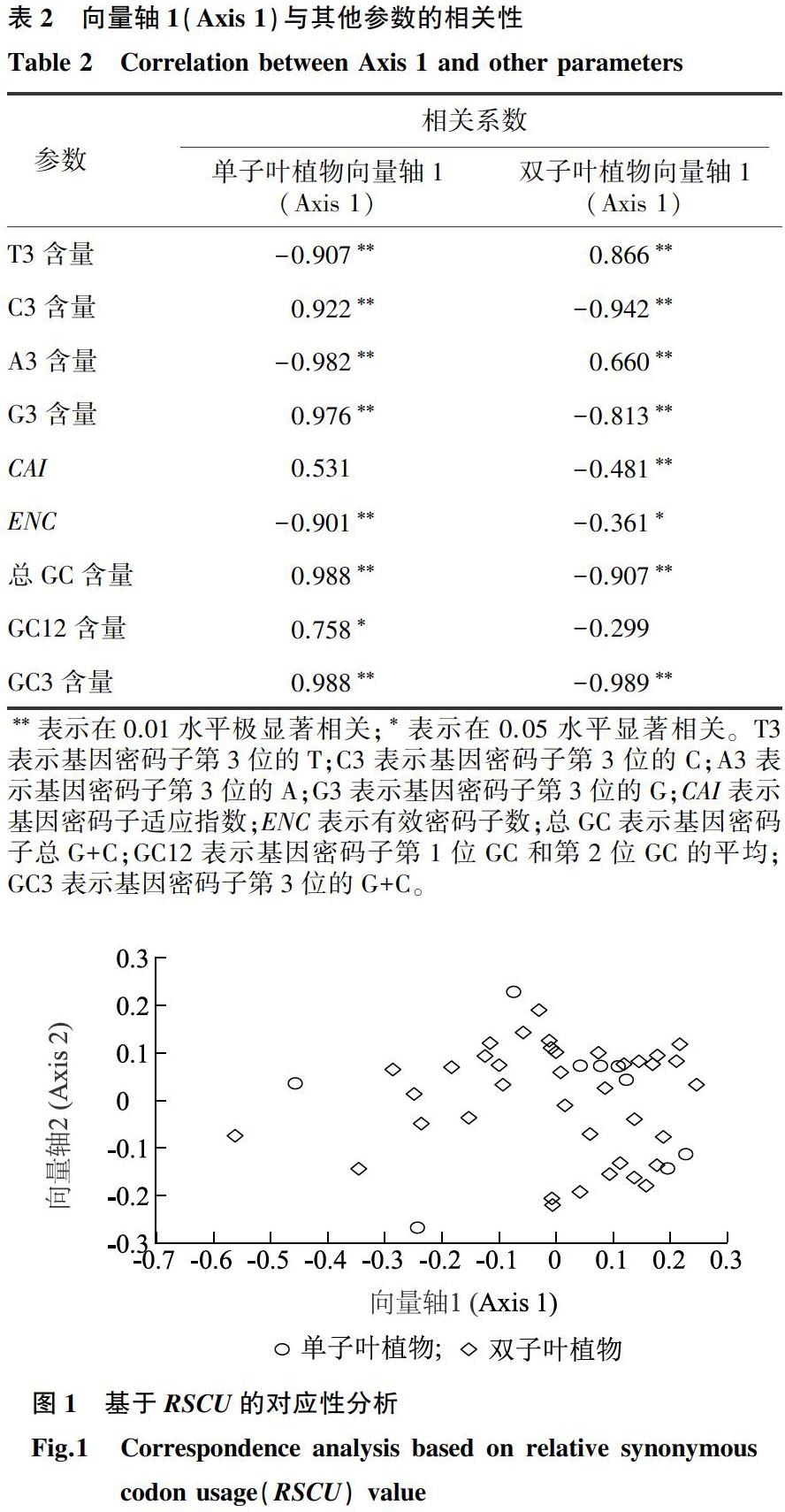
基于RSCU的对应性分析结果显示,在单子叶植物中,前4个向量轴可解释的变异分别占总变异的42.25%、18.60%、15.35%和9.54%,累计可解释总变异的85.74%,其中Axis 1(向量轴1)的值大于其他向量轴,Axis 1对单子叶植物FER基因密码子偏好性的影响最大。在双子叶植物中,前4个向量轴可解释的变异分别占总变异的28.56%、11.93%、8.94%和8.15%,累计可解释总变异的57.58%,其中Axis 1也是影响双子叶植物密码子使用偏好性的主要因素。由表2可以看出,在单子叶植物中,Axis 1与C3、G3、GC3、总GC含量的相关系数分别为0.922、0.976、0.988、0.988,呈正相关且都达到极显著水平,但Axis 1与T3、A3含量和ENC的相关系数分别为-0.907、-0.982和-0.901,呈负相关且达到极显著水平。在双子叶植物中,Axis 1与T3、A3含量的相关系数分别为0.866、0.660,呈正相关且均达到极显著水平,然而Axis 1与C3、G3、GC3、总GC含量和CAI的相关系数分别为-0.942、-0.813、-0.989、-0.907和-0.481,呈负相关且达到极显著水平。由以上结果可以看出,碱基组成是植物FER基因密码子使用偏好性形成的主要因素,但是在单子叶植物和双子叶植物中,影响密码子使用偏好性的碱基组成不同。由图1可以看出,单子叶植物和双子叶植物在坐标系内的位置分布趋于一致,表明影响单子叶植物和双子叶植物FER基因密码子使用偏好性的因素可能是相同的。
2.3植物中FER基因密码子使用偏好性的PR2-plot分析
由图2 FER基因的PR2-plot分析结果看出,多数植物FER基因的G3/(G3+C3)或A3/(A3+T3)都偏离中心值0.5,当G3/(G3+C3)或A3/(A3+T3)不等于0.5时,表明碱基突变和自然选择在FER基因密码子使用偏好性形成过程中的作用不对等。由图2还可以看出,大部分FER基因分布在G3/(G3+C3)<0.5的左侧区域,说明植物中FER基因偏好使用以C结尾的密码子;绝大部分FER基因分布在A3/(A3+T3)<0.5的下侧区域,说明植物中FER基因偏好使用以T结尾的密码子。综上可知,植物FER基因密码子在第3位使用嘧啶多于嘌呤。
2.4植物中FER基因密码子使用偏好性的中性绘图分析
相关性分析结果显示,在单子叶植物和双子叶植物中,GC3与GC12的相关系数分别为0.699和0.321,且都呈显著正相关(P<0.05)。由图3可以看出,在单子叶植物中,GC3与GC12的回归系数为0.118 8,说明突变压力造成的效应只占11.88%;在双子叶植物中,GC3与GC12的回归系数为0.081 5,说明突变压力造成的效应只占8.15%。由此可以看出,突变在FER基因密码子使用偏好性上起的作用比较小,并且其在双子叶植物中的作用小于在单子叶植物中的作用。
2.5植物中FER基因密码子使用偏好性的ENC-plot分析
在ENC-plot绘图分析中,如果密码子使用偏好性仅受GC3密码子组成影响而非受选择效应的影响,那么所有基因都会分布在期望曲线上[4,7]。从图4可以看出,所有基因都分布在期望曲线以下的区域,绝大多数单子叶植物的GC3含量为50.00%~80.00%,而绝大多数双子叶植物的GC3含量为30.00%~60.00%,2种类型植物的GC3含量分布范围都很窄。以上结果说明,FER基因密码子使用偏好性主要是由进化过程中的自然选择引起的。
2.6植物中FER基因的聚类分析
由系统进化树(图5)可以看出,9种单子叶植物聚在一起;在双子叶植物中,蔷薇科中的4种植物聚在一起,豆科植物(除蒺藜苜蓿)聚在1个分支上,茄科植物(除烟草)聚在1个分支上。此外,大戟科植物在系统进化树上分布得比较分散。总体上来看,基于CDS的系统进化分析结果与人们对植物分类的传统认知较一致。
基于RSCU的聚类分析结果(图6)显示,双子叶植物中的茄科、豆科、十字花科和大戟科植物分别聚为1类,而单子叶植物却聚成不同的2类。尤其需要注意的是,单子叶植物和大戟科植物的聚类结果与基于CDS的系统进化树结果不同。植物学分类地位不同的植物反而聚在一起,可能与不同物种在FER基因的使用模式上趋于一致有关。
3讨论
本研究结果显示:单子叶植物中FER基因的GC3含量均值为64.91%,而双子叶植物中FER基因的GC3含量均值为45.82%;单子叶植物偏好使用以G/C结尾的密码子,而双子叶植物偏好使用以A/T结尾的密码子,与Wang等[25]对5种单子叶植物和6种双子叶植物转录组的研究结果一致。FER基因会参与植物生长发育、激素应答反应和环境胁迫反应等生命过程[19,26]。ENC在单子叶植物和双子叶植物中都较高,说明在2种类型的植物中,FER基因的密码子使用偏好性不强,这可能与FER基因功能的保守性相关。CAI是衡量基因表达水平的重要指标[27],在本研究中,单子叶植物和双子叶植物中的CAI都比较低,可能与FER不是管家基因且只在特定时期或特定组织中表达有关。
已有的关于茄属植物叶绿体基因组密码子的研究发现,碱基组成是影响密码子使用偏好性的重要因素之一[8]。G+C碱基含量和位置是影响密码子使用偏好性的主要因素,这也在植物CaM1基因的研究中得到证实[24]。关于非禾本科单子叶植物的研究结果显示,G、C偏好的基因转换(gBGC)被认为是造成密码子使用偏好性差异和核苷酸组成差异的主要因素[7]。以上研究结果显示,在影响密码子使用偏好性的各因素中,碱基组成是重要因素。本研究结果也揭示,碱基组成是影响植物FER基因密码子使用偏好性的重要因素,但是在单子叶植物和双子叶植物中,FER基因密碼子的碱基组成差异显著,对G、C的偏好性现象可能在单子叶植物中广泛存在。
植物在长期进化过程中所形成的密码子使用偏好性,取决于突变压力、自然选择的作用强度和方向[25]。本研究中的PR2-plot分析结果也证实,突变压力和自然选择可能影响植物FER基因密码子使用偏好性。中性绘图结果显示,突变在FER基因密码子使用偏好性上的作用很小。ENC-plot分析结果表明,自然选择才是形成FER基因密码子使用偏好性的主要因素。本研究结果与形成人类密码子使用偏好性的原因主要是突变不同[28],而与酵母[29]、线虫[30]等物种的密码子使用偏好性形成模式受自然选择作用主导类似。此外,自然选择还可通过影响蛋白质翻译的速度和准确性来起作用[25]。一般来说,亲缘关系越近的物种,其密码子使用偏好性越趋于一致。但是由于某些基因具有特殊的密码子使用偏好性,使得聚类结果并不能真实反映亲缘关系和分类地位[27]。本研究发现,基于CDS的聚类分析结果与基于RSCU的聚类分析结果并不完全一致,基于CDS的聚类分析结果更接近传统的分类结果,与植物ICE1基因的聚类分析结果类似[31]。基于CDS的聚类能将单子叶植物聚在一起,而基于RSCU的聚类结果与传统分类结果的差别较大,可能与基于RSCU的聚类分析只能在较小的分类单元中提供较为可靠的结果,而在较大的分类单元中只能作为传统分类的补充有关[27]。进一步研究发现,要想对物种的进化关系进行更为准确的描述,需要对不同分析方法得出的结果进行综合分析。
本研究通过生物信息学方法对植物FER基因密码子使用偏好性特性进行分析,可为进一步挖掘FER基因功能和基因表达调控研究奠定基础。
參考文献:
[1]HERSHBERG R, PETROV D A. Selection on codon bias[J]. Annual Review of Genetics, 2008, 42: 287-299.
[2]PLOTKIN J B, KUDLA G. Synonymous but not the same: the causes and consequences of codon bias[J]. Nature Reviews Genetics, 2011, 12(1): 32-42.
[3]CHIAPELLO H, LISACEK F, CABOCHE M, et al. Codon usage and gene function are related in sequences of Arabidopsis thaliana[J]. Gene, 1998, 209(1/2): GC1-GC38.
[4]WRIGHT F. The effective number of codons' used in a gene[J]. Gene, 1990, 87(1): 23-29.
[5]BULMER M. The selection-mutation-drift theory of synonymous codon usage[J]. Genetics, 1991, 129(3): 897-907.
[6]SUEOKA N. Directional mutation pressure and neutral molecular evolution[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(8): 2653-2657.
[7]MAZUMDAR P, BINTI O R Y, MEBUS K, et al. Codon usage and codon pair patterns in non-grass monocot genomes[J]. Annals of Botany, 2017, 120(6): 893-909.
[8]ZHANG R Z, ZHANG L, WANG W, et al. Differences in codon usage bias between photosynthesis-related genes and genetic system-related genes of chloroplast genomes in cultivated and wild Solanum species[J]. International Journal of Molecular Sciences, 2018, 19: 3142.
[9]SONG H, LIU J, CHEN T, et al. Synonymous codon usage pattern in model legume Medicago truncatula[J]. Journal of Integrative Agriculture, 2018, 17(9): 2074-2081.
[10]尚明照,刘方,华金平,等. 陆地棉叶绿体基因组密码子使用偏性的分析[J]. 中国农业科学, 2011, 44(2): 245-253.
[11]续晨,蔡小宁,钱保俐,等. 葡萄基因组密码子使用偏好模式研究[J]. 西北植物学报, 2012, 32(2): 409-415.
[12]HE W, WANG N N, TAN J M, et al. Comprehensive codon usage analysis of porcine deltacoronavirus[J]. Molecular Phylogenetics and Evolution, 2019, 141: 106618.
[13]ESCOBAR-RESTREPO J M, HUCK N, KESSLER S, et al. The FERONIA receptor-like kinase mediates male-female interactions during pollen tube reception[J]. Science, 2007, 317(5838): 656-660.
[14]ROTMAN N, ROZIER F, BOAVIDA L C, et al. Female control of male gamete delivery during fertilization in Arabidopsis thaliana[J]. Current Biology, 2003, 13(5): 432-436.
[15]HUCK N, MOORE J M, FEDERER M, et al. The Arabidopsis mutant feronia disrupts the female gametophytic control of pollen tube reception[J]. Development, 2003, 130(10): 2149-2159.
[16]YU F, QIAN L C, NIBAU C, et al. FERONIA receptor kinase pathway suppresses abscisic acid signaling in Arabidopsis by activating ABI2 phosphatase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(36): 14693-14698.
[17]KANAOKA M M, TORII K U. FERONIA as an upstream receptor kinase for polar cell growth in plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(41): 17461-17462.
[18]DESLAURIERS S D, LARSEN P B. FERONIA is a key modulator of brassinosteroid and ethylene responsiveness in Arabidopsis hypocotyls[J]. Molecular Plant, 2010, 3(3): 626-640.
[19]YU F, LI J, HUANG Y, et al. FERONIA receptor kinase controls seed size in Arabidopsis thaliana[J]. Molecular Plant, 2014, 7(5): 920-922.
[20]SHIH H W, MILLER N D, CHENG D, et al. The receptor-like kinase FERONIA is required for mechanical signal transduction in Arabidopsis seedlings[J]. Current Biology, 2014, 24(16): 1887-1892.
[21]KESSLER S A, SHIMOSATO-ASANO H, KEINATH N F, et al. Conserved molecular components for pollen tube reception and fungal invasion[J]. Science, 2010, 330(6006): 968-971.
[22]張欣悦,李佳婷,邹婷,等. 花生响应干旱胁迫的FERONIA类受体蛋白激酶基因克隆与表达分析[J]. 植物生理学报, 2018, 54(2): 297-304.
[23]王凉洁,秦云霞,罗红丽. 巴西橡胶树FERONIA类受体激酶基因(HbFER)的克隆及表达分析[J]. 热带作物学报, 2014, 35(7): 1347-1353.
[24]李晨辉,赵子捷,陈文烨,等. 43种植物CaM1基因密码子使用特征及遗传差异分析[J]. 江苏农业科学, 2019, 47(5): 28-32.
[25]WANG L J, ROOSSINCK M J. Comparative analysis of expressed sequences reveals a conserved pattern of optimal codon usage in plants[J]. Plant Molecular Biology, 2006, 61(4/5): 699-710.
[26]YU F, TIAN W, LUAN S. From receptor-like kinases to calcium spikes: what are the missing links?[J]. Molecular Plant, 2014, 7(10): 1501-1504.
[27]赵春丽,彭丽云,王晓,等. 苋菜AtGAI基因密码子偏好性与进化分析[J]. 中国农业大学学报, 2019, 24(12): 10-22.
[28]SUEOKA N, KAWANISHI Y. DNA G+C content of the third codon position and codon usage biases of human genes[J]. Gene, 2000, 261(1): 53-62.
[29]COGHLAN A, WOLFE K H. Relationship of codon bias to mRNA concentration and protein length in Saccharomyces cerevisiae[J]. Yeast, 2000, 16(12): 1131-1145.
[30]DURET L, MOUCHIROUD D. Expression pattern and, surprisingly, gene length shape codon usage in Caenorhabditis, Drosophila, and Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96: 4482-4487.
[31]孙继峰,韩太利,徐立功,等. 萝卜ICE1密码子使用偏性分析[J]. 核农学报, 2018, 32(3): 477-485.
(责任编辑:徐艳)

