基于益生菌调节的太平鸡回肠SNP位点分析
雒林通 马芳 唐德富
摘要 为挖掘益生菌调节下太平鸡回肠SNP位点,将120只太平鸡随机分为对照组与益生菌组,采用高通量测序技术对太平鸡回肠转录组的SNP位点进行分析。结果显示:SNP类型中,转换类型明显高于颠换类型,对照组中转换类型占22.55%~27.45%,颠换类型占11.43%~13.87%;益生菌组与对照组结果相似,转换类型(22.53%~27.46%)明显高于颠换类型(11.37%~13.94%)。SNP位置分析表明,对照组和益生菌组总SNP位点分别为4 684 072和4 647 171个,其中位于内含子和基因间区的SNP位点最多。另外,在SNP功能分析中,同义突变SNP占比最高,在对照组和益生菌组中分别占7.01%和7.10%;非同义突变SNP在对照组和益生菌组中占比分别为2.97%和2.98%。该研究结果可为今后太平鸡的分子标记开发、遗传连锁图谱构建以及輔助育种等提供基础数据。
关键词 太平鸡;益生菌;回肠;转录组测序;SNP
中图分类号 S831 文献标识码 A
文章编号 0517-6611(2020)21-0086-05
Abstract In order to excavate SNP sites in ileum of Taiping Chicken regulated by probiotics,120 Taiping chickens were randomly divided into control group and probiotic group.The SNP sites in the transcriptome of Taiping chickens ileum were analyzed by high throughput sequencing technique.The results showed that the transition proportion was significantly higher than that of the transversion.The transition of control group accounted for 22.55%-27.45%,and the transversion accounted for 11.43%-13.87%.The results of probiotic group were similar to those of control group,and the transition proportion (22.53%-27.46%) was significantly higher than that of the transversion (11.37%-13.94%).The analysis of SNP locations showed that the total number of SNP sites in the control group and the probiotic group was 4 684 072 and 4 647 171 respectively,among which the most SNP sites were located in introns and intergenic.In the analysis of SNP function,the proportion of synonymous mutations of SNP was the highest,which was 7.01% in the control group and 7.10% in the probiotic group.Nonsynonymous mutations of SNP accounted for 2.97% in the control group and 2.98% in the probiotic group.This study could provide basic data for the development of molecular markers,the construction of genetic linkage map and the assistant breeding of Taiping Chicken in the future.
Key words Taiping Chicken;Probiotics;Ileum;Transcriptome sequencing;SNP
基金项目 甘肃省民生科技计划项目(1503FCME005);天水市科技支撑计划项目(2020-SHFZKJK-7879)。
作者简介 雒林通(1973—),男,甘肃天水人,高级实验师,硕士,从事动物遗传育种与繁殖方面的研究。*通信作者,副教授,博士,从事动物遗传育种方面的研究。
收稿日期 2020-06-02;修回日期 2020-06-23
太平鸡具有较强适应性和丰富的基因库,是我国优良家禽品种和甘肃三大优良地方鸡种之一,主要分布于甘肃康县太平乡(现为阳坝镇)因而得名[1]。太平鸡以黑羽红冠为主要特征,体格高大,体型紧凑,生产性能以肉用为主,肉蛋兼用,具有肉嫩骨细、皮脆味鲜、肉质特佳等特点,在市场上具有较高的价值[2]。近年来,由于应对鸡群混养而导致太平鸡鸡种退化[3]、免疫性能低下等问题,部分饲料生产企业和养殖户为了追求养鸡业效益的最大化以及疾病防治中用药的误区,往往存在大剂量使用抗生素[3],使得药物残留、耐药菌株大量出现、损伤消化免疫系统等问题频发[4],对人与自然的可持续发展造成严重威胁,成为食品安全和绿色可循环畜牧业发展中最突出、最严峻的问题[5],所以积极寻找绿色饲料添加剂,对太平鸡进行遗传改良、选育新品种已成为提高养殖效益和健康化养殖水平的当务之急。
益生菌(probiotic)指的是在人和动物肠道内,通过调节动物肠道微生物区系平衡对宿主健康有益的活性微生物菌种[6]。研究表明,益生菌可以维持动物机体肠道菌群平衡,抵抗病原菌入侵,分泌抗菌物质及营养活性物质和改善肠道黏膜免疫功能[7-8]。益生菌进入机体肠道后,刺激宿主肠道分泌多种消化酶,通过充分降解宿主体内未被消化吸收的抗营养因子而提高饲料利用率,对动物生长发育和生产性能的提高具有促进作用。益生菌在宿主肠道内借助体内发酵作用产生的有机酸(乙酸、乳酸和丙酸)来有效降低肠道pH,这不利于有害菌的生长,且益生菌分泌并诱导产生抑菌蛋白对致病菌生长具有抑制作用[9]。此外,益生菌通过刺激宿主肠道分泌免疫抗菌物质,调节机体的免疫系统[10]。因此,益生菌制剂作为一种新型的微生物饲料添加剂,在其调节下能更好地促进养殖业的健康和稳定发展。
作為第三代分子标记的单核苷酸多态性(single nucleotide polymorphisms,SNPs)是由单个核苷酸的变异而导致基因组层面DNA序列的多态性,通常表现为单个碱基的转换或颠换、缺失或插入,但实际上发生的只有转换和颠换2种[11]。由于覆盖密度大,具有遗传稳定性和代表性,便于高通量自动化检测等优点,已经广泛应用在构建高密度遗传连锁图谱构建、关联分析、分子辅助育种、群体遗传系统、品种鉴定等方面,并表现出良好的应用前景[12-14]。笔者以太平鸡为研究对象,通过在饮水中添加益生菌制剂,通过高通量测序分析其对太平鸡回肠多态性位点的干预作用,利用分子标记进行辅助选择从而获得更大的遗传进展,以期为太平鸡的遗传与育种提供科学依据。
1 材料与方法
1.1 试验材料
试验用益生菌制剂由安徽诺伟康饲料有限公司生产;试验动物为60日龄太平鸡,由陇南市康县梅园太平山鸡养殖专业合作社提供。
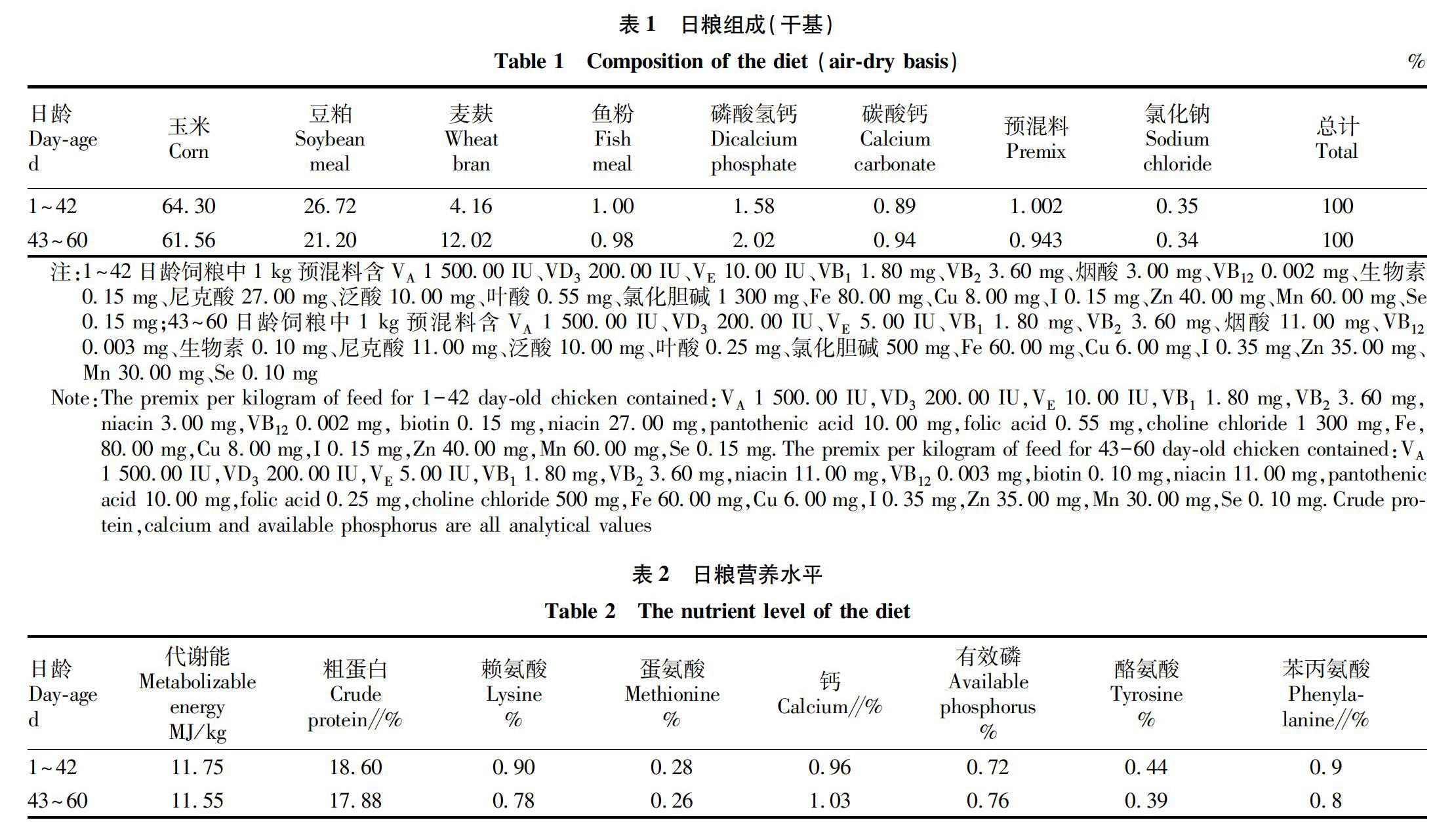
1.2 试验设计与饲料 选择0日龄太平鸡120只,随机分为2组,每组3个重复,每个重复20只。对照组(CT)饲喂基础日粮,试验组(MP)在饲喂基础日粮的基础上每升饮水中加入2 mL益生菌。饲料采购严格按照GB13078—2017《饲料卫生标准》和NY 5032—2006《无公害食品畜禽饲料和饲料添加剂使用准则》执行。参照《鸡饲养标准》(NY/T 33—2004)配制玉米-豆粕型基础饲粮,其组成及营养水平见表1~2。
1.3 饲养管理及样品采集
第1~3天,温度保持在33~35 ℃,此后每7 d降低2 ℃,直到最终室温在18~21 ℃。光照时间1~3日龄为23~24 h,4~15日龄为23~16 h,16~60日龄为16~12 h;1~3日龄光照强度为10~30 lx,4~15日龄为5 lx,16~60日龄逐渐接近自然光照。室内相对湿度55%~70%,自由采食和饮水。
试验用太平鸡饲养至60日龄,各组每个重复取1只鸡,即每组共3只鸡。对照组(CT)样品编号分别为CT1、CT2、CT3,试验组(MP)样品编号分别为MP1、MP2、MP3。称重后心脏注射空气致死,用新洁尔灭进行鸡体消毒后,拔掉腹部羽毛,开腹后分离回肠,液氮中速冻,然后转入-80 ℃低温冰箱内保存。
1.4 RNA提取
用TRIzol试剂盒(Invitrogen,Carlsbad,CA,USA)分别提取MP1、MP2、MP3和CT1、CT2、CT3 6组回肠样品的总RNA。RNA的降解和污染程度用1%琼脂糖凝胶电泳检测,RNA纯度使用Nanodrop微量分光光度计检测,RNA浓度和完整性用Qubit 2.0 Flurometer(Life Technologies,CA,USA)和Bioanalyzer 2100系统(AgilentTechnologies,CA,USA)进行检测。
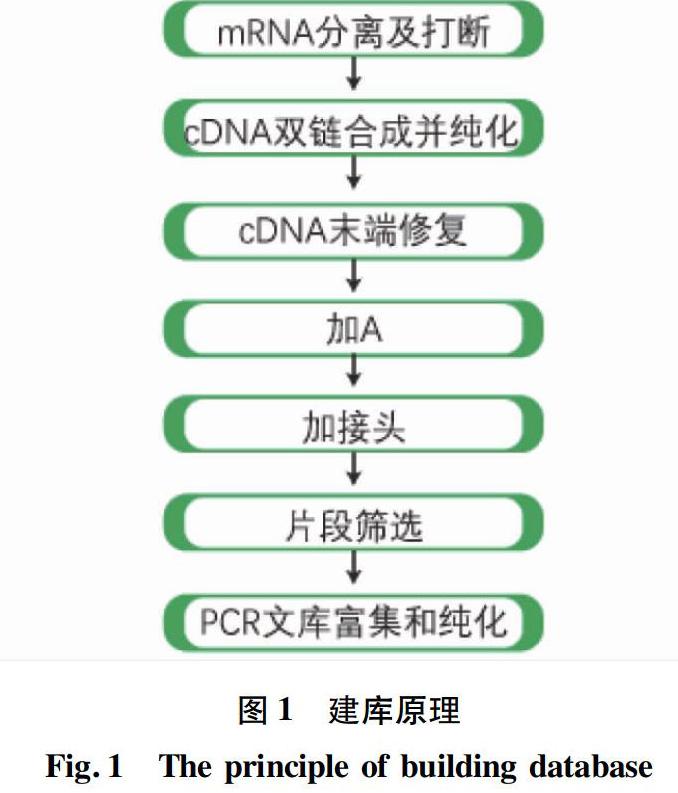
1.5 转录组CDNA文库构建及质检 试验流程按照样品制备试剂盒操作说明的标准步骤执行。每个样品取3 μL的RNA用作RNA样品准备的输入材料构建6个测序文库。流程如下:通过带有Oligo(dT)的磁珠富集具有polyA尾巴的真核mRNA后,用超声波将mRNA打断。以片段化的mRNA为模版,随机寡核苷酸为引物,在M-MuLV逆转录酶体系中合成cDNA第一条链,随后用RNaseH降解RNA链,并在DNA polymerase Ⅰ 体系下,以dNTPs为原料合成cDNA第二条链。纯化后的双链cDNA经过末端修复、加A尾并连接测序接头,用AMPure XP beads筛选200 bp左右的cDNA,进行PCR扩增并再次使用AMPure XP beads纯化PCR产物,最终获得文库。建库原理如图1所示。
1.6 转录组测序
文库质检合格后用Illumina HiseqTM2500平台进行pairedend测序,测序完成后将得到的序列进行去接头、去低质量阅读框和去污染处理,得到干净序列。对得到的序列进行GC含量和Q30分析,以保证测序质量。
1.7 SNP挖掘及统计分析
为了保证数据质量,首先通过去除含adapter的reads,含N比例大于10%的reads,含N比例大于10%的reads,全部都是A碱基reads和低质量reads (质量值Q≤20的碱基数占整条read的50%以上),对原始数据进行数据过滤。然后利用GATK2软件包进行SNP检测。
2 结果与分析
2.1 RNA提取和质量检测
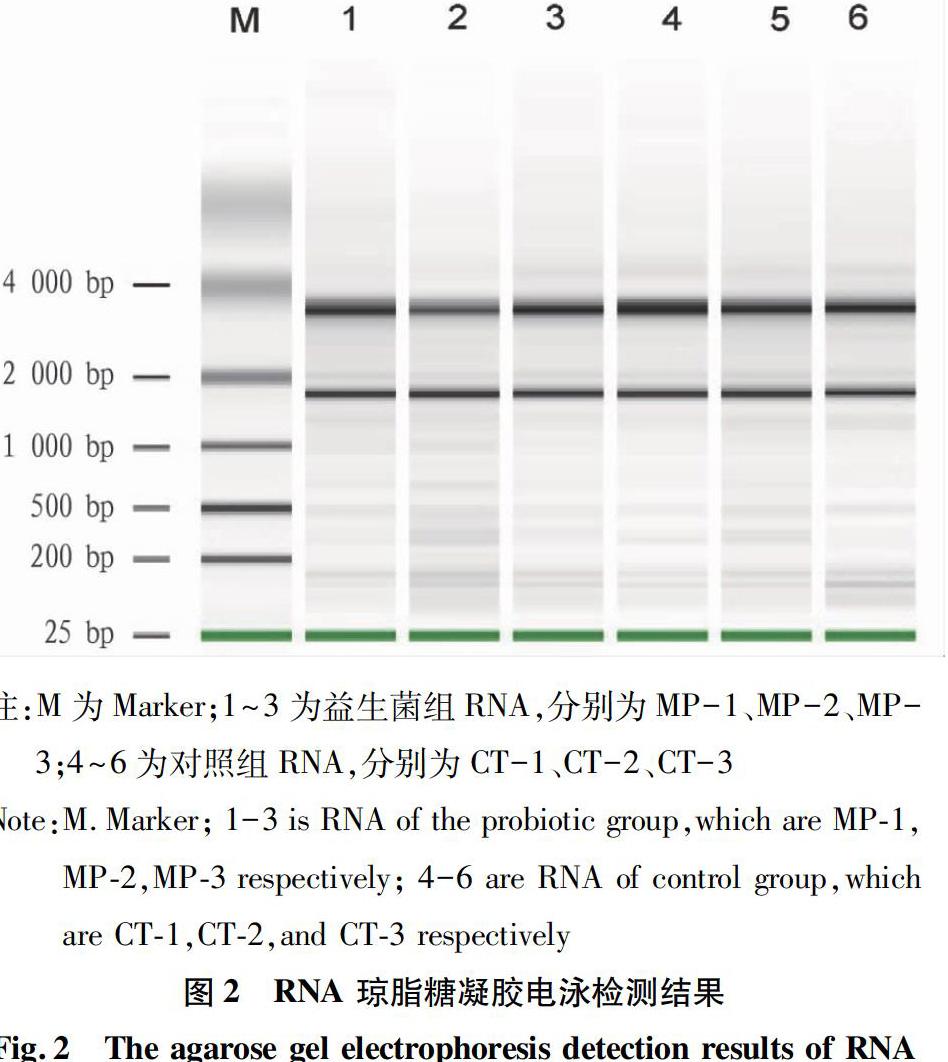
该研究提取的RNA如图2所示,RNA样品28S、18S、5S条带清晰无降解;紫外分光光度法和Nanodrop结果表明OD260 nm/OD280 nm为1.8~2.0,RNA的完整性和纯度都较好,达到Illumina的测序要求(表3),可用于后续的基于 HiseqTM2500平台的转录组测序。
2.2 转录组数据分析
样品测序产出数据质量评估情况见表4。对得到的测序数据进行错误率检查,结果表明在测序长度范围内,6个文库中碱基的错误率均低于0.1%;对GC含量分布进行检测,GC含量比例也在正常范围内;用Fast QC分析软件对Reads文件进行测序质量评估,每碱基质量的Q20值和Q30值最低为92.47%,表明此次测序质量较高,满足后续分析的要求。测序所得的原始序列经过质量检测后,过滤剔除带接头的、低质量的reads,6个文库分别得到了7.3~8.7 G的clean reads,用于后续转录组分析。
2.3 SNP位点分析
2.3.1 SNP颠换、转换信息。
对太平鸡SNP类型的分析(表5)发现,CT组中转换类型占22.55%~27.45%,颠换类型占11.43%~13.87%。转换类型明显高于颠换类型,MP组与CT组结果相似,转换类型(22.53%~27.46%)明显高于颠换类型(11.37%~13.94%)。转换类型中,2组中G→A发生频率最高,分别为27.46%和27.45%。颠换类型中,G→C发生频率最高,分别为13.94%和13.84%%。可能是因为CG中的C(胞嘧啶)常为甲基化的,自发地脱氨后即成为T (胸腺嘧啶)。
2.3.2 SNP位置分析。对太平鸡回肠SNP转录组序列位置变异类型进行分析,CT组和MP组总的SNP位点分别为4 684 072和4 647 171个,其中位于内含子和基因间区的SNP位点最多,位于内含子的SNP位点在CT组占59.69%,MP组中占59.94%。位于基因间区的SNP位点在CT组占20.56%,MP组中占20.12%。另外,在基因组中不同的2个位置间,存在着共有的SNP位点,CT组中,3′-UTR与5′-UTR中共有的SNP位点为603个,上游区与下游区共有的有3940个,外显子与剪接位点中的为66个;MP组中,3′-UTR与5′-UTR中共有的SNP位点为606个,上游区与下游区共有3 879个,外显子与剪接位点中的SNP位点为70个,如图3所示。
2.3.3 SNP功能类型分析。
对太平鸡回肠SNP的功能类型进行分析,发现主要存在一些小规模的突变,其中同义突变SNP占比最高,CT组为7.01%,MP组占比为7.10%;非同义突变SNP在CT组中占比为2.97%,MP组占比为2.98%。其他突变既移码缺失、终止密码突变、非移码缺失、移码插入、非移码插入的SNP位点相继减少,另外CT组和MP组中分别存在未知功能的58个SNP位点(图4)。
3 讨论
新一代的测序技术出现后,通过转录组测序筛选获得SNP已成为开发应用于遗传多样性分析、分子育种等分子标记的重要技术手段,此技术相较于基因组筛序获得分子标记,不仅能大大降低成本,且能快速精且能快速精准地获得具有较高通用性的有用序列,使得开发利用分子标记更加有效便捷[15-16]。
该研究对太平鸡转录组进行了初步研究,采用高通量测序技术对太平鸡的回肠组织进行转录组测序。对太平鸡SNP位点进行分析,MP共获得SNP位点数4 647 171个,CT中含SNP 4 684 072个,SNP类型中转换类型明显高于颠换类型,MP和CT组中转换类型分别达到27.46%和27.45%,理论上发生转换的概率与发生颠换概率的比值应该等于0.5 (1∶2),但有些生物的比值常常>0.5,这种差异被称为“转换偏差”[17]。该研究发现转换概率约为颠换的1.98倍,大于理论值,说明存在转换偏差,这可能与碱基组成及进化过程的选择机制有关,表明碱基转换突变并不是随机产生[17]。另外,该研究发现G/A发生频率在转换类型中最高,这可能与在CG序列上出现的高频率,胞嘧啶极易甲基化有关。
对太平鸡回肠转录组序列SNP位置进行分析,CT组和MP组总的SNP位点分别为4 684 072和4 647 171个,其中位于内含子和基因间区的SNP位点最多,位于内含子的SNP位点在CT组占59.69%,MP组中占59.94%。位于基因间区的SNP位点在CT组占20.56%,MP组中占20.12%。SNP可在DNA、RNA和蛋白质不同水平影响基因的功能,尤其是位于编码区域内的编码SNP (cSNP)与基因的表达相关,直接影响着功能基因的作用途径[18]。通过cSNP分布情况以及功能分析可以得到许多与物种生长发育性状相关的重要资料[19]。位于基因调控区的SNP(pSNP)则会影响基因表达量[20]。因此,研究这两类SNP在太平鸡的功能和疾病发生发展方面具有更重要的意義。
对太平鸡突变类型的功能SNP进行分析,同义突变SNP占比最高,CT组中为7.01%,MP组中占7.10%;非同义突变SNP在CT组中占比为2.97%,MP组中占2.98%。其他突变既移码缺失、终止密码突变、非移码缺失、移码插入、非移码插入的SNP位点相继减少。编码区内的同义SNP造成的编码序列的变化不会引起氨基酸序列变化;而非同义SNP则会影响蛋白质序列,导致生物性状改变[21]。因此,开发太平鸡编码区非同义SNP具有重要的生物学意义。
从太平鸡回肠转录组获得的SNP位点标记可以进一步用来完成后续群体的进化分析和特异性SNP标记的开发,对太平鸡进行遗传改良,资源鉴定分析、高密度遗传连锁图谱的构建、提高养殖效益和健康化养殖水平以及分子标记辅助育种提供依据。
参考文献
[1]刘媚媚,徐魏芹,李金录,等.太平鸡遗传多样性及系统发育研究[J].基因组学与应用生物学,2016,35(7):1731-1737.
[2]巨玉鑫,郭志明,史兆国,等.太平鸡品种资源现状及发展建议[J].畜牧兽医杂志,2019,38(3):55-56,59.
[3]万太军.太平鸡饲养管理技术[J].农村养殖技术,2013(8):39-42.
[4]陈永亮,刘丹丹.抗生素在鸡体内残留及控制技术研究[J].家禽科学,2018(12):55-58.
[5]伏春燕,张燕,姚启蒙,等.益生菌对肉鸡生产性能和免疫机能的影响[J].山东农业科学,2020,52(2):111-116.
[6]刘维,励飞,聂勇.益生菌的作用机理及在养猪生产中的应用研究进展[J].饲料博览,2019(4):23-26.
[7]VANDEPLAS S,DUBOIS D R,BECKERS K,et al.Salmonella in chicken:Current and developing strategies to reduce contamination at farm level[J].Journal of food protection,2010,73(4):774-785.
[8]侯成立,季海峰,周雨霞,等.益生菌的作用机制及其在母猪生产中的应用[J].中国畜牧兽医,2011,38(7):20-22.
[9]葛影影,何国戈,郑经成,等.益生菌对禽消化道疾病防治作用研究进展[J].安徽农业科学,2020,48(6):23-24,29.
[10]LANDER E S.The new genomics:Global views of biology[J].Science,1996,274(5287):536-539.
[11]黄琳.牙鲆免疫相关基因及热休克蛋白基因的转录表达[D].青岛:中国科学院研究生院(海洋研究所),2015.
[12]郭晓峰,郑先虎,匡友谊,等.利用GoldenGate分析的高通量SNP芯片对鲤基因进行分型[J].水产学杂志,2016,29(6):19-25.
[13]金方彭,周睿,李光華,等.基于SLAF-seq技术的鱼康浪白鱼SNP位点开发[J].水产科技情报,2018,45(2):85-89.
[14]CHEN S F,ZHOU Y Q,CHEN Y R,et al.fastp:An ultrafast allinone FASTQ preprocessor[J].Bioinformatics,2018,34(17):i884-i890.
[15]SYVNEN A C.Accessing genetic variation:Genotyping single nucleotide polymorphisms[J].Nature reviews genetics,2001,2(12):930-942.
[16]孙扬,郭宝英,祁鹏志,等.基于转录组的曼氏无针乌贼SSR与SNP位点信息分析[J].浙江海洋大学学报(自然科学版),2019,38(2):100-106.
[17] LI H J,LIU M,YE S,et al.De novo assembly,gene annotation,and molecular marker development using Illumina paired-end transcriptome sequencing in the clam Saxidomus purpuratus[J].Genes & genomics,2017,39(6):675-685.
[18]化文平,韩立敏,魏磊,等.基于盾叶薯蓣转录组的SNP和SSR位点分析[J].分子植物育种,2017,15(10):4003-4009.
[19]聂鸿涛,姜力文,郑梦鸽,等.大竹蛏高通量转录组测序数据组装和分析[J].大连海洋大学学报,2017,32(6):658-663.
[20]ZHAO H,LI Q Z,LI J,et al.The study of neighboring nucleotide composition and transition/transversion bias[J].Science in China series C:Life sciences,2006,49(4):395-402.
[21]田义珂,王彩虹,白牡丹,等.基于梨贝壳杉烯氧化酶基因PpKO序列的功能性SNP标记[J].园艺学报,2012,39(10):1876-1884.
——中国益生菌信息与技术交流平台

