食源性致病菌高光谱识别模型构建
段朋生 许金钗 陈屹耒 叶大鹏 翁海勇
(1. 福建农林大学机电工程学院,福建 福州 350002;2. 现代农业装备福建省高校工程研究中心,福建 福州 350002;3. 福建农林大学生命科学学院,福建 福州 350002)
金黄色葡萄球菌(Staphylococcusaureus,S.aureus)、单增李斯特菌(Listeriamonocytogenes, LM)、致泻大肠埃希氏菌(Diarrheagenice.coli, DEC)、肠炎沙门氏菌(Salmonellaenteritidis,SE)和福氏志贺菌(Shigellasppflexneri,S.flexneri)等食源性致病菌是引起食品安全问题的主要原因[1]。由食源性致病菌所引发的急性食物中毒常出现恶心、腹痛、腹泻、发烧等症状,严重者威胁呼吸、循环、神经系统,甚至留下后遗症[2]。因此,及时快速诊断出食源性致病菌并判别出菌种属对食品安全至关重要。
目前,微生物检验技术是食源性致病菌检测中最常用的技术之一。该技术准确性高,但操作复杂、专业性强,检测时间长,一般需要7~10 d[3]。近年来,光谱技术能获取丰富的样本波谱信息,结合化学计量学方法在食品微生物快速检测领域具有巨大的应用前景[4-6]。刘斌等[7]对菌株富集培养后,获取了经冷冻干燥制成的菌粉的傅里叶近红外光谱信息,建立了偏最小二乘法模型,识别准确率达90%。Yoon等[8]利用高光谱成像技术对显色培养基上培养的17种弯曲杆菌亚种和非弯曲杆菌亚种进行了鉴别,识别准确率达99.29%,建立了17种菌的光谱库,并开发了一种可应用于其他琼脂平板上病原菌的分类鉴别方法。Mehrubeoglu等[9]研究发现,高光谱成像技术能表征细菌随培养时间而变化的光谱特征,以及实现细菌的鉴别。Seo等[10]发现,在显色培养基上应用高光谱成像技术能够实现肠炎沙门氏菌和鼠伤寒沙门氏菌的鉴别。William等[11]研究表明,高光谱成像技术对致病因子中含045和0121的分类准确率为98%,而对致病因子含026、0111、0103和0145的分类准确率为8%~100%。Gu等[12]利用高光谱技术建立了支持向量机(SVM)识别模型,对大肠杆菌、金黄色葡萄球菌和沙门氏菌的准确率>98%。石吉勇等[13]利用高光谱对乳酸菌专用培养基上常见的3种致病菌和5种乳酸菌种进行了鉴别与计数,发现标准正态变换(SNV)为最佳预处理方法,最小二乘支持向量机(LS-SVM)为最佳鉴别模型,识别率为91.88%。上述研究均要求对致病菌进行培养24 h,一定程度上限制了检测效率。
研究拟利用高光谱成像技术获取通用培养基上培养12,18,24 h的不同培养期5类致病菌(金黄色葡萄球菌、单增李斯特菌、致泻大肠埃希氏菌、肠炎沙门氏菌和福氏志贺菌)高光谱图像,探究其在通用培养基上不同生长期的图谱特性,并结合化学计量法建立通用培养基上的源性致病菌种的识别模型,旨在为食品中致病菌的快速检测提供依据。
1 材料与方法
1.1 试验样本制备
将菌种放置在无菌超净台中,活化,用接种环挑取S.aureus、DEC、SE和S.flexneri至营养肉汤培养基(蛋白胨10 g,牛肉膏3 g,NaCl 5 g,超纯水1 000 mL,pH 7.0),挑取LM至李斯特菌增菌液(胰蛋白胨17 g,大豆蛋白胨3 g,K2HPO42.5 g,NaCl 5 g,葡萄糖2.5 g,酵母浸粉6 g,超纯水1 000 mL,pH 7.0),37 ℃恒温培养24 h。将活化后的增菌液按10-1~10-9中的9个梯度进行稀释,用移液枪吸取增菌液至通用平板培养基上再次培养12,18,24 h。
1.2 食源性致病菌高光谱图像采集
高光谱成像系统(HIS)主要包括分辨率为1 024×472 pixels的sCMOS相机、波长范围为400~1 000 nm、分辨率为2.8 nm光谱仪、线光源、电控移动平台、暗箱和控制计算机等(图1)。试验前,开机预热20 min。设定物距、移动平台的速度和曝光时间等参数。经反复测试,最终确定的曝光时间为3 ms,物距为320 mm,移动平台速度为8.38 mm/s。采集样品的高光谱数据前,先扫描反射率为99%标准白板得到全白的标定图像Iwhite;拧上镜头盖,采集全黑的标定图像Idark;并按式(1)计算校正后的图像Icorrection。获取培养至12,18,24 h的5类致病菌(每类病菌在每个时间段各4个平板)的高光谱图像I,以期获得不同生长期的食源性致病菌波谱信息。对校正后的高光谱图像利用ENVI5.1软件,以单个菌落为感兴趣区域,并计算该区域的平均光谱曲线,以此作为一个样本。分别从培养12,18,24 h的培养基上选择647,799,769个样本,其相关信息见表1。
(1)
1.3 食源性致病菌高光谱数据处理
1.3.1 连续投影算法(SPA) 原始高光谱数据信息丰富,但存在数据冗余、维度大等问题,因此,有必要从高维度的图谱数据中提取出对食源性致病菌敏感的特征波段。利用SPA提取不同生长期致病菌敏感波段。SPA从原始的光谱信息中提取共线性最小的特征波长变量组合,以使输入数据的冗余信息达到最小[14]。
1.3.2 最小二乘支持向量机(LS-SVM) 支持向量机(SVM)是一种流行的机器学习算法,其是使定义在特征空间上的不同样本之间的间隔最大化,即寻找最优超平面,将不同样本分割开来,以达到分类和识别的目的[15-16]。而LS-SVM则是以最小二乘线性系统作为损失函数,将原有SVM中的不等式约束问题转化成等式约束,简化计算的复杂性,并提高计算效率[17-18]。

1. 光源 2. 相机 3. 光谱仪 4. 光源控制器 5. 镜头 6. 样品 7. 电脑 8. 样品架 9. 移动平台
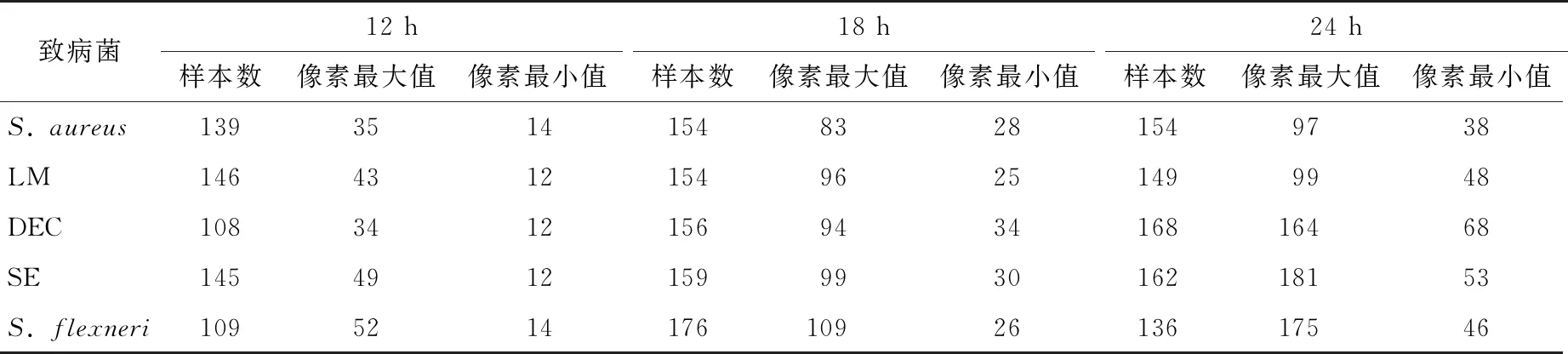
表1 样本统计
LS-SVM算法求解的目标函数为:
(2)
约束条件为:
yi=ωTφ(x1)+b+ei,
(3)
式中:
ω——权重;
γ——正则化参数;
ei——误差;
xi——输入的光谱矩阵,代表第i个样本;
yi——模型的输出变量代表,输出对应的标签值;
n——样本数。
对式(2)和式(3)进行求解,可引入如式(4)的Lagrange函数:
(4)
其中,αi为Lagrange系数,求解式(4)的最优解,将其转化为求解式(5),求解线性方程:
(5)
式中:
I——单位矩阵;
m=<φ(xi),φ(xi)>=K(xi,xj),i,j=1,…,n;
ɑ=[ɑ1, ɑ2,…, ɑn]T;
y=[y1,y2, …,yn]T;
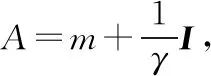
(6)
α=A-1(y-bl)。
(7)
对任意输入x,可得到LS-SVM判别函数:
(8)
选取径向基作为LS-SVM的核函数,采用网格搜索算法和交叉验证相结合方式实现LS-SVM模型中γ和sig2(σ2)的寻优。分类过程中,S.aureus、LM、DEC、SE和S.flexneri的标签分别赋值为1、2、3、4和5。应用Kennard-Stone(KS)算法将光谱数据按2∶1分成建模集和预测集。采用Unscrambler10.1(CAMO AS, Oslo, Norway)、MATLAB R2014a(MathWorks, Inc., Natick, MA, USA)和ENVI5.1(ITT Visual Information Solutions, Bounder, USA)软件进行光谱数据处理。
2 结果与分析
2.1 光谱分析
由图2可知,随着培养时间的增长,各类菌的反射率值均有所增大。可见光区域细菌的反射率值变化比近红外区域780~980 nm的变化更为明显,5类菌在400~1 000 nm 的光谱反射率值变化趋势相似,但不同种类的菌对不同波长光的反射能力不同,这与不同菌的内部物质成分不同有关[9],从侧面说明了高光谱成像技术对食源性致病菌的快速检测具有可行性。此外,由于细菌细胞中化学成分的含氢基团因不同振动方式的合频和倍频在近红外区域产生的谱带重叠,也会引起这些光谱反射率较为接近[19]。因此,无法仅依赖某单个波段的反射率来实现微生物种属的判别,需进一步提取更多的波谱特征,以实现5类致病菌种属的快速判别。
2.2 主成分分析
由图3可知,5类致病菌在培养12,18,24 h的PC1和PC2的累积贡献率分别为89.9%,96.6%,95.0%,说明PC1和PC2能够解释原始数据的绝大部分信息。当培养至12 h时,DEC的光谱与其他4类菌的光谱差异性已经凸显。当培养至18 h时,5类致病菌各自聚成一类的趋势更加明显。当培养至24 h时,5类致病菌的光谱特性差异性进一步增加,重叠部分逐渐减少。
2.3 食源性致病菌敏感波段筛选
由图4可知,当培养至12 h时,SPA算法选取了13个敏感波段(421,422,964,425,968,428,425,435,471,978,497,849,462 nm)用于构建5类致病菌的判别模型,RMSE最小为0.002 4。当培养至18 h时,SPA算法选取了9个敏感波段(421,422,450,853,505,763,431,649,978 nm)用于构建5类致病菌的判别模型,RMSE最小为0.001 9。当培养至24 h时,SPA算法选取了24个敏感波段(421,423,422,425,426,449,443,499,516,555,433,431,458,431,936,445,450,650,437,921,979,978,977,448 nm)用于构建5类致病菌的判别模型,RMSE最小为0.001 7。综上,421,422,978 nm在培养12,18,24 h均被选中为敏感波段,并且有些被选中的波段在不同的生长期非常接近,说明这些波段能较好地反映不同生长期的食源性致病菌的动态信息。
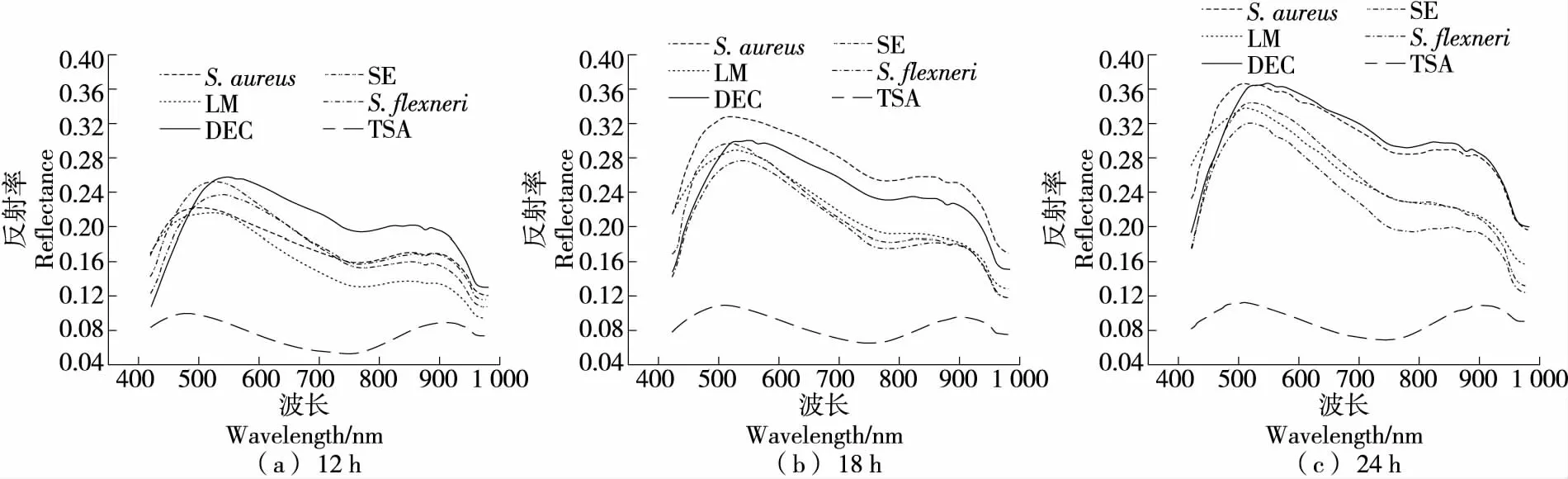
图2 5类致病菌的平均光谱曲线
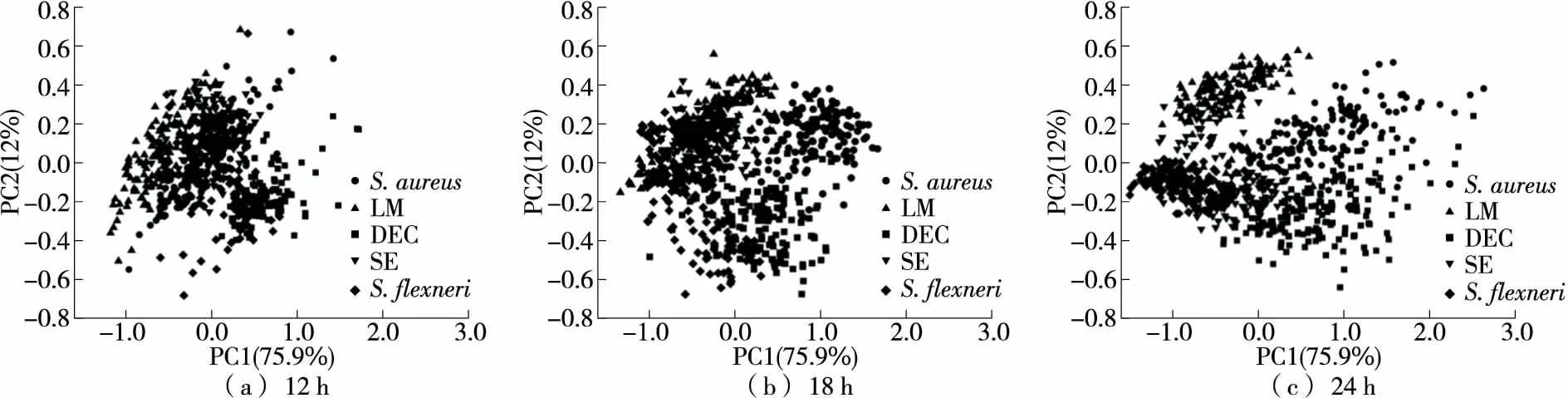
图3 5类致病菌的主成分分析得分图
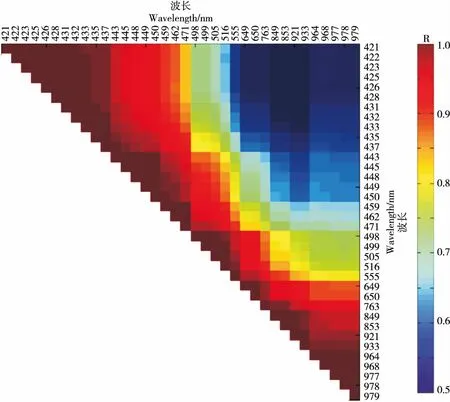
图4 SPA提取得到的36个敏感波段反射率的相关系数
为了更好地全面获取不同生长期食源性致病菌的波谱信息,有必要将培养12,18,24 h的被SPA选中的敏感波段组合起来,共获得36个敏感波段。当两个波段反射率的相关系数>0.95时,视为高度相关,可以去除其中一个。最终,选择462,498,649,853,979 nm 5个波段作为不同生长期食源性致病菌的敏感波段。
2.4 模型的识别效果分析
基于SPA-CA筛选的462,498,649,853,979 nm 5个波段反射率建立的LS-SVM模型对5类食源性致病菌的识别效果如表2所示。由表2可知,革兰氏阳性菌(S.aureus和LM)与革兰氏阴性菌(DEC、SE和S.flexneri)的总体识别准确率分别为99.6%和99.8%,与同属于革兰氏阳性菌或革兰氏阴性菌的鉴别较容易,这可能是由革兰氏阳性菌与革兰氏阴性菌在细胞壁中的主要成分肽聚糖和磷壁酸的差异引起的[20-21]。同属类别的细菌因化学组分相似,造成谱带重叠,易引起误判,如SE和S.flexneri最容易发生误判,其中SE被误判成S.flexneri的概率为11.2%,S.flexneri被误判成SE的概率为19.9%。总体而言,SPA-CA-LS-SVM模型的总体识别准确率为90.9%,说明高光谱成像技术结合化学计量学分析方法能实现食源性致病菌的快速检测。
3 结论
建立了基于高光谱成像技术的食源性致病菌检测方法。结果表明,5类致病菌对不同波长光的反射能力不同。连续投影算法结合相关分析筛选出的5个敏感波段(462,498,649,853,979 nm)反射率可较好地反映5类致病菌在不同生长期的波谱特性。基于5个敏感波段反射率构建的连续投影算法结合相关分析—最小二乘支持向量机模型对金黄色葡萄球菌、单增李斯特菌和致泻大肠埃希氏菌3类致病菌能够有效地判别,而肠炎沙门氏菌和福氏志贺菌两种属容易被互相误判,肠炎沙门氏菌被误判成福氏志贺菌的概率为11.2%,福氏志贺菌被误判成肠炎沙门氏菌的概率为19.9%。最小二乘支持向量机模型对5类致病菌的总体识别正确率为90.9%。综上,基于高光谱成像技术结合最小二乘支持向量机模型能够很好地对这5类食源性致病菌进行识别。由于食品受微生物的污染时有可能存在多种致病菌共同存在的情形,后续将针对混合菌种进行识别。
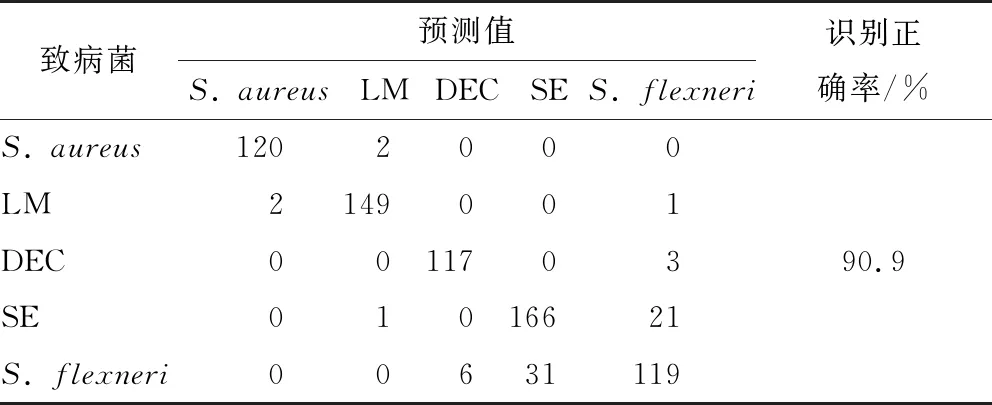
表2 SPA-CA-LS-SVM模型的识别效果

