一株多重耐药猪源ST37型肺炎克雷伯菌铁载体因子的遗传多样性分析
闫鹤 梁慧贤
(华南理工大学 食品科学与工程学院,广东 广州 510640)
肺炎克雷伯菌(Klebsiellapneumoniae)为革兰氏阴性菌,是引起人与动物感染的条件性致病菌[1]。现成为继大肠埃希氏菌(Escherichiacoli)后,临床排名第2位导致感染性疾病发生的肠杆菌科细菌[2]。2015—2016年,在澳大利亚的维多利亚州(Victoria)和昆士兰州(Queensland)发生肺炎克雷伯菌感染,引起新生仔猪发生败血症,死亡率分别为60%和100%[3]。在我国沧州不同猪场收集的45株致病性肺炎克雷伯菌中,耐10种药物的菌株占28.9%,β-内酰胺耐药基因blaTEM、氨基糖苷耐药基因ant(3”)-Ia和aph(3’)-IIa、磺胺类耐药基因sul2和sul3 5种耐药基因检出率在 80.0% 以上,β-内酰胺耐药基因blaSHV、氟苯尼考耐药基因floR、喹诺酮耐药基因qnrA3种耐药基因检出率在 37.8%~46.7%[4]。
肺炎克雷伯菌可分为经典型(cKP)和高毒力型(hvKP)。铁元素是其生长的必需元素,为了在宿主细胞中生长和增殖,肺炎克雷伯菌通过分泌铁载体因子,进而从宿主铁螯合蛋白中获得铁元素[5]。更为重要的hvKP通过分泌多种铁载体因子,获得更多的铁元素,进而增强致病性。肺炎克雷伯菌编码几种铁载体,包括肠抑菌素、耶尔森杆菌素、沙门菌素和气杆菌素[6],以上4种铁载体对毒力的表达和贡献不同[7]。研究表明,肠杆菌素是肺炎克雷伯菌主要使用的铁吸收系统,几乎存在于所有类型的肺炎克雷伯菌中[8]。肠杆菌素合成由位于基因组上entABCDEF基因簇所编码,而fepABCDG基因簇编码蛋白介导其转运[9]。研究发现,仅有6%的cKP合成气杆菌素[10],而气杆菌素是hvKP增强侵入性感染的主要毒力因子[11]。
多位点序列分型(MLST)是一种基于7个管家基因(rpoB、gapA、mdh、pgi、phoE、infB和tonB)的核苷酸序列比对分析方法,被广泛用于肺炎克雷伯菌菌株的分型[12]。全基因组序列正迅速成为预测肺炎克雷伯菌的抗生素耐药性和致病潜力的强大工具,尤其是对了解相关菌株特征的持续进化和地理分布具有重要意义[13]。目前针对猪源多重耐药模式的肺炎克雷伯菌毒力基因的研究较少,本研究以分离自腹泻的猪肝脏的ST37型肺炎克雷伯菌KP200为研究对象,进行比较基因组学分析,并研究铁载体因子的遗传多样性,旨在为动物源肺炎克雷伯菌的进化及毒力研究提供理论依据。
1 材料与方法
1.1 材料
1.1.1 菌株
多重耐药肺炎克雷伯菌KP200分离自某养猪场腹泻猪的肝脏,该菌株KP200表现出对β-内酰胺类、氨基糖苷类、喹诺酮类和磺酰胺类抗生素的多重耐药性。
1.1.2 相关试剂与仪器
Luria-Bertani(LB)液体培养基,购自广州环凯微生物科技有限公司;细菌基因组DNA快速提取试剂盒,购自北京博迈德生物技术有限公司。Pacbio RS II 测序仪,Pacific Biosciences公司生产;Illumina MiSeq测序仪,Illumina MiSeq公司生产。
1.2 方法
1.2.1 基因组测序与注释
将菌株KP200划线接种至LB琼脂培养基,37 ℃培养过夜,再挑取单菌落接种至LB液体培养基中,37 ℃培养12~16 h。DNA 提取按照细菌基因组 DNA 快速提取试剂盒说明书进行。将得到的总DNA通过 Pacbio RS II 和Illumina MiSeq测序仪进行全基因组测序。全基因组测序数据使用SPAdes v3.9.0和Canu v1.4软件进行组装,将完整的基因组序列提交至美国国立生物技术信息中心(NCBI)GenBank数据库,获得该菌株的登录号为 CP055293,并通过原核基因组注释(PGAP)进行基因预测与功能注释。采用 Glimmer 3.02 软件进行开放阅读框(ORF)预测,将所有预测蛋白序列与非冗余蛋白数据库 NR、Swiss-Prot 数据库、eggNOG 数据库进行BLASTp(E<10-10,E表示随机情况下,获得与当前比对分数相等或更高分数的可能比对条数)比对,完成蛋白序列功能注释。
1.2.2 MLST分析
根据PasteurMLST 数据库,确定7个管家基因(rpoB、gapA、mdh、pgi、phoE、infB和tonB)的等位基因号,得出肺炎克雷伯菌KP200菌株的ST型别。
1.2.3 单拷贝基因组进化
在NCBI GenBank数据库查找已经注释的ST37型肺炎克雷伯菌全基因组,加上KP200共获得161株全基因序列。统一使用prokka v1.14.6[14](默认参数)进行注释得到基因文件和蛋白文件。使用orthofinder v2.4.0[15](推荐参数)获得所有(161株)菌株的单拷贝同源蛋白序列文件,使用muscle v3.8.31[16](推荐参数)进行多蛋白序列比对,MEGA X[17]使用默认参数构建单拷贝核心同源蛋白序列最大似然树(Maximum-likelihood,ML),Bootstrap值设置为1 000次。
1.2.4 铁载体因子分析
使用毒力因子VFDB数据库,对肺炎克雷伯菌KP200和160株ST37型(NCBI GenBank数据库)肺炎克雷伯菌进行铁载体因子序列分析。根据比对结果,展示161株ST37型肺炎克雷伯菌中有差异的铁载体因子。
1.2.5 气杆菌素转运基因iutA分析
仅有16株ST37型肺炎克雷伯菌携带气杆菌素转运基因iutA,以KP200为参考菌株,进行基因同源性比较,并根据iutA基因的核苷酸序列,利用软件MEGA X构建ML进化树(1 000 Bootstrap)。将高毒力肺炎克雷伯菌NTUH-K2044携带的iutA基因和KP200携带的iutA基因编码的氨基酸序列进行对比分析,研究KP200携带的iutA基因的碱基突变情况。
2 结果与讨论
2.1 肺炎克雷伯菌KP200基因组的基本特征
肺炎克雷伯菌KP200的基因组全长为5 257 665 bp,平均碱基GC含量为58.78%,共有5 106个编码基因。通过与eggNOG数据库比对,5 106 个编码基因中有4 812个基因编码的蛋白获得COG(直系同源蛋白簇)功能注释,占比为94.24%。COG功能注释分布于22个COG的条目中,大多数注释的基因与代谢有关,共占比38.856%。
2.2 MLST分析结果
根据PasteurMLST数据库比对7个管家基因(rpoB- 1、gapA- 2、mdh- 2、pgi- 1、phoE- 13、infB- 9 和tonB- 16),确定肺炎克雷伯菌KP200的分型为ST37。
2.3 单拷贝基因组进化分析
从NCBI数据库中收集不同来源的160株ST37型肺炎克雷伯菌,其中人源109株、鸡源9株、未知来源33株、环境来源8株以及食品来源1株;主要来自美国(34株)和中国(32株)。
基于全基因组水平的单拷贝基因分析表明,161株ST37型肺炎克雷伯菌共检测到2 563个单拷贝基因。基于每株菌的单拷贝基因构建系统进化树,进化树分析结果表明,161株ST37型肺炎克雷伯菌主要位于4个不同的分支(见图1)。KP200与人源肺炎克雷伯菌WCHKP030295的中国四川菌株位于相邻的进化分支,分离时间分别为2017年和2016年,相似度为99.8%,亲缘关系较近。
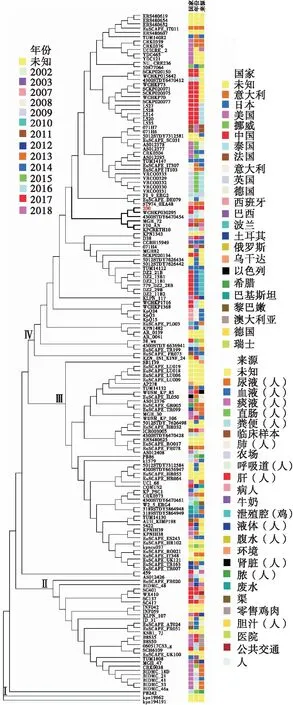
图1 161株ST37型肺炎克雷伯菌的最大似然树Fig.1 Maximum-likelihood phylogeny of 161 Klebsiella Pneumoniae isolates
2.4 铁载体因子分析
经毒力因子VFDB数据库对161株肺炎克雷伯菌基因组进行30个铁载体因子的分析,结果显示不同分支的ST37型肺炎克雷伯菌之间铁载体因子的分布存在一定的差异(见图2),对比结果发现,KP200含有3种铁载体因子相关基因。
161株肺炎克雷伯菌基因组上均携带肠杆菌素基因entABCEF和fepABC,仅entD、fepD和fepG在161株肺炎克雷伯菌中的分布存在差异,结果表明肠杆菌素存在于所有的ST37型肺炎克雷伯菌中。
沙门菌素是通过染色体或者质粒编码的iroA基因簇iroBCDE进行肠杆菌素的c-葡萄糖基化修饰而产生[9,18],基因IroN介导了铁负载形式的转运[19]。重要的是,这种修饰可防止沙门菌素被蛋白Lcn2结合[20],从而增强肺炎克雷伯菌在含有充足蛋白Lcn2的宿主中定植[21],含有沙门菌素的肺炎克雷伯菌更具有毒性。对比结果表明,KP200携带iroE和iroN基因。仅有KP200和另外15株肺炎克雷伯菌携带iroN基因,对16株肺炎克雷伯菌携带iroN沙门菌素基因进行同源性比较,结果表明该基因在核苷酸水平上高度保守,相似度达100%。

同一类型的铁载体因子的毒力基因用不同颜色高亮显示,其余使用橘黄色显示,仅显示161株中有差异的铁载体因子
耶尔森杆菌素是耶尔森杆菌高致病性因子的一部分,发现在肺炎克雷伯菌中也存在[22]。尽管在肺炎克雷伯菌中尚有待充分表征,但是普遍认为合成耶尔森杆菌素蛋白是由基因irp编码,分泌铁载体需要的转运蛋白由ybt和fyu基因编码,摄取受体由ybtQ基因编码[23]。前人的研究表明,仅18%的经典型肺炎克雷伯菌(cKP)携带耶尔森杆菌素[24],KP200不携带耶尔森杆菌素相关基因。
气杆菌素是一种柠檬酸盐-异羟肟酸酯铁载体[5],由iucABCD基因簇编码和iutA基因转运[25]。
对比结果表明,肺炎克雷伯菌KP200和另外15株肺炎克雷伯菌携带气杆菌素转运基因iutA。
2.5 KP200与15株肺炎克雷伯菌携带的转运基因iutA的比较分析
iutA基因常作为识别高毒力肺炎克雷伯菌的特征基因之一[26- 27]。探究肺炎克雷伯菌KP200与另外15株肺炎克雷伯菌携带的iutA基因的进化关系,结果见表1,由表1可知,16株肺炎克雷伯菌携带的iutA基因大小不同,KP200携带的iutA基因为2 190 bp;进一步比较分析发现,KP200携带的iutA基因序列与其他15株肺炎克雷伯菌携带的iutA基因序列核苷酸相似性为73.49%~100%。
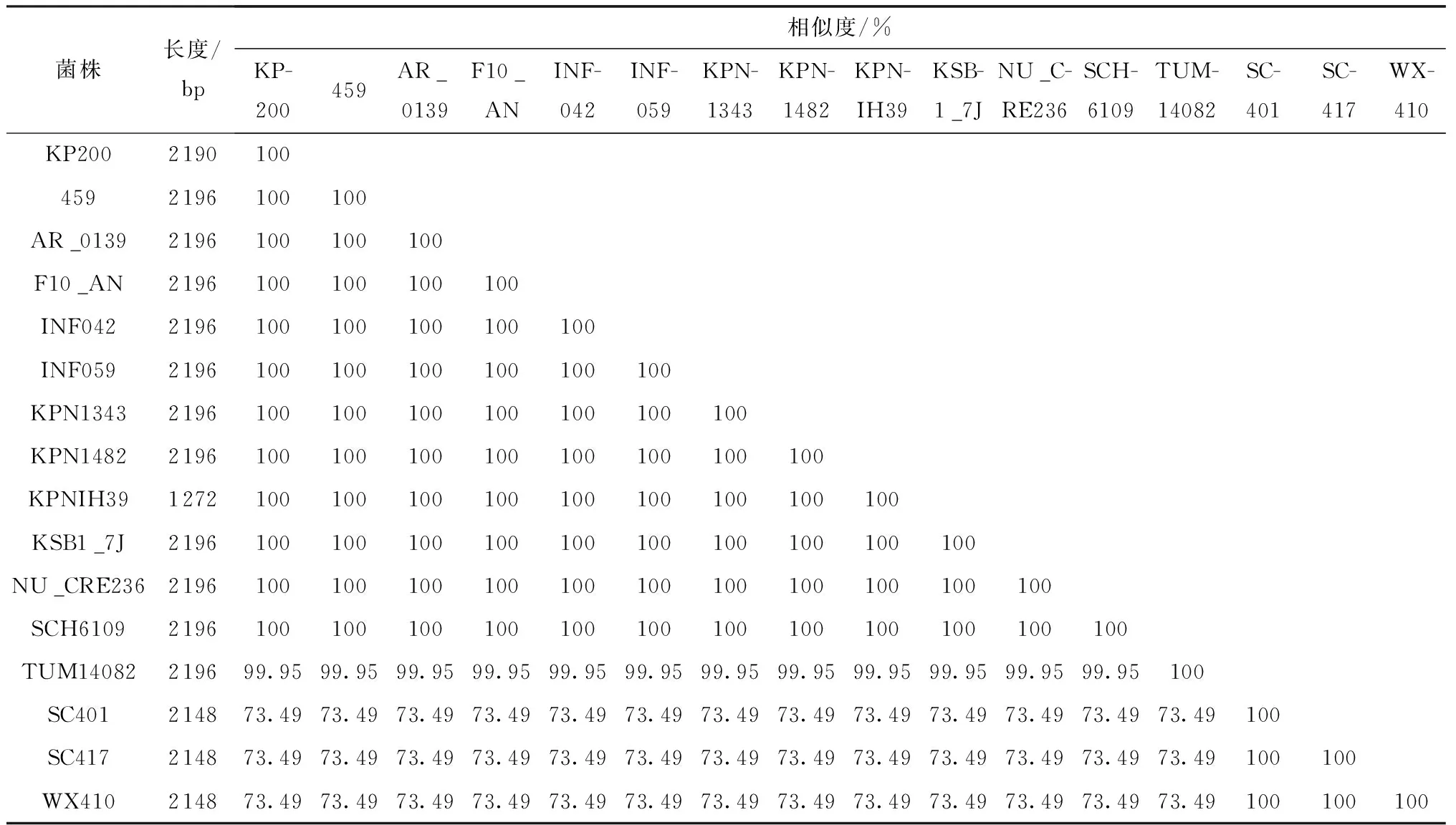
表1 15株分离株与KP200携带的iutA基因的比较结果
根据16株肺炎克雷伯菌携带的iutA的核苷酸序列构建系统进化树。该进化树含有2个簇,与KP200携带的iutA核苷酸序列相似度高的序列聚在一个分支,与KP200携带的iutA核苷酸序列相似度低的序列聚在另一个分支。图3表明,KP200携带的iutA核苷酸序列与肺炎克雷伯菌F10_AN携带的iutA核苷酸序列聚在一起,这与单拷贝基因组进化分析结果相似。对比KP200与肺炎克雷伯菌F10_AN携带的iutA核苷酸序列发现,KP200丢失前端6个碱基(ATGGAT),推测在遗传进化过程中导致了碱基的丢失,是否影响iutA基因表达还需进一步研究。
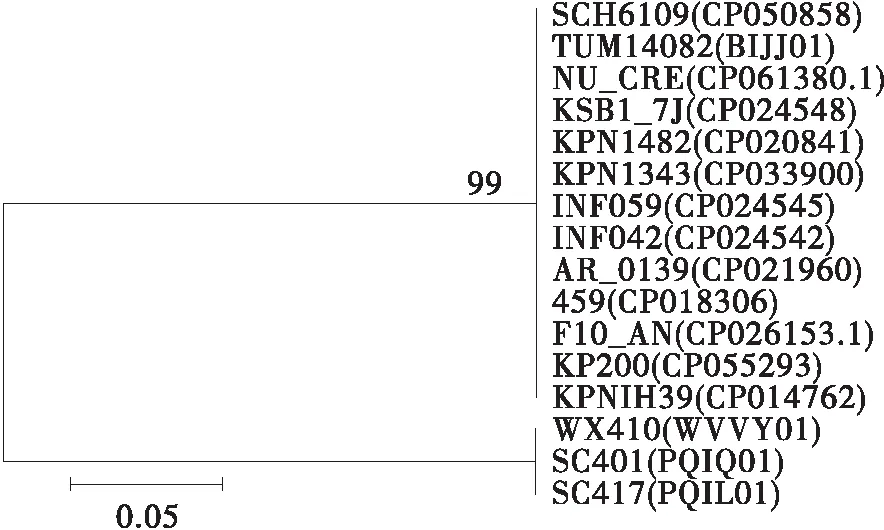
图3 基于16个肺炎克雷伯菌的iutA序列的最大似然树Fig.3 Maximum likelihood tree of 16 Klebsiella Pneumoniae isolates based on iutA sequence
2.6 KP200与NTUH-K2044携带的iutA基因的比较分析
肺炎克雷伯菌铁载体因子发生突变可能影响其抵御宿主清除的能力[28]。研究表明,不同的毒力因子在肺炎克雷伯菌中存在不同的基因突变。ST37型肺炎克雷伯菌携带的iutA基因碱基突变情况尚无研究。3型菌毛毒力基因mrkD在不同型别的肺炎克雷伯菌中存在6种类型的基因突变[29]。气杆菌素基因iuc谱系和肺炎克雷伯菌的型别在采样点之间呈差异性分布[30]。比较KP200与高毒力肺炎克雷伯菌(hvKP)携带的iutA基因突变情况,将NTUH-K2044作为参考菌株[9,31- 32]。分析发现,KP200菌株携带的iutA基因前端缺少21个碱基,即缺少7个氨基酸。进一步分析发现,KP200菌株携带的iutA基因的碱基突变导致其编码氨基酸序列也发生了相应的变化(见图4),与NTUH-K2044相比较,KP200仅有两个氨基酸不同:亮氨酸(L)替换成苯丙氨酸(F),缬氨酸(V)替换成亮氨酸(L)。

仅显示氨基酸缩写,红色表示突变氨基酸
进一步研究碱基突变和氨基酸替换发现,KP200由于碱基替换,C835T导致了亮氨酸变为苯丙氨酸(L279F),G2032C导致缬氨酸变为亮氨酸(V678L)。研究表明,在三甲医院爆发的耐碳青霉烯的ST258型别肺炎克雷伯菌,携带的iutA基因的碱基突变为非同义的单核苷酸突变,突变的结果导致翻译终止[33],与本研究的分析结果不同。
3 结论
单拷贝基因组进化分析表明,分离自病猪肝脏的ST37型别肺炎克雷伯菌KP200与1株来自中国人源的菌株具有较近的亲缘关系。通过铁载体因子的比较分析发现,所有ST37型肺炎克雷伯菌菌株携带肠杆菌素,KP200与其他15株肺炎克雷伯菌携带气杆菌素转运基因iutA,这些iutA基因序列间核苷酸相似性为73.49%~100%。基于iutA基因的序列的进化树结果表明,与KP200携带的iutA核苷酸序列相似度高的序列进化关系更近。与高毒力肺炎克雷伯菌NTUH-K2044携带的iutA核苷酸序列比较,结果发现KP200携带的iutA核苷酸由于两处碱基突变,导致两个氨基酸替换。猪源ST37型肺炎克雷伯菌KP200携带的铁载体因子毒力实际效果还需进一步探究,有望为养殖场感染肺炎克雷伯菌问题提供研究方向和理论依据。

