中文医学知识图谱研究及应用进展
范媛媛,李忠民
中南大学 生命科学学院,长沙410013
受语义网(semantic web)概念的启发,Google公司于2012 年提出了知识图谱(knowledge graph,KG),目的是为了提高搜索引擎的效率和精确度,提供更好的搜索质量和用户体验。随后,这一概念得到了传播并广泛应用于电商、金融、教育和医疗等行业中,推动人工智能从感知智能向认知智能跨越。知识图谱是一种用图模型来描述知识和建模世界万物之间的关联关系的技术方法,它吸收了本体和语义网在知识组织和表达方面的理念,以符号形式描述物理世界中的概念及其相互关系,使得知识更易于在计算机之间和计算机与人之间交换、流通和加工。目前,国内外已经涌现出一大批通用知识图谱,如DBpedia、Freebase、Yago、CN-DBpedia、Zhishi.me以及OpenKG等都很有代表性。由于通用知识图谱具有规模大、领域宽、知识广、技术成熟等特点,在综合性搜索引擎和常识性智能问答方面已经得到了广泛应用。因此,研究人员将目光逐渐聚焦到领域知识图谱的构建及应用上。
在卫生信息化进程不断深入、医学数据规模指数增长的背景下,知识图谱作为机器认识世界的基石,无疑会在医学人工智能的实现上发挥重要的作用。早期与医学知识图谱相关的研究成果已有许多,国外有一体化医学语言系统(unified medical language system,UMLS)、医学系统命名法-临床术语(systematized nomenclature of medicine-clinical terms,SNOMED-CT)和生物医学领域语义数据集成平台(linked life data)等,国内则有中文一体化医学语言系统(Chinese unified medical language system,CUMLS)、中医药学语言系统(traditional Chinese medicine language system,TCMLS)等。有关医学知识图谱近期的研究成果如何,笔者对近五年的综述文献进行了梳理,发现国外学者比较关注知识图谱构建技术的发展,倾向于将基因组学的内容也纳入医学知识图谱中,侧重知识图谱在用药推荐、新药发现等方面的应用。国内学者通常从知识图谱架构、技术及在医疗服务中的应用场景等方面展开综述,也有学者用文献计量学的方法探讨了中文医学知识图谱研究热点的变化。纵观现有的综述文献,少有学者关注通用技术在中文医学知识图谱构建方面的研究进展,也少有学者对中文医学知识图谱已有的研究和应用成果进行系统梳理。因此,本研究将从以下三方面对中文医学知识图谱的研究现状进行综述:(1)对医学知识图谱构建的关键技术进行梳理,发现新的技术进展以及难点;(2)从医学本体构建、全科医学知识图谱构建和单病种医学知识图谱构建三方面对中文医学知识图谱已有的研究成果进行总结,以便学者寻找新的研究方向;(3)对中文医学知识图谱已有的应用成果进行梳理,并探讨未来新的应用场景。
在CNKI、PubMed、WOS、Elsevier 以及谷歌学术等数据库中,采用“知识图谱(knowledge graph)”“知识表示(knowledge representation)”“知识抽取(knowledge extraction)”“知识融合(knowledge fusion)”“知识推理(knowledge reasoning)”与“医学(medicine)”“医疗(medical)”“疾病(disease)”进行组配检索,文献类型选择非综述,时间跨度限近五年,同时利用追溯法对重要文献的引文进行扩展检索,共检索出472篇文献。对外文文献进行初步的整理和归纳后,发现除英文之外还有针对印尼语、阿拉伯语、瑞典语和西班牙语等语种的医学命名实体识别研究,但未涉及医学知识图谱构建及应用。因此本文仅保留代表知识图谱技术进展的经典文献以及相关度较高的医学知识图谱中英文文献进行综述。
1 知识图谱构建的关键技术
知识图谱的构建指通过从大量的结构化或非结构化的数据中提取出实体、关系、属性等元素绘制成图谱,并选择合理高效的方式进行存储。根据知识图谱的逻辑结构可将知识图谱划分为两个层次:模式层和数据层。模式层存储的是用于规范实体、属性和关系,经过提炼的知识,通常借助本体库来管理,指导数据层的构建;数据层存储的则是以三元组为基本表达单元的一系列具体事实。正是由于这一逻辑结构,知识图谱衍生出了自顶向下和自底向上两种构建方式,前者常用于领域知识图谱的构建,后者常用于通用知识图谱的构建。
无论何种知识图谱构建方式,都涉及知识表示、知识抽取、知识融合以及知识推理这些关键技术,即需要首先确定知识表示模型,然后对不同来源的数据选择不同的手段进行知识抽取,利用知识融合和知识推理技术提升知识图谱的质量,最后根据具体的应用场景设计不同的知识访问与知识呈现方式。具体流程如图1 所示。本文从知识图谱的全生命周期出发,对知识图谱关键技术的研究进行分析。
1.1 知识表示
知识表示是对现实世界的一种抽象表达,知识必须经过合理的表示才能被计算机处理。从图1可以看到,知识表示主要有符号表示和向量表示两种形式。

图1 知识图谱构建流程Fig.1 Construction process of knowledge graph
以符号逻辑为基础的知识表示方法主要包括产生式表示法、框架表示法、语义网络表示法等,由于这几种方法都缺少严格的语义理论模型和形式化的语义定义,Baader等提出了描述逻辑语言以提升知识表示的能力,进而满足复杂程度更高的推理需要。在Tim Berners-Lee提出语义网概念后,业界需要一套标准语言来描述Web的各种信息。W3C就以描述逻辑为基础提出了资源描述框架(resource description framework,RDF)、RDF 模式(resource description framework schema,RDFS)和网络本体语言(Web ontology language,OWL)来规范互联网中的知识表示,使信息可以被计算机应用程序读取并理解。
由于符号化的表示无法满足计算的需要,向量化表示很快成为了知识表示的主流形式,即将语义信息表示为稠密、低维、实值向量,通过计算习得自然语言中的复杂语义模式,以解决知识图谱面临的计算效率低和结构稀疏等问题。自Word2vec问世以来,以深度学习为代表的知识表示学习(knowledge graph representation learning,KRL)研究获得广泛关注。在Word2Vec 的启发下,Bordes 等提出了翻译模型TransE,许多学者在这一经典模型上进行研究和改进,先后提出了TransH、TransD、TransR和TransG等基于复杂关系建模的知识表示模型。国外有学者对不同的知识表示模型在生物医学领域的应用进行了研究,在关系抽取和链接预测任务中,利用TransE 进行嵌入表示的效果都优于其他常用的知识表示模型。随着知识表示和知识外延的扩充,越来越多的知识表示模型不断被提出,如Deep-Walk、Node2Vec以及SDNE(structural deep network embedding)等。
针对知识表示学习在中文医学数据上的应用,国内学者也进行了研究。Zhao等在其研究中使用TransE 模型对中文电子病历中的医学实体进行分布式表示,实验结果表明向量表示确实有利于挖掘医学知识之间的关系,并有利于推理计算。Li等还基于TransH 模型提出了一种将知识三元组的不确定性引入到翻译学习算法中的增强模型PrTransH,并利用该模型学习中文疾病实体的嵌入向量,对从电子病历中抽取到的疾病实体进行聚类,完成了实体排序任务,实验证明该模型在中文表示学习方面优于TransH。沈思等以中文肿瘤期刊全文为研究对象,用主题词嵌入表示模型(topic word embedding,TWE)进行词向量和主题向量的词嵌入表示,然后基于孪生神经网络模型进行相似度计算,实验结果表明嵌入主题层面的语义信息有利于挖掘中文医学文本中的关联知识。
与国外研究相比,中文医学知识表示的研究大多采用单一模型,缺乏对不同模型的对比研究,也未见针对中文医学知识表示的新模型提出。但现有的研究也证实了知识表示学习能有效提升计算机对中文医学文本的处理能力,未来仍值得深入研究。
1.2 知识抽取
知识抽取是实现自动化构建知识图谱的重要技术,其目的在于从不同来源、不同结构的数据中进行知识提取并存入知识图谱中。由图1 可以看出,知识抽取包括了实体抽取、关系抽取和属性抽取,其中实体抽取和关系抽取最为关键。
实体抽取又称命名实体识别,常用的方法有基于词典及规则的方法、基于统计模型的方法和基于深度学习的方法。基于词典及规则的方法需要事先编制词典或制定规则,虽精确度高,但召回率低。基于统计模型的方法则过分依赖人工标注语料的质量。由于缺乏中文标注语料,现有研究多在国外的公开语料GENIA 和BioCreative 大赛的语料库上进行。基于深度学习的方法直接以文本中的词向量作为输入,可以有效地减少模型对人工标注数据的依赖,目前在命名实体识别方面的研究较多。
由于上述三种方法均存在一定的局限性,就有学者对混合实体抽取方法进行了探索。栗伟等提出了机器学习与规则结合的方法对医学实体进行抽取,以中文电子病历为数据集进行实验,取得了不错的结果。2016 年,Lample等开创性地提出了长短期记忆网络(long short-term memory,LSTM)与条件随机场(conditional random fields,CRF)模型相结合的实体抽取方法,并在实验中取得了与传统统计方法最好结果相近的结果,很快这种模型就成为了学界研究的热点。国内许多学者在此模型的基础上结合不同的词向量预训练模型进行中文医学实体识别工作都取得了较好的结果。在不同的预训练模型中,基于Transformer 的双向编码器表示模型(bidirectional encoder representations from transformers,BERT)能很好地处理中文医学文本中常见的一词多义问题,因此BERT-BiLSTM-CRF 模型在中文医学实体识别中得到了更广泛的应用。当学界聚焦于基于深度学习的实体抽取时,Ramachandran等提出了基于词典和深度学习混合的命名实体识别方法。他们在研究中构建了医学词典,依据词典对文本进行标注,用标注数据训练深度学习模型,再用词典验证模型识别的结果。利用该混合方法进行生物医学文献命名实体识别,准确率比基线模型提升了约0.15。
关系抽取一般是在实体抽取完成之后,通过从文本中抽取实体之间的关联关系,将识别出的一系列离散实体联系起来。早期的关系抽取方法大多基于模板匹配实现,由领域专家手工编写模板,从文本中匹配具有特定关系的实体。但由于人工构建的模板数量有限,覆盖范围较小,在系统中召回率普遍不高,因此学界开始尝试采用基于监督学习的关系抽取方法,包括最大熵方法、核函数方法和特征工程方法等,这些方法本质上还是依赖标注数据对统计模型进行训练从而实现关系抽取。为了能进一步减少模型训练对标注数据的依赖,基于弱监督学习的关系抽取方法也逐渐成为了学界的一大研究热点。目前比较有代表性的模型有Ji等提出的基于句子级注意力和实体描述的神经网络关系抽取模型(attention piecewise convolutional neural networks,APCNNs)以及Feng等基于卷积神经网络(convolutional neural network,CNN)提出的强化学习关系分类模型(convolutional neural networks reinforcement learning,CNN-RL),还有Carlson等提出的一种基于Bootstrap 算法的半监督学习方法等。
目前中文医学实体抽取的研究常用基于深度学习的方法。曹春萍等使用BioCreative V 大赛的语料库与数据库进行实体关系抽取,针对长文本中存在核心实体关系不精确的问题,提出了双向简单循环神经网络与带注意力机制的卷积神经网络相结合的模型,实验验证该模型在化学物质与疾病的关系抽取中具有良好表现。丁泽源等利用公开的英文生物医学标注语料,结合翻译技术和人工标注方法构建了中文生物医学实体关系语料,然后使用结合注意力机制的双向长短期记忆网络抽取实体间的关系。实验结果表明,该方法可以准确地从中文文本中抽取生物医学实体及实体间关系。此外,高峰等在BiGRU-2ATT 模型之上融合了关系发现词算法,将关系发现词作为模型的额外特征输入对诊疗关系进行抽取,有效提升了模型性能。武小平等根据中文语义中主要以词而不是字为基本单位的特点,提出了改进的基于全词掩膜的BERT-CNN 模型。这两项实验均提升了中文语料关系抽取的性能,但所用数据集均为学者自主构建,难免影响模型的可移植性。
1.3 知识融合
知识图谱中的数据由于来源不同常存在异构现象,导致了知识质量的参差不齐。知识融合就是通过映射和匹配使不同来源的知识在同一框架规范下进行整合、消歧和加工。知识融合对提升知识图谱的质量、知识复用以及实现异构数据源之间的语义互通都具有重要意义。知识融合的主要任务包括实体对齐和实体消歧。
知识在不同的数据源中常出现多元共指现象,实体对齐就是用于解决异构数据中的实体冲突、指向不明等不一致问题。传统的实体对齐方法主要依赖众包技术或者利用维基百科的信息框等结构良好的模式进行。由于人工成本较高且难以大规模应用,基于机器学习和深度学习的实体对齐方法的研究很快就在学界兴起。决策树算法很早就被用来解决实体对齐问题,近年仍有学者在此算法上结合知识嵌入进行深入探索。深度学习方面,国内学者李文娜等利用TransE 模型表示实体的结构信息,利用BERT 模型表示实体的语义信息,并据此设计了联合语义表示模型完成了不同知识库之间的实体对齐任务。Zhang等提出了一种基于语义和结构嵌入的相关性预测方法(semantic&structure embeddings-based relevancy prediction,S2ERP),该方法在使用BERT 模型获取实体语义嵌入的同时使用图卷积网络(graph convolutional network,GCN)获取术语库中实体同义词和下位词的结构嵌入,从而完成电子病历与知识库之间的实体对齐。
实体对齐解决了同义异名的问题,而实体消歧则用来解决不同知识库之间实体的同名异义问题。实体消歧的核心思想就是聚类,关键在于如何定义实体对象与指称项之间的相似度。较为常用的一种方法为词袋模型,将当前实体指称项周边的词构建成特征向量,利用余弦相似度进行比较从而完成聚类。然而这种方法没有考虑上下文的语义信息,在性能上就会有一定的损失,而后就有学者提出了基于语义上下文相似度的实体消歧方法。现有的研究大多依赖外部知识库进行实体消歧,如Han 和Zhao选择以维基百科作为背景知识,将各词条之间的关联关系融合进了实体指称项的相似度计算中,提升了实体消歧的效果。王静等基于DBpedia 知识库生成候选实体指称,再利用概率模型计算实体上下文和实体指称上下文之间的相似度,选取相似度最大的实体作为目标实体,完成生物医学领域文献中的实体消歧并在实验中取得了83%的准确率。为了减少实体消歧对外部资源的依赖,Duque等开发了一个实体消歧系统,先以PubMed 上下载的文献摘要为数据源,采用无监督的方法自动构建知识图谱,然后使用PageRank 算法进行词义消歧。在深度学习技术方面,Vretinaris等对图神经网络(graph neural networks,GNN)模型进行了改进,将来自医学知识库的领域知识引入到查询图中,并在负采样过程引入了生成对抗网络(generative adversarial network,GAN),以避免梯度消失的问题,从而获得更好的性能,有效解决了医学领域的实体消歧问题。
知识融合对医学知识图谱质量的提升具有重要意义,然而目前中文医学知识融合的研究相对较少,高效且可扩展性强的中文医学知识融合算法仍有待深入研究。
1.4 知识推理
知识推理指通过计算从图谱中已有的实体关系中挖掘出隐含信息。知识图谱也正是由于具备可推理性而广泛应用于不同领域的具体业务中。传统的知识推理方法有基于描述逻辑推理、基于规则推理与基于案例推理等。Bousquet等使用DAML(DARPA agent markup language)+OIL(ontology inference layer)描述逻辑语言对监管活动医学词典(medical dictionary for regulatory activities,MedDRA)执行术语推理来改进药物警戒系统中的信号检测。Chen等采用基于规则推理的方法开发了糖尿病诊断系统以提供用药建议。由于案例推理与医疗诊断具有极高的相似性,符合医学专家求解新问题的思维过程,在医学领域的应用更为广泛,国内相关研究也较多。沈亚诚和舒忠梅提出了患者病历的多元式表示法,并结合归纳索引法与最近邻法构建了基于病历的案例推理系统。Ping等提出了基于多重测量值的案例推理方法(multiple measurements case-based reasoning,MMCBR)来建立肝癌复发预测模型,该模型综合患者在一定时间序列的多个测量指标来进行案例匹配,实验表明模型性能优于单测量值的案例推理。陈延雪等以医疗领域的突发事件为主体,结合基于规则和基于案例的推理方法构建了医疗应急响应决策支持系统。
随着知识数量的激增以及复杂程度的不断加深,传统知识推理方法表现出了学习能力不足、准确率较低等缺陷,因而基于神经网络的推理和基于图的推理很快引起学界的关注。英文医学知识推理在这方面已有一定的研究积累,相关工作包括利用图神经网络预测蛋白质功能,利用卷积神经网络进行药物组合预测,判断患者当前用药的合理性等。此外,Woensel等研究了如何基于知识图谱推理出电子病历中缺失的字段。中文医学知识推理方面较有代表性的研究有陈德华等将临床数据的时序特征融入到知识推理中,通过构建基于LSTM 的序列增量学习层,以端到端的方式提取三元组时序特征,实现了对糖尿病时序知识图谱的链接预测,为临床决策提供更具价值的参考。Gong等提出了一种安全药物推荐框架,将药物推荐分解为一个考虑临床诊断和药物不良反应的链接预测过程,为患者提供最佳的药物推荐。利用深度学习技术对知识图谱进行推理计算有利于对知识进行挖掘,以提升知识的利用价值,未来需对深度学习在中文医学知识推理方面的应用进行深入探索。
通过对知识图谱构建的关键技术进行梳理,可以发现近年来深度学习方法在医学知识图谱构建中的使用得到了学者的广泛研究,其中知识表示和知识抽取方面的相关研究较多,而知识融合和知识推理方面的研究则较为欠缺。就中文医学知识图谱而言,突出的问题在于公开的中文医学标注语料较少,许多学者在研究中仍使用英文数据集或自行构建数据集,这会因标注数据的差异影响技术的泛化,阻碍技术的深入研究。此外,随着客观世界知识量的不断累积以及知识图谱规模的不断扩大,实体间的关系也逐渐趋于复杂,如何提升深度学习模型的算力以及精确度仍是医学知识图谱走向应用的一大挑战,因此深度学习在医学知识融合与推理方面的研究潜力还有待挖掘。
2 医学知识图谱构建
通用知识图谱知识覆盖范围广且数据量大,通常采用自底向上的方式构建,自动化程度较高。医学知识图谱属于领域知识图谱,构建的关键技术与通用知识图谱存在共性,但构建流程则有所区别。领域知识图谱构建的流程如图2所示,其中模式层对后续领域知识的获取和组织有着重要的指导意义。
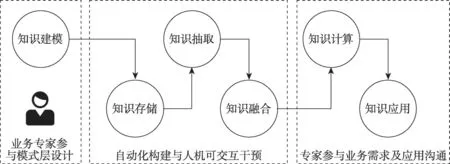
图2 领域知识图谱构建流程Fig.2 Construction process of domain knowledge graph
由于医学领域具有相对完备的知识体系,而且医学知识具有术语多样、结构复杂、专业性强且应用场景容错率低等特点,由医学专家参与构建的医学本体对医学知识图谱模式层的构建极具参考价值,也对医学知识图谱的快速发展起到了很大的促进作用。本文将从医学本体构建、全科医学知识图谱构建和单病种医学知识图谱构建三个角度对中文医学知识图谱构建的相关工作进行总结。
2.1 医学本体构建
本体这一概念最早来源于哲学领域,后在知识工程领域作为知识组织的一种形式被广泛使用。本体指利用基本术语表达领域知识,确定领域内共同认可的概念和概念间的关系,以用于领域内不同主体之间的交流与知识共享的形式化规范说明。目前本体采用国际通用的形式化语言OWL 来规范描述领域的概念及其语义关系,使得这些知识可被人机共同理解,从而解决了人机之间、机器之间信息传递和交流的障碍。本体作为一种重要的知识组织方法,为医学领域的知识图谱构建工作提供了坚实基础,生物医学领域也一直处于本体研究的前列。国外成熟的医学本体包括SNOMED-CT、基因本体(gene ontology,GO)、疾病本体(disease ontology,DO)和人类表型本体(human phenotype ontology,HPO)等。近年来,仍有不少学者在进行这些权威本体的改良细化研究,也有学者通过参考或复用它们来构建专科疾病本体,如Shepherd等基于SNOMED-CT 构建了一个本体并将其作为边界对象,以解决照顾慢性病患者的多学科卫生保健小组成员之间的语义互操作鸿沟。国内也有学者对医学本体构建进行了积极的探索。牟冬梅等基于SNOMED-CT 和形式概念分析构建了甲状腺疾病本体,并利用该本体对电子病历进行标注,验证了其有用性。李晓瑛等复用了UMLS 和SNOMED-CT 中的语义关系,并结合从文献中获取的疾病与药物之间的治疗关系,构建了呼吸系统肿瘤本体。任慧玲等构建了中医疾病本体,并完成了与ICD-11 中文版的语义映射,为中医疾病分类统计的规范化和标准化奠定了基础。
由于国内还尚未形成权威的医学术语标准,目前学者大多参考UMLS、SNOMED-CT 以及MeSH(medical subject headings)词表等国际权威术语构建中文医学本体。近年来,国内也有机构致力于中文医学术语标准化的研究并取得了一定成效,如开放医疗与健康联盟(open medical and healthcare alliance,OMAHA)于2019 年在HiTA 知识服务平台上发布了“七巧板”医学术语集;中国医学科学院医学信息研究所于2020 年在BioPortal 平台公开了他们的研究成果——精准医学本体(precision medicine ontology,PMO),这都为中文医学领域知识图谱的构建工作提供了极富价值的参考。
2.2 全科医学知识图谱构建
全科医学知识图谱旨在搜集各类医学知识,通常包含大量的疾病、症状、检查、治疗、用药等多方面的实体及语义关系,通过对这些数据进行分析和整合构建成知识图谱,为医学领域的智能化发展提供帮助。
开放资源是早期全科医学知识图谱构建的主要数据来源,Lin等以公共医疗网站上爬取的数据和医院的电子临床数据为数据源,搭建了MED-Ledge系统,该系统可对医学数据进行有效的处理和分析,并绘制成知识图谱以支持各种真实的医疗保健应用。刘燕等和魏自强等以垂直性医疗网站中的医学知识为数据基础构建了医疗知识图谱,并对其应用进行了探讨。此外,Shi等还利用某城市卫生信息系统中的医疗服务数据构建了一个语义健康知识图谱,以便从这些离散的医疗文本数据中挖掘有价值的信息,使医疗信息系统中积累的数据得以充分利用。近年来,不断积累的电子病历数据也引起了学界的广泛关注,其中有些学者就利用电子病历构建了知识图谱,为临床决策提供支持。聂莉莉等还以权威的医学文献和书籍为数据源,在医学专家的帮助下梳理了呼吸系统常见疾病及其症状之间的关系,以“疾病-症候-特征”为模型构建了呼吸系统医疗诊断知识图谱。阮彤等利用上海曙光医院已有的中医临床知识库作为数据基础,利用文本信息抽取和关系数据转换(database to RDF,D2R)等信息技术,通过领域专家构建的模式层将疾病库、症状库、中草药库和方剂库进行了融合,构建了一个中医药知识图谱。
全科医学知识图谱的构建是医学数据向知识化转变的重要尝试,不同渠道的医学信息的积累也使得全科医学知识图谱的规模不断扩大。目前,中文全科医学知识图谱相关研究已有一定的积累,但如何提升数据的质量以满足医学具体应用场景的需求仍是亟待解决的关键问题。
2.3 单病种医学知识图谱构建
由于医学应用场景对知识精确度要求较高,全科医学知识图谱在数据精度方面的缺陷导致了其应用的局限性。近年来许多学者展开了对单病种医学知识图谱构建的研究。单病种医学知识图谱往往以某一疾病为核心节点,通过梳理该疾病的临床指南构建某疾病的知识模型,再结合一系列技术手段完成知识图谱的构建。
目前单病种知识图谱涉及的疾病种类已经非常丰富,如Weng等提出一种基于语义分析的医学知识图谱自动构建框架,并基于此框架利用886 例高血压患者病历构建了高血压知识图谱。糖尿病知识图谱的构建也有学者进行了研究。精神疾病方面,Huang等依据UMLS 的概念层级和医学术语对从科研文献、临床指南、维基百科和电子病历中获取的抑郁症相关数据进行了整合,构建了抑郁症知识图谱,并开发了相应的系统对图谱进行管理和更新。马欢欢则基于癫痫患者的电子病历构建了癫痫知识图谱。此外,还有Chai利用某三甲医院的甲状腺疾病患者的电子病历,结合医院已有的知识库进行甲状腺疾病相关实体和关系的抽取,构建了甲状腺疾病知识图谱,并采用样例数据测试了其辅助诊断的可用性。Fang等从电子病历和医学网站(寻医问药网、百度百科和春雨医生)中抽取了垂体腺瘤相关信息,在临床专家的帮助下构建了垂体腺瘤知识图谱,为临床决策提供支持。另外,慢性肾脏病、心血管疾病以及近年突发的新冠肺炎,均有学者在其知识图谱构建及应用方面进行了研究。然而中文的单病种医学知识图谱大多针对较常见的疾病,国外已经有学者对罕见病知识图谱的构建与应用展开了研究,这也是中文医学知识图谱未来值得研究的方向。
近年来,中文医学知识图谱构建的研究成果不断增加并呈现以下特点:一是图谱构建的数据来源趋于多样化,包括科研文献、临床指南、医疗百科、电子病历等;二是图谱类型从全科医学知识图谱发展到单病种医学知识图谱,且涉及的疾病种类日益丰富,在应用层面也取得了较好的成果。然而中文医学知识图谱的研究仍存在一些难点和挑战。首先,医学本体对医学知识图谱的构建具有重要的指导意义,然而目前国内尚未形成权威的中文医学术语,不同研究采用的知识结构并不统一,这阻碍了现有医学知识图谱的融合,不利于研究的深入。其次,现有的中文单病种医学知识图谱大多针对常见病和多发病,如何利用知识图谱辅助罕见病的诊断和治疗也是未来亟待解决的问题。
3 医学知识图谱应用
通用知识图谱的应用方向在医学领域大都适用,但医学知识图谱也因医学领域的不同业务而延伸出了更广泛的应用场景,本文将对医学知识图谱在语义搜索、决策支持、智能问答及其他方面的应用进行分析。
3.1 语义搜索
Google 提出知识图谱时就是用于优化搜索引擎的检索质量,通过语义关系分析为用户匹配更精确的检索结果,并将结果结构化地展示给用户。
在医学领域也有许多专用的搜索引擎,美国的在线健康网站Healthline 就是一个基于知识库的医学信息搜索引擎,用户可以利用疾病名称、症状名称、药物名称和治疗手段等字段进行检索,还可以查询当地的医院和医生信息等,涵盖的医学信息非常全面。国内主流的医学搜索引擎有搜狗明医、寻医问药网、春雨医生、医脉通等,还有一些客户端产品,如腾讯医典、科大讯飞与学习强国联合推出的讯飞健康平台等,这些平台都在使用知识图谱相关技术来优化其语义搜索功能。
中文医学知识图谱在语义搜索方面的应用也有一些代表性的研究:其一为于彤等开发的一个大型语义搜索平台TCMSearch,该平台融入了语义视图和基于领域本体的语义索引,可以为领域专家提供更智能的信息检索服务;另外一项则是中国中医科学院的贾李蓉等开发的中医药学语言系统,该系统中也使用了包含12 万余个概念、60 万余个术语以及127 万余个语义关系的中医药知识图谱,通过在检索系统中嵌入“知识卡片”以及一个“知识地图”展示系统,将中医领域的概念进行可视化展示。近年来,有学者在搜索的基础上进行了扩展研究。Wang等开发了一个基于知识的医学信息检索系统,不仅从UMLS 中提取信息作为背景知识库以优化搜索结果,还对该系统在医学临床决策和个性推荐等方面的应用进行了研究。刘崇从寻医问药网和39 健康网等网站采集数据构建了医学知识图谱,并开发了医疗知识搜索系统。该系统可借助知识图谱理解用户的意图,以更直观、精确的方式返回用户所需的医疗知识,还能向用户推荐相关的社区问答链接供用户查阅。
3.2 决策支持
知识图谱可以实现对各类医学知识的关联与整合,通过一定规则的逻辑推理从已有的知识中得出一些新的结论,为用户制定决策提供支持。目前医学知识图谱在临床诊疗决策支持、药物研发决策支持和应急响应决策支持方面均有应用。
国外医学知识图谱早期在临床诊疗决策支持方面的应用较多,近年来的研究集中在了药物研发方面,如利用知识图谱实现药物重定位或揭示药物之间的相互作用,为药物研发提供决策支持。此外,Gentile等利用药物说明书构建了知识图谱,通过对药物说明书进行解析并与知识图谱进行匹配,能快速识别并标注出新版说明书中变更的字段,为药物审查人员提供决策支持,提升审查效率。
中文医学知识图谱的应用主要还是集中在临床诊疗决策支持上,如王昊奋等将其构建的医学知识图谱应用于上海林康医疗信息技术有限公司的医疗质量与患者安全辅助监控系统中,检测抗生素的不合理使用情况。Zhao开发了一个临床决策支持系统,该系统可以持续监测患者的生命体征参数,并在几个级别上计算风险分级,结合知识图谱识别患者存在的风险,以便医护人员及时做出干预。除了此类面向医护人员的决策支持研究,也有学者在其研究中考虑了患者的需求。武家伟等利用互联网开放数据构建了“疾病-症状”知识图谱,并融合深度学习技术设计实现了问诊推荐系统,在患者查询疾病相关问题时可以为其推荐合适的医生和医院,以便患者做进一步的诊断和治疗。
此外,中文医学知识图谱在医疗突发事件应急响应方面的应用也有研究。根据医疗突发事件知识图谱可以推理实际救援的资源调配方案,辅助应急决策者做出更高效的决策措施。
3.3 智能问答
智能问答系统可以通过自然语言处理技术理解用户的提问,从海量数据中查询用户所需的答案并反馈给用户。基于医学知识图谱开发智能问答系统可以帮助患者实现自查自诊,缓解医护人员人手不足的压力。
Watson 机器人是最早在医学领域应用的智能问答平台,而后诸多学者开始对医学智能问答系统展开研究。郑懿鸣等构建了中医药知识图谱,并基于自然语言处理技术开发了智能问答系统,然而该系统仅针对疾病和症状提供用药推荐,问答类型过于单一。王继伟等在其开发的智能问答系统中使用了基于共享层的卷积神经网络与词频-逆文本频率(term frequency-inverse document frequency,TF-IDF)结合的混合算法,以保证系统能准确地获取用户输入的问句类型并匹配最接近的模板,从而实现更丰富的问答交互。此外,针对单病种的智能问答系统也有学者研究,如田迎等从抑郁症论文摘要中抽取其知识三元组,构建了抑郁症知识图谱,并采用模板匹配的方法开发了抑郁症智能问答系统。在新冠肺炎持续肆虐的当下,也有学者开发了新冠肺炎智能问答系统,既有助于民众获取最新疫情信息以避免恐慌,也能帮助疫情防控相关部门的咨询人员缓解压力。
除以上应用场景之外,Gopez等还基于医保政策构建了知识图谱,用于辅助医保审查,减少医保欺诈事件的发生。黄智生等还将知识图谱用于微博平台进行自杀监控预警。也有学者对临床指南的图谱化表示进行了尝试。
4 总结与展望
医学知识图谱的研究将不断推进海量医学数据的智能化处理,推动医学智能化的脚步。本文通过对医学知识图谱的关键技术、构建及应用进行分析,发现中文医学知识图谱的研究存在医学术语标准化程度不高、标注语料缺乏、技术研究不够深入以及应用场景有局限性等问题,现对中文医学知识图谱未来的研究方向做出以下展望。
(1)中文医学术语的标准化问题需进一步研究。标准的医学术语不仅能从模式层上指导医学知识图谱的构建,还能促进现有医学知识图谱的融合,以实现医学知识的互联互通,这对中文医学知识图谱的研究和应用有着重要意义。其次,中文医学标注语料的研究及共享将成为新的发展方向。医学领域语料标注需要耗费大量的人力、物力,而医学知识图谱的研究又依赖高质量的标注语料。在保证数据质量的前提下,未来学界和业界应该更注重中文医学标注语料的研究和共享,以减少研究成本,提升研究效率。
(2)人工智能技术在医学知识图谱构建中的应用需更加深入,特别是加强深度学习在中文医学知识融合和知识推理方面的研究,通过提升模型的性能及泛化能力,形成中文医学知识图谱构建的技术体系或通用平台,以满足更多研究工作的需要。此外,在知识表示和知识抽取方面,不同语种在语言结构和表达上的差异理论上会对深度学习模型的效果造成一定的影响,未来可以从语言学的角度对中文特征进行深入分析,探索针对中文医学知识表示和抽取的新技术。
(3)中文医学知识图谱未来需要探索更广阔的应用前景。随着互联网中医学数据的不断积累,医学知识图谱的可用价值已经远远超出了疾病知识的查询和辅助诊断,药物研发、临床指南的图谱化以及突发公共卫生事件的应对等都将是未来医学知识图谱值得探索的应用场景。

