Streptomyces lavendulae X33遗传转化体系的建立及其内源性启动子活性的比较
孙亚楠,王园秀,丁忠涛,张 斌,吴晓玉,2*
(1.江西农业大学 生物科学与工程学院/江西省农业微生物资源开发与利用工程实验室,江西 南昌 330045;2.江西省果蔬采后处理关键技术及质量安全协同创新中心,江西 南昌 330045)
【研究意义】淡紫灰链霉菌(Streptomyces lavendulae)X33是课题组前期筛选的一株防治柑橘青霉病的生防菌,其所产的活性物质具有广谱抑菌性能,对多种病原菌均有良好的抑制效果[1],且小鼠毒性试验表明其安全、无毒,在农业和医药等领域具有广泛的应用前景[2]。菌株X33 对指状青霉抑菌机制的研究证实:其发酵提取物寡肽通过干扰病原菌的多种代谢途径、影响细胞结构及降低产孢能力等方式有效地抑制柑橘青霉病的发生[3]。为开展提高菌株产活性物质性能的研究,课题组已对菌株X33进行了全基因组测序,拟通过建立菌株X33 稳定的遗传转化体系、探寻高效启动子,为次级代谢产物生物合成基因簇研究及构建高产菌株奠定基础。【前人研究进展】目前链霉菌导入外源基因的方法主要有聚乙二醇(PEG)介导的原生质体转化法[4]、接合转移法[5]和电转化法[6]。大多数链霉菌具有自身的限制修饰系统,外源遗传物质难以进入[7],而接合转移能够避开胞外核酸酶,在一定程度上可以克服宿主对外源DNA 的限制性修饰[6-7]。自Bierman 等[8]首次成功利用接合转移法将质粒由大肠杆菌转入变铅青链霉菌后,接合转移法逐渐成为广泛应用于链霉菌的一种高效基因转移方法[9-10]。如:黄霉素产生菌加纳链霉菌[11]、Xinaomycins产生菌诺尔斯链霉菌xinao-4[12]、丰加霉素产生菌淀粉产色链霉菌1628[13]、卡那霉素B 产生菌卡那霉素链霉菌ATCC12853[14]及固氮链霉菌WZS021[15]等的接合转移体系构建。在淡紫灰链霉菌中,徐萌萌等[16]、Kitani 等[17]成功建立了菌株FRI-5 及菌株gCLA4 的遗传转化体系。【本研究切入点】国内外已建立了多种链霉菌的遗传转化体系[11-15],为淡紫灰链霉菌X33 的遗传转化体系的建立提供了参考。Myronovskyi等[18]、Siegl等[19]、Xu等[20]证实:以β-葡萄糖苷酸酶(GUS)基因作为报告基因,通过测定GUS 蛋白可准确反映启动子的转录活性强弱。故选择以β-葡萄糖苷酸酶(GUS)基因作为报告基因比较红色糖多孢菌来源的permE 启动子及4 个内源性启动子的转录活性。【拟解决的关键问题】利用接合转移法初步建立菌株X33的遗传转化体系,并对接合转移过程中的关键影响因素进行优化,包括热激温度、预萌发时间、供受体比例、接合转移培养基及抗生素覆盖时间等,同时评估不同启动子在菌株X33 的启动活性,为后续菌株X33生物合成基因簇研究及分子改造提供参考。
1 材料与方法
1.1 试验材料
1.1.1 菌株及质粒菌株:Streptomyces lavendulaeX33、克隆菌株E.coliDH5α、接合转移供体菌E.coliET12567(pUZ8002)均来自实验室保藏。质粒:大肠杆菌-链霉菌穿梭质粒pIB139,多克隆位点上游含启动子permE,安普霉素抗性(Apr),由实验室保藏;含报告基因质粒pUC57-gusA,由南京擎科生物科技有限公司合成,氨苄青霉素抗性(Amp)。
1.1.2 培养基及缓冲液高氏一号培养基[21](g/L):可溶性淀粉20.0,NaCl 0.5,KNO31.0,K2HPO40.5,MgSO40.5,FeSO40.01,琼脂20.0,pH 7.4~7.6;2×YT 培养基[21](g/L):胰蛋白胨16.0,酵母粉10.0,NaCl 5.0,pH 7.0;LB 培养基[22](g/L):酵母粉5.0,胰蛋白胨10.0,NaCl 10.0,pH 7.0;MS 培养基[23](g/L):黄豆饼粉20.0,甘露醇20.0,琼脂20.0,含10 mmol/L MgCl2;TSB培养基[24](g/L):胰蛋白胨17.0,大豆蛋白胨3.0,葡萄糖2.5,NaCl 5.0,K2HPO42.5,含10 mmol/L MgCl2,pH 7.5;发酵培养基(g/L):淀粉20.0,牛肉膏3.0,鱼粉蛋白胨10.0,NH4Cl 0.6,NaCl 10.0,CaCO30.02,pH 7.0。以上培养基均121 ℃灭菌20 min。TES缓冲液:称取TES 11.46 g,溶解在1 L ddH2O,调pH为8.0,121℃20 min灭菌后备用。
1.1.3 试剂氯霉素(Chloramphenicol,Chl)、卡那霉素(Kanamycin,Kan)、安普霉素(Apramycin,Apr)、萘啶酮酸(Nalidixicacid,Nad)购于上海阿拉丁生化科技股份有限公司。限制性内切酶XbaⅠ、NdeⅠ、快速裂解液Lysis Buffer for Microorganism to Direct PCR 购于北京宝日医(Takara)生物技术有限公司;DNA 聚合酶Phanta Max Super-Fidelity DNA Polymerase、一步克隆试剂盒ClonExpress II One Step Cloning Kit 购于南京诺唯赞生物科技股份有限公司;金牌Mix(green)Golden Star T6 DNA PCR Mix、DNA 分子量标准物均购于北京擎科生物科技有限公司。三羟甲基甲胺基乙磺酸(TES)、GUS染色液等其他试剂均购于北京索莱宝科技有限公司。
1.2 试验方法
1.2.1 大肠杆菌及链霉菌的基本分子操作大肠杆菌的培养方法、质粒载体的提取克隆与转化以及感受态细胞的制备等操作方法参照《分子克隆实验指南》[22]。链霉菌的培养和接合转移方法等参照《链霉菌遗传操作手册》[21]。
1.2.2 菌株X33 对安普霉素的抗性检测配制浓度为0,1,2,3,4,5,6,7,8,9 mg/mL 安普霉素的高氏一号培养基,分别倒入9 cm平皿制成平板,涂布接种菌株X33孢子悬液(含量为108CFU/mL),30 ℃培养5 d后观察其生长情况[5]。
1.2.3 大肠杆菌和菌株X33 的接合转移供体菌株E.coilET12567(pUZ8002,pIB139)制备[21]:将整合型质粒pIB139 通过电转化法导入E.coilET12567(pUZ8002)中,得到供体菌株ET12567(pUZ8002,pIB139)。挑取单克隆接种至LB 液体培养基(含Apr 50 mg/L、Chl 25 mg/L 及Kan 50 mg/L)中,使用振荡培养箱(ZQLY-180N,上海知楚仪器有限公司)在37 ℃、180 r/min振荡培养12 h以上,按1%的接种量再转接至50 mL 含上述3种相同抗生素的LB 液体培养基中,37 ℃、180 r/min 振荡培养;当菌浓度OD600约为0.4时,4 ℃离心收集菌体,用等体积不含抗生素的预冷LB液体培养基洗涤菌体3次,菌体重悬于1 mL的LB 培养基中,稀释至108CUF/mL 备用。受体菌X33 孢子悬液制备[21]:将高氏一号平板上生长4 d 的菌株X33孢子用TES缓冲液洗脱下来,涡旋振荡后用8层纱布过滤,50 ℃水浴热激10 min,待冷却后加入等体积的2×YT 培养基,30 ℃、180 r/min 预萌发振荡培养3.5 h,使用高速离心机(TGL-20M,上海卢湘仪离心机仪器设备有限公司),5 000 r/min 常温离心收集,重悬后测定孢子浓度,稀释至108CUF/mL 备用。接合转移获得接合子[21]:将经上述备用的供体菌E.coilET12567(pUZ8002、pIB139)和受体菌X33孢子悬液按浓度比1∶10的比例混匀,涂布于高氏一号培养基,30 ℃培养14 h后,再用含Apr(50 mg/L)和Nad(60 mg/L)的无菌水覆盖,继续培养4~5 d至长出接合子。
1.2.4 接合子的PCR 验证阳性接合子均应获得Apr 抗性。根据安普霉素抗性基因序列设计引物Papr-F和Papr-R(表1),由北京擎科生物科技有限公司合成。将接合子在含Apr(50 mg/L)和Nad(60 mg/L)的高氏一号培养基上划线纯化3 次以上,用枪头随机挑取8 个单菌落,采用快速裂解液(Lysis Buffer for Microorganism to Direct PCR)提取接合子的基因组DNA,将裂解后的上清液作为PCR模板,以菌株X33的裂解上清液为阴性对照,以质粒pIB139为阳性对照,利用Mix(green)Golden Star T6 DNA PCR Mix 进行扩增,片段大小应为783 bp。

表1 试验所用引物Tab.1 Primers used in study
1.2.5 接合转移体系的优化对接合转移过程中的部分关键条件[11-15,23-24]进行优化。接合转移培养基设置MS、高氏一号、发酵培养基及TSB 培养基;菌株X33 的孢子热激温度设置为40,45,50,55,60,65 ℃;孢子预萌发时间设置为0,0.5,1,1.5,2,2.5,3,3.5,4,4.5,5 h;供受体比例设置为100∶1、10∶1、1∶1、1∶10、1∶100;抗生素覆盖时间设置为10,12,14,16,18 h。优化实验中,供体菌E.coliET12567(pUZ8002、pIB139)的浓度保持为108CFU/mL,受体菌菌株X33 孢子浓度保持为108CFU/mL。接合子的计数以肉眼可分辨的接合子菌落为准,接合转移效率=接合子单菌落数量/受体菌孢子数量。
1.2.6 启动子活性检测载体的构建将pIB139质粒进行NdeI和XbaI双酶切,1%琼脂糖凝胶电泳跑胶回收线性片段,备用[22]。根据NCBI数据库公布的GUS(β-葡萄糖苷酸酶)基因及氨基酸序列进行密码子优化,委托北京擎科生物科技有限公司合成目的基因,交付克隆质粒pUC57-gusA。根据合成的gusA基因序列设计引物,并在引物5’端引入pIB139载体双酶切末端的同源序列及相应的酶切位点,如表1所示引物为PgusA-F 和PgusA-R。以pUC57-gusA为模板,利用DNA 聚合酶(Phanta Max Super-Fidelity DNA Polymerase)扩增目的片段,产物大小约为1 850 bp。PCR产物经1%琼脂糖凝胶电泳检测并回收,与备用的线性化pIB139通过一步克隆试剂盒(ClonExpress II One Step Cloning Kit)进行连接,将重组后的质粒以热激法转入E.coliDH5α感受态,分子验证后将质粒送至北京擎科生物科技公司测序。将测序结果正确的重组质粒pIB139-gusA及其克隆菌株保藏。pIB139-gusA重组质粒图谱如图1所示。
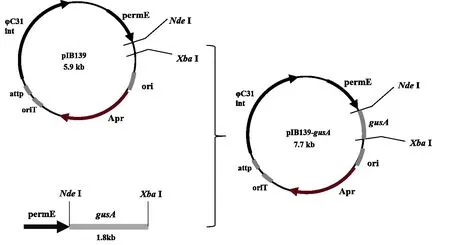
图1 pIB139-gusA重组质粒图谱Fig.1 Map of pIB139-gusA recombinant plasmid
以含报告基因的pIB139-gusA为出发质粒,NdeI单酶切处理后回收,根据质粒序列设计引物PpIB139-gusA-TF 和P-pIB139-gusA-TR(表1),以线性化的pIB139-gusA为模板扩增出不含启动子permE的DNA片段回收备用。
根据已测序的菌株X33 全基因组,使用启动子预测工具Softberry,随机筛选出内源性启动子4 个,P116、P4565、P5092、P5500分别为β-半乳糖苷酶、苹果酸脱氢酶、高丝氨酸脱氢酶、GTP 焦磷酸激酶的启动子。片段大小截取400 bp,根据启动子片段序列及经Nde I单酶切处理的线性化质粒两端序列设计扩增启动子的引物(表1),扩增回收备用。
将4个启动子片段分别与线性化质粒片段通过一步克隆试剂盒(ClonExpress II One Step Cloning Kit)进行连接,转化及验证方法与1.2.6 同。获得4 个含不同启动子的活性检测载体pIB139-116-gusA、pIB139-4565-gusA、pIB139-5092-gusA和pIB139-5500-gusA,质粒构建过程如图2所示。
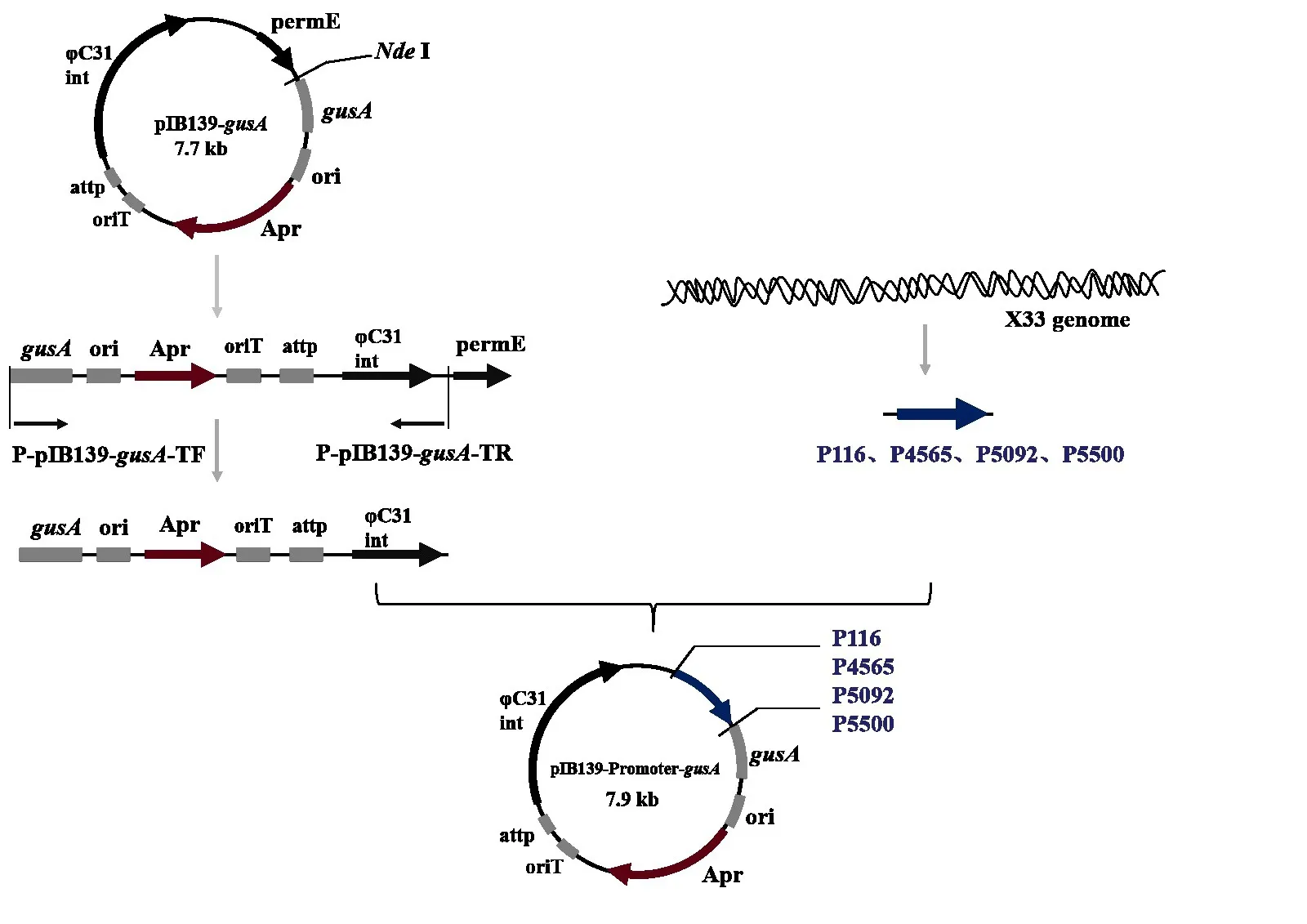
图2 启动子活性检测质粒的构建Fig.2 Construction of plasmid for promoter activity detection
1.2.7 启动子表征菌株的构建将经验证的表征启动子质粒按照方法1.2.3制备供体菌后与菌株X33接合转移,获得启动子表征菌株(X33-116G、X33-4565G、X33-5092G、X33-5500G、X33-permEG),3次纯化单菌落后进行PCR验证,保藏备用。
1.2.8 GUS 的活性检测分析gusA基因的表达产物为β-葡萄糖苷酸酶(GUS),以5-溴-4-氯-3-吲哚-β-葡萄糖苷酸(X-Gluc)作为反应底物,GUS 可将X-Gluc 水解呈现蓝色[25],此方法可用于GUS 的初步定性分析。以4-硝基苯基-β-D-葡糖醛酸(CAS:10344-94-2)为底物进行酶解,底物水解后产生的对硝基苯酚含量可使用可见光分光光度计在415 nm处进行比色测定,此方法可用于GUS的定量检测[26]。
将含不同启动子的重组菌株与野生型菌株X33 及含空载质粒的X33-pIB39 划线于高氏一号固体培养基上,30 ℃培养1~2 d 后,向其中均匀加入过滤除菌的GUS 染色液[27],避光培养观察颜色变化;将以上不同菌株分别接种至50 mL的发酵培养基中30 ℃、180 r/min下培养,至48 h时取样500 μL于1.5 mL离心管中,并加入500 μL GUS染色液避光培养培养24 h,观察其颜色变化;同时在培养至48 h时进行取样,根据Myronovskyi 等[18]、Siegl 等[19]、Xu 等[20]描述的方法进行定量GUS 活性检测,所得吸收曲线的斜率用于计算每克菌体干重的酶活性。以上试验均设置3个平行进行测定。
2 结果与分析
2.1 菌株X33遗传转化体系的建立和优化
2.1.1 菌株X33对安普霉素的抗性检测试验所用接合转移质粒pIB139上携带安普霉素抗性基因。参考1.2.2 的方法,结果见表2。菌株X33 对安普霉素的最小抑制浓度为3 mg/mL,对安普霉素较为敏感。为便于后续筛选和纯化接合子,参考文献报道[5,11-15],在以下接合转移实验中选用Apr浓度50 mg/L作为后续抗性筛选标记。

表2 菌株X33对不同浓度安普霉素的敏感性Tab.2 Sensitivity of strain X33 to different concentrations of apramycin
2.1.2 接合子的PCR 鉴定及稳定性检测采用1.2.3 方法,将整合型质粒pIB139 通过大肠杆菌E.coliET12567(pUZ8002)接合转移到菌株X33,获得接合子。随机挑取8 个单菌落的X33 接合子(X33-pIB139)扩大培养后,提取其基因组DNA,以安普霉素抗性基因序列为引物分别进行PCR 扩增,PCR 检测结果如图3 所示,接合子及阳性对照pIB139 均扩增出略大于750 bp 的片段,而野生菌株X33 未扩增出目的片段,表明pIB139 质粒已成功导入到菌株X33中。
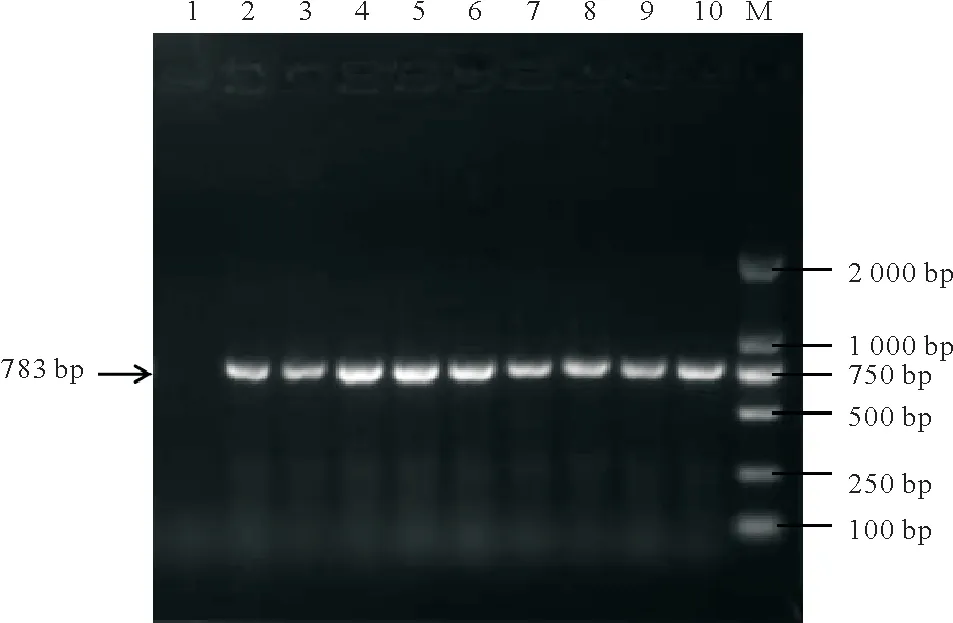
图3 菌株X33接合子的PCR鉴定Fig.3 The PCR identification of strain X33 exconjugants
将经PCR 鉴定的8 个接合子在含或不含Apr 的高氏一号培养基上,采用抗性交替传代10 次后,接合子X33-pIB139 的生长速度、菌落形态与野生型菌株均无明显差异。将第10 代的接合子单菌落随机挑选10 个,提取基因组DNA 进行PCR 扩增鉴定,均可获得略大于750 bp 的目的片段,表明质粒pIB139 在菌株X33 中可以稳定遗传。
2.1.3 接合转移培养基对接合转移效率的影响根据前期实验中菌株X33在不同培养基上的生长速度以及产孢情况,同时参考其他链霉菌的最适接合转移培养基[11-15],选择MS、高氏一号、发酵培养基及TSB的固体培养基进行接合转移效率比较。结果如图4所示,高氏一号、发酵培养基、MS上均有接合子生长,且在高氏一号上接合效率最高可达2.68×10-5,而在TSB 培养基上几乎没有接合子生长,故高氏一号为菌株X33的最佳接合转移培养基。
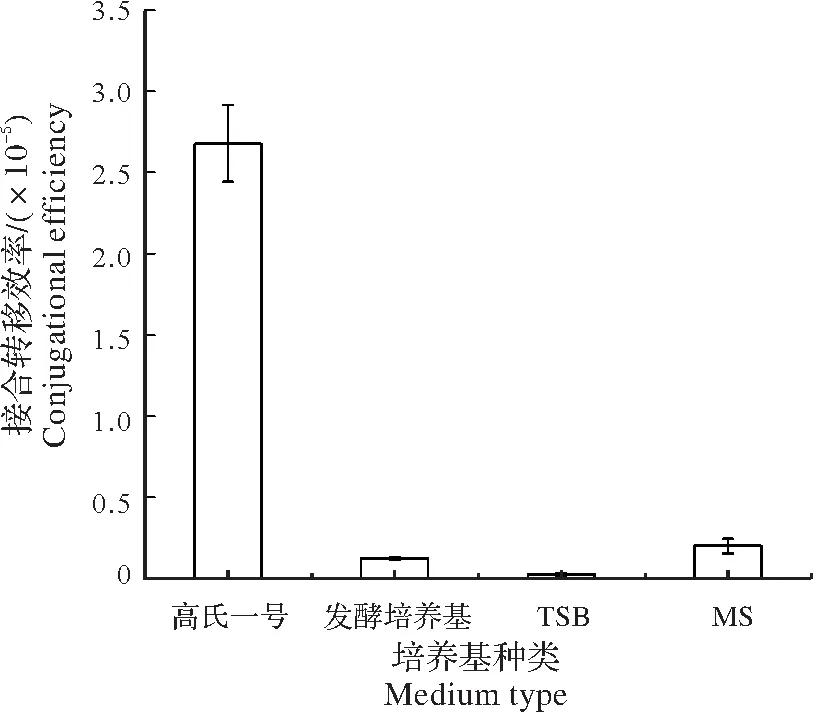
图4 不同种类接合培养基对菌株X33接合转移效率的影响Fig.4 Effect of different kinds of mediums on conjugation transfer efficiency of Strain X33
2.1.4 热激温度对接合转移效率的影响将孢子分别在40,45,50,55,60,65 ℃6 个热激温度下水浴热激10 min,促进孢子的萌发。待冷却后加入等体积2×YT培养基,30 ℃、180 r/min预萌发培养3.5 h。接合转移效率如图5 所示,热激温度在40~65 ℃范围内,接合转移效率随温度升高而逐渐增大,至55 ℃时达最大值,为2.78×10-5,随后不断下降。因此确定孢子的最佳热激温度为55 ℃。
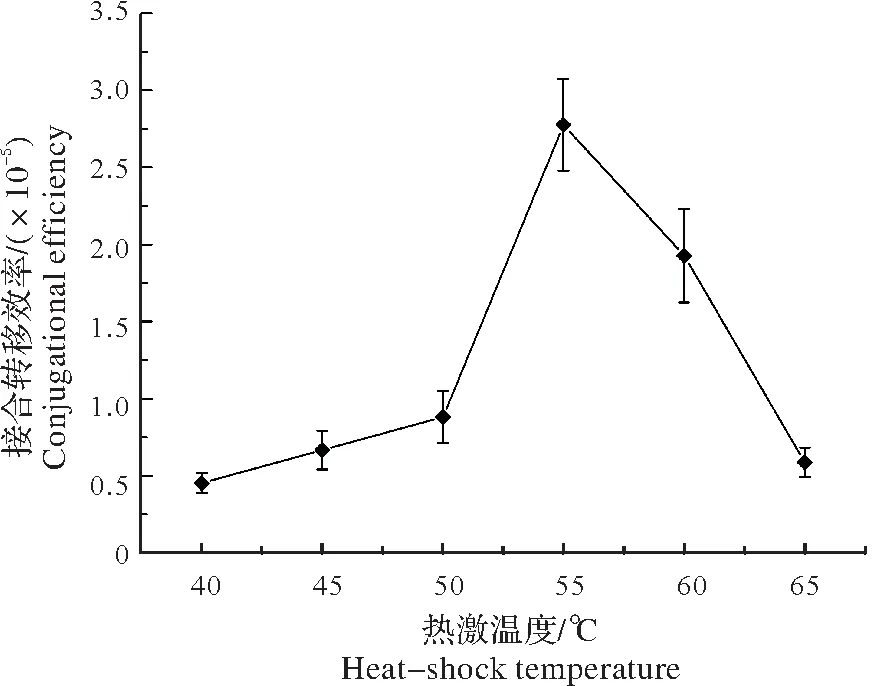
图5 不同热激温度对菌株X33接合转移效率的影响Fig.5 Effect of different heat shock temperature on conjugation transfer efficiency of strain X33
2.1.5 孢子预萌发时间对接合转移效率的影响热激后对孢子进行预萌发,预萌发培养时间过长会使孢子芽管萌发过长,菌体聚集结团[11],接合转移时难以充分与供体菌接触,造成接合效率下降。实验中设置预萌发时间为0,0.5,1,1.5,2,2.5,3,3.5,4,4.5,5 h 共11 个处理,热激温度55 ℃。结果如图6 所示,孢子经预萌发处理1 h 的接合效率最高,为2.62×10-5。随着预萌发时间的增加,接合效率呈下降趋势。因此确定菌株X33孢子最佳预萌发培养时间为1 h。
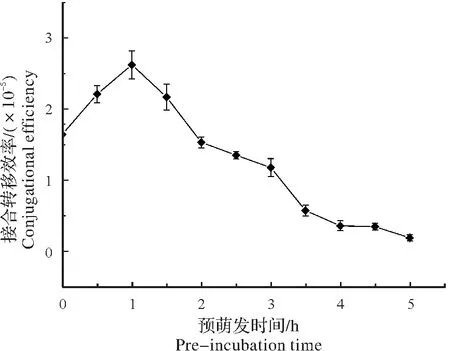
图6 不同预萌发时间对菌株X33接合转移效率的影响Fig.6 Effect of different pre-germination time on conjugation transfer efficiency of strain X33
2.1.6 供受体比例对接合转移效率的影响供受体比例在链霉菌接合转移过程中是一个关键因素,且不同链霉菌的最佳供受体比例存在差异[5,7,11]。分别将供体菌ET12567(pUZ8002,pIB139)与受体菌菌株X33的浓度比值以100∶1、10∶1、1∶1、1∶10、1∶100进行混匀后涂布培养。结果如图7所示,供受体比例为10∶1 时接合转移效率最高,随着供受体菌浓度比值的降低,接合转移效率不断下降。
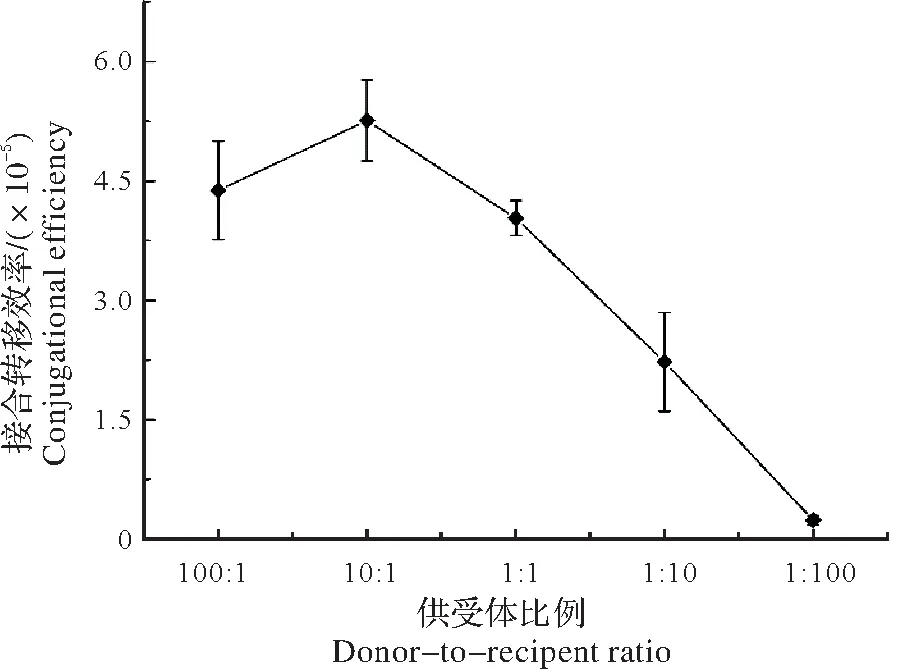
图7 不同供受体比例对菌株X33接合转移效率的影响Fig.7 Effect of different donor-to-recipent ratio on conjugation transfer efficiency of strain X33
2.1.7 抗生素覆盖时间对接合转移效率的影响不同覆盖抗生素的时间会影响接合转移效率以及对阳性接合子的筛选,抗生素覆盖过早会导致接合转移的效率降低,而覆盖过晚则会使得假阳性增多影响纯化。抗生素覆盖时间分别设置10,12,14,16,18 h,计算接合转移效率,结果如图8 所示。混匀涂布培养后,在10~12 h随着覆盖Apr 时间后延,接合转移效率增大,在12 h 时可达3.26×10-5;而12 h 后,随着覆盖时间的推迟,接合转移效率下降,因此12 h 左右覆盖抗生素为最佳。
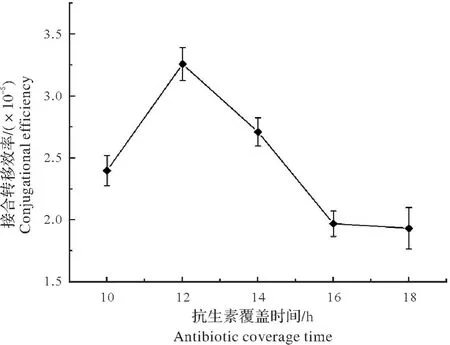
图8 不同抗生素覆盖时间对菌株X33接合转移效率的影响Fig.8 Effect of different antibiotic coverage time on conjugation transfer efficiency of strain X33
2.2 不同来源启动子在菌株X33中的启动活性比较
2.2.1 pIB139-gusA 载体与启动子活性检测载体的构建质粒pIB139 在多克隆位点前有链霉菌常用的组成型强启动子permE,采用1.2.6 方法将报告基因gusA插入到启动子permE 后,构建可表征启动子permE的质粒pIB139-gusA。重组质粒pIB139-gusA的双酶切验证胶图如图9所示。
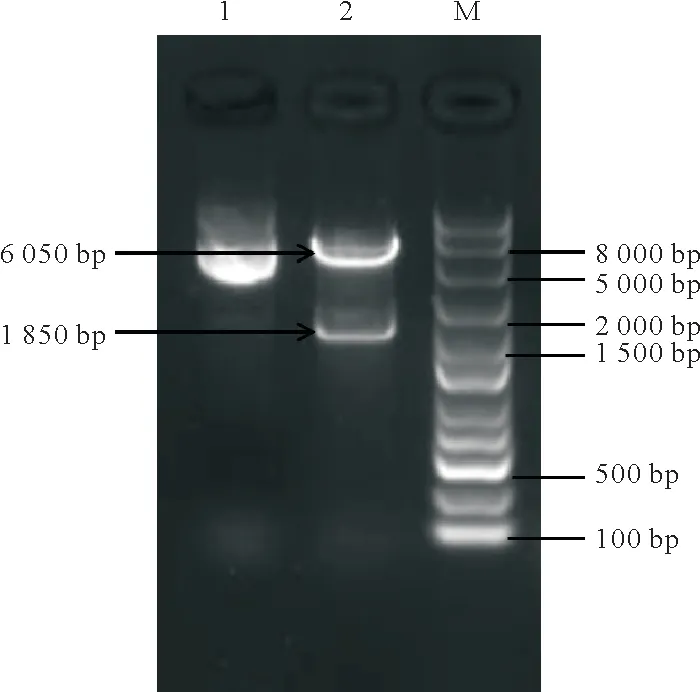
图9 重组质粒pIB139-gusA的双酶切验证Fig.9 Analysis of double enzymes digestion of recombinant plasmid pIB139-gusA
以线性化的pIB139-gusA为模板扩增出不含启动子permE 的片段回收备用,与扩增出的4 个内源性启动子片段分别连接,转化至克隆菌株DH5α,菌落PCR验证胶图如图10所示。
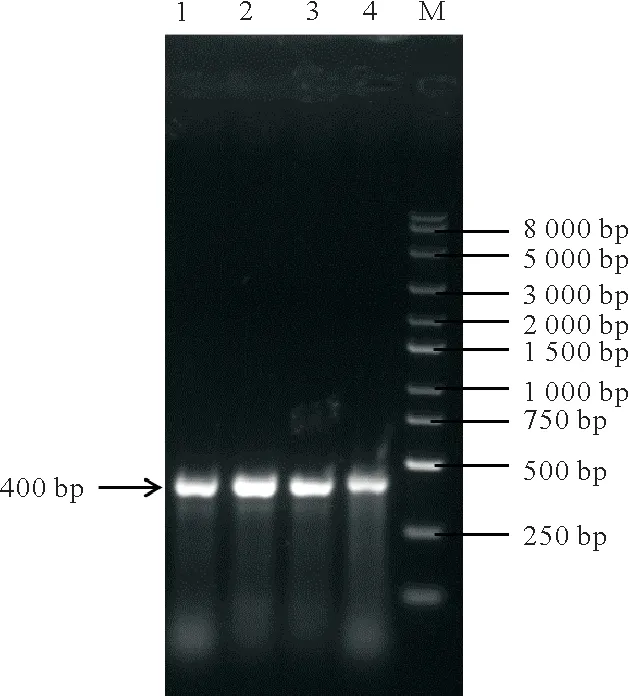
图10 启动子片段PCR特异性验证结果Fig.10 The PCR product of promoter fragment
2.2.2 以GUS 为报告基因的启动子活性比较采用1.2.7 方法获得5 个表征启动子的重组菌株X33-permEG、X33-116G、X33-4565G、X33-5092G、X33-5500G,接种至固体和液体培养基培养,以野生型菌株X33 和含空载的X33-pIB139 作为阴性对照,根据1.2.7 的方法,GUS 染色后,观察颜色变化,结果如图11的A和B所示。启动子表达菌株中,菌株X33-permEG产生的蓝色较其他菌株更深,而菌株X33-116G未观察到明显的颜色变化。
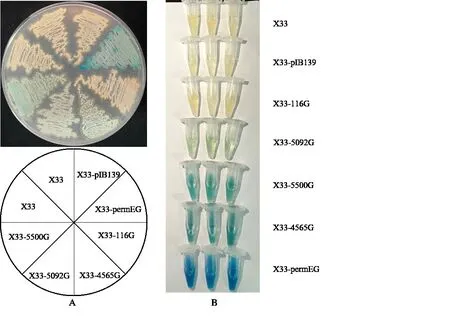
图11 野生型菌株X33和不同重组菌株GUS活性的可视化比较Fig.11 Visual observation of GUS activity of wild type strain X33 and different recombinant strains
液体培养至48 h时取样进行GUS定量的活性检测,根据所得吸收曲线的斜率来计算每克菌体干重的酶活性。结果如图12 所示,与GUS 染色法的结果相一致。在淡紫灰链霉菌X33中,启动子permE的转录活性相对较强,而筛选出的4个内源性启动子的转录活性均小于permE。
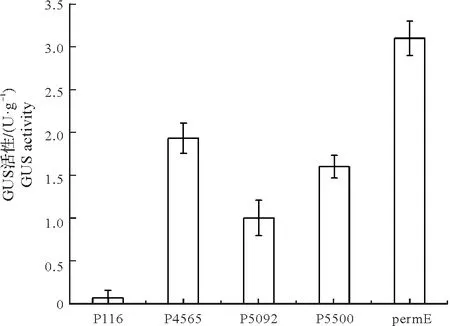
图12 不同启动子下GUS活性测定Fig.12 Detection of GUS activity under different promoters
3 讨论
虽然多种链霉菌的遗传转化体系已经建立,但由于链霉菌菌种遗传代谢背景不同,即使同一种链霉菌的不同株系间其转化效率也存在较大差别[5]。与其他导入外源基因的方法相比,接合转移法具有明显优势,其原因在于(1)链霉菌具有较强的限制性修饰系统,外源DNA 在其胞内难以稳定存在,而预先导入外源DNA 到甲基化修饰缺陷的大肠杆菌(Dam-),再与链霉菌进行接合转移时,可有效降低链霉菌对外源DNA 的限制作用,提高链霉菌的转化效率[29];(2)插入oriT 位点的穿梭载体,可借助供体菌大肠杆菌内pUZ8002 质粒上的tra基因区进行接合转移,实现跨属种可控性的基因转移[29]。另外,在链霉菌中,由于种间变异,链霉菌的接合转移条件具有高度菌种特异性,需要为不同种类菌株找到适当的接合转移条件[12,14,26]。
Du等[30]采用链霉菌S.lincolnensisATCC25466孢子作为受体与大肠杆菌接合时,未获得接合子,而选择菌丝作为受体后,成功建立了高效的属间转化体系。Rocha等[28]对不产孢的链霉菌S.peucetiusvar.caesiusNRRL B-5337 同样使用气生菌丝作为受体,获得了高效的接合效率,为其他不产孢链霉菌的遗传操作提供了参考。Song 等[24]发现龟裂链霉菌M527 可使用菌丝作为接合转移的受体菌,但效率远低于孢子。前期课题组同样尝试使用菌株X33 的菌丝作为受体菌进行接合转移,但未成功获得接合子。表明不同种的链霉菌在接合转移过程中适用的受体形态不同。
MS培养基是一般链霉菌产孢和接合转移的通用培养基[22]。淡紫灰链霉菌gCLA4[17]和链霉菌769[10]在MS 上进行接合转移的效率最高,在高氏一号培养基上几乎无法获得接合子;而固氮链霉菌WZS021[15]在高氏一号上的接合效率明显高于MS;另一个淡紫灰链霉菌菌株FRI-5[16]的最佳培养基为ISP(含10 mm MgCl2)。表明不同种甚至不同株系链霉菌最适的接合转移培养基不同,且对效率影响较大。接合转移培养基既要适合链霉菌生长,又要使大肠杆菌生长放缓,缩小两种菌的生长速度的差距有利于两者接合转移过程的发生[10]。淡紫灰链霉菌X33在高氏一号上的接合转移效率最佳,因此选择高氏一号为接合转移培养基。
对受体菌进行适当温度的热激处理可以提高孢子的萌发频率,进而提高接合转移效率[11],而温度过高可能会导致部分孢子的损伤或失活。淡紫灰链霉菌FRI-5[16]的最适热激温度为40 ℃,固氮链霉菌WZS021[15]、杨凌链霉菌KM-1-2[5]和淡紫灰链霉菌gCLA4[17]的最适热激温度均为50 ℃,而55 ℃处理后,以上四者的接合转移效率都几乎为0。不同的是,淡紫灰链霉菌X33 在55 ℃热激处理时的接合转移效率最高,在60 ℃和65 ℃处理后仍有一定的接合效率。推测不同种系链霉菌孢子的耐热性不同,菌株X33在55 ℃处理10 min 的孢子存活率无明显下降,且提高了孢子的萌发率。故菌株X33 的最佳热激处理为55 ℃10 min。
孢子在预萌发时长出芽管,利于接合转移的发生,但不同种的链霉菌最适预萌发时间区别较大。加纳链霉菌ATCC 14672 预萌发3 h 的接合效率相对较高[11];链霉菌S-28 不进行预萌发处理的接合转移效率最高,预萌发时间越长,反而使接合转移效率越低[23];而菌株X33的预萌发为1 h时接合效率最高,超过1 h后接合效率持续下降。
Xu 等[20]通过以β-葡萄糖苷酶(GUS)为报告基因筛选到较强转录活性的启动子SPL-21,通过构建toyF基因过表达菌株,进一步证明了SPL-21 较强的转录活性且适用于功能基因的表达。Siegl 等[19]通过以gusA为报告基因表征人工合成的启动子库,发现gusA基因的转录水平与GUS 蛋白活性高度相关,证实gusA可作为表征启动子转录水平的报告系统。试验选择以GUS 蛋白为报告基因,成功获得了相应的4个内源性启动子及permE启动子表征菌株,通过染色法及酶活测定比色法对不同启动子下表达GUS进行活性测定,表明启动子permE 的活性相对较高,后续考虑利用该启动子在菌株X33 中进行其他目的基因的表达。
淡紫灰链霉菌X33遗传转化系统的建立与优化是对其进行分子生物学研究和验证基因功能的必要前提,为提高活性物质的产量、实现基因工程菌株的构建及进一步对菌株资源的开发等奠定基础。GUS报告系统可继续用于筛选其它适用于菌株X33表达的强启动子。
4 结论
通过接合转移的方法成功建立淡紫灰链霉菌X33 的遗传转化体系,并优化接合转移条件,包括:培养基、热激温度、预萌发时间、供受体比例及抗生素覆盖时间等5个因素,确定了最佳接合转移条件,即:菌株X33 的孢子于55 ℃热激10 min,预萌发1 h,供受体比例为10∶1,以高氏一号为接合转移培养基,混合培养12 h 后覆盖含抗生素Apr 和Nad 的无菌水,接合转移效率可达5.8×10-5。通过分析内源性启动子及permE 的启动活性,表明链霉菌常用启动子permE 的启动活性远高于4 个内源性启动子P116、P4565、P5092、P5500。

