生物共现网络原理及其在淡水生态系统评估中的应用*
万凌琳,陈芷凡,郭 佳,佟立辉, 任丽娟,韩博平,吴庆龙
(1:暨南大学生命科学技术学院生态学系,广州 510632) (2:广东粤港供水有限公司, 深圳 518021) (3:中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室,南京 210008) (4:中国科学院大学中丹学院,北京 101408)
淡水生态系统在陆地和水体中占比较低,却承载着地球上很高的物种多样性,具有多样化的生态系统功能,为人类提供重要的生态服务功能[1]. 但随着人类活动的加剧,淡水生态系统面临着气候变化与富营养化等多重环境压力,导致生物多样性减少和生态系统功能退化,及淡水生态系统的服务功能降低[2-3]. 为了维持社会经济发展和生态系统可持续性之间的平衡,减缓淡水生态系统的退化,许多国家和地区已经制定了各种法案和相关指令,如联合国的《生物多样性公约》《欧盟水框架指令》《欧盟地下水指令手册》、美国环境保护署发布的《清洁水法案》等. 这些条例使用一系列指标对所监测的水环境进行评估,如使用化学指标来评估环境污染风险水平[4],或使用生物指标来衡量生态系统的状况[5-6]. 其中,关注生态系统功能变化的生物监测与评估是保护、管理和恢复淡水生生态系统的核心.
目前,淡水生态系统的生物评估大多基于形态鉴定的指示生物或类群[7-8],这类方法往往局限于对特定生物类群的观察,例如鱼类[9-11]、水生大型无脊椎动物[12-14]、浮游植物[15-19]、原生生物[20]等,而针对不同指示生物的观察方法几乎没有一致性,因而无法进行不同生物类群间的比较[21]. 不仅如此,传统评估方法主要利用指示生物的出现率和多度信息,忽略了水体环境中“生物”与“生物”,以及“生物”与“环境”间相互作用的复杂关系[22],而这些相互作用却深刻影响着淡水生态系统的生物多样性、生态系统服务功能[23-24]以及生态系统对环境变化的响应[25]. 因此,迫切需要建立一套灵敏且有效的淡水生态系统监测和评价工具[26-28].
生态网络是对生态系统中物质、能量流动进行模拟的结构模型[29],被达尔文称为“树木交错的河岸(tangled bank)”,描述了物种间的相互作用、群落的基本结构以及生态系统的功能和稳定性[30-31],为人们完整地认识生态系统及其组织提供了一个多维度的视角[32]. 生态网络的结构特性揭示了生态系统的组织规律及其面对干扰时发生进化和补偿机制[33-36],并与生态系统的长期动态(例如恢复力和稳定性)密切相关[37-40]. Thompson等提出将生态网络作为协调生物多样性和生态系统功能研究的概念框架[24]. 因此,生态网络及其分析是一种强大且有潜力的生物监测和评价工具[41]. 为了简化复杂的生态网络,通常只用一种主要相互作用关系来表示,可分为营养网络(trophic networks,也称为食物网)和互惠网络(mutualist networks). 然而,在群落尺度上,物种间可以同时以多种方式发生相互作用,共同组成了一个生物共现网络(biological co-occurrence networks)[32]. 生物共现网络是群落水平物种互作的结构模型,通过物种在群落出现及丰度数据,描述了物种间潜在的相互作用、群落的基本结构,反映了群落在生态系统的功能和结构特性.
在这篇综述中,我们首先回顾了淡水生态系统中生物体间不同类型的相互作用,以及这些相互作用被用来定义不同类型的生态网络. 然后重点介绍表现最全面也最容易实现的生物共现网络:(1)如何构建生物共现网络,(2)如何解析共现网络的拓扑结构,以及(3)如何利用生物共现网络重要的拓扑结构参数作为淡水生态系统质量监测的生物指标.
1 淡水生态系统中的生物相互作用
在生态系统中,空间和时间上共存的不同物种通过物质、能量和信息的交换而相互联系,形成了复杂的相互作用[42-44],从根本上影响着种群、群落和生态系统过程[45]. 生物多样性丧失、入侵物种以及人类活动都会改变生物相互作用,对重要的生态系统功能产生级联影响[45]. 生物相互作用可分为直接相互作用(direct interaction)和间接相互作用(indirect interaction). 生物间的直接相互作用是指一个物种在没有中间物种参与的情形下对另一个物种产生的正面或负面影响,可以概括为两类:正相互作用(positive interaction)和负相互作用(negative interaction)[46]. 对于生物体而言,正相互作用可能是由于有益的生物相互作用(互惠作用、偏利共生、促进作用或其他原因)产生的,而负相互作用可能是由于对抗性的互作(捕食关系、竞争等原因)造成的[47]. 物种间的正相互作用和负相互作用往往同时存在[48],这种复杂的生态关系可以表示为以物种为节点,以物种之间的相互作用关系为连接的生态网络[49-50]. 物种间的直接相互作用可以借助其他物种导致物种间产生明显的间接相互作用,如营养级联(trophic cascade)、似然竞争(apparent competition)等[46].
1.1 种间的正相互作用
种间的正相互作用(positive interspecific interaction)在淡水生态系统中广泛存在,是维持物种丰度和群落结构形成的主要驱动力[45,51-54]. Stachowicz将其定义为“生物体间的相遇,至少对其中一方有利,但对双方都没有伤害”[55]. 淡水生态系统中的正相互作用主要可分为3种类型:互惠作用(mutualism)、偏利共生(commensalism)以及促进作用(facilitation)[55]. 大多数正相互作用都至少提供或交换3种关键资源或者服务,即营养方面、运输方面或是保护作用中的一种[56]. 此外,促进作用还可通过改善栖息地产生积极效应[57].
1.1.1 互惠作用 互惠作用是一种双赢的局面, 两个物种都从相互作用中获利. 这种相互作用关系不受种间遗传距离的限制,动植物之间、微生物和动物之间、微生物和植物之间等都可能发生互惠作用[51]. 营养交换关系是互惠作用中最为常见的[51]. 例如淡水海绵(Spongillalacustris)为其内共生绿藻提供栖所和营养物质,以交换绿藻分泌的葡萄糖[58]. 在互惠作用中,运输能力也是一种常见的交换资源. 例如植物为鱼类提供果实(也是营养交换的一种体现),以交换鱼类进行种子的传播[59-60]. 这种运输方面的互惠作用可发生在多种鱼类和多种植物之间,由此形成了一个互惠网络(mutualist network)[54]. 互惠作用也可提供保护方面的功能. 例如,淡水生态系统中的有根大型植物为底栖生物提供了栖身之所,支持了附生无脊椎动物及其捕食者的多样性[61]. 作为回报,附生的无脊椎动物为大型植物去除附生藻类[62]. 微生物之间也存在互惠作用. 不同类群细菌的相互作用会形成生物膜,生物膜中的细菌可因某些类群的功能而受益. 例如某些细菌种类具有对抗生素的抗性,从而使生物膜中的其他细菌获得耐药性[63].
1.1.2 偏利共生 偏利共生指两个不同生物间发生一种对一方有益,而对另一方没有影响的关系[46]. 在偏利共生关系中,一方从互动中获益,但不会给另一方造成损失. 例如摇蚊幼虫可与各种水生昆虫和鱼类共生[64-65]. 这些与之共生的水生生物能为摇蚊幼虫提供“搭便车”服务,称之为携播(phoresy). 生活在这些“载体(carrier)”上为摇蚊幼虫提供了更好的觅食机会,增加其移动性,还能降低其被捕食的风险[66],但对这些“载体”似乎并没有任何影响.
1.1.3 促进作用 促进作用通常是一种“单向”(unidirectional)作用,指两生物体相遇时,至少有一方受益,但对任何一方都没有伤害. 若两个物种都从相互作用中受益时,则是互惠作用. 当“一个物种直接使当地环境对另一种生物更有利时”,就会发生促进作用[67]. 例如淡水生态系统中的固着生物,如大型植物和贻贝,除了可为附生于其表面的植物和无脊椎动物提供栖息地外,还具有稳定基质、改善底栖生物生境条件的功能[68-69].
1.2 种间的负相互作用
种间的负相互作用(negative interspecific interaction)是指在两个物种的相互作用中,至少对两者的一方会产生不利影响的作用,主要包括捕食作用(predation)、寄生(parasitism)、偏害共生(amensalism)和种间竞争(competition)等[46].
1.2.1 捕食作用 通过典型的捕食作用,淡水生态系统中的捕食者可以限制猎物的丰度,改变猎物的大小、行为、年龄结构或其他特征,并可能通过营养网络的级联效应产生间接影响[70]. 例如,底栖食肉鱼类的捕食作用会显著降低牧食性螺类的生物量,从而减少了螺类对附生植物的牧食,对附生植物的生物量产生了间接的促进作用[71]. 捕食作用还会改变猎物所提供的生态功能. 例如,褐鳟(Salmotrutta)通过捕食碎屑生物(Zelandopsycheingens)而降低了溪流中凋落物的分解率[72].
1.2.2 寄生 与典型的捕食作用一样,寄生生物消耗寄主资源,不同的是,寄生生物通常不会杀死寄主[73]. 一些寄生生物对寄主的伤害相对较小,例如淡水贻贝的幼体仅暂时侵染鱼类[74]. 而大部分的寄生作用对寄主的伤害很大. 例如,成年的海七鳃鳗(Petromyzonmarinus)对湖鳟(Salvelinusnamaycush)的攻击和寄生作用会导致其流失大量的组织和体液,这可能会导致慢性应激反应、继发性伤口感染,甚至死亡[75]. 寄生生物能够改变寄主的行为、颜色或是形态特征,这些改变可能有利于其进一步的传播[76-79].
1.2.3 偏害共生 在偏害共生中,一种生物对另一种产生抑制、伤害作用,甚至杀死对方,但本身却也无法从中获得益处或害处[80]. 例如,水体中的铜绿假单胞菌(Pseudomonasaeruginosa)能产生大量的抗生素来抑制其他细菌和藻类的生长[81].
1.2.4 种间竞争 种间竞争是研究最多的种间负相互作用,是指两物种或更多物种共同利用相同资源时产生的竞争. 可以发生在同一营养级的物种之间,也可以发生在不同营养级的生物体之间.种间竞争方式可分为利用性竞争(exploitative competition, EC)和干涉性竞争(interference competition, IC)[46]. 例如,水体中食物资源丰富时,不同种类的轮虫和枝角类能够共存,反之则会发生争夺食物的资源利用性竞争[82]. IC则包含广泛的生物相互作用,主要包括化学、生存空间和遭遇竞争[83-84].
似然竞争是物种间的间接相互作用,指某一个物种数量的增加将会导致捕食者数量增加,从而加重了对另一物种的捕食强度,反之亦然[46]. 例如,食鱼动物对拟鲤(Rutilusrutilus)和鲈鱼(Percafluviatilis)的取食会导致这两种非竞争性鱼类产生似然竞争[85]. 湖泊中,杂食性鱼类会同时在水体和底栖生境取食无脊椎动物,导致了浮游和底栖无脊椎动物之间的似然竞争[86].
1.3 生物相互作用的动态变化
不论是正相互作用还是负相互作用,都是动态的,随着环境中生物和非生物因素的变化而变化[52,54]. 物种间的相互作用是一个从对抗到互惠的连续统一体[52,87]. 环境变量的改变会使生物相互作用发生转变. 例如,在自然条件下,淡水螯虾与蠕虫是偏利共生关系,只有蠕虫受益[88]. 蠕虫在淡水鳌虾的鳃上生活,以附生寄生虫和生物膜为食[89-90],并降低了其被捕食的风险. 但若水体中污垢(如表面活性物质和有机碎屑颗粒)变多,蠕虫会帮助淡水鳌虾清洗鳃,使淡水鳌虾受益,两者转变为互惠关系[88]. 当然,在污垢变多的情况下,蠕虫的食物资源也更丰富,也将有利于其生长繁殖[91]. 生物因素(如种群密度)的变化也会导致生物相互作用发生转变. 例如,当环境资源有限而蠕虫密度较高时,蠕虫会以淡水鳌虾的鳃为食,使两者转变为寄生关系[90,92].
2 淡水生态系统中的网络类型
淡水生态系统具有很高的生物多样性,存在多个营养级,各个生物体不是孤立存在的,可同时以直接或间接途径与多个其他生物体发生错综复杂的相互作用,构成一个极为复杂的生态网络[29,42]. 为了简化复杂的交互网络,通常只用一种主要相互作用关系来表示,可分为营养网络(trophic networks,也称为食物网)和互惠网络(mutualist networks). 生物共现网络(biological co-occurrence networks)则展现了环境中所有生物体之间复杂相互作用网络的综合视图[32].
2.1 营养网络
营养网络是生态学中研究最早、最多的网络类型,是以物种间的营养关系作为连接的复杂生态网络[93],体现了生态系统中的物质循环和能量流动[24]. 一个营养网络是由多个营养级(trophic level)组成. 营养级呈现了物种在生态系统中的营养生态位,营养级的大小主要取决于初级生产者把能量传递到该级别所需的转换次数. 初级生产者位于生态系统的第一营养级,它们主要利用各种无机形式的能量(如阳光)把CO2转换成生物量;食草动物和食碎屑生物为初级消费者,位于第二营养级;食肉动物以食草动物和食碎屑生物为食物,是第三营养级;捕食食肉动物的动物位居第四营养级. 每一营养级都包含几个,甚至几百个物种[46]. 营养网络的结构非常复杂,主要体现在杂食性和模块性两个方面,其中杂食性增加了营养网络垂直结构(跨营养级的结构)的复杂性,然而模块性不仅增大了营养网络中物种间相互作用的异质性,还增大了营养网络水平(某一营养级内的结构)和垂直结构的复杂性[94]. 鉴于营养网络的高度复杂性,通常使用理论模型的方法来研究其结构和功能[95]. 由于在生态系统水平的研究中,不可能观察或估量系统内所有生物间的营养相互关系,所以基于生态系统水平的营养网络研究本质上都是启发式的(heuristic). 因此,营养网络的构建需要从已发表的文献中收集先验信息(如分类群的相互作用关系、体型和营养习惯等),这无疑是一个费力、艰难而又很难精确的过程[41].
2.2 互惠网络
互惠网络体现了生物体间的互惠作用(mutualism),包括植物-传粉者网络、植物-种子散布者网络以及植物-蚂蚁互惠网络[96]. 互惠网络最显著的特征是模块化(modular)和嵌套结构(nested pattern). 模块化组织意味着大多数互惠连接可以包含在若干子集中. 嵌套结构则是指网络中相对特化物种(specialist)的连接伙伴是相对泛化物种(generalist)的连接伙伴的一个子集[97]. 以人们最为熟知的传粉网络为例,蝶类只能够访问少数植物,而蜜蜂则能够为所有植物传粉,蝶类传粉的植物种类只是蜜蜂传粉植物种类的一个子集[98]. 淡水生态系统中的互惠网络研究较少,主要是集中于鱼类对植物种子的传播作用. 鱼类传播种子是一种普遍的生态相互作用,涉及超过275种鱼类和许多植物物种. 通过食用植物果肉,鱼类获得丰富的碳水化合物、脂类和蛋白质来源,因此这些鱼类通常具有较高的流动性,体型较大并具有较长的寿命,在种子散播过程中发挥重要作用[60,99-100].
2.3 生物共现网络
研究基于单一关系类型的网络(如营养网络和互惠网络)简化了群落的概念,使我们能更好地理解群落的组成. 然而,自然群落的物种之间除了营养关系和互惠关系,还存在竞争、寄生等种间相互作用. 因此,这些网络难以体现生态系统真正的复杂性,基于此的分析结果往往比较片面或产生偏差[32]. 与营养网络和互惠网络不同,生物共现网络是群落水平物种互作的结构模型,通过物种在群落出现及丰度数据,描述了物种间潜在的相互作用、群落的基本结构,反映了群落在生态系统的功能和结构特性[101]. 两个物种可以同时以多种方式发生相互作用,所有这些相互作用的综合结果会使得生物体间发生联合进化(正相关关系,即共同出现co-occurrence),或反向进化(负相关关系,即彼此排斥co-exclusion),或缺乏相互作用(可能是生物体间没有相互作用,或是生物体间的多种相互作用的综合效应为零)[47,102](图1). 在群落尺度上,这些正相关和负相关关系共同组成了一个共现网络. 生物共现网络的多项拓扑结构属性(如连接度、模块性、中心性等)体现了生物间以及生物与非生物(环境因素)间潜在的交互关系,可能是生态系统健康状态的有力指标,为评价淡水生态系统的健康状态和功能提供了一个生物监测框架[103-104]. 生物群落共现网络的研究已经在多种淡水生态系统中展开,包括湖泊[105]、河流[106]、水库[107-109]等.

图1 物种间相互作用关系(根据Faust等绘制的图片修改[47])Fig.1 Summary of ecological interactions between different species(modified from Faust et al[47])
3 构建生物共现网络
由于取样问题、物种分类或物种互作的错误鉴定等限制,很难用传统方法在生态系统水平上构建系统内所有生物的共现网络[104,110]. 环境 DNA(environmental DNA,eDNA)宏条形码(metabarcoding)技术可直接从环境介质(如水体、土壤、沉积物等)中提取DNA,采用通用引物扩增eDNA中特定基因片段(即DNA条形码)并通过高通量测序(high-throughput sequencing,又称“下一代”测序技术 “next-generation” sequencing technology,NGS)获得DNA条形码序列,然后比对参考物种数据库获取环境中生物的有无或丰度信息,由此可实现对自然中多生物群落的同步监测[110-111]. 多项研究证实eDNA宏条形码技术是评估淡水环境生物多样性的可靠手段[112-113],可用于监测底栖动物[114]、鱼类[115]、两栖动物[116]等. Gibson 等的研究表明,基于形态学的传统方法和基于eDNA宏条形码技术的监测方法之间具有良好的一致性[117]. 通过eDNA宏条形码技术能够高效便捷地获取淡水环境中的群落大数据信息,结合环境数据可用于构建生物共现网络[118](图2).

图2 基于eDNA宏条形码技术构建生物共现网络流程图Fig.2 Flow chart of constructing biological co-occurrence network based on eDNA metabarcoding technology
通过测序得到的数据可以用矩阵的形式表现,矩阵的每一行表示一个样品,每一列表示一个物种或类物种[119],数值的高低表示该物种或类物种在样品中含量的高低,矩阵中 0 或者 NA(空缺)表示该物种或类物种没有在该样品中被检测到. 基于这种环境基因组数据构建共现网络的方法很多,而这些方法的效率、精度、计算速度和数据要求各不相同. 简单的如基于成对Pearson或Spearman相关度量的方法,复杂的如基于多元回归和高斯图模型的方法[120]. 这里主要讨论一些构建生物共现网络的常用方法(附表Ⅰ).
3.1 基于相异度的方法
以物种或类物种数据构建共现网络最简单快速的方法是使用配对相异度指数,如Bray-Curtis 指数或Kullback-Leibler指数. 通常用置换检验(permutation test)评估相异度分数的显著性,所有显著的关系将用于构建网络. 比利时佛兰德生物技术研究所(Flemish institute for biotechnology)的Faust等综合多个指标,如相关性系数(如Spearman指数)、相似性(如交互信息)和相异度(如Kullback-Leibler指数),开发了一套集合流程[47],已用于预测海洋浮游生物群落中的相互作用[121].
3.2 基于相关性的方法
这类方法使用相关系数(如Pearson积矩相关系数或Spearman秩相关系数)来检测物种或类物种之间显著的成对相互作用,主要通过物种在不同样本中相对丰度的变化,计算两两物种间的相关系数,然后计算相关显著性P值,结合相关系数和P值筛选数据后绘图,由此形成共现网络[122]. 该方法已成功应用于研究海洋和湖泊等生态系统的微生物共现模式,这些研究不仅探究了微生物之间的联系,还阐明了微生物与环境变化之间的联系[122-123]. 但这类方法存在一定的局限性,如在处理存在大量0的数据集时,低丰度的物种或类物种之间会产生虚假相关关系(spurious correlation),或是对测序得到的组成数据较为敏感[124]. Weiss等[125]以模拟数据和真实数据测试了8种基于相关性方法的性能,并评估了各种方法区分信号和噪声以及检测一系列生态和时间序列关系的能力. 他们总结了各种方法的性能水平和局限性,并确定了稀疏性是这8种方法未能充分解决的关键问题,建议在构建共现网络之前过滤掉极其罕见的物种信息.
3.3 基于回归的方法
基于相异度和相似性的分析方法简单易行,是推断生物共现网络最常用的方法,但局限性在于它们只能推断成对关联度量(如Bray-Curtis指数和Pearson系数)之间的关系,没有考虑这些关系是否会被其他变量所影响. 因此,这类分析方法可能会导致虚假相关性[126]. 可使用多元回归分析进行替代,即将某个分类单元的丰度设置为响应变量,使用所有其他分类单元的丰度作为解释变量. 在多元回归模型中,每个分类单元的系数作为两个分类单元相互作用强度的线性度量[127]. 这类方法简单易行,但回归结果的含义可能难以进行合理的解释. 例如,通过多元回归分析预测得到的连接(links)可能并不代表存在什么生物学关联. 此外,回归模型中由于特征数量较多,容易出现过度拟合(overfitting)的问题,过度拟合随着解释变量的数量增加而增加,并且假阳性数量也会相应增加. 可以通过稀疏回归(sparse regression)和交叉验证(cross-validation)来校正过度拟合[127]. 目前,基于回归的方法已被广泛用于构建生物共现网络. 例如,van den Bergh等使用逻辑斯蒂回归模型研究了幼儿上呼吸道中细菌、病毒和其他危险因素之间的关联性[128]. 回归模型主要优势在于能够结合相关的协变量信息,并有多种统计工具适用于该算法,研究者可根据特定的分析需求灵活选择. 然而,大多数基于回归的方法需要将每个特征(或特征类型)设置为响应变量或解释变量. 若所研究对象的基础生物学知识相对匮乏时,这一点较难实现.
3.4 概率图模型
概率图模型(probabilistic graphical models, PGMs)是一类用图形模式表达基于概率相关关系的模型,利用计算机科学中离散数据结构的思想,利用概率论有效地计算高维数据中的不确定性[120]. 简而言之,PGMs分别使用概率论和图形来处理不确定性和复杂性. PGMs在形式上是由图结构组成的. 图的每个节点(node)都关联了一个随机变量,而图的边(edge)则被用于编码这些随机变量之间的关系. 最常用的两种PGMs包括贝叶斯网络(Bayesian network)和马尔可夫随机场(Markov random field, MRFs). 两者的主要区别在于采用不同类型的图来表达变量之间的关系:贝叶斯网络采用有向无环图(directed acyclic graph)来表达因果关系,MRFs则采用无向图(undirected graph)来表达变量间的相互作用[120].
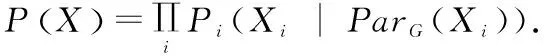
(1)

3.5 对组成数据偏差具有鲁棒性的网络推导方法
淡水生态系统中的生物体(尤其是微生物)不是独立存在的,且生物群落结构的变化与环境密切相关,使得通过NGS测序所得到的物种或类物种数据是组成数据,即生物计数是相互依赖的,并非完全独立. 这类数据反映的是物种的相对丰度信息而非绝对丰度信息,具有稀疏性和内部关联度高等特征. 使用经典的相关性方法(如Pearson相关系数)对这类数据进行网络推导时常常产生不合理甚至错误的结果. 最近,已开发了许多新的算法来提高网络推导的准确性.
3.5.1 生态关联的稀疏逆协方差估计与统计推断(sparse inverse covariance estimation for ecological association and statistical inference, SPIEC-EASI) SPIEC-EASI是一种从16S rRNA扩增子测序数据集推断微生物生态网络的新工具. SPIEC-EASI将为组成数据分析开发的数据转换与图形化的模型推理框架相结合,该框架假设底层的生态关联网络是稀疏的,并依赖于稀疏邻域和逆协方差选择算法构建网络[139]. 目前,SPIEC-EASI除了应用于16S细菌群落测序,同样适用于对ITS、18S,或者蛋白质组学等数据的分析.
3.5.2 组分数据的稀疏相关分析(sparse correlations for compositional data, SparCC) 对于识别群落中生物相互作用,基于物种丰度组分数据的相关性分析是一种常见方法,但是这种分析可能会产生与真实情况相违背的虚假关系,归因于测序获得的物种丰度或基因丰度等很难绝对定量. 群落多样性是调节这种组合效应的剧烈程度的关键因素,由此开发了SparCC方法,它能够将组分数据进行Aitchison对数转换,计算转换后的线性Pearson相关并得到相关系数[140]. SparCC是一种适合测序数据特征的新颖方法,可以推断物种或基因之间的相关性,以及构建物种或基因功能相互作用网络. 例如,Bell等利用SparCC共现网络分析研究了普通小球藻(Chlorellavulgaris)和假单胞菌(Pseudomonassp.)在(用于生产藻类生物燃料的)露天池塘中的共现关系[141]. 但应谨慎使用基于对数转换的方法,对数转换无法处理微生物组数据中常见的0. 通常用一个很小的计数代替0来解决此问题,但伪计数值的选择可能会严重影响结果.
3.5.3 共现网络法(co-occurence network,CoNet) CoNet会同时计算Pearson和Spearman相关性以及Kullback-Leibler和 Bray-Curtis相异性距离. 然后,使用特定的方法将4个分析结果进行合并,得到一个统一的相关系数和P值,校正P值后,得到的结果用于构建共现网络[142]. 另外,CoNet作为Cytoscape软件的插件,其结果可以直接在Cytoscape中可视化呈现,简化了操作过程[109].
3.5.4 基于组成数据的基础协方差的正则化估测方法(regularized estimation of the basis covariance based on compositional data,REBACCA) REBACCA是近年来比较新的一种方法,通过寻找缺陷秩的系统的稀疏解来识别显著的共现模式[143]. 具体而言,REBACCA使用计数或比例数据的对数比转换来估算丰度之间的相关性.
3.5.5 基于套索的组成数据的相关推断方法(correlation inference for compositional data through Lasso,CCLasso) CCLasso是另一种从组成数据中推断相关性的方法[144]. CCLasso对原始组分数据进行对数比变换后,使用具有1罚分的最小二乘法(即Lasso)通过潜变量模型来推断生物之间的相关性.1正则化通常用于防止因协变量的共线性或高维性而导致的过度拟合.
3.5.6 分子生态网络分析通道(molecular ecological network analysis pipeline,MENAP) MENAP 是一种基于随机矩阵理论(random matrix theory, RMT)的方法,旨在解决阈值的选择问题[145]. 该方法自动识别阈值并在此基础上定义邻接矩阵. 最后由邻接矩阵构建无向网络图. 由此得到的网络是自动定义的,对噪声具有很强的鲁棒性,从而可以解决NGS测序数据的几个常见问题.
3.5.7 微生物相互作用法(microbial interaction,MInt) MInt是一种多元泊松正态分层模型,通过控制Poisson层的混杂预测变量,并使用1罚分矩阵,捕获多元正态层中直接的微生物相互作用,因此可从宏基因组计数数据中找到分类单元的相互作用[146].
上文简要介绍了目前常用于生物共现网络构建的方法,这些方法在解析生物之间以及生物与环境之间的相互作用方面具有巨大的潜力,但也各有其适用范围以及局限性(附表Ⅰ,图3). 基于相异度和相关性的分析方法简单易行,是推断生物共现网络最常用的方法,但局限性在于它们只能推断成对关联度量(如Bray-Curtis指数和Pearson系数)之间的关系,没有考虑这些关系是否会被其他变量所影响,容易产生虚假相关性. 基于回归的方法能够结合相关的协变量信息,解决虚假相关的问题,但大多数基于回归的方法需要将每个特征(或特征类型)设置为响应变量或解释变量. 若所研究对象的基础生物学知识相对匮乏时,这一点较难实现. 贝叶斯网络可推断因果关系并整合先验知识进行建模,但计算成本高昂,不适合大样本量数据. MRFs很适合从观测(存在-不存在)数据中获取有关物种相互作用强度的信息,同时展示物种间的间接相互作用,但也存在计算复杂的局限性. 因此,研究者通常需要根据所分析数据的独特特征(如组成性、稀疏性等)量身定制分析工具.

图3 生物共现网络构建流程图(根据Faust等绘制的图片修改[47] )Fig.3 Flow chart of biological co-occurrence network construction(modified from Faust et al[47])
4 生物共现网络结构特征
生物共现网络构建好以后,可以根据图论指标对其结构特征进行描述[148]. 共现网络由节点(nodes)和连接(links)组成. 节点,即网络线条末端的对象,代表物种或类物种、属、科、类、门、界或是功能群. 节点还可以代表其他感兴趣的环境变量[149]. 连接,即节点之间的边,表示通过统计分析所确定的节点间的重要关系,可根据相互作用关系分为正相关连接和负相关连接[150]. 进一步可使用多种网络分析工具,如Cytoscape[151]或Gephi[152]等对生物共现网络进行可视化,并通过不同的衡量标准来评估共现网络的结构体系. 例如以分类单元(即节点)为中心的评估标准,提供了网络中分类单元的类型、多样性以及相互关系的信息. 另一类评估标准则是以整个网络组织为中心,提供了生态系统中群落复杂性的信息[32].
4.1 基于网络的评价指标
4.1.1 平均度数 度(degree)是指某一节点(或分类单元)相邻边(连接)的数量. 平均度数(average degree)是指网络中所有节点的平均连接数量[120].
4.1.2 连接度 连接度(connectance)是指网络中已实现的连接数量与潜在连接数量之间的比率,体现了群落的组织层次,是网络复杂性的一个指标[120].
4.1.3 聚类系数 聚类系数(clustering coefficient)又称传递性(transitivity),是指一个节点的邻点(和这个节点有连接的节点)间的连接数量与邻点间可能存在的最大连接数量的比值,用来反映一个节点的邻点之间联系的紧密程度,提供了网络连接性的度量. 节点的聚类系数总是介于0~1之间,如果说所有的邻点两两之间都相连,则值为1;如果所有的邻点都没有连接,则值为0[153]. 聚类系数值越高,则说明节点的聚集程度越高. 平均聚类系数是网络中所有节点聚类系数的平均值,Watts等发现具有较高平均聚类系数的网络具有模块结构[153].
4.1.4 节点度分布 节点度分布(node degree distribution)是指网络中每个节点的连接数量的分布,在随机网络中,其分布服从泊松分布[154]. 然而,对于大多数生物网络(如无标度网络),常用幂律分布来描述度分布[155]. 节点度分布体现了网络的连通性,常用于推断网络的稳健性(robustness).
4.1.5 模块性 模块(module)是指具有密集连接的一组节点,与其他组的节点连接稀疏[156]. 生态位重叠、生境异质性和系统发育相关性可用于解释模块的形成[157-159]. 模块性(modularity)用来衡量一个网络能否自然地划分成模块[160]. 可参考 Newman等[150,160]的方法对网络的模块性值进行计算,一般数值在 0~1 之间,当模块性值大于 0.4 时,则认为网络具有模块结构. 现在发现几乎所有已分析的生态网络都是模块化的[161].
4.1.6 平均最短路径长度和网络直径 平均最短路径长度(average shortest path length, AL)为网络中所有节点对的平均最短距离. 这里节点间的距离指的是从一个节点到另一个节点要经历的边的最小数目,其中所有节点之间的最大距离称为网络直径(network diameter),平均最短路径长度和网络直径可用于衡量网络的传输性能和效率[120].
4.1.7 网络密度 网络密度(network density)是网络中实际存在的边数与可容纳的边数上限的比值. 因此,网络密度是一个从0~1的比率. 该指标反映了网络中节点之间的联系程度,网络密度越大,表明网络节点之间的关系越密切[162].
4.2 基于节点的评价指标
可利用图论中的衡量指标(如介数betweenness、度degree、亲和度closeness与特征向量中心性eigenvector centrality,或模块性)来评价节点重要性的不同方面,并可由此来确定代表关键分类群的关键节点(keystone nodes)[163-164].
4.2.1 度中心性 度中心性(degree centrality)是网络中每个节点的连接总数,或与其他节点的共享连接总数,这是分类单元水平上复杂性的一个指标[165].
4.2.2 介数中心性 介数中心性(betweenness centrality)指一个网络里通过某节点的最短路径条数,反映了该节点连接子网的能力[166]. 节点的介数中心性与其在群落中的丰度无关,因此,一些丰度较低的分类群可能在群落中具有重要的位置. 具有高介数中心性的分类单元是那些在模块之间共享连接,而在模块内部共享连接较少的节点,代表了模块之间资源共享的潜在途径[167]. 如在某一网络中某节点表现出最大的介数中心性,则可认为此节点是维持网络结构的关键节点. 关键节点可能是群落中最具影响力的成员,对于稳定群落结构,维持生态系统功能具有重要作用.
4.2.3 紧密度中心性 紧密度中心性(closeness centrality)则表示关键节点与网络中所有其他节点的相对距离,可用于识别与其他节点关联度较大的节点. 紧密度(closeness)包含了网络中所有节点的距离信息[166].
4.2.4 特征向量中心性 特征向量中心性(eigenvector centrality)是测量节点对网络影响的一种方式. 针对连接数相同的节点,相邻节点分数更高的节点会比相邻节点分数更低的节点分数高,依据此原则给所有节点分配对应的分数. 特征向量得分较高意味着该节点与许多自身得分较高的节点相连接[166].
4.2.5 模块内连通度和模块间连通度 在网络模块化的基础上衍生出了两个重要的节点特征,即模块内连通度(within-module connectivity, Zi)和模块间连通度(among-module connectivity, Pi),其中Zi衡量一个节点在模块中与其他节点的连接程度,值越高表明其在模块中的作用越大,Pi衡量一个节点参与其他模块的程度,值越高表明和其他模块关系越密切[168]. 根据Zi和Pi的值可将节点分为4种类型,包括(1)模块中心点(module hubs),在模块内部具有高连通度的节点,Zi>2.5且Pi<0.62;(2)连接节点(connectors),在两个模块之间具有高连通度的节点,Zi<2.5且Pi>0.62;(3)网络中心点(network hubs),在整个网络中具有高连通度的节点,Zi>2.5且Pi>0.62;(4)外围节点,在模块内部和模块之间均不具有高连通度的节点,Zi<2.5且Pi<0.62[168]. 在生物共现网络模块中,可根据Zi和Pi值识别关键节点,这些节点往往代表了维持生物群落结构稳定的重要关键物种.
4.2.6 关键节点 生物共现网络是无标度(scale-free)的,即网络中的大部分节点只有少数连接,仅有几个节点具有高度连接,这种节点称之为关键节点(keystone nodes)[47,169]. 综合上述几个中心性指标和模块化指标(Zi和Pi值)可以确定共现网络的关键节点. Steele等将关键节点与关键物种(keystone species)的生态概念联系起来[122].
4.3 网络性质
基于随机图论构建起来的分子生态网络都具有小世界(small world)、无标度、模块化(modular)等基本特征[170-171],而这些特性可能对生态系统的结构和功能稳定性有重要影响[172-173].
4.3.1 小世界 许多网络具有小世界行为,即两个均匀随机选择的节点之间的典型距离与节点数的对数成比例增长,同时表现出较高的平均聚类系数[153]. 简而言之,小世界网络中的大多数节点间并不相邻,但大多数节点可通过少量的步骤从其他节点相连[170]. 小世界模式有助于系统内不同成员之间高效、快速的通信,因此系统可以对环境变化做出快速响应. 另一方面,小世界模式允许环境扰动迅速波及整个网络,从而改变网络的结构和功能[174].
4.3.2 无标度 如果网络中节点的度k满足无标度的幂分布p(k)~k-γ,其中γ为常数,则该网络为无标度网络[155]. 无标度网络的连通性遵循幂律,即网络中仅有少数节点与其他节点有许多连接(有大量的边),但大多数节点只有少数的边[174].
4.3.3 模块化 模块化是许多大型复杂系统的固有特征. 在生态学中,模块可以是一组物种,它们之间相互作用强烈,但与其他模块中的物种相互作用很小[175]. 生态群落的模块性可能反映了生境异质性、物理接触、功能联系、差异选择和/或密切相关物种的系统发育聚类[159]. 模块及其组成物种甚至可能是共同进化的关键单元[174]. 许多生态网络都具有模块化(层次化)的拓扑结构,这种网络可以划分为若干模块(组),同一模块内的节点间紧密连接,而不同模块间的节点之间连接稀疏[176]. 各个模块间的分层组织确保了模块和枢纽节点间快速的通信,因此网络的模块化特性将有助于降低环境变化对整个系统的影响[173].
5 生物共现网络拓扑结构参数可作为淡水生态系统健康评价指标
在进行生态系统状态评价时,应从能反映其组成、结构和功能等多方面来选择评价指标. 理论上理想的评价指标需满足以下几个要求(1)简单直观,即能直接反映生态系统的内在特性;(2)评估方法标准化,使用一些相对简单的特征参数作为评估指标;(3)可操作性,即评估设备易于设置,成本适中,基于现有的技术手段能够获取相关参数;(4)对环境变化具有敏感性和特异性;(5)评估结果可用于指导管理决策[32]. 传统的淡水生态系统生物监测和评估方法都是基于劳动密集型的形态学观察,成本高,鉴定困难,难以得到广泛应用. 随着eDNA宏条形码技术的不断完善和测序成本的持续下降,可简便快捷地利用eDNA数据构建生物共现网络(图2). 分析网络和群落在空间和时间上的变化,可以检测和解释生态系统功能和服务的变化[177-181]. 这种分析方法不依赖于节点本身的物种特性和生态特征[182],避免了物种鉴定的困难. 不仅如此,环境因子也可作为节点放入生物共现网络中,以研究环境因子与物种以及群落组成的关系[148]. 生物共现网络的拓扑结构特征提供了与生物群落结构有关的信息,可用于评估群落对自然扰动和人为影响的抵抗力以及恢复力[181,183]. 因此,生物共现网络的拓扑结构参数可能是生态系统功能和完整性的有力指标[178,184-186]. 近年来,人们开展了一系列研究,从生物群落数据中推断生物共现网络,以探索网络结构特性(如连接度、中心性、模块性等)与生态系统功能之间的联系[187-191],并用以评估环境变化对生态系统的影响. 因此可以利用生物共现网络拓扑结构的一些关键参数作为淡水生态系统健康的评价指标.
5.1 网络复杂性指标
研究表明,生物共现网络拓扑结构的变化会影响生态系统的功能和稳定性[93,192-193]. 网络复杂性越高代表网络稳定性越高,可能说明生态系统越稳定[32,193]. 例如,Zappelini等发现,人为导致的土壤汞污染将显著降低土壤微生物共现网络的复杂性[188]. Lupatini等也发现土地利用对网络的连接度和网络密度有影响[191]. 在淡水生态系统中,人为影响使得网络的连接度降低[194-196],并使得正向连接的比例降低[194,197]. Al等对真核微生物共现网络的分析发现,城市水体污染会对网络的复杂性和相异性产生显著的不利影响[198]. 综合这些研究结果会发现,当环境受到干扰时,生物共现网络的复杂性会降低. 因此,代表网络复杂性的拓扑结构参数可作为淡水生态系统健康的潜在评价指标.
5.2 模块性
生物共现网络由多个节点和连接组成,较难直观地获取信息,可使用模块划分等方法简化网络[199]. 模块化是生态系统结构的重要特征,可识别高度连接的分类群,反映了复杂网络中的协同关系、竞争互动和生态位分化[159]以及一些进化特性[200]. 将网络划分为多个模块可以揭示执行不同功能的不同节点组[201]. 例如,Zhao等对南京城区6个湖泊中浮游细菌群落的研究中将共现网络分为几个主要的模块[199]. 同一模块中的细菌连接紧密,具有相似生态位,共同行使某一功能特性. 研究还发现,不同的环境因子影响不同的模块. Abdullah等发现,城市水体污染会打破微生物共现网络结构的模块化,相较于正常水体,网络模块性指数显著下降[198]. Ford等也发现,海洋鱼类共现网络模块化更高的群落对物种损失等干扰的抵抗力更强[183]. 对亚热带水库蓝藻水华的研究中发现,蓝藻生物量的变化强烈影响了水库微型真核浮游生物共现网络的模块性,不同模块对应于蓝藻水华生消的不同时期,表明蓝藻水华生消可导致环境异质性,创造不同的生态位,从而改变了水库微型真核浮游生物的共现模式[107-108]. 上述研究均说明,共现网络模块性能够体现环境变化对生物群落组成的影响[202-204],模块性的增加使得共现网络的结构更加稳定,体现了更高的生物群落稳定性[30,193]. 因此,模块性指数也可作为淡水生态系统健康的潜在评价指标.
5.3 关键节点
网络结构中的关键节点可能代表了对生态系统具有重要作用的关键物种(keystone species),可通过生物共现网络的中心性指标、模块内连通度(Zi)和模块间连通度(Pi)对其进行推断[122]. 例如,Ford等选择向量中心性指标确定了澳大利亚海域鱼类共现网络中的关键物种是该海域的特有品种[183]. Yang等综合Zi和Pi值,确定了4个细菌类群(假单胞菌属Pseudomonas、寡养单胞菌属Stenotrophobacter、博斯氏菌属Bosea和生丝微菌属Hyphomicrobium)和5 个真核藻类类群(单胞藻属Monodus、四链藻属Tetradesmus、栅藻属Scenedesmus、单针藻属Monoraphidium和双眉藻属Amphora)是维持城市河流微生物共现网络稳定性的关键物种[205]. 利用中心性指标可以确定亚热带水库蓝藻水华暴发时期的关键真核微生物[107-108],并且Liu等还发现这些高中心性节点与蓝藻生物量的相关性也越高,表明这些浮游生物在共现网络中可能发挥了决定性作用[108]. 研究表明,关键物种(节点)为共现网络结构提供了完整性,移除这些物种将导致网络结构的巨大改变[206-207]甚至崩溃[208]. 因此,在生态系统监测中应优先关注关键物种,以避免生态系统内的连锁灭绝或生态系统功能发生改变[37,209-210]. 因此,生物共现网络中能够代表关键物种的关键节点也可作为淡水生态系统健康的潜在评价指标. 例如,真核微生物共现网络中关键物种的相对丰度会随着城市水体污染程度的升高而下降,这些关键物种可作为水质状态监测和诊断的生物指标[198].
综上所述,体现网络复杂性的指标(如平均度数、连接度和网络密度等),体现关键物种的关键节点指标(中心性指标、Zi和Pi值)以及共现网络模块性参数可能是监测淡水生态系统状态的合适指标(图4).
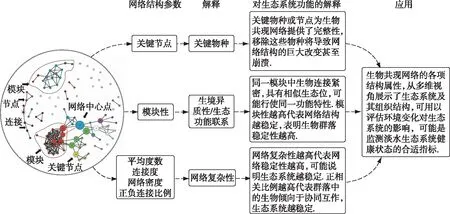
图4 生物共现网络的部分结构参数可能是监测淡水生态系统健康状态的合适指标Fig.4 Some topological metrics of biological co-occurrence networks could be used as indicators of the state and stability of freshwater ecosystem
6 展望
虽然利用生物共现网络进行淡水生态系统质量监测和评价具有很大的应用前景,但基于eDNA宏条形码技术推断网络结构特性与生态系统状态和功能的联系仍处于初级阶段[121,190,195-196]. 一些方法学问题限制了共现网络的稳健推断.
首先,eDNA宏条形码技术尚未标准化和规范化. 主要表现在(1)eDNA采样及提取方法缺乏统一的标准,使用不同方法获得的研究结果之间没有可对比性[211]. 此外,在eDNA样品采集和提取过程中应尽量避免环境污染和样品之间的交叉污染. 尽量增大样本量和设置重复(尤其是生物重复)以保证样品中物种分布的全面性[212]. (2)通用引物的选择. 目前用于原核生物识别的 DNA 条形码区域主要为核基因的16S rDNA基因. 而用于真核生物不同分类群的DNA 条形码区域则差别很大. 例如,用于真核浮游植物的包括核基因的ITS、18S rDNA、28S rDNA 和叶绿体rbcL基因等[213-214];用于鱼类的包括核基因的12S rDNA[215]、16S rDNA[216],线粒体细胞色素b(Cytb)基因[217];用于识别两栖类的包括12S rDNA[218]、Cytb基因[219]、COI基因[220];用于识别节肢动物的有COI基因[221]等. 不同通用引物的扩增效率以及对应数据库的物种覆盖率差别较大. 因此研究者需对监测的水体系统中物种组成有所了解,选择适宜的通用引物应用于不同分类群eDNA的扩增. (3)测序平台和测序深度的选择. 由于不同生态系统中物种丰富度不同,所需的测序深度也有所不同,因此不同研究中样品的测序深度相差较大. (4)通过eDNA估计物种丰度或生物量的准确性. 虽然现已证明eDNA在评估物种存在与否方面特别有效,但使用eDNA检测估计物种丰度或生物量(尤其是大型生物)尚处于起步阶段,迫切需要建立分析eDNA 浓度与物种丰度之间关系的全面而普适性的方法[222].
其次,前文简要介绍了目前常用于生物共现网络构建的方法,这些方法在解析生物之间以及生物与环境之间的相互作用方面具有巨大的潜力,但也各有其适用范围以及局限性. 研究者通常需要根据所分析数据的独特特征(如组成性、稀疏性等)量身定制分析工具. 幸运的是,已开发了越来越多的统计方法用于生物共现网络的构建,这些新方法有可能解决目前阻碍测序数据分析的一些主要限制[126].
第三,尽管生物共现网络已广泛应用于同一营养级内生物种类(群落)的分析,如微生物群落、鱼类或真核浮游生物等,而在淡水生态系统中同时存在多个营养级,迫切需要发展新的分析技术以展现不同营养级之间的关系. 不仅如此,生物共现网络分析只是体现了生物之间和生物与非生物(环境因素)间潜在的交互关系,研究者还需要通过行为观察、操纵实验或生物自然史等来确定网络模型识别出的相互作用的确切性质. 因此,在实际应用时,还应考虑不同淡水水体环境的背景特征,综合使用各项生物共现网络指标并结合传统的评价指标和其他群落梯度分析方法,如广义非相似性模拟(generalized dissimilarity modelling, GDM)[223],对淡水生态系统状态进行更全面的评价.
上述几点均影响了生物共现网络分析在淡水生态系统生物监测和评价中的推广应用. 不仅如此,推断稳健网络需要更多的样品,这也增加了时间和经济成本[181,224]. 然而,这些限制和局限性也促进了研究者不断完善研究设计和开发合适的分析工具. 这些设计和技术发展有可能使得生物共现网络作为淡水生态系统生物监测和评价指标得以实现.
7 结论
开发快速有效的淡水生态系统监测和评估工具,对保护淡水生态环境具有重要意义. 传统的基于形态学鉴定的评估方法存在耗时长、劳力投入高的限制,不利于实施大规模的生物监测. 基于eDNA宏条形码技术能够高效便捷地获取淡水环境中的群落大数据信息,借助于生物共现网络构建与分析技术,为研究淡水生态系统的响应和功能提供了一个生物监测和评价框架. 尤其是生物共现网络的拓扑结构参数可与特定的生态系统状态相关联,可作为早期的、敏感的生物指标,是一种很有应用前景的评估淡水生态系统状态和稳定性的工具. 更可靠的生态系统评估,需要基于多营养级的下一代网络构建与分析技术的发展.
8 附录
附表Ⅰ见电子版(DOI: 10.18307/2022.0601).

