DNA甲基化测量技术及准确性评估研究进展
杨 怡, 杨佳怡, 高运华, 董莲华, 杨靖亚
(1.上海海洋大学食品学院,上海201306;2.中国计量科学研究院前沿计量科学中心,北京100029)
1 引 言
表观遗传学是在DNA序列不发生改变的情况下,基因表达可遗传性改变的一门遗传分支学科,主要表现在DNA甲基化、组蛋白修饰和非编码RNA[1]。其中DNA甲基化是人们深入研究的最重要的表观遗传机制,在许多关键的生物学过程中发挥着重要作用[2,3]。基于测序的DNA甲基化分析为描绘比较完整的DNA CpG图谱奠定了基础。在过去的几十年里,大量DNA甲基化测量技术的涌现使基因组甲基化分析研究得到了极大的发展。但是由于缺乏评估基因组甲基化准确性的相关研究,目前尚不明确基因组甲基化特异性是否如通常识别序列所暗示的那样精确。因此,本文对DNA甲基化测量技术的进展以及甲基化测量的准确评估所存在的问题展开讨论,以期为基因组甲基化测量的准确可比性提供可能的解决方案。
2 DNA 甲基化的概述
DNA甲基化是DNA的一种天然修饰方式,具有多态性、随年龄变化[4]、组织特异性[5]、亲源特异性[6]等特点。其主要是指通过甲基转移酶(DNA methyltransferases,DNMTs)将S-腺苷甲硫氨酸(S-Adenosylmethionine,SAM)提供的甲基转移到DNA的胞嘧啶(C)或腺嘌呤(A)上,对DNA进行修饰而发生的一系列表观遗传现象[7]。研究发现有不同的DNA甲基化修饰,如5-甲基胞嘧啶(5-mC)、5-羟甲基胞嘧啶(5-hmC)和N6-甲基腺嘌呤(6mA)等,其中最为常见且研究最充分的碱基甲基化是5-mC[8~10]。DNA的甲基化可以调节基因活性并影响许多关键过程,如染色质结构、细胞分化、DNA构象和转录调控[11]。因此,DNA的甲基化也是实现基因沉默和基因印记的重要途径。
3 DNA甲基化的测量技术
DNA甲基化的测量就是用于区分DNA序列中的C和5-mC的能力。检测基因组DNA甲基化水平的方法有很多种,根据目的的不同可分为全基因组和特定位点的DNA甲基化水平测量[12]。但大多数DNA甲基化测量技术均是基于PCR方法,即模板是经亚硫酸氢盐处理过的DNA,根据需求选择引物进行测量。在这里,根据采用其技术类型的不同分为3大类[13]:基于甲基化敏感限制性内切酶(methylation sensitive restriction enzymes,MRE)的测量、基于亚硫酸氢盐的测量和基于亲和富集方法的测量。表1中,对本文所列举的方法进行了归纳对比,后文将对重点关注的测量技术原理以及优缺点进行简要介绍。
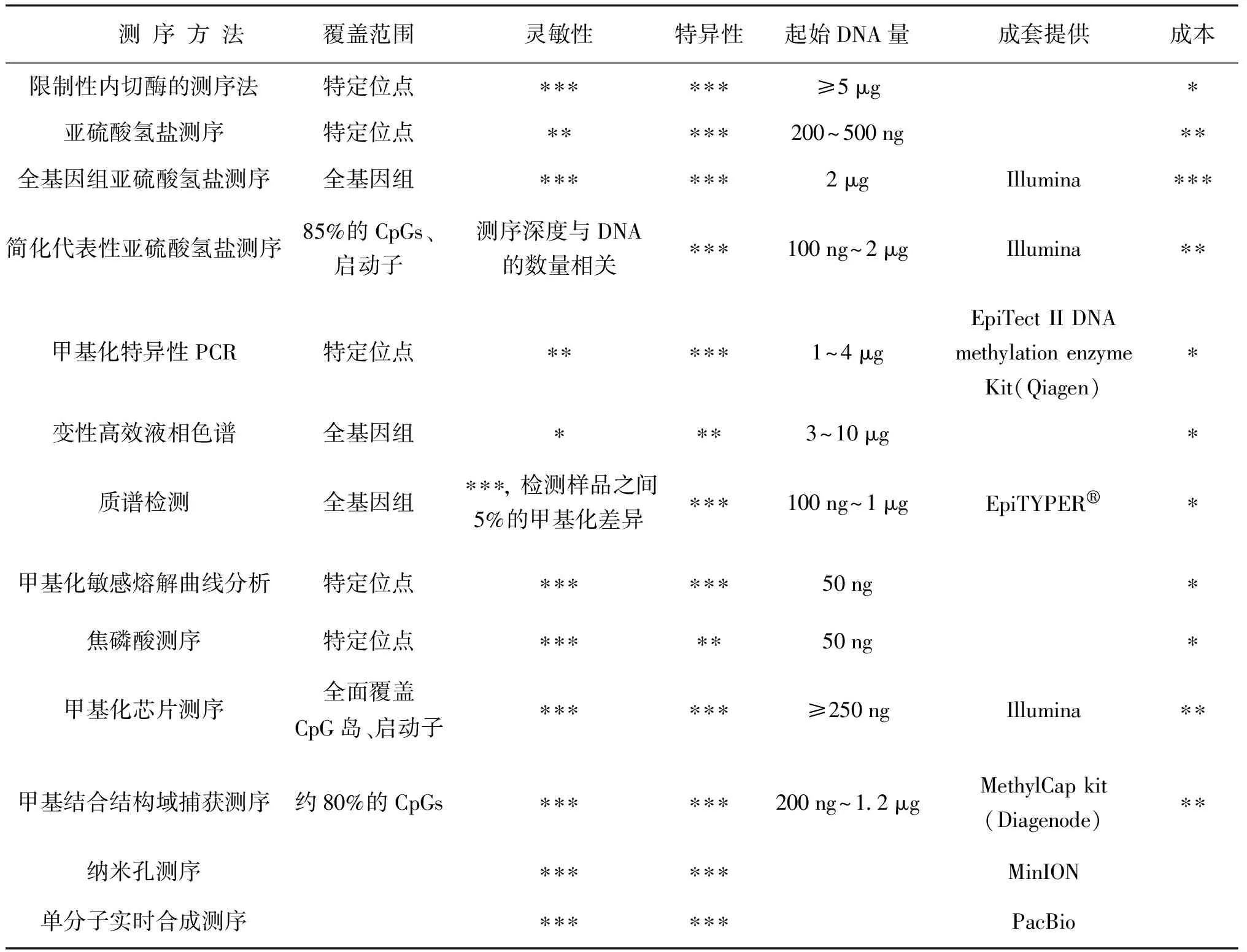
表1基于测序的DNA甲基化分析方法对比表Tab.1 Comparison table of DNA methylation analysis methods based on sequencing
3.1 基于MRE的测序
MRE是一组只针对非甲基化DNA片段的酶(如BstU l、Hpa ll、Not l)[13]。根据这一特性将其应用于DNA甲基化的测量,主要原理是利用MRE识别且切割未甲基化C位点,假设未甲基化DNA完全裂解,不能被扩增;甲基化DNA不会被消化从而保持完整,通过DNA扩增来确定基因组甲基化[14]。该方法测定条件温和、操作简单快速。不足之处在于:一是引入了识别位点偏差,分辨率相对较差,并且存在消化不完全而造成的假阳性;二是酶只能识别特定的位点,即识别CpG位点(CCGG)之前的C,不能完全并且准确地反应甲基化全貌。Sun[14]等使用了一种新型的GlaI,其能结合等温指数扩增反应(isothermal exponential amplification reaction,EXPAR)测量出特异性DNA甲基化。GlaI以极好的选择性切割甲基化的靶位点,而保留未甲基化DNA,这与MRE特性完全相反,而暴露出来的甲基化DNA末端片段触发EXPAR,放大了其高效信号。因此,GlaI-EXPAR对测量DNA甲基化具有高度特异性和灵敏性的特点,弥补了传统基于MRE的测量造成假阳性结果的缺陷。然而,GlaI也是一种酶,也只能识别特定的DNA甲基化靶点,不能准确的反映出的全基因组范围的甲基化状态。
3.2 基于亚硫酸氢盐的测量
3.2.1 亚硫酸氢盐测序(BSP)
亚硫酸氢盐测序(bisulfite sequencing PCR,BSP)是目前公认的主流DNA甲基化测量技术之一,是评估DNA甲基化的“金标准”技术。该方法首先是由亚硫酸氢盐处理DNA,使未甲基化的C化学转化为胸腺嘧啶(T),而甲基化的C保持不变;进而PCR扩增,对PCR产物测序,比较测序结果与未处理序列。因为C只来源于5-mC,由此可确定DNA序列中甲基化位点[15,16]。此法可对目的基因的每一个CpG甲基化位点精准定位,但样本的准备过程过于繁琐,并且需要纯度较高的DNA以避免残余的蛋白质对亚硫酸氢盐的转化造成干扰。
BSP是对特定位点DNA甲基化的测量,不能测量出全基因组DNA的甲基化。因此,基于二代测序的全基因组甲基化测序(whole genome bisulfite sequencing,WGBS)随之问世, 它结合了亚硫酸氢盐转化和高通量测序,提供了单碱基分辨率的全覆盖,以随机的方式覆盖人类基因组中超过90%的CpGs[17],但需达到一定的测序深度才能保证覆盖范围和水平。简化代表性亚硫酸氢盐测序(reduced representation bisulfite sequencing,RRBS)是另一种在单碱基分辨率下分析DNA甲基化的技术,它结合了MRE(如MspI)对基因组DNA进行消化、制备文库和亚硫酸氢盐处理的测序,以丰富CpG含量较高的区域[17]。RRBS的优势在于增加了基因组特定区域的测序深度,降低了成本以及基因组数据量;缺点是不能测量全基因组范围内DNA甲基化[17,18]。
为了解决WGBS和RRBS的局限性,Bonora等[18]开发了一种新的方法,即甲基化敏感性限制酶亚硫酸氢盐测序(methylation-sensitive restriction enzyme bisulfite sequencing,MREBS),降低了RRBS的测序要求,且显著扩大了基因组中CpG位点的覆盖范围。其基本原理是:利用3种MRE(如HpaII(CCGG),HinP1I(GCGC)和AciI(CCGC))并行消化DNA非甲基化区域,基于MRE-seq读图覆盖率与切割位点附近的DNA甲基化水平之间的反比关系,直接测量其侧翼区域的DNA甲基化水平。
3.2.2 甲基化特异性PCR(MSP)
甲基化特异性PCR(methylation-Specific PCR,MSP)是由Herman于1996年首次提出的一种针对特定位点DNA甲基化的测量技术[19]。其主要原理是DNA片段由亚硫酸氢盐处理后,根据目的基因修饰前后的改变分别设计出非甲基化特异性引物和甲基化特异性引物,随后目的基因经PCR扩增,根据自身甲基化情况将结果通过琼脂糖凝胶电泳图像显现出来。若甲基化引物扩增出条带,则该位点发生甲基化;若无甲基化引物扩增条带,则该位点没有发生甲基化。因此,该方法操作简便快捷且特异性高。由于需要设计引物,所以只能对已知序列和部分甲基化DNA进行测量[16,20]。
Fackler等[21]创建了一种定量多重甲基化特异性PCR(quantitative multiplex methylation-specific PCR,QM-MSP)的技术,具有灵敏性强且能定量多重分析DNA甲基化水平的特点。该方法的特殊之处在于运用了两步PCR方法:第一步反应是基因特异性引物在其PCR反应中能同时多重扩增同一基因的甲基化和非甲基化拷贝,不依赖于甲基化扩增步骤中产生的扩增产物;第二步反应是可以使用实时PCR和两个独立的荧光团通过标准曲线对第一步反应的扩增片段进行定量,以测量同一孔中每个基因的甲基化/未甲基化DNA。
数字PCR(digital PCR,dPCR)是可以对单个DNA分子进行计数且对核酸进行绝对定量的第三代PCR技术。Cui等[22]开发了一种基于微孔芯片dPCR的甲基荧光分析法(MethyLight)测量DNA甲基化。与14种传统定量PCR相比,dPCR达到了93.3%的最高检出率。因此,dPCR是一种很有潜质的测量DNA甲基化的工具。其主要原理是:通过微液滴化,将其随机分配至微反应单元中,PCR循环扩增和读取后,根据泊松分布来进行绝对定量[22]。该方法灵敏度强、精准度高且重复性好,但液滴的控制和读出需要专门的设备,这增加了程序的成本,并限制了其广泛应用。
3.2.3 变性高效液相色谱(DHPLC)
变性高效液相色谱(denaturing high-performance liquid chromatography,DHPLC)是一种常用于检测基因突变位点的新型检测技术。Deng[23]等将发生甲基化的多个CpG位点作为多位点突变,首次利用DHPLC测量DNA甲基化。其主要原理是:DNA甲基化的差异是通过监测部分变性条件下亚硫酸氢盐处理的DNA扩增产物的保留时间来揭示的。也就是说,序列中所有CpG位点完全甲基化导致PCR产物的G/C含量增加,进而需要更高的熔融温度,从而增加了在DHPLC分析中保留的时间。但单个异常甲基化的CpG位点不能被精准定位。如果发生不完全CpG岛甲基化,MSP和MRE-seq测定的非甲基化的结果可能就没有意义了,而DHPLC则将目标基因的所有CpG位点甲基化状态显现了出来。因此,此方法只适用于CpG岛甲基化模式的筛选[24,25]。
3.2.4 质谱检测(mass spectrometry,MS)
质谱检测(mass spectrometry,MS)是由亚硫酸氢盐处理的DNA,经PCR扩增并且转录成单链RNA分子后被特异性地裂解成碱基,利用基质辅助激光解吸电离飞行时间质谱法(matrix assisted laser desorption ionization-time of flightmass spectrometry,MALDI-TOF-MS)进行质谱分析,检测出原DNA甲基化的胞嘧啶位点的方法[16,25,26]。因为在亚硫酸氢盐处理过程中,未甲基化的C转化为U将产生碱基特异性切割产物,而这些产物能反映潜在的甲基化模式,所以可以很容易地用MALDI-TOF-MS进行分析,进而测量出甲基化位点。Suchiman等[27]开发了一种基于质谱的亚硫酸氢盐测序技术平台,即EpiTYPER®。它能对100~600个碱基对的基因组区域进行甲基化检测,在很大程度上以单核苷酸分辨率对甲基化水平进行定量测量,实现了定量和高通量的区域特异性DNA甲基化分析。因此,该方法特别适合大规模或者全基因组DNA甲基化的研究验证。
3.2.5 甲基化敏感熔解曲线分析(MS-HRM)
甲基化敏感熔解曲线分析(methylation-sensitive high-resolution melting,MS-HRM)是一种检测单核苷酸变异的新型技术,具有灵敏性高、特异性强且更为有效的特点[28]。它为临床研究中筛选基因甲基化案例提供了敏感性和快速性,主要原理是基因组DNA经亚硫酸氢盐处理后,保留了甲基化DNA中CG碱基对,未甲基化DNA中CG碱基对则转变成了AT碱基对,通过熔解曲线分析,CG含量越高的双链DNA螺旋的熔融温度(Tm)越高,进而检测出特定位点的DNA甲基化水平[29]。通过对熔解温度及峰型的分析,可以区分基因组甲基化的程度,包括完全甲基化、部分甲基化和未甲基化。因此,该方法可以区分甲基化中极其微小的差距,缺点是不能测量出基因组甲基化的碱基序列。MS-HRM灵敏性高的原因:一是特别设计的引物可以与甲基化等位基因互补,因此可在未甲基化背景下检测出低至(0.1~1)%的甲基化等位基因;二是特定的退火温度可以使这些引物能够同时对甲基化和未甲基化等位基因进行退火,所以提高了测量的灵敏度[30]。MS-HRM的高灵敏度已经被证实可用于无创检测膀胱癌患者尿液、结直肠癌患者粪便以及乳腺癌患者颊粘膜中的癌症生物标记物[30]。
3.2.6 焦磷酸测序(PSQ)
焦磷酸测序(pyrosequencing,PSQ)是一种边合成边测序的实时DNA序列分析技术[31]。其被用来分析DNA甲基化模式,首先是将基因组DNA经亚硫酸氢盐处理,使其未甲基化C转化,保持甲基化C不变,PCR扩增后,DNA甲基化水平就是CT单碱基多态性位点中等位基因C的频率[32]。此方法反应过程直接,且绝对定量甲基化水平,操作简便、特异性高;不足之处在于检测CpG数量有限[32]。Reed等[33]于2010年首次对BSP和PSQ进行评估,通过实验检测和量化各种药物敏感性和耐药性基因组启动子的甲基化状态,结果表明BSP和PSQ均能检测到DNA的低甲基化和混合甲基化,但BSP检测强甲基化DNA更为敏感。
3.2.7 甲基化芯片测序( Methy-chip-seq)
甲基化芯片测序(methylation chip sequencing, Methy-chip-seq)是一种基于亚硫酸氢盐或酶处理DNA序列杂交的信号探测技术[16]。其主要原理是:基因组DNA经过亚硫酸氢盐处理,将未甲基化C变成U,保持甲基化C不变,然后再将U转化成A,最后进行芯片杂交,根据荧光信号来确定某一位点的甲基化水平[16]。该方法可以进行跨物种甲基化片段测量,且不需要基因组序列信息,可归属于全基因组甲基化测量技术;但芯片杂交要求设备昂贵,数据处理依赖生物信息学知识。Wu等[34]设计了一个基于微流控芯片的新型的dPCR装置,将甲基化测量的dPCR过程集成在一个芯片上,能在低至1%甲基化基因的样品中成功测量到甲基化,检测限为0.52%。该方法不仅灵敏度高、特异性强且重复性好,芯片还降低了dPCR的成本,简化了dPCR的过程,不需要专门的dPCR设备,属于甲基化的全芯片测量。
3.3 基于亲和富集方法的测量
3.3.1 甲基化DNA免疫共沉淀测序(MeDIP-seq)
甲基化DNA免疫共沉淀测序(methylated DNA immunoprecipitation sequencing, MeDIP-seq)是一种经济高效的用于基因组特异性和全基因组分析的方法[35]。最先是由Weber等[36]提出,设计原理是:首先将双链DNA超声波处理并变性后得到片段单链DNA,然后使用抗5-mC的单克隆抗体来富集选择大小(通常为150~300 bp)的甲基化DNA片段,并对富集片段进行建库、测序和分析,以测量CpG和非CpG甲基化。随后,有研究者在此基础上进行方案优化,提出了能在输入DNA量为50~5,000 ng范围内显示出可比的特异性(>97%)和富集(>100倍)以及在5 GB测序时达到饱和的方案[37,38]。该方法的优点是:不经过亚硫酸氢盐处理,所获得的数据量较小,降低了数据处理难度的同时也提高了该方法的性价比;缺点是不能确定富集片段中每个位点的胞嘧啶甲基化状况和无法实现单碱基分辨率,同时测量不出绝对的甲基化水平,只适合样本间的相对比较,该抗体只能在单链DNA中捕获5-mC[39]。当然,研究的步伐永不停歇。Jia等[40]开发了一种名为MB-seq的新型DNA甲基化基因组分析技术,这是一种结合MeDIP-seq和亚硫酸氢盐转化的新颖DNA甲基化组分析技术,可以精确检测5-mC位点并以单碱基分辨率确定其DNA甲基化水平,并且成本较低。它只需要7~8 Gbp的数据来测量具有足够覆盖范围和测序深度的人类甲基化组,加速了人类疾病中DNA甲基化机制的解码过程。
3.3.2 甲基结合结构域捕获测序(MBD-seq)
甲基结合结构域捕获测序(methyl-binding protein sequencing,MBD-seq)的原理与MeDIP-seq极其相似,区别在于将抗5-mC的单克隆抗体富集甲基化DNA片段替换成特异性结合甲基化DNA的MBD蛋白富集[41,42]。MBD之所以能用于DNA甲基化分析是其能特异性结合甲基化DNA。因此,基因组DNA的MBD沉淀与大规模平行测序相结合,可以实现对多个样本的高通量分析,且不会引起序列偏倚[43]。MBD分离的基因组测序(MBD-isolated genome sequencing,MiGS)特异性高,灵敏性强且高通量,适合于研究全基因组DNA甲基化模式,既能有效地测量已知的DNA甲基化水平又可鉴定许多新型DNA甲基化位点。它结合了MBD2蛋白的重组甲基CpG结合域对甲基化DNA的沉淀,并通过大规模平行测序仪对分离的DNA进行测序,以鉴定基因组规模上的差异甲基化区域[41]。
3.4 基于第三代测序的甲基化测量
3.4.1 基于单分子纳米孔技术的甲基化测量
纳米孔测序(nanopore sequencing,Na-seq)是一种可以直接测量DNA甲基化修饰的新兴的第三代测序技术。其主要原理是:基因组DNA经过外切酶消化成单链DNA分子,在电泳作用下,不同碱基(A、T、C、G、5-mC等)的化学性质差异在穿越纳米通道时,会引起不同变化的电流信号,最后可以通过测序仪快速读出碱基类型[44]。简而言之,纳米孔测量基因组甲基化就是根据穿过蛋白孔的离子电流变化来确定DNA甲基化位点[45,46]。该方法从成本和时间上看,具有无法比拟的优越性,同时还具有灵敏度高、特异性强且重复性好的特点。因此,纳米孔测序技术可能会成为未来发展的主流方向。
3.4.2 基于单分子实时测序的甲基化测量
单分子实时合成测序(single molecule real time sequencing,SMRT)是由FJusberg等[47]提出利用DNA聚合酶进行边合成边收集荧光信号的方法进而测序的第三代测序技术。它是最新开发的方法,通过对模板链复制来获得序列信息[48]。基本原理如下:将待测DNA片段化,使其两端连接发夹接头形成闭合的环状单链模板(称其为SMRTbell),DNA聚合酶捕获文库DNA序列[49],锚定在零模波导孔底部(zero-mode waveguides,ZMWs),4色荧光标记的dNTP与DNA模板碱基配对,形成聚合酶-引物-SMRTbell复合物,通过其荧光信号的颜色和脉冲间隔时间获得酶动力学信息,从而直接检测DNA模板上的核苷酸修饰,包括甲基化[50]。该方法的优势在于单分子测序,不需要PCR扩增,覆盖高CpGs区域,延长了测序读长,最大限度地保持了聚合酶的活性;劣势在于:错误率较高(可达15%),且出错随机。目前已经通过提高酶的活性、增加测序的深度等手段极大地降低了随机错误率。
4 甲基化测量准确性评估
4.1 目标基因甲基化准确性评估研究现状
DNA甲基化是肿瘤诊断的理想标志物[51~53],但是如果DNA甲基化测量结果不可靠,将会严重影响其在临床中的应用,因此DNA甲基化测量结果的准确性评估非常关键。由于不同实验室的设备、人员、操作等方面存在很大差异,导致测量结果在不同实验室、不同平台间测定结果的比较性不高,为此国际计量局物质量咨询委员会于2005年、2009年和2014年分别组织了3次关于DNA甲基化的测量比对。由主导实验室韩国标准科学研究院(Korea Research Institute of Standards and Science,KRISS)协同不同国家计量机构之间进行国际比较研究。在对人工合成的DNA样品中总甲基胞嘧啶含量定量测量和CCQM P94.2的细胞周期蛋白依赖性激酶抑制剂2A(cyclin-dependent kinase inhibitor 2A,CDKN2A)基因DNA甲基化定量测量的国际比对研究中,各机构实验平台分别采用不同的方法(如毛细管电泳法、MALDI-TOF-MS、序列测定技术、熔点法、液相色谱同位素稀释质谱法和MRE-seq等)进行测量,然而,不同实验室之间的测量结果与KRISS所设定的参考值存在差异[54,55]。差异的来源可能是实验室之间的设备、人员操作存在问题,也可能是采用的测量方法原理的不同导致对甲基化测量的特异性和灵敏度存在差异。期间,仍有研究学者为了研究DNA甲基化检测方法的准确度和灵敏性,分别采用BSP和PSQ两种检测方法来检测和量化各种药物敏感性和耐药性基因组启动子的甲基化状态,结果表明BSP和PSQ均能检测到DNA的低甲基化和混合甲基化,但BSP检测强甲基化DNA更为敏感[33]。由于缺乏与DNA甲基化有关的参考分析系统来进行准确性评估,DNA甲级化测量结果的可信度需要进一步验证。因此,急需建立一套规范的目标基因甲基化测量分析流程或研制出甲基化测量标准物质,以减少甲基化测量中的假阳性或假阴性结果的产生,提高目标基因甲基化相关疾病诊断的准确性。
4.2 基因组甲基化准确性评估研究现状
随着研究的深入,大量基因组检测技术浮出于世,应用于一定范围的同时也取得相应的成就。但基因组甲基化测序技术非常复杂,包括样本的制备、建库和测序,以及生物信息学分析算法和软件等。每个步骤都会引入不确定性,导致最后获得结果可靠性降低。因此,急需建立评估基因组甲基化分析流程或模型。
Simpson等[45]在纳米孔检测胞嘧啶甲基化的实验中设计了由PCR扩增的基因组DNA作为完全未甲基化的阴性对照和由CpG甲基转移酶(CpG methyltransferase,M.Sssl)处理的基因组DNA作为完全甲基化的阳性对照,但该酶是否可以完全将其进行甲基化尚不清楚;随后,利用大肠杆菌数据训练的隐马尔可夫模型(hidden markov model,HMM)和对数似然比对阴阳性对照样本的每个位点进行甲基化调用,评估甲基化调用的准确性,发现使用纳米孔R7.3数据的甲基化位点的准确率为83%,使用R9数据的准确率为87%。由此可见,不同的芯片产生的结果存在差异,这也表明了基因组甲基化准确性评估的重要性。然而,由于训练数据集的限制,HMM无法识别非CpG甲基化或甲基化和未甲基化的混合物。随后Rand等[46]建立了一个更完整的检测甲基化位点调用准确性的模型,称其为HMM-HDP。它是由HMM和层次狄利克雷过程(hierarchical dirichlet processes,HDP)组成,除了训练并记忆1 784个典型碱基的6-聚体离子流分布,还要训练2 868个新的具有甲基化碱基的6-聚体离子流分布,通过单链上正确甲基化调用的比例来衡量每次读取的准确性。结果表明,HMM-HDP对pUC19质粒和大肠杆菌的纳米孔R9数据在基因组水平上的准确率为86%~95%。McIntyre等[56]提出一种新的核酸修饰呼叫器(称其MCaller),并结合国际空间站(international space station,ISS)所生成的样本DNA甲基化的正交PacBio数据,用其测量信号和预期信号的偏差作为特征模型训练且测试二元分类器,利用训练后的4种分类器(神经网络、随机森林、朴素贝叶斯和逻辑回归)检测小鼠、大肠杆菌MG1655K12和λ噬菌体三种基因组DNA的等摩尔混合物数据中的6 mA,在15倍或更高的覆盖率下,它们在基因组水平上达到了94%的准确率。Ni等[44]开发了一种深度学习方法,称为深度信号(DeepSignal),他们利用pUC19质粒的5-mC和6 mA数据集对DeepSignal进行检验,均获得比HMM更高的读数水平和基因组水平的准确率。为了进一步评估DeepSignal的性能,还使用大肠杆菌和人类的CpG甲基化(5-mC)数据集进行测试,在5倍覆盖率下,它们在基因组水平上的准确率超过90%。以上基于模型的方法中,经过对模型的优化改进,其结果均能提高基因组甲基化测量的可行性和准确性。但它们需要进行大量的先前训练数据集,因此不能测量不同类型的基因组甲基化修饰,特别是人为引入的修饰。
Stoiber等[57]使用一种可实现基因组序列可视化的软件包(Nanoraw)来解析带有基因组甲基化位置的原始纳米孔信号。其原理主要是使用改良DNA测序(modified DNA sequencing, MoD-seq)对天然的和匹配的扩增DNA样本进行纳米孔测序(其中利用扩增来产生未甲基化修饰的DNA)。Nanoraw通过碱基检出、错误校正等处理这些数据,并在全基因组范围内比较重新处理的原始信号,从而识别出甲基化修饰的碱基。为了评估Nanoraw的准确性及可行性,Mann等[58]通过MoD-seq的曼-惠特尼U检验(Mann-Whitney U-test)对大肠杆菌中由7个不同纯化甲基酶构建的已知目标位点的基因组甲基化进行测量,结果表明它们在基因组水平上的准确率为(84~90)%。Liu等[59]开发了一种名为NanoMod的新型计算工具,其使用从纳米孔长读长测序技术产生的原始电信号来测量基因组甲基化。主要原理是:用含甲基化碱基和不含甲基化碱基的两组读数作为NanoMod的输入,然后进行碱基检出,错误校正和检验已知位置的信号,从而测量出甲基化修饰的碱基。他们根据已公布的大肠杆菌甲基化数据集对结合了柯尔莫哥洛夫-斯米尔诺夫检验(Kolmogorov-Smirnov test,K-S test)[60]的NanoMod进行准确性评估,其结果表明在基因组水平上的准确率为70%。这两种基于统计的方法都能在没有进行先前训练数据集的情况测量甲基化水平。与基于模型的方法相比,基于统计的方法测量的准确率较低。
科学合理的分析流程或模型的建立,依赖于准确可靠的基因组甲基化标准物质或标准数据集,通过使用标准物质或标准数据集进行质控和参数的训练优化,得到的流程或模型才能可靠。然而在基因组甲基化标准物质方面的研究除上述团队外,目前还没有更多的报道。对于全基因组甲基化标准物质的准确定值是其中的难点和关键所在,这也是我们在未来的研究中需要攻克的难题。
5 总结与展望
分析基因组中DNA的甲基化对理解其在正常的生物学功能和肿瘤疾病中的影响至关重要。DNA的甲基化改变可以为肿瘤疾病的诊断和治疗提供一条重要的途径,而且准确可靠的甲基化检测结果对疾病早期诊断、发病风险评估、疗效评价以及复发预测具有重要的意义。现今DNA甲基化的检测方法有全基因组分析的方法,如第三代测序技术,已经被用来评估大部分的甲基化基因组;以及以亚硫酸氢盐为基础的方法,如典型的BSP、PSQ等,已经建立了许多不同的定量方法来绘制DNA表观基因的单碱基分辨率图谱。DNA甲基化的检测方法众多,各有优劣,应多方比较,结合实际情况后选取。
随着生物技术的发展,DNA甲基化测量技术势必会不断更新,未来可能会开发出具有更高准确性和更高灵敏度的甲基化测量技术。然而,对现有甲基化测量技术和未来新技术的可靠性和准确性评估,是使其能否得到更广泛的临床应用的关键。这就需要攻克基因组甲基化标准物质的定值关键技术,开发准确可靠的基因组甲基化标准物质,用来评价不同检测方法的准确性,保证检测结果的可靠性。

