苹果锈果类病毒(ASSVd)的种群及遗传变异分析
杜亚杰 张煜泓 李世访 朱英波 张志想
摘要 苹果锈果类病毒apple scar skin viroid(ASSVd)引起苹果锈果病,是限制我国苹果生产的重要因素之一。然而,目前对ASSVd全球种群的组成及遗传变异仍缺乏足够的了解。为此,本研究对GenBank中登录的212条基因组序列进行了比较、变异分析以及系统发育分析。确认ASSVd的种群符合准种模式,由165种序列相似但不相同的变体组成。以来自我国苹果的MW315909和MW302328为代表的两种变体的数量最多,是主流变体。依据参考序列(NC001340)分析碱基变异,发现碱基变异偏好于某种碱基,并且偏好于基因组上的一些特定位点。变异不仅发生在基因组二级结构的左末端区、致病区、中央区,也发生在可变区和右末端区。值得指出的是,基因组上有4个区域极少发生变异,为保守区。其中的两个保守区(末端保守区和中央保守区)是已知的,而在可变区与右末端区交界处的两个保守区是新发现的,它们对ASSVd的复制和移动可能具有重要作用。这些结果不仅有助于掌握ASSVd的发生及流行,而且为研发RT-PCR/qPCR检测技术提供了参考,为研究ASSVd与寄主的相互作用提供了线索。
关键词 苹果; 类病毒; 种群; 遗传变异; 变异
中图分类号: S 436.611.1
文献标识码: A
DOI: 10.16688/j.zwbh.2021656
Abstract Apple scar skin viroid (ASSVd) is the causal agent of apple scar skin disease and is a major pathogen limiting apple production in China. However, the composition and genetic variation of the global population of ASSVd remain unclear. Here, we aligned 212 genome sequences of ASSVd retrieved from GenBank and analyzed their genetic variation and phylogenetic relationships. The global population of ASSVd fitted the model of quasispecies. It consisted of 165 highly related but nonidentical variants with two master variants represented by MW315909 and MW302328 from apple in China. In addition, genetic variation analysis based on the reference sequence (NC001340) showed obvious bias towards certain bases for mutation types and towards several certain positions for the distribution of mutations on the genome. Mutations occurred not only in terminal left (TL), pathogenicity (P), and central (C) regions in the secondary structure of ASSVd, but also in variant (V) and terminal right (TR) regions. It should be noted that there were four conserved regions in the secondary structure of ASSVd, two of which, terminal conserved region (TCR) and central conserved region (CCR), are known and the other two located in the junction of V and TR regions are new. The two new conserved regions may play important roles in ASSVd replication and movement. Together, these results are helpful for understanding the occurrence and epidemic of ASSVd, and provides a reference for developing detection technology of RT-PCR/qPCR and new clues for studying the interactions between ASSVd and its hosts.
Key words apple; viroid; population; genetic variation; variation
蘋果锈果类病毒(apple scar skin viroid, ASSVd)是苹果锈果病的病原[1]。侵染苹果后可在果实上引起锈果和花脸等症状,使果实产量下降,品质变劣。除苹果外,还可侵染梨[2]、杏[3]、桃[4]和樱桃[5]等果树。ASSVd是限制我国苹果主产区,尤其是北方产区苹果生产的主要因素之一[1,6]。近年来,其发生面积不断扩大,危害也日趋严重[7]。山东[8-10]、陕西[11]和河北[7]等地常有发生及危害的报道。在陕西咸阳,‘富士苹果上ASSVd的检出率高达65.2%[6]。在山东烟台,2007年-2008年约有11%的果园发生ASSVd[12];2018年的检测发现,病株率为27.7%[10]。ASSVd引起的锈果病已成为烟台苹果种植区的主要病害[10,12-13]。因此,需要重视对ASSVd的防控。
ASSVd的防控需要明确其种群组成及遗传变异。同马铃薯纺锤形块茎类病毒科Pospiviroidae的其他成员类似[14],ASSVd在复制过程中易产生变异。这意味着ASSVd种群由多种基因组序列互不相同的个体,即变体组成。这些变体在种群中的数量不同。数量多,在种群中比例高的变体通常被称为主流变体,是防控的重点。明确了主流变体后,需要进行快速、准确地检测。最常用的技术是RT-PCR/qPCR[15-17],其关键是引物设计。引物要尽可能匹配种群的不同变体,尤其是主流变体,这就需要了解变体的遗传变异。
虽然已有许多ASSVd种群和遗传变异分析的报道,但是仍无针对全球种群的分析。早期的一些研究[18-22]受测序技术和成本的限制,样本数量少,获得的基因组序列也较少。随着测序技术的发展,虽然获得的基因组序列数量有所增加[5,23-24],多的达210条[25],但序列大多来自一个国家或地区的一个寄主,难以从全球层面了解ASSVd的种群组成和遗传变异。本研究基于GenBank中来自不同国家和地區、不同寄主的ASSVd的基因组序列,进行了序列比对、变异分析和系统发育分析,明确了全球ASSVd的种群组成、主流变体、遗传变异的特征及规律,为ASSVd的防控和基础研究提供了重要参考。
1 材料与方法
1.1 材料
GenBank中登录的ASSVd全基因组序列共215条(截至2021年6月22日)。其中,MG546298未说明来源,只表明实验室寄主为动物细胞;KP772687和KP772685与其余序列的序列相似性低于70%,不符合类病毒种划分的标准[26],不是ASSVd的序列。去除这3条序列,剩余212条序列,其中参考序列为NC001340。
1.2 方法
1.2.1 序列比对及相似性分析
使用软件SDT 1.2比较两两序列之间的相似性[27]。虽然该软件不计算碱基的插入缺失对相似性的影响,但是使用DANMAN验证发现,ASSVd基因组中插入缺失的碱基数量少,不影响计算结果。
1.2.2 种群组成分析
为找出相同的序列,根据大小将212条序列分类,然后使用SDT 1.2计算各类序列相互之间的相似性。相同的序列记为同一种变体,主流变体用M1和M2表示,由此可得出212条序列的变体种类及每种变体包含的序列数量,即种群组成。
1.2.3 碱基变异分析
以参考序列NC001340为依据,使用软件Cluxtal X2比对这212条序列。利用软件BioEdit[28]分析碱基变异。为保证变异的真实性,只选择至少在两条序列中重复出现的变异。碱基的位置以参考序列为准。变异分析包括:1) 变异的种类及其数量。种类包括碱基替换(包括转换和颠换)和插入缺失,数量指在所有序列中出现的次数,或包含该变异的序列的数量;2)变异的频率,指包括该变异的序列的数量与所有序列数量的百分比;3)变异的选择,主要是ASSVd二级结构(见1.2.4)对变异的选择作用。将变异逐一标注在ASSVd的二级结构上,看其分布情况。
1.2.4 二级结构预测
根据最小自由能(ΔG)原理,使用RNAfold(http:∥www.unafold.org/mfold/applications/rna-folding-form.php)预测ASSVd参考基因组(NC001340)的二级结构。使用软件Rnaviz 2.0.3,并参考Giguere等通过SHAPE(selective 2′-hydroxyl acylation analyzed by primer extension)技术测定的ASSVd(来源于日本的M36646)的二级结构[29],对预测的结构进行编辑。
1.2.5 系统发育树构建
采用邻接法,使用软件MEGA X[30]构建系统发育树,并用自展法(重复1 000次)评估构建的系统发育树。使用最大似然法计算遗传距离。将与ASSVd同属的苹果凹果类病毒(apple dimple fruit viroid, ADFVd)的参考序列(NC003463)作为外类群。
2 结果与分析
2.1 ASSVd的基因组序列
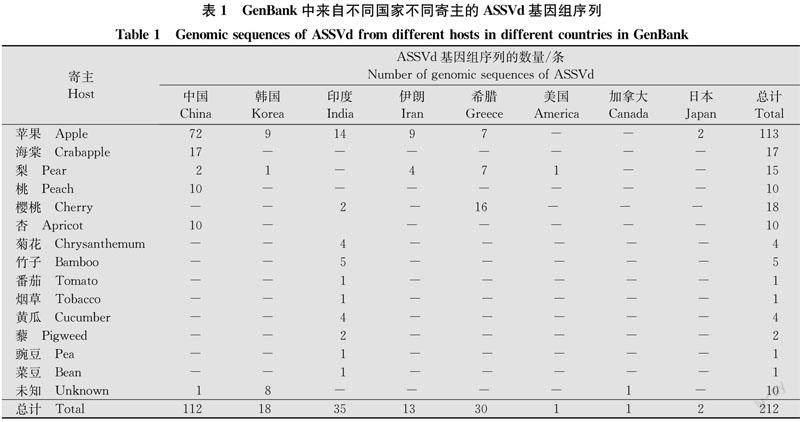
本研究使用的212条ASSVd基因组序列来自8个国家(表1)。中国的最多,为112条,随后依次为印度35条;希腊30条;韩国18条;伊朗13条;日本2条;美国与加拿大各1条。寄主主要是苹果(113条),随后依次为樱桃(18条)、海棠(17条)和梨(15条)。另外,寄主未知的有10条序列。
序列长度为315~340 nt,主要是330 nt和331 nt,数量分别为74条和66条,占总数的66.0%。329 nt和334 nt的序列数量较少,分别有23条和12条。其余大小的数目均不足10条。说明ASSVd的基因组大小较稳定。
2.2 ASSVd序列的相似性
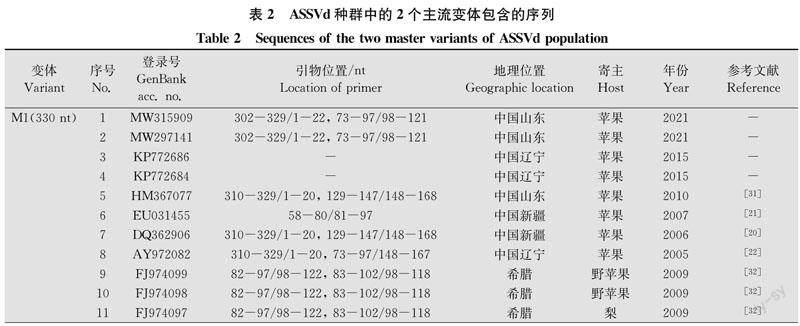
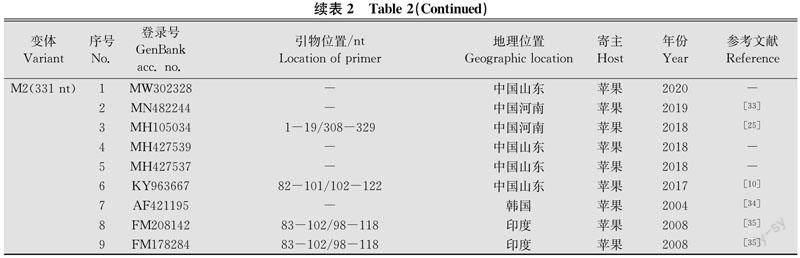
这212条序列两两之间的相似性绝大部分大于80%。进一步分析可知,这212条序列中,有些序列相同。如以MW315909为代表的11条序列,记为变体M1;以MW302328为代表的9条序列,记为变体M2(表2)。此外,分别比较不同国家的序列,发现同一国家的序列之间的相似性与不同国家的序列之间的相似性没有明显的差异。
2.3 全球ASSVd的种群结构
根据序列相似性分析结果,将相同序列归入同一种变体,共得到165种变体。由它们的频率(每种变体包含的序列数量占序列总数量的百分率)分布可知,全球ASSVd的种群结构符合准种模式。绝大部分变体(149种)的频率较低(0.47%),即只有1条序列;仅有16种变体的频率高于0.47%,即至少含有2条序列。频率最高的2种变体分别含有11条和9条序列,来自于中国、希腊、印度和韩国的苹果或梨(表2),是全球范围内流行的主流变体。
2.4 中国ASSVd的种群结构
这212条ASSVd序列有一多半(112条,占52.8%)来自中国。来自中国的序列包含93种变体,种群结构与全球ASSVd的种群结构类似。绝大多数变体(86种)的频率很低(0.89%),即只有1条序列;仅有7种变体的频率高于0.89%,即至少含有2条序列。频率最高的2种变体分别包含8条和6条序列,均来自苹果(表2),但来自不同地区,包括山东、辽宁、新疆和河南,是中国流行的主流变体。值得指出的是,这两种变体与全球流行的两种主流变体相同。
2.5 ASSVd种群中的变异
与164种变体相比,参考序列中有多半的位点发生了变异,包括碱基插入(13个位点),碱基缺失(35个位点)以及碱基替换(178个位点)(表3)。碱基转换的4种类型发生的位点数量无明显差异,为18~21。而碱基颠换的不同类型发生的位点数量差异较大,为7~18。CA颠换发生最多,TG颠换发生最少。表明ASSVd种群中的变异具有明显的偏好性。
计算ASSVd种群(212条序列)中不同变异发生的次数发现,碱基转换中的嘧啶间转换的次数(374次)稍多于嘌呤间转换的次数(339次)。碱基颠换中的GT颠换和AT颠换的次数较多,分别为236次和176次;而TG颠换和CG颠换的次数较少,分别只有29次和38次。进一步说明ASSVd种群中的变异具有碱基偏好性。
2.6 变异在ASSVd基因组上的分布及其频率
从图1可知,变异在ASSVd基因组上的分布不均匀。有的连续10多个位点均产生了变异,如40-55 nt和250-270 nt;也有连续多个位点严格保守,如180-193 nt。这应该是选择的结果。选择压力可能来自与寄主的相互作用或者来自基因组的二级结构(见2.7)。此外,不同变异的频率(种群中含有该变异的序列的数量所占的百分比)差异较大。多数变异的频率低于25%,即含有该变异的序列少于50条;而个别变异的频率较高,频率最高的两个变异(第125位和126位之间插入G,219位和220位之间插入U)的频率分别为97.2%和57.5%,即含有这两个变异的序列分别有206条和122条(图1)。
含有上述频率最高的两个变异的序列,除来自苹果外,还有桃、杏、海棠和樱桃等。说明这两个变异具有广泛的寄主适应性。此外,有些变异表现出寄主特异性。如第2位点的G变为U和第173位点的A变为G的变异只出现在梨树的序列中,第55位点的A变为G、第145及146位点间插入C以及第196位点的C变为U的突变仅出现在樱桃的序列中。
2.7 二级结构对变异的选择
ASSVd基因组(参考序列NC001340)的二级结构整体上呈棒状(图2),左末端的上、下两侧各有一个凸出的小发夹。由双链结构(连续的碱基对)和环状(上下两侧均有未配对碱基)或凸起(仅上侧或下侧有未配对碱基)结构相互间隔构成。基因组中的一多半碱基(共220个,占66.9%)互补配对。对变异在二级结构上的分布进行了分析,发现双链结构对变异具有明显的选择作用。首先,雙链结构中变异发生的频率明显低于环状结构。互补配对的碱基发生变异的次数为1 326次,未配对碱基发生变异的次数为1 156次,各自除以相应的碱基数目(分别为220个和109个)可知,互补配对碱基发生变异的频率为6.02次/碱基,明显低于未配对碱基(10.61次/碱基)。其次,保持碱基互补配对的变异更易于被保留下来。例如第167位碱基A,虽然可变为G、U或C中的任意一个,但只有变为G才不改变与第156位U的配对。实际上,A变为G的频率最高,为14.7%(31/211),A变为U和A变为C的频率分别只有3.3%(7/211)和1.4%(3/211)。类似的变异也发生在第43、48、61、74、244、264、277等位置。此外,存在共突变。如第32位的C变为U,与此同时,与其配对的第293位的G变为了A。这一共突变存在于9条序列中(HQ326087, HQ326088, HQ326090-HQ326092, HQ326094, HQ326095, HQ326098, KM213397)。第159位(G变为U)和第163位(C变为A)的共突变出现于另外4条序列(FJ974100, FJ974073, FJ974091, FJ974092)中。
除碱基互补配对的双链结构外,二级结构中的一些结构功能区对变异也有选择作用。从图2可知,二级结构上存在4个变异较少的区域,即保守区。其中,两个是已知的,分别为末端保守区(terminal conserved region, TCR)和中央保守区(central conserved region, CCR),它们在类病毒复制过程中具有重要功能;另外两个是新发现的,在106-119位点处和179-198位点处,分别命名为可变保守区(variable conserved region, VCR)和右端保守区(right conserved region, RCR)。
2.8 ASSVd的系统发育分析
构建由165种ASSVd变体的序列以及外类群ADFVd组成的系统发育树,其中,包括两种主流变体和参考序列(NC001340)在内的77条序列聚在一起,且所在分支的自展值低于60%。为便于呈现结果,将这77条序列所在分支合并为一个分支,以黑色圆点表示(图3)。其余的88种变体中,有些来自某一个国家或某一种寄主的变体聚在一起,表现出明显的地理特异性和寄主特异性。例如从中国的桃和杏分离的变体。此外,也有来自不同国家或寄主的变体聚在一起,如从印度苹果上分离的变体(AM993160)与中国杏树上分离的变体聚在一起,从中国苹果或海棠上分离的变体与从伊朗的梨或苹果上的分离的变体也聚在一起。说明存在着地区间以及寄主间的传播。
3 结论与讨论
本研究通过分析GenBank中来自于8个国家的212条ASSVd基因组序列,明确了全球ASSVd种群的组成结构以及遗传变异的特点和规律。种群符合准种模式[36],由165种序列高度相似但不相同的变体组成,有两个主流变体。种群中的变异明显受到了选择,因为无论是碱基变异的类型,还是变异在基因组及二级结构上的分布,均具有明显的偏好性。
以我国苹果上的两条序列MW315909和MW302328为代表的两种变体,是我国种群以及全球种群的主流变体。其发生分布广泛,遍布于不同国家,包括中国、韩国、印度和希腊,以及中国的不同产区。需要指出的是,这两种变体并非是所有自然种群的主流变体。例如,李紫腾等[25]从7个不同品种的苹果上获得17种变体,除MH105034外,都不是这两种主流变体。遗传漂变、传播过程中的瓶颈效应以及寄主的选择等均会影响自然种群的组成。尽管如此,严密监测这两个主流变体的发生和分布,可快速掌握ASSVd的流行和传播。
这两个主流变体与参考序列(NC001340)[19]并不相同。与参考序列相比,均在第125位和126位之间插入了G;此外,一种主流变体在第219位和220位之间插入了U,249位置的G变为了U。其中,G和U插入的频率很高,出现在种群中的大多数变体中。需要指出的是,这两个变异并非最近才出现,因为与参考序列同时期被测定的日本分离物的序列Y00435和M36646[37]以及加拿大分离物的序列X71599[18],在第125位和126位之间也插入了G,而日本的两条序列在第219位和220位之间也插入了U。尽管如此,早期测定的序列在近几年出现的频率很低,只分离到与加拿大分离物相同的3条序列MN598208[38],MH105028和MH105020[25],且均非主流变体。说明一些变异的频率是动态变化的,而这种变化可能与ASSVd对环境或寄主的适应性有关。
虽然朱慧[38],查富蓉[39],陈冉冉等[40]和李紫腾等[25]的研究已发现了很多变异,但大多位于基因组二级结构的左末端区、致病区和中央区。除上述3个区,我们在可变区和右末端区也发现了很多变异。
一方面是因为我们使用了更多的序列,尤其是国外的序列;再者是因为所用的参考序列不同,如查富蓉[39]和李紫腾等[25]以分离物中的主流序列或共有序列作为参考,我们则使用ASSVd的参考序列。
变异分析发现了两个新保守区(VCR和RCR),分别位于可变区以及可变区与右末端区的交界处。如果不是RNA聚合酶的保真性在这两个区域具有偏好性[41],那么就意味着这两个区域可能具有重要功能,比如与类病毒的重组[42-44]或移动[45]有关。这两个新保守区的发现为研究ASSVd与寄主的相互作用提供了线索,也为设计能够涵盖更多变体的RT-PCR/qPCR检测技术提供了参考。
参考文献
[1] 王树桐, 王亚南, 曹克强. 近年我国重要苹果病害发生概况及研究进展[J].植物保护, 2018, 44(5): 13-25.
[2] 王钰婷, 金珠, 董芳园, 等. 新疆梨树上苹果锈果类病毒的检测与全序列分析[J]. 新疆农业科学, 2013, 50(1): 94-99.
[3] 赵英, 牛建新. 新疆杏树苹果锈果类病毒的檢测与全序列分析[J].园艺学报, 2008, 35(6): 805-810.
[4] 赵英, 牛建新. 新疆桃树上苹果锈果类病毒(ASSVd)的检测与全序列分析[J]. 果树学报, 2008, 25(2): 274-276.
[5] WALIA Y, DHIR S, BHADORIA S, et al. Molecular characterization of apple scar skin viroid from Himalayan wild cherry [J]. Forest Pathology, 2012, 42(1): 84-87.
[6] 郝璐, 叶婷, 陈善义, 等. 我国北方部分苹果主产区病毒病的发生与检测[J]. 植物保护, 2015, 41(2): 158-161.
[7] 郗娜娜, 赵珂, 杨金凤, 等. 苹果花脸病田间病情发展及ASSVd在组培条件下的传播[J]. 园艺学报, 2020, 47(12): 2397-2404.
[8] 张富军, 张振鲁, 张蕊芬, 等. 苹果锈果类病毒火焰海棠分离物全基因组克隆及序列分析[J]. 果树学报, 2019, 36(8): 1005-1012.
[9] 刘洪玉, 孙子豪, 李保华, 等. 侵染‘舞美苹果的苹果锈果类病毒检测与全序列分析[J]. 植物保护, 2019, 45(4): 176-179.
[10]赵玲玲, 刘娟, 宋来庆, 等. 烟台市富士苹果上苹果锈果类病毒分子变异分析[J]. 植物保护学报, 2018, 45(4): 856-863.
[11]吴宽, 赵磊, 孙丽英, 等. 苹果锈果类病毒陕西秦冠分离物的鉴定与序列分析[J]. 植物保护学报, 2016, 43(5): 877-878.
[12]于青, 刘美英, 宋来庆, 等. 烟台市苹果病毒病的发生与防治[J]. 山东农业科学, 2009(9): 54-56.
[13]苏佳明, 段小娜, 于强, 等. 烟台市主要果树病毒调查与检测鉴定初报[J]. 山东农业科学, 2010(6): 86-88.
[14]LOPEZ-CARRASCO A, BALLESTEROS C, SENTANDREU V, et al. Different rates of spontaneous mutation of chloroplastic and nuclear viroids as determined by high-fidelity ultra-deep sequencing [J/OL]. PLoS Pathogens, 2017, 13(9): e1006547. DOI: 10.1371/journal.ppat.1006547.
[15]吴然, 李君英, 邵建柱, 等. 苹果锈果类病毒实时荧光PCR检测方法的建立[J]. 果树学报, 2015, 32(1): 150-155.
[16]郗娜娜, 李紫腾, 张静怡, 等. 苹果锈果类病毒实时荧光定量反转录PCR检测及其在苹果树体内的扩散转移规律[J]. 植物保护学报, 2020, 47(6): 1304-1312.
[17]HEO S, CHUNG Y S. Rapid real-time detection method of ACLSV and ASSVd for apple quarantine field [J]. Plant Biotechnology Reports, 2021, 15(2): 187-195.
[18]ZHU Shuifang, HADIDI A, YANG Xicai, et al. Nucleotide sequence and secondary structure of pome fruit viroids from dapple apple diseased apples, pear rusty skin diseased pears and apple scar skin symptomless pears [J]. Acta Horticulturae, 1995, 386: 554-559.
[19]PUCHTA H, LUCKINGER R, YANG Xicai, et al. Nucleotide-sequence and secondary structure of apple scar skin viroid (ASSVd) from China [J]. Plant Molecular Biology, 1990, 14(6): 1065-1067.
[20]郭瑞, 李峰, 李明福, 等. 苹果锈果类病毒新疆分离物的核苷酸序列分析[J]. 农业生物技术学报, 2007, 15(6): 1076-1077.
[21]赵英, 牛建新. 苹果锈果类病毒新疆分离物的克隆和序列分析[J]. 果树学报, 2006, 23(6): 896-898.
[22]郭瑞, 李世访, 董雅凤, 等. 苹果锈果类病毒辽宁分离物的克隆与序列分析[J]. 植物病理学报, 2005, 35(5): 472-474.
[23]KAPONI M S, SANO T, KYRIAKOPOULOU P E. Natural infection of sweet cherry trees with apple scar skin viroid [J]. Journal of Plant Pathology, 2013, 95(2): 429-433.
[24]SHAMLOUL A M, YANG Xicai, HAN Lijuan, et al. Characterization of a new variant of apple scar skin viroid associated with pear fruit crinkle disease [J]. Journal of Plant Pathology, 2004, 86(3): 249-256.
[25]李紫腾, 曹钰晗, 李楠, 等. 苹果锈果类病毒在7个品种苹果上的分子变异及系统发育关系[J]. 中国农业科学, 2021, 54(20): 4326-4336.
[26]DI SERIO F, OWENS R A, LI Shifang, et al. ICTV virus taxonomy profile: Pospiviroidae [J]. Journal of General Virology, 2021, 102(2): 1-2.
[27]MUHIRE B M, VARSANI A, MARTIN D P, et al. SDT: A virus classification tool based on pairwise sequence alignment and identity calculation [J/OL]. PLoS ONE, 2014, 9(9): e108277. DOI: 10.1371/journal.pone.0108277.
[28]ALZOHAIRY A M. BioEdit: An important software for molecular biology [J]. GERF Bulletin of Biosciences, 2011, 2(1): 60-61.
[29]GIGUERE T, ADKAR-PURNSHOTHAMA C R, PERREAULT J P, et al. Comprehensive secondary structure elucidation of four genera of the family Pospiviroidae [J/OL]. PLoS ONE, 2014, 9(6): e98655. DOI: 10.1371/journal.pone.0098655.
[30]KUMAR S, STERCHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms [J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549.
[31]馬伟, 姜冬梅, 陈丽芬, 等. 苹果锈果类病毒山东栖霞分离物的分子鉴定及序列分析[J]. 植物保护, 2011, 37(2): 91-94.
[32]KAPONI M, LUIGI M, KYRIAKOPOULOU P E. Mixed infections of pome and stone fruit viroids in cultivated and wild trees in Greece [J/OL], New Disease Reports, 2012, 26:8. DOI: 10.5197/j.2044-0588.2012.026.008.
[33]JIAO Jiao, KONG Kangkang, HAN Jinmeng, et al. Field detection of multiple RNA viruses/viroids in apple using a CRISPR/Cas12a-based visual assay [J]. Plant Biotechnology Journal, 2021(19): 394-405.
[34]LEE J H, PARK J K, LEE D H, et al. Occurrence of apple scar skin viroid-Korean strain (ASSVd-K) in apples cultivated in Korea [J]. The Plant Pathology Journal, 2001, 17(5): 300-304.
[35]WALIA Y, KUMAR Y, RANA T, et al. Molecular characterization and variability analysis of apple scar skin viroid in India [J]. Journal of General Plant Pathology, 2009, 75(4): 307-311.
[36]DOMINGO E, SHELDON J, PERALES C. Viral quasispecies evolution [J]. Microbiology and Molecular Biology, 2012, 76(2): 159-216.
[37]HASHIMOTO J, KOGANEZAWA H. Nucleotide-sequence and secondary structure of apple scar skin viroid [J]. Nucleic Acids Research, 1987, 15(17): 7045-7052.
[38]朱慧. 来源梨的苹果褪绿叶斑病毒和两种类病毒的分子特性研究[D]. 武汉: 华中农业大学, 2014.
[39]查富蓉. 侵染苹果的类病毒与果实症状间的相关性及分子特性研究[D].武汉: 华中农业大学, 2015.
[40]陈冉冉, 谢吉鹏, 叶婷, 等. 我国部分苹果产区苹果锈果类病毒的检测和全序列分析[J]. 植物保护, 2017, 43(6): 97-102.
[41]WU Jian, BISARO D M. Biased Pol Ⅱ fidelity contributes to conservation of functional domains in the potato spindle tuber viroid genome [J/OL]. PLoS Pathogens, 2020, 16(12): e1009144. DOI: 10.1371/journal.ppat.1009144.
[42]KEESE P, SYMONS R H. Domains in viroids: evidence of intermolecular RNA rearrangements and their contribution to viroid evolution [J]. Proceedings of the National Academy of Sciences of the United States of America, 1985, 82(14): 4582-4586.
[43]SEMANCIK J S, SZYCHOWSKI J A, RAKOWSKI A G, et al. A stable 463-nucleotide variant of citrus exocortis viroid produced by terminal repeats [J]. Journal of General Virology, 1994, 75(4): 727-732.
[44]HASELOFF J, MOHAMED N A, SYMONS R H. Viroid RNAs of cadang-cadang disease of coconuts [J]. Nature, 1982, 299(5881): 316-321.
[45]GOZMANOVA M, DENTI M A, MINKOV I N, et al. Characterization of the RNA motif responsible for the specific interaction of potato spindle tuber viroid RNA (PSTVd) and the tomato protein Virp1[J]. Nucleic Acids Research, 2003, 31(19): 5534-5543.
(責任编辑:杨明丽)

