机器学习方法用于建立乙酰胆碱酯酶抑制剂的分类模型
杨国兵 李泽荣 饶含兵 李象远 陈宇综
(1四川大学化学学院,成都 610064; 2四川大学化学工程学院,成都 610065; 3Department of Pharmacy,National University of Singapore,Singapore 117543)
机器学习方法用于建立乙酰胆碱酯酶抑制剂的分类模型
杨国兵2李泽荣1,∗饶含兵1李象远2陈宇综3
(1四川大学化学学院,成都 610064;2四川大学化学工程学院,成都 610065;3Department of Pharmacy,National University of Singapore,Singapore 117543)
我们构建了表征乙酰胆碱酯酶抑制剂分子组成、电荷、拓扑、几何结构及物理化学性质等特征的1559个描述符,通过Fischer Score排序过滤和Monte Carlo模拟退火法相结合进行变量筛选得到37个描述符,然后分别用支持向量学习机(SVM)、人工神经网络(ANN)和k⁃近邻(k⁃NN)等机器学习方法建立了乙酰胆碱酯酶抑制剂的分类预测模型.对于训练集的515个样本,通过五重交叉验证,各机器学习方法对正样本,负样本和总样本的平均预测精度分别为87.3%-92.7%,67.0%-81.0%和79.4%-88.2%;通过y⁃scrambling方法验证SVM模型是否偶然相关,结果正样本,负样本和总样本的平均预测精度分别为72.7%-82.5%,41.0%-53.0%和62.1%-69.1%,明显低于实际所建模型的预测精度,表明所建模型不存在偶然相关;对172个没有参与建模的外部独立测试样本,各机器学习方法对正样本,负样本和总样本的预测精度分别为93.3%-100.0%, 74.6%-89.6%和86.1%-95.9%.所建模型中,SVM模型预测精度最好,且明显高于其它文献报道结果.
乙酰胆碱酯酶抑制剂;机器学习方法;变量筛选;应用域
阿尔茨海默病(Alzheimer disease,AD)即老年性痴呆症,是慢性进行性神经退化疾病,严重影响患者的认知功能、记忆功能、个人生活能力和情感人格等,65岁以上人群中大约10%患有此病.一直以来,世界上较为接受的AD病理为“胆碱能缺失学说”,学说认为老年性痴呆症患者大脑内神经递质——乙酰胆碱的缺失是导致AD疾病的关键原因[1-2].乙酰胆碱酯酶(AChE)会催化乙酰胆碱的裂解反应,导致乙酰胆碱的缺失、神经信号传递失败.目前对老年痴呆的药物治疗主要是通过用乙酰胆碱酯酶抑制剂(AChEIs)来减少乙酰胆碱的消耗,从而提高胆碱能神经元的兴奋性,达到治疗的效果[3].所以乙酰胆碱酯酶抑制剂作为主要治疗手段已经被广泛研究.1993年,第一个乙酰胆碱酯酶抑制剂他克林(tacrine)用于临床治疗AD[4],此后,越来越多的AChEIs被开发出来,比如多奈哌齐(donepezil)[5],加兰他敏(galantamine)[6],利凡斯的明(rivastigmine)[7].
事实上,实验方法开发AChEIs是比较盲目的,因此用计算的方法在实验之前先进行针对性的筛选AChEIs是必要的,可以为实验提供理论指导,挑选出一些有用的分子供实验分析,这样减少了药物开发的盲目性,大大节省了开发的时间和资金.到目前为止,人们已经开发出了许多计算方法或模型去预测AChEIs,比如定量构效关系(QSAR)[8-11],分子对接(docking)[12-14].最近,Lv等[15]把三种机器学习方法SVM,k⁃NN和决策树(DT)用于建立AChEIs分类模型,对抑制剂的预测精度在76.3%-88.0%,非抑制剂的预测精度在74.3%-79.6%,但是他们用的数据不够多,预测结果也不够理想.为此,本文着力于用机器学习方法(SVM[16],ANN[17],k⁃NN[18])使用更多的样本去开发预测精度更高的模型,为筛选出有用的AChEIs提供更有效的手段.
1 原理与方法
1.1 数据收集
本文收集的717个抑制剂化合物分子(抑制剂的基本结构单元见Scheme 1)及它们的实验半数抑制浓度IC50主要来源于参考文献[12,19-55].同文献[15]一样,我们将IC50≤400 nmol·L-1的化合物划分为抑制剂(正样本),将IC50≥600 nmol·L-1的化合物划分为非抑制剂(负样本).这样,717个分子包含了420个抑制剂和267个非抑制剂,此外,还有30个化合物的IC50在400和600 nmol·L-1之间,和文献[15]一样,这里也不把他们归入抑制剂或非抑制剂,不对它们进行研究.本文所研究分子的稳定几何结构均用HyperChem 7.0(http://www.hyper.com/)中的MM+力场优化得到.
1.2 数据集的结构多样性
数据集中化合物的结构多样性可以通过多样性指标(DI)来评定,DI值是指数据集中成对化合物间的不相似性[56]:
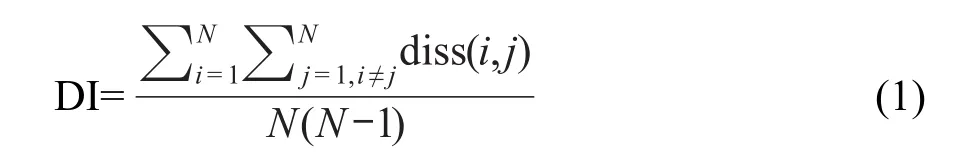
其中N是数据集中化合物的个数,diss(i,j)是化合物i和j之间不相似度的度量.因为不相似度和相似度是互补的,所以经常定义为:
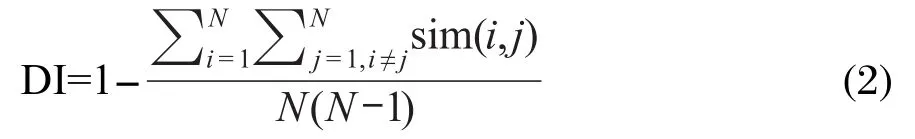
其中,sim(i,j)是化合物i和j之间的相似度的度量,本文中,相似度采用常用的Tanimoto系数[57],定义如下:
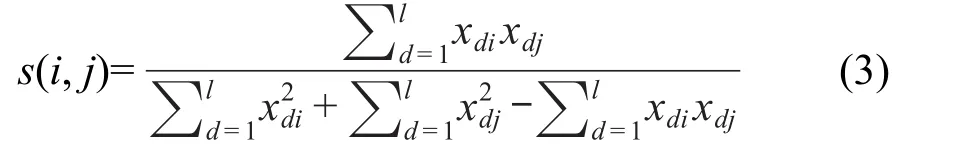
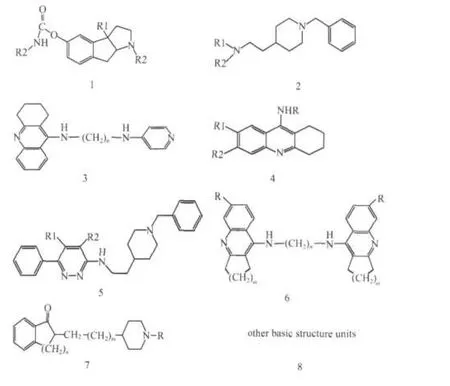
Scheme 1 Basic structure units ofacetylcholinesterase inhibitors
其中l是描述符的个数,xdi和xdj分别是化合物i和j的第d个描述符,s(i,j)为Tanimoto系数.由计算可知连续的Tanimoto系数的变化范围为(-0.333,1.0),所以将Tanimoto系数归一化,使其范围为(0,1.0).归一化的过程通过下式实现:
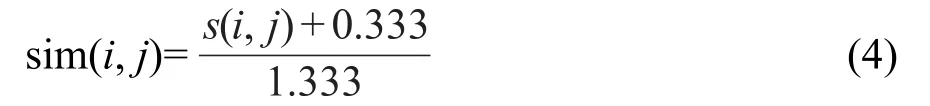
这样代入式(2)得到的DI值的变化范围为(0,1.0),数据集化合物的结构多样性随着DI值的增大而增加.
本文研究的687个分子的DI值为0.738,表明所研究对象具有足够的结构多样性.
1.3 分子描述符的计算及筛选
分子描述符是构建模型的关键,本研究我们采用MODEL软件[58]进行描述符的计算,此软件在网址(http://jing.cz3.nus.edu.sg/cgi⁃bin/model/model.cgi)可寻,是免费的学术软件.我们对每个分子计算了1559个描述符,其中包含20个组成描述符、21个电荷相关描述符、2个物理化学性质描述符、482个拓扑描述符和1034个几何描述符.
很显然,并不是所有1559个描述符都与分子的活性相关,而且有些描述符之间存在线性相关,所以需要选择合适的描述符以使建立的模型达到最好的预测能力.本文采用一套混合描述符筛选方法来寻找最优的描述符子集,其步骤如下.
(1)预处理:首先,如果有90%样本的同一个描述符值是相同的,那么这个描述符对区别分子的活性无效,将它去除;其次,对任意一个描述符,遍及所有样本计算该描述符的相对标准偏差,如果其值小于0.05,也将它删除;最后,如果两个描述符的Pearson相关系数[59]大于0.9,说明这两个描述符高度相关,删除其中之一.
(2)描述符排序:经过预处理后的描述符,按照Fischer Score(F⁃Score)[60]值递减的顺序排序.F⁃Score值F(i)是单个描述符的分辨能力的简单度量,定义为:

其中μ+i和μ-i分别是第i个描述符的正、负样本的平均值,σ+i和σ-i分别是它们的标准偏差.描述符的F⁃Score值越大,它对样本的区分能力就越强.具体的筛选步骤如下:①计算每一个描述符的F⁃Score值,并将所有描述符按照F⁃Score值从大到小的顺序排列;②把训练集用五重交叉验证的方法得到SVM模型的平均推广误差,并调节模型参数σ使推广误差最小;③删除排序靠后5个描述符,回到第②步,直至模型的推广误差最小.由此,可选择出F⁃Score值最好的描述符子集及对应的模型参数σ.
(3)Monte Carlo模拟退火算法:描述符在经过上述两个步骤过滤后,进一步用Monte Carlo模拟退火法结合SVM进行筛选,文献[61-62]有类似描述.首先我们给定一较高的模拟退火起始温度T0,以及每一温度的平衡步数和终止温度,然后具体的筛选步骤如下:①给定SVM高斯指数的初值σ;②在初始温度T0下随机选择一组描述符子集作为初始解;③用五重交叉验证计算SVM模型的平均推广误差Eold;④对描述符子集给一随机微扰,得到一新描述符子集,按上述方法计算模型的平均推广误差Enew;⑤如Enew<Eold,则接受新的描述符子集代替原来的描述符子集.反之,则以概率P=exp{-(Enew-Eold)/ T}接受为新描述符子集;⑥回到步骤④,直到达到此温度下的最大平衡步数;⑦降低模拟退火温度T,重复上步温度的筛选过程,直到达到指定的终止退火温度;⑧系统地调整σ值,返回步骤②,直至SVM模型的平均推广误差最小.由此,可得到最优的描述符子集和对应的最佳模型参数σ.
1.4 数据集的划分和模型的验证
本研究采用较流行的KS方法[63]来设计训练集和外部独立测试集.所构成的训练集包含315个正样本和200个负样本,外部独立测试集包含了105个正样本和67个负样本.训练集用于模型的构建,外部独立测试集用于客观评价已建模型的预测能力.
本文中我们用三种方法去验证分类模型.第一种是五重交叉验证,把训练集随机分为5个数量接近的子集,每次用其中4个作为训练集,另外一个作为测试集,这样重复5次,保证每个子集都用作测试集一次.在此文中,我们用五重交叉验证去优化预测模型:选出最优的描述符子集,优化模型参数如SVM中的高斯指数σ,ANN中隐含层的个数,k⁃NN中的k.第二种验证方法是y⁃scrambling,用来评估所建模型是否偶然相关.保持训练集样本输入变量不变,随机将部分样本的分类标号打乱,用打乱类别的训练集建立新的分类模型,新模型的预测能力应该大大低于基于原始数据建立的实际模型[64],否则就是偶然相关,偶然相关的模型没有预测能力.第三种验证方法是外部独立测试[65],把训练集中所有样本基于选出的最优描述符和最佳参数建立预测模型,用外部独立测试集来评价已建模型的预测能力.因为外部独立测试集中的样本没有参与模型的开发,所以这种验证方法比交叉验证更为严谨,更能客观评价模型的预测能力.
1.5 机器学习方法
1.5.1 SVM方法
SVM方法是基于结构风险最小化准则,即通过对推广误差风险最小化,从而实现模型的最大推广能力.该方法在文献[16]中有详细介绍,这里我们只做简述.
给定一训练集{xi,yi},i=1,2,…,n,yi∈{-1,+1},其中n是训练集样本的个数,yi∈{-1,+1}是类别标号.如果训练集是线性可分的,SVM就是构建超平面

使正样本和负样本可分,且使其边界上的点到该超平面的距离最大,这可转化为下面的条件

限制下求函数
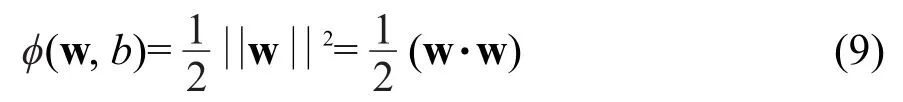
的最小值.由Lagrange乘数法可得解

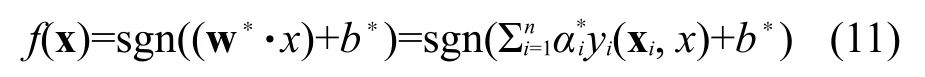
其中,sgn()为符号函数.如果训练集是非线性可分的,则将向x通过非线性函数ϕ(x)投影到某个高维特征空间,使其线性可分.非线性函数ϕ(x)的引入是通过核函数K(xi,x)来实现的,即K(xi,x)=(ϕ(xi)· ϕ(x)).此时的分类判别函数变为:
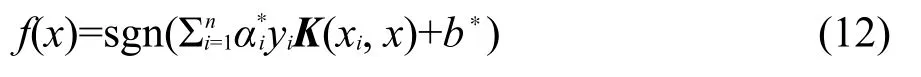
核函数K(xi,x)可以有多种形式,本文使用Gaussian核函数:
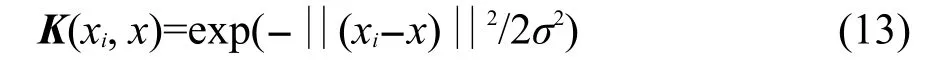
式中高斯指数σ称为模型参数.
1.5.2 其他机器学习方法
此外,还分别将三层(包括输入层、隐含层和输出层)反向传播ANN[17]和k⁃NN[18]方法用于建立乙酰胆碱酯酶抑制剂的分类预测模型.各机器学习方法的模型参数,即SVM中的高斯指数σ,ANN中隐含层的个数,k⁃NN中的k,均是通过五重交叉验证使模型的推广误差最小来优化.上述方法均采用本小组自编的程序来实现.
1.6 结果评价
我们采用普遍使用的方法来评估分类模型对测试集的预测能力[66]:
灵敏性(sensitivity,SE):
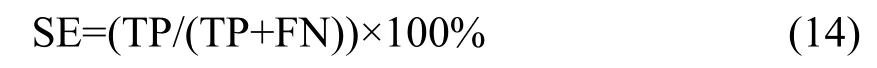
特效性(specificity,SP):
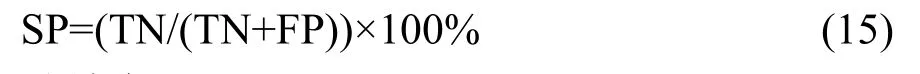
总精度(accuracy,Q):
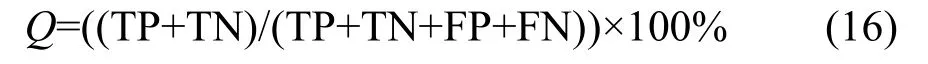
Matthews相关系数(MCC)[67]:
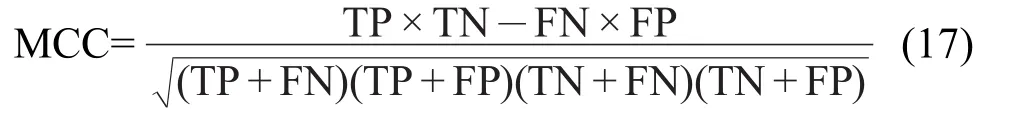
其中TP代表预测正确的正样本数,TN代表预测正确的负样本数,FP代表预测错误的负样本数,FN代表预测错误的正样本数.模型的敏感度分析SE用于评估正样本的预测正确率,特效性分析SP用于评估负样本的预测正确率.Q用于评估总体样本的预测正确率.
2 结果和讨论
2.1 描述符筛选对SVM预测性能影响
本研究中我们用SVM方法执行描述符变量的筛选,筛选结果列于表1.最初每个训练集分子的描述符数目为1559个,经过预处理后,描述符的数目减少到564个,说明在最初的1559个变量中存在大量信息含量低或与其它描述符高度相关的描述符,经过预处理可以去掉.用这564个描述符通过五重交叉验证方法建立SVM的分类预测模型,正样本,负样本和总样本的平均预测正确率分别是84.4%, 72.0%和79.6%,MCC为0.573.进一步用F⁃Score值递减排序筛选后,描述符数量减少到295个,通过五重交叉验证建立SVM模型,正样本,负样本和总样本的平均预测正确率分别是87.9%,70.5%和81.2%,MCC为0.602.对比两组数据可知,F⁃Score排序步骤虽然没有明显提高模型的预测精度,但大大减少了描述符的数量,有利于提高模型的计算效率.由于描述符的个数还很多,故进一步用Monte Carlo模拟退火法进行筛选,最终得到37个描述符,此时SVM分类预测模型对正样本,负样本和总样本的平均预测正确率分别为92.7%,81.0%和88.2%, MCC为0.749.由此可见,模拟退火法在大量减少描述符数量的同时能够大大提高模型的预测精度,说明干扰变量和噪音变量已经被排除,得到了与分子活性相关性很强的描述符.最初的1559个描述符及筛选出的37个描述符列于Supporting Information中的表S1和S2.
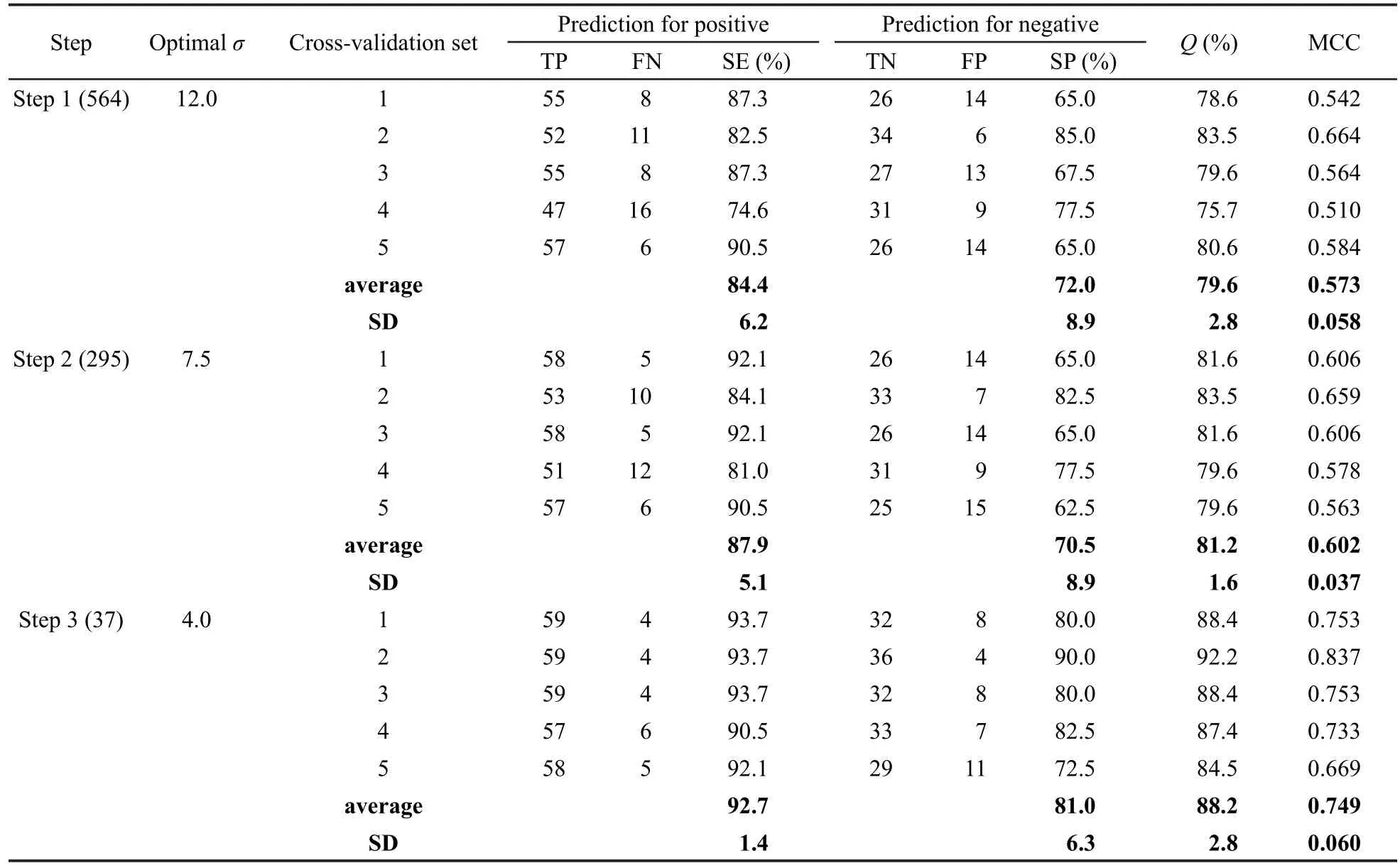
表1 变量筛选对SVM预测精度的影响Table 1 Effect of feature selection on the performance of SVM
2.2 SVM模型y⁃scrambling验证
为了评估所建模型对偶然相关的依赖程度,本文采用y⁃scrambling方法对SVM所建的模型进行验证.首先将训练集样本中的输入变量x保持不变,随机将部分样本对应的分类标号y改变类别,使这些样本的输入变量和类别号不再对应,以消除两者之间存在的内在定量关系;随后,针对上述改变类别的训练集,采用本文的混合描述符筛选方法结合SVM通过五重交叉验证建立新的预测模型;重复上述过程30次,并将其预测结果与基于原始样本数据建立的实际预测模型进行比较,其预测结果见Sup⁃porting Information中的表S3.
从附表S3可以看出,通过y⁃scrambling方法验证SVM模型,正样本,负样本和总样本的平均预测精度分别在72.7%-82.5%,41.0%-53.0%和62.1%-62.1%-69.1%,很明显,预测精度远低于基于原始样本数据建立的实际预测模型.这说明我们构建的SVM模型不存在偶然相关性.
2.3 其他机器学习方法
同时,为了检验所选描述符能否真正区别开不同类别的样本,我们把SVM方法筛选出的37个描述符直接用于建立ANN和k⁃NN的分类模型,结果见表2.从表中可知,机器学习方法对正样本、负样本、总样本的平均预测精度和MCC分别在87.3%-92.7%、67.0%-81.0%、79.4%-88.2%和0.561-0.749,SVM模型给出了最好的预测结果:SE= 92.7%,SP=81.0%,Q=88.2%,MCC=0.749.上面的结果表明我们所选择的描述符也能用于其它机器学习方法建立分类模型并且可以得到良好的预测结果,所选的描述符是与乙酰胆碱酯酶抑制剂活性真正相关的.
2.4 外部独立测试集
把所有的训练集化合物用筛选出来的最优描述符子集和最优参数建立模型,然后用外部独立测试集来评价模型.在讨论外部测试之前,先讨论模型的应用域.
机器学习方法所建立的模型是基于特定的训练集而产生的,所以必定有其应用的范围,只有确定模型的应用范围,我们才知道所建模型对未知事物预测的可信度.模型的应用域就是用来讨论模型应用范围的.本论文中关于应用域的研究主要采用描述符空间(即训练集化合物所覆盖的描述符空间的组合,也称之为描述符域[68])的方法,从建立模型所使用的训练集和描述符出发进行研究.本文根据欧氏距离的方法来表征描述符域,Tropsha等[69]定义了应用域距离的阀值DT:

其中Z为阈值参数,〈D〉和s是训练集中所有化合物间欧氏距离平均值和标准差.我们根据类似的方法定义应用域ST为:

〈S〉和σ是训练集中所有化合物的归一化Tanimoto相似性系数和标准差,Z为任意的阈值参数,目的是调控得到最大的应用域,我们设置Z缺省值为0.5.如果用来测试的化合物的Tanimoto相似性系数低于ST,则该化合物被认为是超出了应用领域,此时预测是不可靠的.
把筛选后37个描述符用于训练集的计算,然后对所得的分子描述符数据进行标准化处理,即对每个变量先减去样本平均值,再除以样本方差,从而消除不同量纲对变量的影响,然后通过主成分分析(principal component analysis,PCA)消除多余的信息,并选取信息含量的加和大于90%的主成分(17个)用于样本间相似性系数的计算.对于训练集,经过归一化的Tanimoto系数平均值为0.778,标准偏差为0.106,由式(19)计算出ST为0.725.对于172个独立的测试集分子,严格地说只有9个化合物没有在应用域范围内,它们距离训练集样本最近分子的Tanimoto系数值分别为:0.656,0.682,0.720,0.680, 0.661,0.696,0.687,0.634和0.711.尽管这些Tanimoto系数低于ST值(0.725),但是彼此很接近,所以我们认为这9个分子也在所建模型的应用域范围内.
表3列出了模型对独立测试集的预测结果,可以看到正样本,负样本,总样本的预测精度和MCC分别在93.3%-100.0%,74.6%-89.6%,86.1%-95.9%和0.704-0.916.结果表明机器学习方法所建模型对外部独立测试分子取得了良好的预测结果,所建模
型不存在过拟合的现象,同时也可以看出基于SVM方法所建模型的预测精度要明显好于基于k⁃NN和ANN建立的模型.
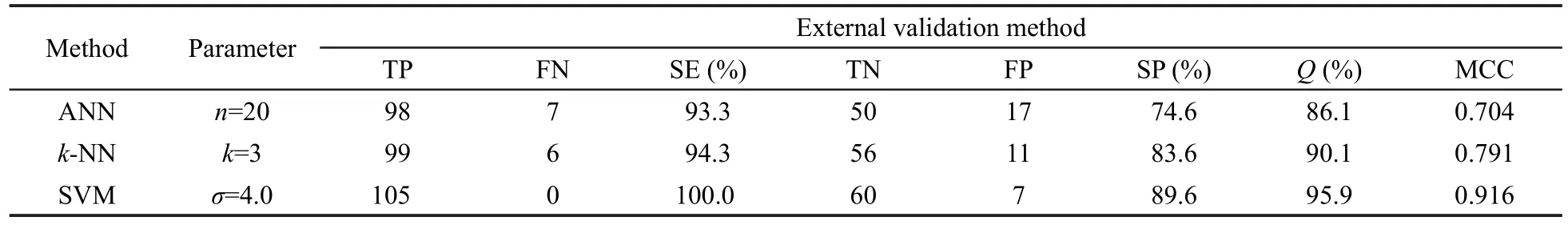
表3 3种机器学习方法对外部独立测试集预测结果的比较Table 3 Comparing predicted results based on three machine learning methods using independent validation sets
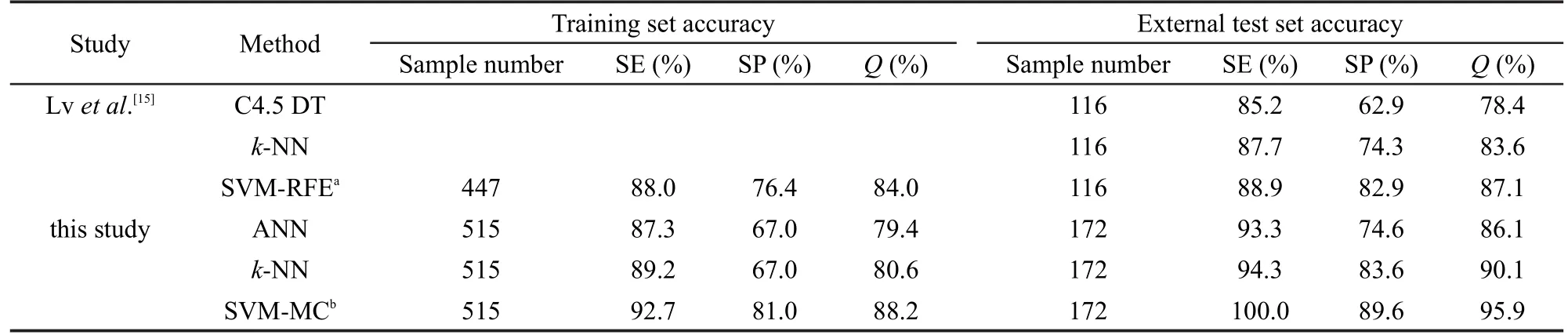
表4 本文与文献[15]预测精度的比较Table 4 Comparison of the prediction accuracies of our models with literature[15]
2.5 文献结果比较
将我们建立的模型的预测结果与文献[15]预测的结果进行比较,以便检验我们模型的预测水平.但是需要指出的是直接和不同模型的预测结果相比较不太合理,因为不同的模型采用了不同的机器学习方法、不同的训练集和测试集样本、不同的分子描述符和不同的验证方法.但是可以进行尝试性的比较,以便对乙酰胆碱酯酶抑制剂的预测精度进行大概的评估.本研究主要与Lv等[15]建立的模型进行比较,他们也使用了三种机器学习方法建立了分类模型并取得了良好的预测结果.比较结果列于表4,从表中可以看出,本文的机器学习方法所建模型的预测精度要接近或者好于文献报道的结果,尤其是SVM⁃MC模型,无论是交叉验证还是外部独立测试的结果,都明显好于文献报道结果,而且我们所用样本的数量更多,化合物结构更具有多样性,应用域覆盖了更大的化学空间.所以我们建立的模型尤其是SVM⁃MC模型更加适用于对未知的乙酰胆碱酯酶抑制剂进行预测.
3 结论
本文对结构多样性较大的乙酰胆碱酯酶抑制剂化合物数据集计算了1559个分子描述符,然后通过一套混合描述符筛选方法筛选出和抑制剂活性相关性很强的37个描述符,最后用SVM,k⁃NN和ANN建立乙酰胆碱酯酶抑制剂的分类预测模型.研究结果表明:变量筛选在大大减少描述符个数的同时能大大提高模型的预测能力;本文所建的SVM⁃MC模型要优于其他机器学习方法建立的模型,且预测精度明显好于文献已报道的结果,从而可以用于计算机辅助筛选具有乙酰胆碱酯酶抑制活性的先导化合物.
Supporting Information Available: Table S1 listed 1559 molecular descriptors.The selected molecular descriptors were listed in Table S2.Table S3 listed the results of y⁃scrambling test on SVM model.This information is available free of charge via the internet at http://www.whxb.pku.edu.cn.
1 Dekosky,S.T.;Scheff,S.W.Ann.Neurol.,1990,27:457
2 Terry,R.D.;Masliah,E.;Salmon,D.P.;Butters,N.;DeTeresa, R.;Hill,R.;Hansen,L.A.;Katzman,R.Ann.Neurol.,1991,30: 572
3 Rösler,M.;Anand,R.;Cicin⁃Sain,A.;Gauthier,S.;Agid,Y.;Dal⁃Bianco,P.;Stähelin,H.B.;Hartman,R.;Gharabawi,M.Br.Med. J.,1999,318:633
4 Whitehouse,P.J.Acta Neurologica Scandinavica Supplement, 1993,149:42
5 Kelly,C.A.;Harvey,R.J.;Cayton,H.Br.Med.J.,1997,314:693
6 Scott,L.J.;Goa,L.K.Drugs,2000,60:1095
7 Gottwald,M.D.;Rozanski,R.I.Expert Opinion on Investigational Drugs,1999,8:1673
8 Hasegawa,K.J.Chem.Inf.Comput.Sci.,1999,39:112
9 Recanatini,M.;Cavalli,A.;Belluti,F.;Piazzi,L.;Rampa,A.; Bisi,A.;Gobbi,S.;Valenti,P.;Andrisano,V.;Bartolini,M.; Cavrini,V.J.Med.Chem.,2000,43:2007
10 Sheng,R.;Shen,Y.H.;Lin,X.;Luo,Y.;Fan,Y.J.;Li,J.Y.;Xia, H.R.;Hu,Y.Z.Chinese Journal of Medicinal Chemistry,2007, 17:348 [盛 荣,申艳红,林 肖,罗 蕴,范永剑,李静雅,夏海蓉,胡永洲.中国药物化学杂志,2007,17:348]
11 Liu,A.L.;Guang,H.M.;Zhu,L.Y.;Du,G.H.;Li,M.Y.;Wang, Y.T.Sci.China Ser.C⁃Life Sci.,2007,37:503 [刘艾林,光红梅,朱莉亚,杜冠华,李铭源,王一涛.中国科学C辑:生命科学, 2007,37:503]
12 Mizutani,M.Y.;Itai,A.J.Med.Chem.,2004,47:4818
13 Jiang,Y.R.;Xu,H.;Chen,F.J.;Ma,G.J.Acta Phys.⁃Chim.Sin., 2009,25:1379 [蒋玉仁,许 慧,陈芳军,马贯军.物理化学学报,2009,25:1379]
14 Zheng,Q.C.;Chu,H.Y.;Niu,R.J.;Sun,C.C.Sci.China Ser.B⁃Chem.,2009,39:1454 [郑清川,楚慧郢,牛瑞娟,孙家锺.中国科学B辑:化学,2009,39:1454]
15 Lv,W.;Xue,Y.European Journal of Medicinal Chemistry,2010, 45:1167
16 Czerminski,R.;Yasri,A.;Hartsourgh,D.Quant.Struct.⁃Act. Relat.,2001,20:227
17 Tetko,I.V.;Luik,A.I.;Poda,G.I.J.Med.Chem.,1993,36:811
18 Ruiz,V.E.Pattern Recognition Letters,1986,4:145
19 Ishihara,Y.;Hirai,K.;Miyamoto,M.;Goto,G.J.Med.Chem., 1994,37:2292
20 Villalobos,A.;Blake,J.F.;Bigger,C.K.;Butler,T.W.;Chapin, D.S.;Chen,Y.L.;Ives,J.L.;Jones,S.B.;Liston,D.R.;Nagel, A.A.;Nason,D.M.;Nielsen,J.A.;Shalaby,I.A.;White,W.F. J.Med.Chem.,1994,37:2721
21 Chen,Y.L.;Nielsen,J.;Hedberg,K.;Dunaiskis,A.;Jones,S.; Russo,L.;Johnson,J.;Ives,J.;Liston,D.J.Med.Chem.,1992,5: 1429
22 Sugimoto,H.;Tsuchiya,Y.;Sugumi,H.;Higurashi,K.;Karibe, N.;Iimura,Y.;Sasaki,A.;Araki,S.;Yamanishi,Y.;Yamatsu,K. J.Med.Chem.,1992,35:4542
23 Villalobos,A.;Butler,T.W.;Chapin,D.S.;Chen,Y.L.; DeMattos,S.B.;Ives,J.L.;Jones,S.B.;Liston,D.R.;Nagel,A. A.;Nason,D.M.;Nielsen,J.A.;Ramirez,A.D,;Shalaby,I.A.; White,W.F.J.Med.Chem.,1995,38:2802
24 Rampa,A.;Bisi,A.;Valenti,P.;Recanatini,M.;Cavalli,A.; Andrisano,V.;Cavrini,V.;Fin,L.;Buriani,A.;Giusti,P.J.Med. Chem.,1998,41:3976
25 Contreras,J.M.;Rival,Y.M.;Chayer,S.;Bourguignon,J.J.; Wermuth,C.G.J.Med.Chem.,1999,42:730
26 Nagel,A.A.;Liston,D.R.;Jung,S.;Mahar,M.;Vincent,L.A.; Chapin,D.;Chen,Y.L.;Hubbard,S.;Ives,J.L.;Jones,S.B.; Nielsen,J.A.;Ramirez,A.;Shalaby,I.A.;Villalobos,A.;White, W.F.J.Med.Chem.,1995,38:1084
27 Vidaluc,J.L.;Calmel,F.;Bigg,D.C.H.;Carilla,E.;Briley,M. J.Med.Chem.,1995,38:2969
28 Carlier,P.R.;Chow,E.S.H.;Han,Y.;Liu,J.;Yazal,J.E.;Pang, Y.P.J.Med.Chem.,1999,42:4225
29 Kenna,M.T.M.;Proctor,G.R.;Young,L.C.;Harvey,A.L. J.Med.Chem.,1997,40:3516
30 Sugimoto,H.;Iimura,Y.;Yamanishi,Y.;Yamatsu,K.J.Med. Chem.,1995,38:4821
31 Rampa,A.;Piazzi,L.;Belluti,F.;Gobbi,S.;Bisi,A.;Bartolini, M.;Andrisano,V.;Cavrini,V.;Cavalli,A.;Recanatini,M.; Valenti,P.J.Med.Chem.,2001,44:3810
32 Contreras,J.M.;Parrot,I.;Sippl,W.;Rival,Y.M.;Wermuth,C. G.J.Med.Chem.,2001,44:2707
33 Hu,M.K.;Wu,L.J.;Hsiao,G.;Yen,M.H.J.Med.Chem.,2002, 45:2277
34 Recanatini,M.;Cavalli,A.;Belluti,F.;Piazzi,L.;Rampa,A.; Bisi,A.;Gobbi,S.;Valenti,P.;Andrisano,V.;Bartolini,M.; Cavrini,V.J.Med.Chem.,2000,43:2007
35 Belluti,F.;Rampa,A.;Piazzi,L.;Bisi,A.;Gobbi,S.;Bartolini, M.;Andrisano,V.;Cavalli,A.;Recanatini,M.;Valenti,P.J.Med. Chem.,2005,48:4444
36 Pilar,M.R.;Rubio,L.;Esther,G.P.;Dorronsoro,I.;Maria,M. M.;Valenzuela,R.;Usan,P.;Austria,C.;Bartolini,M.; Andrisano,V.;Axel,B.C.;Orozco,M.;Luque,F.J.;Medina,M.; Martinez,A.J.Med.Chem.,2005,48:7223
37 Sauvaitre,T.;Barlier,M.;Herlem,D.;Gresh,N.;Chiaroni,A.; Guenard,D.;Guillou,C.J.Med.Chem.,2007,50:5311
38 Cardoso,C.L.;Ian,C.G.;Silva,D.H.S.;Furlan,M.;Epifanio, R.A.;Pinto,A.C.;Rezende,C.M.;Lima,J.A.;Bolzani,V.S. J.Nat.Prod.,2004,67:1882
39 Bolognesi,M.L.;Cavalli,A.;Valgimigli,L.;Bartolini,M.; Rosini,M.;Andrisano,V.;Recanatini,M.;Melchiorre,C.J.Med. Chem.,2007,50:6446
40 Kavitha,C.V.;Gaonkar,S.L.;Chandra,J.N.;Sadashiva,C.T.; Rangappa,K.S.Bioorganic&Medicinal Chemistry,2007,15: 7391
41 Bianchi,D.A.;Hirschmann,G.S.;Theoduloz,C.;Bracca,A.B. J.;Kaufman,T.S.Bioorganic&Medicinal Chemistry Letters, 2005,15:2711
42 Sheng,R.;Lin,X.;Li,J.Y;Jiang,Y.K.;Shang,Z.C.;Hu,Y.Z. Bioorganic&Medicinal Chemistry Letters,2005,15:3834
43 Ishichi,Y.;Sasaki,M.;Setoh,M.;Tsukamoto,T.;Miwatashi,S.; Nagabukuro,H.;Okanishi,S.;Imai,S.;Saikawa,R.;Doi,T.; Ishihara,Y.Bioorganic&Medicinal Chemistry Letters,2005,13: 1901
44 Wang,B.;Mai,Y.C.;Li,Y.;Hou,J.Q.;Huang,S.L.;Ou,T.M.; Tan,J.H.;An,L.K.;Li,D.;Gu,L.Q.;Huang,Z.S.European J. Med.Chem.,2010,45:1415
45 Martinez,A.;Fernandez,E.;Castro,A.;Conde,S.;Isabel,R.F.; Banos,J.E.;Badia,A.J.Med.Chem.,2000,35:913
46 Shao,D.;Zou,C.Y.;Luo,C.;Tang,X.C.;Li,Y.C.Bioorganic& Medicinal Chemistry Letters,2004,14:4639
47 Belluti,F.;Piazzi,L.;Bisi,A.;Gobbi,S.;Bartolini,M.;Cavalli, A.;Valenti,P.;Rampa,A.European Journal of Medicinal Chemistry,2009,44:1341
48 Rampa,A.;Bisi,A.;Belluti,F.;Gobbi,S.;Valenti,P.;Andrisano, V.;Cavrini,V.;Cavalli,A.;Recanatini,M.Bioorganic& Medicinal Chemistry,2000,8:497
49 Toda,N.;Tago,K.;Marumoto,S.;Takami,K.;Ori,M.;Yamada, N.;Koyama,K.;Naruto,S.;Abe,K.;Yamazaki,R.;Hara,T.; Aoyagi,A.;Abe,Y.;Kaneko,T.;Kogen,H.Bioorganic& Medicinal Chemistry,2003,11:1935
50 Zaheer,U.H.;Mahmood,U.;Jehangir,B.Chem.Biol.Drug. Des.,2009,74:571
51 Pan,L.;Tan,J.H.;Hou,J.Q.;Huang,S.L.;Gu,L.Q.;Huang,Z. S.Bioorganic&Medicinal Chemistry Letters,2008,18:3790
52 Marco,J.L.;Rios,C.;Garcia,A.G.;Villarroya,M.;Carreiras,M. C.;Martins,C.;Eleuterio,A.;Morreale,A.;Orozco,M.;Luque, F.J.Bioorganic&Medicinal Chemistry,2004,12:2199
53 Shen,Q.;Peng,Q.;Shao,J.L.;Liu,X.F.;Huang,Z.S.;Pu,X. Z.;Ma,L.;Li,Y.M.;Chan,A.S.C.;Gu,L.Q.European Journal of Medicinal Chemistry,2005,40:1307
54 Martin,L.L.;Davis,L.;Klein,J.T.;Nemoto,P.;Olsen,G.E.; Bores,G.M.;Camacho,F.;Petko,W.W.;Rush,D.K.;Selk,D.; Smith,C.P.;Vargas,H.M.;Winslow,J.T.;Effland,R.C.;Fink, D.M.Bioorganic&Medicinal Chemistry Letters,1997,7:157
55 Sugimoto,H.;Yamanishi,Y.;Iimura,Y.;Kawakami,Y.Current Medicinal Chemistry,2000,7:303
56 Stewart,J.J.P.J.Comput.Chem.,1989,10:221
57 Willett,P.;Barnard,J.M.;Downs,G.M.J.Chem.Inf.Comput. Sci.,1998,38:983
58 Li,Z.R.;Han,L.Y.;Xue,Y.;Yap,C.W.;Li,H.;Jiang,L.;Chen, Y.Z.Biotechnol.Bioeng.,2007,97:389
59 Yang,C.W.;Su,J.Y.;Tsou,A.P.;Chau,G.Y.;Liu,H.L.;Chen, C.H.;Chien,C.Y.;Chou,C.K.Biochem.Biophys.Res. Commun.,2005,330:489
60 Fisher,R.A.Annals of Eugenics,1936,7:179
61 Ajmani,S.;Jadhav,K.;Kulkarni,S.A.J.Chem.Inf.Model., 2006,46:24
62 Li,P.;Tan,N.X.;Rao,H.B.;Li,Z.R.;Chen,Y.Z.Acta Phys.⁃Chim.Sin.,2009,25:1581 [李 平,谈宁馨,饶含兵,李泽荣,陈宇综.物理化学学报,2009,25:1581]
63 Kennard,R.W.;Stone,L.A.Technometrics,1969,11:137
64 Liu,H.;Papa,E.;Gramatica,P.Chem.Res.Toxicol.,2006,19: 1540
65 Liu,H.;Gramatica,P.Bioorganic&Medicinal Chemistry,2007, 15:5251
66 Bhasin,M.;Zhang,H.;Reinherz,E.L.;Reche,P.A.FEBS.Lett., 2005,579:4302
67 Matthews,B.W.Biochim.Biophys.Acta,1975,405:442
68 Dimitrov,S.;Dimitrova,G.;Pavlov,T.;Dimitrova,N.;Patlewicz, G.;Niemela,J.;Mekenyan,O.J.Chem.Inf.Model.,2005,45:839 69 Tropsha,A.;Golbraikh,A.Curr.Pharm.Des.,2007,13:3494
July 22,2010;Revised:August 15,2010;Published on Web:October 11,2010.
Classification Models for Acetylcholinesterase Inhibitors Based on Machine Learning Methods
YANG Guo⁃Bing2LI Ze⁃Rong1,∗RAO Han⁃Bing1LI Xiang⁃Yuan2CHEN Yu⁃Zong3
(1College of Chemistry,Sichuan University,Chengdu 610064,P.R.China;2College of Chemical Engineering,Sichuan University,Chengdu 610065,P.R.China;3Department of Pharmacy,National University of Singapore,Singapore 117543)
A total of 1559 molecular descriptors including constitutional,charge distribution,topological, geometrical,and physicochemical descriptors were calculated to encode acetylcholinesterase inhibitors. The 37 molecular descriptors were selected using a hybrid filter/wrapper approach by combining a Fischer Score and Monte Carlo simulated annealing.Classification models for the acetylcholinesterase inhibitors were then built based on support vector machine (SVM),artificial neural networks(ANN),and k⁃nearest neighbor(k⁃NN)methods.For the 515 samples in the training set,we obtained average prediction accuracies of 87.3%-92.7%,67.0%-81.0%,and 79.4%-88.2%for the positive,the negative, -and the total samples,respectively,by 5⁃fold cross validation.Average prediction accuracies of 72.7%82.5%, 41.0%-53.0%,and 62.1%-69.1%were obtained for the positive,the negative,and the total samples, respectively,by the y⁃scrambling method,indicating that there was no chance correlation in our models.An external test was conducted on 172 samples that were not used for model building and we obtained prediction accuracies of 93.3%-100.0%,74.6%-89.6%,and 86.1%-95.9%for the positive,the negative,and the total samples,respectively.The prediction accuracies obtained by all the machine learning methods especially by the SVM method were far better than previously reported results.
Acetylcholinesterase inhibitor;Machine learning method;Feature selection; Applicability domain
O641
∗Corresponding author.Email:lizerong@scu.edu.cn;Tel:+86⁃28⁃85406139.
The project was supported by the National Natural Science Foundation of China(20973118).
国家自然科学基金(20973118)资助项目

