真菌苯二酚内酯聚酮类化合物生物合成研究进展
王晓婧, 陆 伟, 徐玉泉*, 梁晓东
1.中国农业科学院生物技术研究所, 北京 100081 2.国药一心制药有限公司, 北京 100176
由聚酮合酶(poylketide synthases, PKS)生物合成的真菌聚酮是一类化学结构多样、生物活性广泛的天然产物家族,具有重要的经济价值和药用价值[1,2]。其中包括二羟基苯甲酸内酯(resorcylic acid lactones, RALs)和二羟基苯乙酸内酯(dihydroxyphenylacetic acid lactones, DALs)在内的苯二酚内酯(benzenediol lactones, BDLs)类化合物更是呈现出抗癌、抑制热休克反应和调节免疫系统等重要药用活性,对于这类化合物生物合成的研究是当今该领域的热点之一[3]。本文综述了苯二酚内酯的双聚酮合酶协作合成机制和组合生物合成,以期为更深入的研究真菌苯二酚内酯的作用机理提供参考。
1 真菌聚酮合酶
聚酮合酶在结构上与脂肪酸合成酶类似,它们可能拥有共同的进化起源[4]。聚酮合酶基因大多定位在染色体接近端粒的部位[5],与聚酮合酶后修饰过程的编码基因、调节基因及抗性基因等成簇存在[6]。聚酮合酶根据其结构和催化机制不同可分为3类:模块化Ⅰ型PKS(modular PKS Ⅰ)、迭代Ⅱ型PKS(iterative PKS Ⅱ)和查尔酮Ⅲ型PKS(chalcone PKS Ⅲ)。真菌PKS从结构角度看属于模块化Ⅰ型PKS,但是之后发现这些模块在聚酮链的装配过程中大多是重复使用的,因此将它单独分为一类,即重复的Ⅰ型PKS (iterative PKS Ⅰ, iPKS Ⅰ)。根据在催化过程中对酮基还原程度的不同,真菌聚酮合酶又可分为非还原型PKS(non-reduced, nr PKS)和还原型PKS(highly reduced, hr PKS)[7~9]。
真菌聚酮合酶是由不同的功能结构域组成固定化的模块[9],其中识别起始单元或者延伸单元的酰基转移酶(acyl transferase, AT)、催化双碳单位缩合反应的酮酯酰基合酶(ketosynthase, KS)、结合延长聚酮链的酰基转运蛋白(acyl carrier protein, ACP)是PKS的核心结构单元。hr PKS包含酮还原酶(ketoacyl reductase,KR)、脱水酶(dehydratase, DH)、烯基还原酶(enoy lreductase, ER)等结构域对合成的酮基做进一步的还原。nr PKS含有起始单元酰基转移酶(starter unit ACP acyltransferase, SAT)、产物模板结构域(product template, PT)、硫脂酶(thioesterase, TE),分别负责接受上游起始合成产物、合成产物的折叠和释放[9,10]。
2 苯二酚内酯的双酶协作生物合成机制
大多数真菌聚酮只需要一个PKS催化合成,而合成苯二酚内酯需要一对高度还原型hr PKS和非还原型nr PKS的共同催化,这种合成机制被称为双酶协作机制[11~13]。首先,由hr PKS生成一条线性还原聚酮链,并通过SAT直接传递给nr PKS继续延伸聚酮,通过羟醛缩合反应,PT催化第一个芳香环形成,通过Claisen酯缩合反应,TE催化形成第二个内酯环,并从聚酮合酶上释放产物[1]。在苯二酚内酯生物合成过程中,需要hr PKS和nr PKS之间,PKS内部结构域之间相互识别和协作,程序化地完成从起始底物的识别、聚酮链还原程度和长度的确定、芳香环的折叠模式、产物的释放等生物合成过程(图1)。

图1 苯二酚内酯化合物生物合成Fig.1 Biosynthesis of benzenediol loctone.注:实箭头为必需步骤,虚箭头为非必需步骤
生物合成苯二酚内酯类化合物的聚酮合酶,在结构域的组合方式和氨基酸序列上都有很高的一致性,如hr PKS都含有KS、AT、KR、ER、DH和ACP结构域,nr PKS都含有SAT、KS、AT、PT、ACP和TE结构域[14,15];合成苯二酚内酯radicicol、hypothemycin、zearalenone以及curvularin聚酮合酶之间氨基酸序列的一致性分别可达到55%~61%(hr PKS)和49%~54%(nr PKS)[15]。另外,在基因组中,苯二酚内酯聚酮合酶hr PKS和nr PKS的编码基因一般都相邻成对出现[14]。基于上述苯二酚内酯PKS的结构和编码基因的显著特点,利用SMURF(Secondary Metabolite Unknown Regions Finder)和antiSMASH(antibiotics and Secondary Metabolite Analysis Shell)等工具能够从已测序的基因组中挖掘到更多苯二酚内酯类化合物的生物合成途经[16,17]。但是,由于苯二酚内酯程序化生物合成的精确机制不清楚,如确定聚酮链的长度、酮基的还原程度、中间产物的立体构型等合成机制,仅仅通过基因序列的信息无法预测出绝大部分化合物的结构[17]。另一方面,由于很多真菌产生的苯二酚内酯类化合物数量很少,或者只在特殊的环境下才能合成,所以,在实验室条件下,大部分苯二酚内酯合成基因的功能未知[24,25]。迄今为止,研究重点依然在已知结构苯二酚内酯的生物合成上(表1,图2)。

表1 真菌苯二酚内酯类化合物Table 1 Fungal benzenediol lactones.

图2 苯二酚内酯聚酮类化合物及其类似物结构Fig.2 Structures of BDLs and its analogs.
3 典型苯二酚内酯类化合物的生物合成
3.1 Radicicol的生物合成
Radicicol(图2-5)是一种二羟基苯甲酸内酯类化合物,能够通过抑制Hsp90热休克蛋白调节热激反应阻断多条致癌通路[12,14,18]。多种真菌都可产生Radicicol,其中Chaetomiumchiversii[14]和Pochoniachlamydosporia[13]中的生物合成途径已经得到阐明。
在Chaetomiumchiversii生物合成radicical的过程中,还原型聚酮合酶CcRadS1和非还原型聚酮合酶CcRadS2协同作用合成radicical核心骨架monocillin Ⅱ(图2-1),二者缺一不可[14](图3)。基因敲除上述任何一个聚酮合酶的编码基因,都不能获得预期的产物。通过在酵母表达系统中异源共表达CcRadS1和CcRadS2基因,可以在发酵液中检测到monocillin Ⅱ[14]。另外,将在酵母中表达的合成radicicol的还原型聚酮合酶Rdc5和非还原型聚酮合酶Rdc1纯化后,加入丙二酰CoA和辅因子NADPH,同样可以合成中间产物monocillin Ⅱ[13]。以上基因敲除和体内体外重构活性酶蛋白的实验,都验证了还原型聚酮合酶和非还原型聚酮合酶在radicical生物合成途径中的重要性。
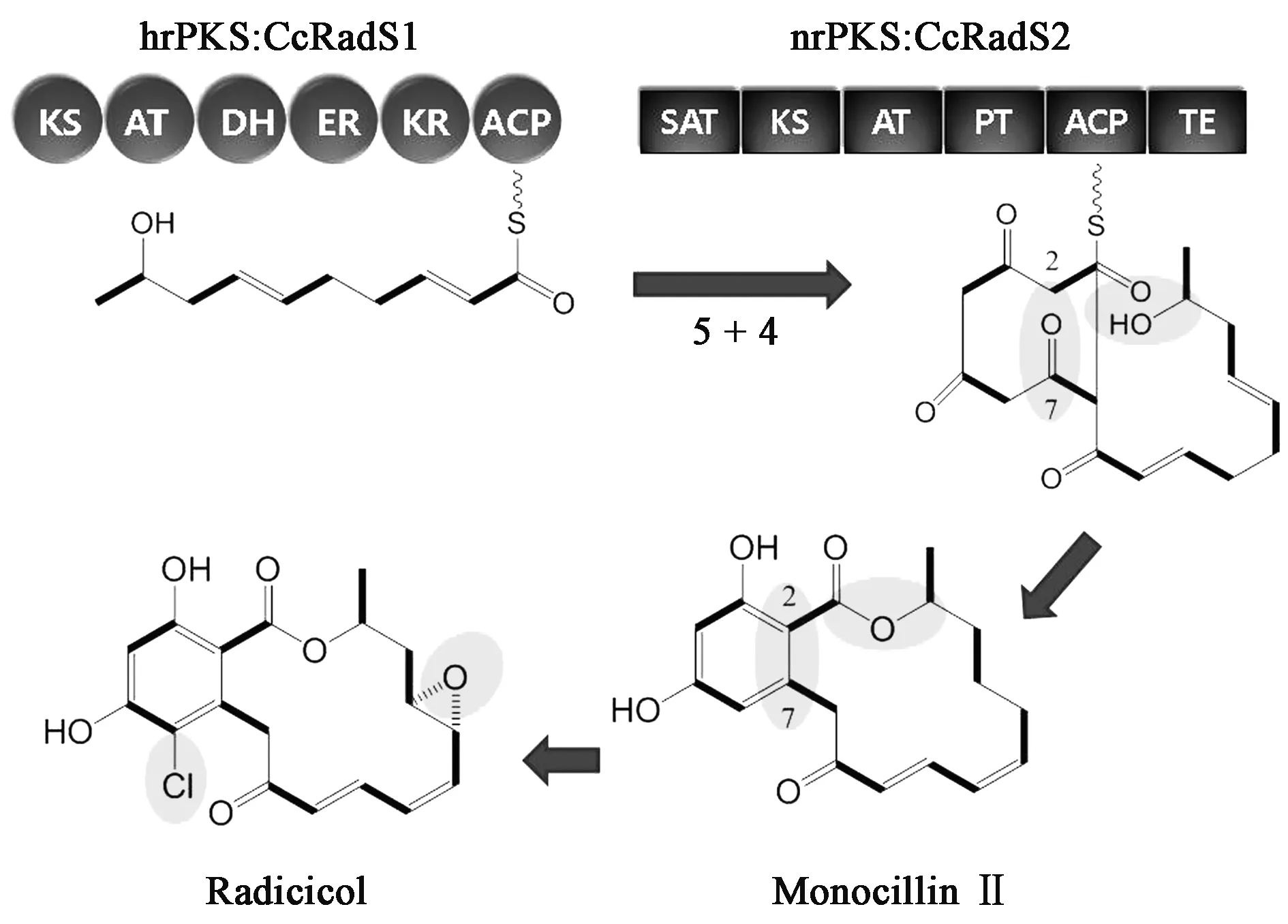
图3 Radicicol的生物合成Fig.3 Biosynthesis pathway of radicicol.
在radicicol的合成基因簇中,含有卤化酶基因,其蛋白序列与细菌中黄素依赖卤化酶一致性很高。敲除卤化酶RadH基因后,得到一个缺少氯原子的radicical类似物monocillin I (图2-2)[14]。在酵母表达系统中异源共表达卤化酶Rdc2、Rdc5和Rdc1的编码基因,可以在发酵液中检测到增加一个氯原子的monocillin Ⅱ类似物pachonin D(图2-3)[13],该物质曾在P.chlamydosporia中分离鉴定出来[26]。在离体实验中,只有加入FAD时,在Rdc2的催化下,monocillin Ⅱ的C6或C4、C6上添加氯原子形成pachonin D(图2-3)或化合物(图2-4),以上实验均验证了黄素依赖卤化酶的功能。
此外,敲除细胞色素氧化酶P450基因RadP得到一个缺少环氧环的radicical类似物pachonin D,说明RadP表达产物具有环氧酶功能。敲除预测的RadR转录因子基因,不能合成radicicol,表明RadR在radicicol合成过程中起正调控作用[12]。
以上的研究证明在radicicol的生物合成中,通过还原型聚酮合酶和非还原型聚酮合酶协同催化合成核心骨架monocillin Ⅱ,然后经过核黄素依赖的卤化酶、细胞色素氧化酶的修饰,monocillin Ⅱ分别在C6、C14-C15处增加一个氯原子和一个环氧原子形成radicical。在生物合成过程中,转录因子对激活合成途径也起到了重要的作用[14]。总之,只有在生物合成radicicol的多个酶的协调作用下,才能生成完整的化合物。
3.2 Hypothemycin 的生物合成
在Hypomycessubiculosus和其他一些真菌中生物合成的hypothemycin(图2-8)是一种MAP激酶抑制剂[18,27,28],其核心骨架DHZ(图2-6)是由还原型聚酮合酶Hpm8和非还原型聚酮合酶Hpm3协同合成。通过在酵母表达系统中异源共表达Hpm8和Hpm3,可以在发酵液中检测到DHZ[13];Hpm8单独表达时,只能在发酵液中检测到吡喃酮类化合物,而不是预期的六酮;Hpm3单独表达时,可以接受天然前体类似物生物合成DHZ,但是不能直接利用丙二酰CoA作为底物[19],这说明在Hypothemycin的生物合成中非还原型聚酮合酶只接受还原型聚酮合酶合成中间产物。通过SAT结构域突变,证明非还原型聚酮合酶上的SAT结构域是连接两个聚酮合酶的重要环节[13],另外,还原型聚酮合酶合成中间产物的速率决定DHZ的生成速率[13]。以上研究说明hypothemycin的生物合成需要还原型聚酮合酶和非还原型聚酮合酶协同完成。
苯二酚内酯的生物合成中,氧甲基转移酶(O-methyltransferase, OMT)能够将苯环上的羟基转化为氧甲基。在离体实验中,H.subiculosus中的氧甲基转移酶基因Hpm5可以将C5的羟基甲基化[18]。黄素依赖的单加氧酶(flavin-dependent monooxygenase, FMO)Hpm7能够在DHZ的C8-C9之间形成环氧键,在NADPH存在时,Hpm7可以将zearalenone(图2-10)或desepoxyhy-pothemycin的C-C双键转化成环氧键。在hypothemycin的生物合成基因簇中,还包含乙醇氧化酶基因和谷胱甘肽-S-转移酶基因,这些基因的确切功能还不清楚[18]。
3.3 Zearalenone 的生物合成
真菌生物合成的zearalenone(图2-10)是一种雌激素抑制剂[20]。在玉蜀黍赤霉Gibberellazeae中,还原型聚酮合酶ZEA2和非还原型聚酮合酶ZEA1协同作用合成核心骨架zearalenol。基因敲除任何一个聚酮合酶的编码基因,都不能合成zearalenol(图2-9)[21]。ZEA1在大肠杆菌中表达纯化后,加入天然前体类似物六烯酮后,可以合成预期的化合物[22]。实验中还证明了ZEA1合成途径可以和脂肪酸生物合成途径相互作用,不加入天然前体类似物六烯酮时,大肠杆菌仍能产生少量酰基二羟基苯甲酸化合物,这些化合物是以大肠杆菌脂肪生物合成途径中的短链脂肪酰基硫酯作为起始单元合成的[22]。
在zearalenone的生物合成中,通过还原型聚酮合酶和非还原型聚酮合酶协同催化合成核心骨架zearalenol[21],之后经过乙醇氧化酶的修饰,zearalenol生成zearalenone,完成整个化合物的生物合成过程。
3.4 Curvularin的生物合成
Curvularin是一种具有Hsp90抑制活性和免疫调节活性的苯二酚内酯类化合物,能够在Alternaria、Aspergillus、Cochliobolus、Curvularia和Penicillium等真菌中合成[15]。上述介绍的苯二酚内酯属于二羟基苯甲酸内酯,而curvularin是二羟基苯乙酸内酯,二者在芳香环的折叠环化方式上有差异[1]。
Curvularin的生物合成同样需要还原型聚酮合酶和非还原型聚酮合酶双酶协作合成核心骨架。通过在酵母表达系统中异源共表达还原型聚酮合酶AtCURS1基因和非还原型聚酮合酶AtCURS2基因,可以在发酵液中检测到10,11-Dehydrocurvularin(图2-11)[15]。AtCURS2单独在酵母中表达时,其SAT结构域可以接受天然前体类似物四烯酮,但是当添加的四烯酮增加为五烯酮时,就不能生成预期的化合物,这说明AtCURS2的SAT结构域在前体长度的选择上有局限性[15]。
苯二酚内酯类化合物的芳香环是通过PT结构域的羟醛缩合反应环化而来的,其PT结构域与细菌芳香化酶/环化酶在氨基酸序列上相似性很低,蛋白折叠模式和活性位点也完全不同,这是一个很典型的趋同进化的例子。真菌苯二酚内酯类化合物通常遵循F-型折叠模式,合成C2-C7、C4-C9或C6-C11型产物,如radicicol等[1];curvularin的合成遵循与细菌类似的S-型折叠模式,在C3-C8处羟醛缩合生成curvularin。尽管折叠模式不同,F-型radicicol和S-型curvularin的PT结构域在活性位点和环化室上有很高的保守性(47%序列一致性和65%相似性)。生物信息学确认PT结构域活性中心上的3个重要氨基酸位点,即curvularin:W1584、F1455和Y1576;radicicol:L1609、Y1478和F1601,氨基酸替换实验表明,curvularin PT中两个氨基酸位点被替换时,能够产生F-型化合物,当3个氨基酸位点都被取代时,S-型化合物完全消失,而只产生F-型化合物。这说明苯二酚内酯非聚酮合酶PT结构域中关键氨基酸的改变可影响第一个芳香环的环化模式[1]。
4 展望
苯二酚内酯双聚酮合酶协同合成的催化机制和聚酮合酶模块化的结构,为组合不同真菌来源的合成模块和模块内的结构域组合生物合成“非天然”的苯二酚内酯类化合物奠定基础[28]。组合生物合成技术是近年兴起的,通过扩展天然产物结构的多样性,形成新生物合成途径,从而产生非天然化合物的新方法。利用该技术合成苯二酚内酯类化合物是当前药物研发的热点,但尚处于起步阶段。通过聚酮合酶亚基重排和随机组合,能够在酿酒酵母中异源表达新型聚酮合酶,实现一系列“非天然的”的聚酮类化合物的一步合成,为新一代药物筛选提供新的候选化合物库,同时为揭示天然聚酮类化合物的程序化合成机制奠定了重要的理论基础。这种“即插即用”的模块化方法可应用于结构多样的全新化学物质的组合生物合成中。利用合成生物学的方法,在现有合成模块的基础上,通过人工设计和智能优化,创造更多新型重要活性的苯二酚内酯聚酮化合物,是未来真菌聚酮合成生物学的发展方向[28~31]。
[1] Xu Y Q, Zhou T, Zhou Z F,etal.. Rational reprogramming of fungal polyketide first-ring cyclization[J]. Proc. Natl. Acad. Sci. USA, 2013, 110(14): 5398-5403.
[2] Chiang Y M, Oakley C E, Hhuja M,etal.. An efficient system for heterologous expression of secondary metabolite genes inAspergillusnidulans[J]. J. Am. Chem. Soc., 2013, 135: 7720-7731.
[3] Xu Y Q, Zhou T, Zhang S W,etal.. Thioesterase domains of fungal nonreducing polyketide synthases act as decision gates during combinatorial biosynthesis[J]. J. Am. Chem. Soc., 2013, 135: 10783-10791.
[4] 周礼红. 红曲霉遗传转化系统及桔霉素、MonaocnilK生物合成相关PKS基因的克隆与功能鉴定[D]. 江苏 无锡:江南大学, 博士学位论文,2005.
[5] Keller N P, Turner G,Bennett J W,etal.. Fungal secondary metabolism-from biochemistry to genomics[J]. Nat. Rev. Microbiol., 2005, 3(12): 937-947.
[6] 魏康霞. 橙色红曲菌聚酮合酶基因的克隆[D].南昌:南昌大学,硕士学位论文,2007.
[7] 严少华, 郭 亮. 真菌聚酮合酶分类进展及其应用[J]. 江西科学, 2006, 24(2): 170-172.
[8] 陈路劼, 赵 薇, 连云阳. 聚酮合酶与药物筛选的研究进展[J]. 中国抗生素杂志, 2012, 37(9): 655-661.
[9] Chiang Y M, Oakley B R, Keller N P,etal.. Unraveling polyketide synthesis in members of the genusAspergillus[J]. Appl. Microbiol. Biotechnol., 2010, 86(6): 1719-1736.
[10] Adrian T, Keatinge C. The structures of type I polyketide synthases[J]. Nat. Prod. Rep., 2012, 29: 1050-1073.
[11] Winter J M, Sato M,Sugimoto S,etal.. Identification and characterization of the Chaetoviridin and Chaetomugilin gene cluster inChaetomiumglobosumreveal dual functions of an iterative highly-reducing polyketide synthase[J].J. Am. Chem. Soc.,2012,134(43): 17900-17903.
[12] Zhou H, Qiao K J, Gao Z Z,etal.. Insights into radicicol biosynthesis via heterologous synthesis of intermediates and analogs[J]. J. Biol. Chem., 2010, 285(53): 41412-41421.
[13] Zhou H, Qiao K J, Gao Z Z,etal.. Enzymatic synthesis of resorcylic acid lactones by cooperation of fungal iterative polyketide synthases involved in Hypothemycin biosynthesis[J]. J. Am. Chem. Soc., 2010, 132(13): 4530-4531.
[14] Wang S H, Xu Y Q, Maine E A,etal.. Functional characterization of the biosynthesis of radicicol, an Hsp90 inhibitor resorcylic acid lactone fromChaetomiumchiversii[J]. Chem. Biol., 2008, 15: 1328-1338.
[15] Xu Y Q, Artiles P E, Schubert V,etal.. Characterization of the biosynthetic genes for 10,11-Dehydrocurvularin, a heat shock response-modulating anticancer fungal polyketide fromAspergillusterreus[J]. Appl. Environ. Microbiol., 2013,79(6): 2038-2047.
[16] 王 芬. 变构菌素生物合成相关基因功能的研究[D]. 辽宁 大连:大连理工大学, 博士学位论文,2013.
[17] Chiang Y M, Lee K, Sancheza J F,etal.. Unlocking fungal cryptic natural products[J]. Nat. Prod. Commun., 2009, 4(11): 1505-1510.
[18] Reeves C D, Hu Z H, Reid R,etal.. Genes for the biosynthesis of the fungal polyketides hypothemycin fromHypomycessubiculosusand radicicol fromPochoniachlamydosporia[J]. Appl. Environ. Microbiol., 2008, 74(16): 5121-5129.
[19] Gao Z Z, Wang J J, Norquay A K,etal.. Investigation of fungal iterative polyketide synthase functions using partially assembled intermediates[J]. J. Am. Chem. Soc., 2013, 135(5): 1735-1738.
[20] Kim Y T, Lee Y R, Jin J M,etal.. Two different polyketide synthase genes are required for synthesis of zearalenone inGibberellazeae[J]. Mol. Microbiol., 2005, 58(4): 1102-1113.
[21] Gaffoor I, Trail F. Characterization of two polyketide synthase genes involved in zearalenone biosynthesis inGibberellazeae[J]. Appl. Environ. Microbiol., 2006, 5: 1793-1799.
[22] Zhou H, Zhan J X, Watanabe K J,etal.. A polyketide macrolactone synthase from the filamentous fungusGibberellazeae[J]. Proc. Natl. Acad. Sci. USA, 2008, 105(17): 6249-6254.
[23] Xu Y Q, Zhou T, Espinosa A P,etal.. Insights into the biosynthesis of 12-membered resorcylic acid lactones from heterologous production inSaccharomycescerevisiae[J]. ACS Chem. Biol., 2014,9(5): 1119-1127.
[24] Sanchez J F, Somoza A D, Keller N P,etal.. Advances inAspergillussecondary metabolite research in the post-genomic era[J]. Nat. Prod. Rep., 2012, 29: 351-371.
[25] Wiemann P, Keller N P. Strategies for mining fungal natural products[J]. J. Ind. Microbiol. Biotechnol., 2014,41: 301-313.
[26] Moulin E, Zoete V, Barluenga S,etal.. Design, synthesis, and biological evaluation of HSP90 inhibitors based on conformational analysis of radicicol and its analogues[J]. J. Am. Chem. Soc., 2005, 127: 6999-7004.
[27] El E T, Figueroa M, Raja H A,etal.. Biosynthetically distinct cytotoxic polyketides fromSetophomaterrestris[J]. Eur. J. Org. Chem.,2015,1: 109-121.
[28] Schirmer A, Kennedy J, Sumati M,etal.. Targeted covalent inactivation of protein kinases by resorcylic acid lactone polyketides[J]. Proc. Natl. Acad. Sci. USA, 2006, 103: 114234-114239.
[29] Xu Y Q, Zhou T, Zhang S W,etal.. Diversity-oriented combinatorial biosynthesis of benzenediol lactone scaffolds by subunit shuffling of fungal polyketide synthases[J]. Proc. Natl. Acad. Sci. USA,2014, 111(34): 12354-12359.
[30] 黄惠娟, 乔建军. 聚酮类抗生素组合生物合成[J]. 细胞生物学杂志,2007, 29:692-696.
[31] 张晓琳. 阿维链霉菌中聚酮合酶基因的遗传改造[D]. 北京:中国农业大学, 博士学位论文,2004.

