基于bootstrap方法的贝叶斯网络结构学习算法在构建基因调控网络中的应用*
哈尔滨医科大学卫生统计教研室(150081) 李海龙 侯 艳 柯朝甫 李 康
基于bootstrap方法的贝叶斯网络结构学习算法在构建基因调控网络中的应用*
哈尔滨医科大学卫生统计教研室(150081) 李海龙 侯 艳 柯朝甫 李 康△
目的探讨基于bootstrap重抽样方法的贝叶斯网络结构学习算法构建网络的性能,并将其应用于卵巢癌基因表达谱数据分析。方法通过模拟实验和实例验证本文给出的算法构建网络的有效性,同时将这种算法应用于构建基因调控网络。结果模拟实验显示,在样本量较小的情况下,基于bootstrap算法构建的贝叶斯网络明显优于普通贝叶斯方法构建的网络;实例分析结果也表明,应用本文的方法能够得到有价值的网络结构。结论应用本文给出的算法能够在样本量较少的情况下得出准确度较高的网络,同时能够给出网络结构中各条边置信度的估计值。
贝叶斯网络 结构学习 bootstrap
贝叶斯网络是一种概率图形模型,它能够发现变量之间潜在的依赖关系。其模型构建可分为三个步骤:①网络变量的确定;②网络结构学习;③参数估计[1-2]。贝叶斯网络的结构学习是根据原始数据,通过一定的搜索策略找到得分最高的网络结构,得分高说明网络结构能够很好地代表数据中变量间的调控关系[3]。然而,实际中由于样本量不足,常常出现一些结构不同而得分相近的网络,难以从得分相近的网络中分辨出哪一种结构更接近真实网络[4-5]。此外,一般的结构学习方法难以根据评价指标来评价网络结构的可靠程度。本文将贝叶斯网络结构学习方法与bootstrap重抽样方法相结合,通过设定阈值得到包含高置信度边的网络,并与一般的结构学习方法相比较,考察其有效性。最后运用本文给出的方法对卵巢癌基因表达谱数据进行分析,做出生物学解释。
原理与方法
1.贝叶斯网络结构学习
贝叶斯网络是一个有向无环图,可以表示成一组随机变量的联合概率分布。形式上一组随机变量X={X1,…,Xn}的贝叶斯网络可以用B=(G,θ)表示,其中第一个成分θ表示一个有向无环图,图中节点代表随机变量,节点之间的边代表变量之间的直接依赖关系。第二个成分θ,代表一组量化网络的参数θ=(θ1,θ1,…,θm′),m′>m以条件概率分布的形式表示,即θi=PB(Xi|pa(Xi)),其中pa(Xi)表示变量Xi在图G中的父节点集。贝叶斯网络B给一组变量X定义的联合概率分布:
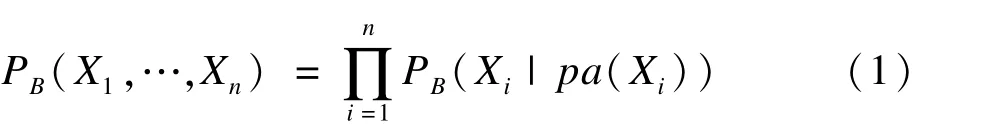
贝叶斯网络结构学习可以归结为:对于给定的数据训练集D,寻找一个网络B使之能与数据集D最匹配。解决这个问题最常用的方法就是引入一个得分函数来评价对应训练集所得网络的拟合程度,然后根据得分搜索到最优网络。本文采用BIC得分函数,并运用贪婪爬山法(greed hill-climbing)结合随机重搜索得分最高的网络结构,这种方法能够避免陷入局部最优。
网络的得分使用BIC准则确定,BIC得分越大,构建的网络越好,其计算公式为
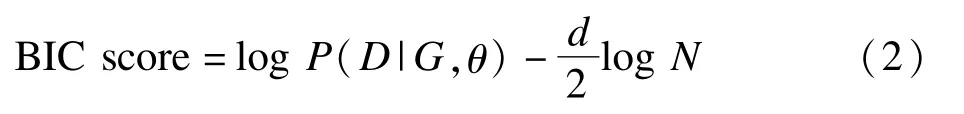
其中N为数据的总例数,d为网络的参数个数。
2.bootstrap方法置信度估计
对于网络G的结构,感兴趣的特征可以是某条有向边X→Y,也可以是无向边X-Y。总之,可以将这些边用字母fij来表示,并通过网络结构的函数转换成集合{0,1}表示,fij=0表示节点Xi和节点Xj不连接,fij=1表示两节点连接,简记为f。
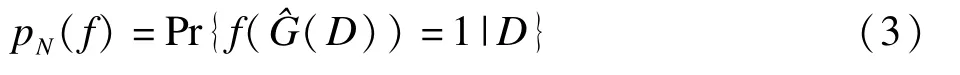
PN(f)表示贝叶斯网络B中抽到一个任意两节点是否相连网络的概率。如果结构学习过程一致,则希望当样本量N足够大时,pN(f)会收敛于f(G)。也就是说,如果真实网络结构G中确实存在节点相连特征f,则它的置信度应该接近1,相反如果不存在则应该接近于0。
使用bootstrap估计置信度的方法是通过对数据集有放回地重抽样,然后通过对多个bootstrap数据集进行学习得出多个网络,在这多个网络中任意两节点相连接(包括方向)的频率就是其置信度估计。算法的过程如下:
M(bootstrap重抽样次数),Fs(得分函数),t(阈值)
Output:G,包含概率大于阈值有向边Xi→Xj的图形Fori=1 toMdo
有放回地从数据D中抽取N个观测得到数据集Di
根据Di通过得分函数Fs指导的学习算法得出得分最高的网络结构Gi
end
模拟实验
模拟数据来源于已知的真实网络结构,目的是检验bootstrap平均模型的有效性,即将bootstrap方法中高置信度特征与真实网络中的特征进行比较,若bootstrap平均模型包含了大部分原真实网络中存在的边,并具有较高置信度,则能说明该方法的有效性。
1.模拟实验1
根据已给定的网络结构产生相应的模拟数据(参见图1)。网络包含7个随机变量(节点)和7条边,根据此网络结构产生10000个观测,其中变量均服从正态分布。每次从数据集中随机抽出100例样本作为结构学习的数据集,重复抽样得到100个数据集,分别用典型的贝叶斯网络结构学习方法以及贝叶斯网络的bootstrap方法分别对一个数据集进行结构学习得出网络,重复实验100次。
采用基于信息准则的BIC得分函数确定最优网络[4],搜索过程采用贪婪爬山法,bootstrap重抽样次数为300次。为了避免陷入局部最优,在搜索过程中结合随机重启搜索。通过这个过程尝试寻找能使得分提高最多的网络结构,直到结构的改变无法继续提高得分为止。一旦爬山法陷入局部最优,算法将随机扰动网络结构中的边(添加、删除和反向)并重新开始搜索。在重启一定次数后终止搜索,选出得分最高的网络作为结果。最后,根据设定的三个不同的阈值t=0.5,0.7,0.9,将pN(f)≥t的所有连接边输出得到最终结果网络。模拟使用R软件包bnlearn[6]和编程实现。
评价构建网络的指标分别使用真阳性数目、假阳性数目、假阴性数目、真阴性数目、灵敏度、特异度和准确度,其中准确度为真阳性边占阳性边的比例,相当于诊断试验中的阳性预测值。计算这些指标时需要对100次实验的结果取平均值,表1给出了使用不同方法和取不同阈值的网络评价结果,即分别使用普通的贝叶斯方法(origin)和取不同阈值t的基于bootstrap的贝叶斯方法。
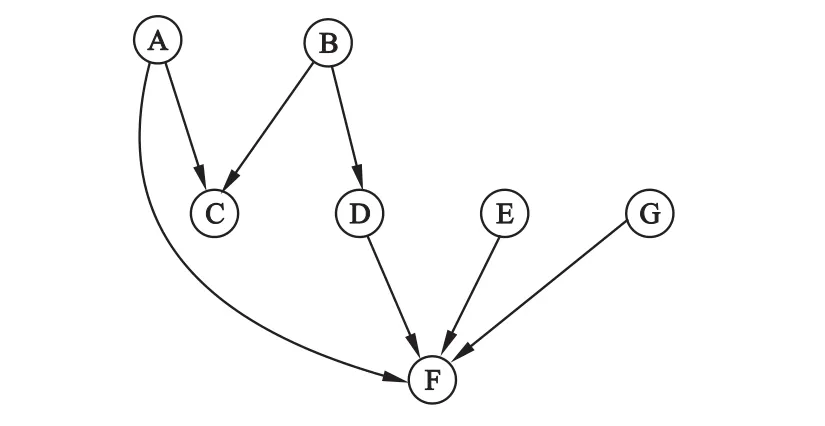
图1 模拟实验1的真实网络关系图
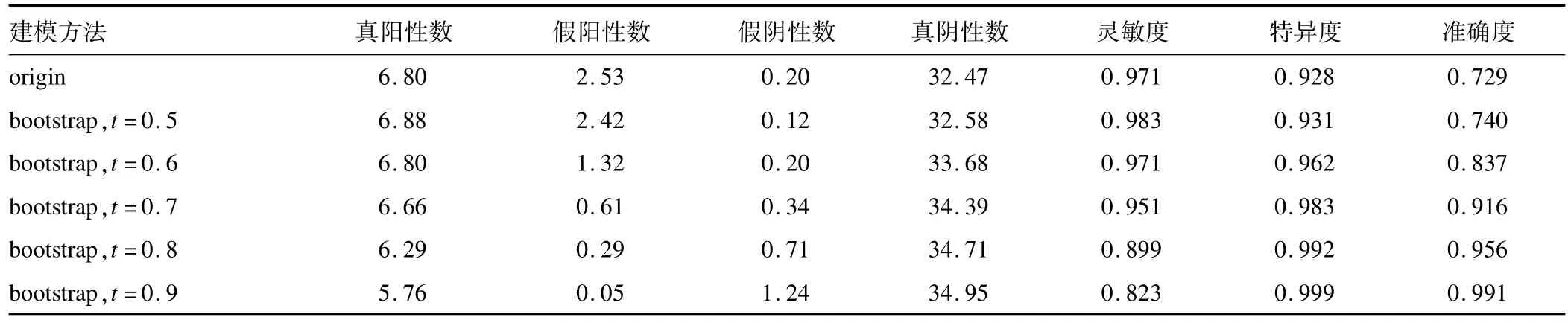
表1 使用不同方法和取不同阈值的网络评价结果
2.模拟实验2
使用ICU-Alarm网络模拟数据。该数据产生于ICU-Alarm网络模型,此模型是机器学习中网络学习问题的经典模型,广泛应用于评价网络学习方法。ICU-Alarm网络模型包含37个随机变量,46条边。在样本量N=100,300,600,1000下比较网络学习的结果,每个样本量下抽取100个数据集,重复实验100次。然后,分别使用普通的贝叶斯网络模型和基于bootstrap的贝叶斯方法构建网络,并对其进行评价。
模拟实验评价结果见图2。结果显示:使用基于bootstrap的贝叶斯方法得到网络模型明显优于使用普通贝叶斯网络模型。同时可以看到,当样本量增加时,构建的网络的结构学习越来越准确,即真阳性边增加,假阳性和假阴性的边减少;另外,提高阈值,真阳性和假阳性边减少,但容易漏掉真实边,说明合理设定阈值的必要性。由于真实边(46条)相对于网络所有可能边(1332条)要少很多,因此不同方法的特异度均接近于1,假阳性率均低于3%。
实例分析
为了研究卵巢癌的分子生物学机制,本研究通过对卵巢癌患者基因表达数据进行分析并构建贝叶斯网络,从网络中得出基因之间的调控关系,并结合生物功能和通路数据库查询以及查阅文献,对网络进行生物学解释,从基因组学的角度为卵巢癌的发病机制提供线索[7]。
本研究从TCGA数据库下载570例卵巢癌患者基因表达谱数据,以及8例健康对照数据[8]。全基因组表达谱数据一共测得12042个基因的表达值,由于基因的数目过多,需要先筛选出与卵巢癌相关的基因,再对这部分基因构建贝叶斯网络。对分析变量的筛选不仅能提高建模的效率,也使构建的网络更加合理,有助于对其进行解释。本研究使用基于Wilcoxon秩和检验的置换检验[8],进行1000次置换,筛选出P<0.05(校正后)的基因一共744个。继而,对这部分基因进行KEGG通路富集分析,结果有12个基因显著富集在p53信号通路中。
将映射上这个通路的12个基因的表达数据整理出来,并对数据构建贝叶斯网络。贝叶斯网络的搜索过程采用贪婪爬山搜索法,再结合bootstrap重抽样方法对网络特征进行置信度估计,重抽样次数设为1000次以保证结果的稳定性。将阈值设定为0.8,结果如图3所示,其中节点代表富集于p53信号通路中的基因,灰色的节点代表枢纽基因(Hub Gene),信度大于0.8小于1的边用虚线表示,信度等于1的边用实线表示。
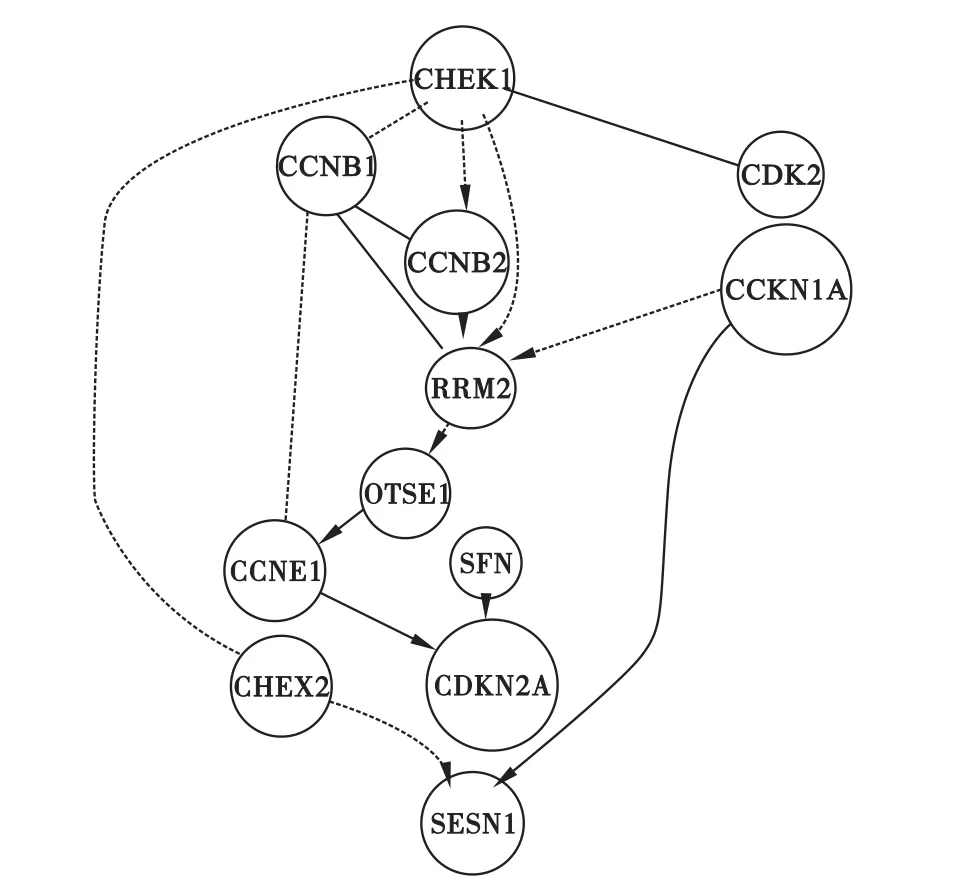
图3 利用bootstrap方法构建的卵巢癌基因调控网络
为了验证bootstrap方法置信度评价的可信程度,本研究通过随机重排列每个基因的测量值产生一个新的数据集。在这样一个数据集中,基因彼此之间是独立的,所以我们并不期望能从中找出真实的边。具体做法如下,对每个基因下的所有测量值随机打乱顺序产生新的数据集,对此数据进行学习构建贝叶斯网络,结合bootstrap方法得出每条边的置信度,重复100次这样的实验。结果见图4,图中实线为真实数据下不同置信度水平下有向边的数目,虚线表示随机重排列数据下有向边的数目,横轴表示置信度阈值,纵轴表示大于等于对应置信度阈值的有向边数目。正如预期,对随机数据集构建的网络中边的置信度普遍比较低。如图4所示,比较原始数据集和随机数据集在不同置信度下的边数,可以看出原始数据的边数分布在高置信度区域有更长更重的尾部。当置信度大于0.2时,两条分布曲线出现间隔,随着置信度的增大间隔也越来越大,即在原始数据上得到的网络关系具有一定的可信度,说明贝叶斯网络的bootstrap估计方法确实能够发现大量的网络关系。
图3中构建的贝叶斯网络反映了基因之间的调控关系,通过查询已有的基因/蛋白互作网络数据库[9-10](如STRING,GENEMANIA等),贝叶斯网络中基因调控关系80%以上得到支持。图3中RRM 2基因受多个基因调控,可以将它定义为枢纽基因。已有大量文献报道该基因与卵巢癌的诊断,预后以及化疗有关[11-12],它所编码的蛋白构成氧化还原酶,能催化核苷酸还原成脱氧核苷酸的反应,为DNA合成提供前体准备。图3中另外一个枢纽基因CHEK1调控多个基因,该基因与卵巢癌有密切联系[13-14],其编码的蛋白属于丝氨酸/苏氨酸蛋白激酶家族,在DNA损伤反应中起着重要作用。
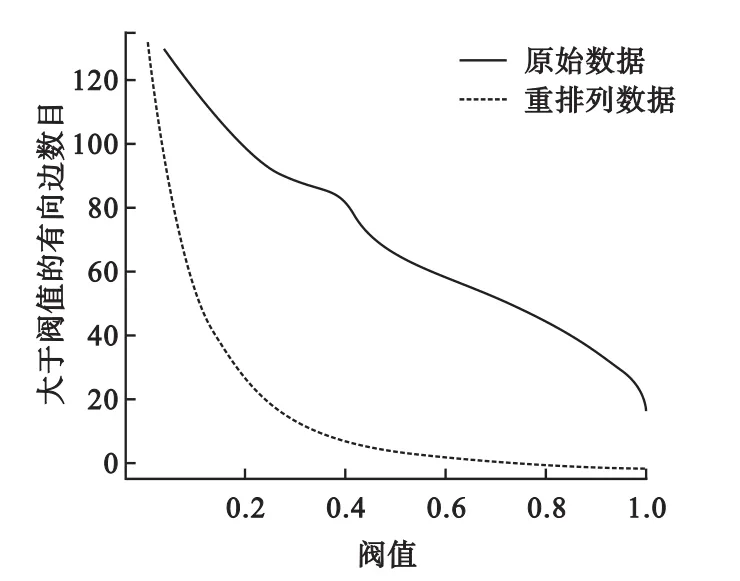
图4 100次重复实验不同阈值下网络中边的数目比较
讨 论
本研究的目的是验证贝叶斯网络的bootstrap估计方法的性能,并将其应用于癌症基因组数据的分析,揭示基因之间的调控关系。通过模拟实验将贝叶斯网络bootstrap方法与一般结构学习方法的结果进行比较,验证了改进后方法的性能。此外,贝叶斯网络bootstrap方法能给出网络中边的置信度,这可以为研究者提供更多的信息。通过模拟实验我们检验了置信度可以作为评价特征真实性的度量。本研究得出以下几点结论:①bootstrap估计是谨慎可靠的,在高置信度的情况下网络几乎不包含假阳性。②当数据集样本量较少(相对其所要推断的模型复杂度)时,本文给出的bootstrap方法相比原始方法新方法能够得出更准确的结果。③阈值的设定十分重要,它直接影响最终结果,要根据实际情况设置,如果实际中想发现更多的网络关系,可选较小的阈值(如t=0.3),如果想得到更可信的网络关系,则应选取大的值(如t=0.7)。总之,建立生物学网络可以更好地验证差异变量,揭示变量之间的因果关系,本文将bootstrap方法应用于贝叶斯网络估计,获得了较为理想的结果,更深入的问题有待进一步研究。
1.游项云,李康.贝叶斯网络方法在基因调控研究中的应用.中国卫生统计,2009,26(1):83-86.
2.范丽珺,游顶云,张旺,等.贝叶斯因果关系网络模型在断面调查数据中的应用.中国医院统计,2010,17(2):97-100.
3.虞慧婷,吴骋,柳伟伟,等.基于贝叶斯网络的原发性肝癌预后影响因素相互关系研究.中国卫生统计,2008,25(1):10-14.
4.Friedman N,Goldszmidt M,Wyner A.Data analysis with Bayesian networks:A bootstrap approach.Proceedings of the Fifteenth conference on Uncertainty in artificial intelligence.Morgan Kaufmann Publishers Inc.,1999:196-205.
5.Broom BM,Do KA,Subramanian D.Model averaging strategies for structure learning in Bayesian networks with limited data.BMC Bioinformatics,2012,13(Suppl 13):S10.
6.Scutari M.Learning Bayesian networks with the bnlearn R package. arXiv preprint arXiv:0908.3817,2009.
7.Friedman N,Linial M,Nachman I,Pe’er D.Using Bayesian networks to analyze expression data.J Comput Biol,2000,7(3-4):601-620
8.Bell D,Berchuck A,Birrer M,et al.Integrated genomic analyses of ovarian carcinoma.Nature,2011,474(7353):609-615.
9.Warde-Farley D,Donaldson SL,Comes O,et al.The GeneMANIA prediction server:biological network integration for gene prioritization and predicting gene function.Nucleic Acids Res,2010,38(Web Server issue):W 214-220.
10.Franceschini A,Szklarczyk D,Frankild S,et al.STRING v9.1:proteinprotein interaction networks,with increased coverage and integration. Nucleic Acids Res,2013,41(Database issue):D808-815.
11.Ferrandina G,Mey V,Nannizzi S,et al.Expression of nucleoside transporters,deoxycitidine kinase,ribonucleotide reductase regulatory subunits,and gemcitabine catabolic enzymes in primary ovarian cancer. Cancer Chemother Pharmacol,2010,65(4):679-686.
12.Zhang M,Wang J,Yao R,et al.Small interfering RNA(siRNA)-mediated silencing of the M2 subunit of ribonucleotide reductase:a novel therapeutic strategy in ovarian cancer.International Journal of Gynecological Cancer,2013,23(4):659-666.
13.Connell CM,Shibata A,Tookman LA,et al.Genomic DNA damage and ATR-Chk1 signaling determine oncolytic adenoviral efficacy in human ovarian cancer cells.J Clin Invest,2011,121(4):1283-1297.
14.Kumar G,Breen EJ,Ranganathan S.Identification of ovarian cancer associated genes using an integrated approach in a Boolean framework. BMC Syst Biol,2013,7:12.
(责任编辑:邓 妍)
The Application of Bayes Network Structure Learning Algorithm Based on Bootstrap Method to the Construction of Gene Regulatory Networks
Li Hailong,Hou Yan,Ke Chaofu,et al.(Department of Medical Statistics,Harbin Medical University(150081),Harbin)
ObjectiveTo explore the performance of Bayes network structure learning algorithm based on bootstrap method in network construction,and to apply it to the analysis of ovarian cancer gene expression data.MethodsThe efficiency of the algorithm given in this article was testified with simulation data and gene expression data,and meanwhile this algorithm was used to construct gene regulatory networks.ResultsBayes network structure learning based on bootstrap method performed better than the general Bayes network in the case of small sample sizes,as shown in simulation tests;the results of gene expression data analysis also indicated that this algorithm could provide valuable network structures.ConclusionBayes network structure learning algorithm based on bootstrap method can establish highly precise network models even with small sample sizes,and meanwhile provide the confidence estimates of each edge in the network.
Bayes network;Structure learning;Bootstrap
*高等学校博士学科专项基金(2012230711004);国家自然科学基金(81172767)
△通信作者:李康,E-mail:likang@ems.hrbmu.edu.cn

