Taqman探针荧光聚合酶链式反应实时同步鉴定动物源性食品中猪肉、鸡肉源性成分
金萍++结莉++陆俊++陈英++丁洪流


摘 要:目的:建立一种能实时同步检测动物源性食品中猪肉源性和鸡肉源性成分的Taqman探针双重荧光聚合酶链式反应(polymerase chain reaction,PCR)方法,应用于动物源性食品的成分掺假快速检验。方法:分别依据猪和鸡的种间保守基因(Cytb)序列设计、合成特异性引物及不同荧光标记(FAM、HEX)的Taqman探针,建立可同步检测动物源性食品中的猪源性和鸡源性成分的Taqman探针双重荧光PCR方法。结果:所建立的Taqman探针双重荧光PCR检测方法特异性强,仅对猪、鸡成分有扩增;灵敏度高,最低检测到猪源、鸡源DNA的含量分别为0.02、0.10 ng;抗干扰性强,当DNA混样中猪源、鸡源性成分含量在2%以上水平时,所建立的混合检测体系均能对DNA混样给出正确判断。结论:所建立的混合检测体系具有高特异性、高灵敏度,能够适用于动物源性食品中猪肉、鸡肉成分的同时快速检测。
关键词:双重荧光聚合酶链式反应;动物源性食品;猪源性成分;鸡源性成分
A Dual Fluorescent Real-Time Polymerase Chain Reaction Method for Concurrently Detecting Pork-Derived and Chicken-Derived Ingredients in Animal-Origin Foods
JIN Ping, JIE Li, LU Jun, CHEN Ying, DING Hongliu*
(Suzhou Quality Supervision and Inspection Center, Suzhou 215128, China)
Abstract: Objective: To establish a dual fluorescent real-time polymerase chain reaction (PCR) assay for the simultaneous detection of pork-derived and chicken-derived ingredients which enables rapid detection of adulteration in animal-origin foods. Methods: With this aim, specific primers and TaqMan probes were designed according to the interspecific conservative sequences. Results: The dual fluorescence PCR method had good specificity and could only amplify pork-derived ingredients and chicken-derived ingredients. The method had high sensitivity and could detect as low as 0.02 ng of porcine DNA and 0.10 ng of chicken DNA. It had strong anti-interference ability and could correctly detect more than 2% pork-derived and chicken-derived ingredients spiked in mixed DNA samples with other species. Conclusion: The dual fluorescent real-time PCR provides a specific and sensitive method for the detection of pork and chicken-derived ingredients in animal-origin foods.
Key words: dual fluorescent real-time polymerase chain reaction (RT-PCR); animal-origin product; pork-derived ingredients; chicken-derived ingredients
DOI:10.15922/j.cnki.rlyj.2016.09.004
中图分类号:TS207.3 文献标志码:A 文章编号:1001-8123(2016)09-0017-06
引文格式:
金萍, 结莉, 陆俊, 等. Taqman探针荧光聚合酶链式反应实时同步鉴定动物源性食品中猪肉、鸡肉源性成分[J]. 肉类研究, 2016, 30(9): 17-22. DOI:10.15922/j.cnki.rlyj.2016.09.004. http://rlyj.cbpt.cnki.net
JIN Ping, JIE Li, LU Jun, et al. A dual fluorescent real-time polymerase chain reaction method for concurrently detecting pork-derived and chicken-derived ingredients in animal-origin foods[J]. Meat Research, 2016, 30(9): 17-22. (in Chinese with English abstract) DOI:10.15922/j.cnki.rlyj.2016.09.004. http://rlyj.cbpt.cnki.net
牛肉享有“肉中骄子”之美称,是中国人的第二大肉类食品。牛肉不仅味道鲜美、消化吸收率高,同时具有高蛋白、低脂肪、富含多种氨基酸以及矿物质等特点,受大众喜爱。牛肉的相对高价促使一些不法分子制作假牛肉,如2013年内蒙古包头公安破获的一起制售假劣食品案,涉案公司经过调查发现,采用鸭等原材料生产假冒的牛肉干、羊肉干,在15 个省市自治区进行销售,涉案价值600多万[1]。2011年人民网报导江苏无锡的犯罪分子利用硼砂和猪血等物品将猪肉伪装成牛肉出售;2013年西安通报了一起“9.10”特大制贩假牛肉案,共查处17 t假牛肉,这些假牛肉其实均为猪肉[2]。食品掺假是指向食物中非法混进外观、物理性状或物种形态相似的物质,混入后的物质在外观上很难鉴别[6]。随着目前食品检测技术不断完善和更新,食品掺假的手段也越来越隐蔽,主要手段可总结为“注”、“混”、“改”、“充”、“提”[3]。肉类掺假主要采用“混”,即以一些低价肉(如猪肉、鸡肉)代替/部分替代高价肉(如牛肉、羊肉),再在高价肉中加入各种调料、香精基料等化学品进行遮掩。由于替代成分与被替代成分均同属肉类,两者在感官、理化特性等方面极其相似,另外肉类制品大都经过高压、煮沸、腌制等加工程序,使得食品本身复杂多变。因此这类型的食品掺假鉴定变得非常困难,也成为食品质量安全保障方面的技术瓶颈之一[4]。
荧光定量聚合酶链式反应(polymerase chain reaction,PCR)方法具备特异性高、样本需求低(少量受检样品即可检测)、耗时短、操作过程简单,同时具有无污染(闭管操作)、无毒性、高通量、多重扩增、可定量分析的优点。因此,对于实时掺假食品的监督和管理,应用以DNA为靶分子的实时荧光PCR技术是未来发展方向之一[15]。通过前期对江苏苏州地区32 家生产单位共计90 份样品的掺假鉴定[5],发现目前不法分子主要是用猪肉以及鸡肉制作假牛肉、假羊肉,因此本研究目的在于建立一种能同时检测动物源性食品中猪源性和鸡源性成分的双重荧光PCR方法,其中涉及2 个扩增体系:猪源特异性扩增体系、鸡源特异性扩增体系。2 个体系的探针用不同的荧光基团标记,在同一反应管内同时进行鉴定,节省检测中试剂的损耗以及时间。
1 材料与方法
1.1 材料与试剂
Taqman反应预混液(ABI Taqman Universal PCR master mix) 英潍捷基(上海)贸易有限公司;天根血液/组织DNA提取试剂盒、焦碳酸二乙酯(diethy pyrocarbonate,DEPC)处理水 苏州阿尔法科技有限公司;引物、Taqman荧光探针 生工(上海)生物工程股份有限公司合成;无水乙醇 美国Thermo-Fisher公司。
1.2 仪器与设备
台式高速冷冻离心机 美国Thermo-Fisher公司;核酸蛋白测定仪(酶标仪) 美国Bio-Tek公司;7500型实时荧光PCR仪 美国ABI公司;旋涡振荡器 德国IKA公司;槽恒温水浴锅 韩国Daihan Scientific公司。
1.3 方法
1.3.1 基因组DNA模板制备
使用一次性组织研磨棒、剪刀、镊子等器具对样品进行均质处理,保证样品的均匀性;不同的样品分开处理,器具使用前后均用酒精棉球擦净,避免不同样品间的交叉污染。按照血液/组织DNA提取试剂盒说明书的步骤提取样品DNA。采用微量核酸蛋白测定仪测定模板DNA的浓度以及纯度,OD260 nm/OD280 nm比值均在1.7~2.0之间的动物组织DNA纯度达到要求,对模板DNA进行分装,冷冻备用。
1.3.2 引物、探针的设计、合成及有效性验证
在Genbank数据库(http://www.ncbi.nlm.nih.gov/)中搜索,得到猪、鸡线粒体细胞色素b基因(Cytb)序列,利用引物探针设计软件(Primer Express 3.0),依据荧光PCR扩增中引物探针设计的相应原则,获得猪、鸡成分特异性的引物和探针。同时将设计得到的引物、探针在NCBI数据库中进行BLAST比对,确定其理论特异性。
引物、探针序列:
猪上游引物:5-GAAAAATCATCGTTGTACTTCAACTACA-3;
猪下游引物:5-GGTCAATGAATGCGTTGTTGAT-3;
猪探针:5-FAM-CAAACATCCGAAAATCACAC CCACTAAT-TAMRA-3。
鸡上游引物:5-CATCTCATCCGACTCTGACAAAATT-3;
鸡下游引物:5-GGGAGAATAGGGCTAGTGTTAGGAA-3;
鸡探针:5-HEX-TCAAAGACATTCTGGGCTTAACTCTC ATACTCACC-TAMRA-3。
1.3.3 双重实时荧光PCR检测
扩增反应体系的体积为50 μL:Taqman PCR master mix 25 μL;10 μmol/L的上游引物1 μL;10 μmol/L的下游引物1 μL;10 μmol/L的荧光标记探针1 μL;模板DNA(0.001~50ng/μL)2 μL;其余不足用DEPC处理灭菌双蒸水补齐。反应循环参数:50 ℃,2 min;95 ℃,10 min;95 ℃,15 s;60 ℃,1 min;40 个循环,于实时荧光PCR仪上进行。
1.3.4 特异性实验
按照血液/组织DNA提取试剂盒的说明书步骤分别提取纯牛、羊、猪、鸡、鸭、鱼的DNA,按照1.3.3节所描述的体系进行实时荧光PCR扩增反应,实时PCR扩增结束,通过观察各体系对不同模板DNA的扩增曲线及临界循环值(Ct值)来判定所设计的双重荧光体系的特异性。
1.3.5 灵敏度实验
按照1.3.1节的方法提取纯猪、鸡DNA,用DEPC处理灭菌双蒸水进行稀释,以0.001~50 ng/μL的猪、鸡核酸质量浓度,按照1.3.3节中所描述的体系分别进行实时荧光PCR反应,根据两体系的Ct值及扩增曲线确定所建立的猪-鸡双重荧光PCR体系中猪源、鸡源的检测灵敏度。
1.3.6 混合样品检测能力验证
为进一步检测所建立的Taqman探针双重荧光PCR检测体系的抗干扰性以及其特异性检测能力,确定该体系对混合样品的检测低限,所以决定从DNA核酸水平进行实验确认。取牛、羊、猪和鸡的纯DNA模板,分别按表1~2所示制备各混合DNA样品,进行Taqman探针双重实时荧光PCR,以确定该体系用于混合样品检测的检出限。
2 结果与分析
2.1 Taqman探针双重实时荧光PCR特异性实验检测结果
图1为双重体系中猪源体系的特异性检测结果,该体系采用FAM荧光标记探针进行检测。猪源体系仅对猪源成分产生扩增,与其他肉源(牛、羊、鸭、鱼、鸡)成分无交叉,阴性样品(牛、羊、鸭、鱼、鸡)的Ct值限制在32 个循环以后,猪源检测体系具有较好的特异性。图2为双重体系中鸡源体系特异性的检测结果,该体系采用HEX荧光标记探针进行检测。鸡源体系仅对鸡源成分产生扩增,与其他成分(牛、羊、鸭、鱼、猪)无交叉,阴性样品(牛、羊、鸭、鱼、猪)的Ct值限制在32 个循环以后,鸡源检测体系具有较好的特异性。
从实验结果中可以看出,所建立的Taqman探针双重实时荧光PCR体系对猪、鸡成分产生有效扩增,而对阴性样品无明显扩增且Ct值均限制在32 个循环以后,因此该双重体系对猪、鸡成分具有特异性,同时Ct值≥32.0视为不含有目标成分。
2.2 灵敏度实验检测结果
取纯猪、鸡肉提取DNA,采用微量核酸蛋白测定仪测定获得模板DNA的浓度,用灭菌双蒸水进行10 倍倍比稀释。取各稀释度的核酸2 μL作为模板,进行猪-鸡Taqman探针双重荧光PCR检测。猪源性DNA模板质量浓度依次为10、1、0.1、0.01、0.001 ng/μL,鸡源性DNA模板质量浓度依次为50、5、0.5、0.05、0.005 ng/μL。
猪源检测中DNA模板质量浓度10、1、0.1、0.01、
0.001 ng/μL的Ct值分别为19.6、23.8、26.4、30.1、
>32.0,其中0.01 ng/μL的DNA模板质量浓度的Ct值
30.1<32.0,是该反应体系能检出猪源性成分的最低限;鸡源检测中DNA模板质量浓度50、5、0.5、0.05、
0.005 ng/μL的Ct值分别为18.9、23.7、28.6、30.4、>32.0,其中0.05 ng/μL的DNA模板质量浓度的Ct值30.4<32.0,是该体系能检出鸡源性成分的最低限。因此在保证特异性的前提下,该猪-鸡Taqman探针双重荧光PCR体系最低检测到猪、鸡源DNA的含量分别为0.02、0.10 ng。
2.3 Taqman探针双重荧光PCR混合样品检测能力验证
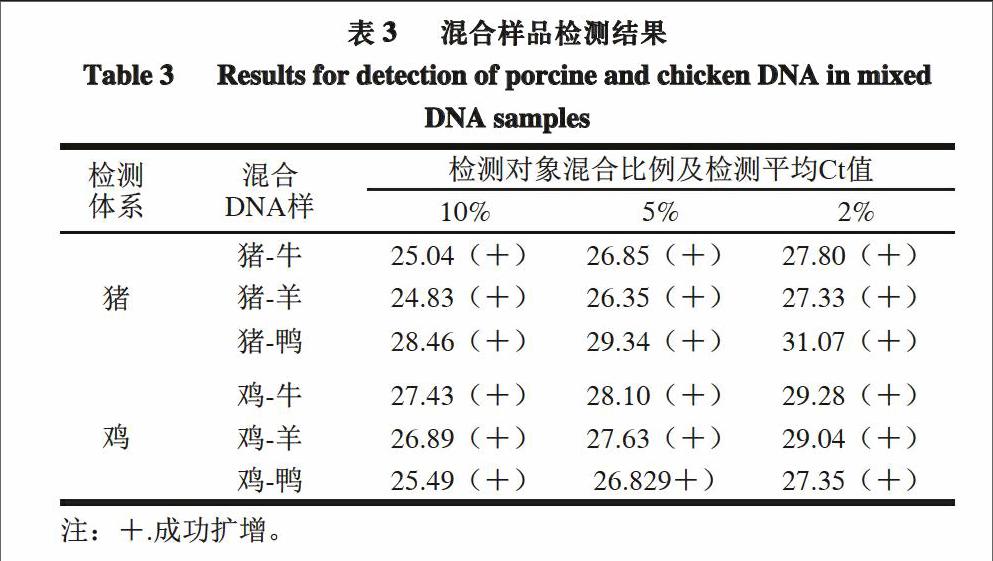
由表3可知,模拟混合样品中所有含有2%~10%猪DNA的混合样都获得了成功扩增,Ct值在31.07~24.83;其中含有2%~10%鸡DNA的混合样也都获得了成功扩增,Ct值在29.28~25.49。结果显示,当DNA混样中猪、鸡含量在2%以上水平时,所建立的混合检测体系均能对DNA混样给出正确判断,DNA混样的猪、鸡成分检测下限为水平2%,证实所设计引物探针体系具有较好的抗干扰能力及较高的灵敏性。
3 讨 论
本研究所建立的双重荧光PCR,特异性强,仅对猪、鸡成分有扩增,Ct值限制在32。该方法灵敏度高,最低检测到猪、鸡源DNA的含量分别为0.02、0.10 ng;抗干扰性强,当DNA混样中猪、鸡含量在2%以上水平时,所建立的混合检测体系均能对DNA混样给出正确判断,适用于动物源性产品中猪、鸡源性成分的鉴别检测,可为食品安全和饲料监管提供有效依据。
与食品标签的食品成分不相符合的食品掺假会误导消费者,对消费者造成各种影响,甚至危害其身体健康,也影响社会的公平竞争和健康发展。当消费者选购食品时,通过观察食品标签就可以知道这种食品的成分,可以根据自己的需要做出合理的判断、选择。食物过敏者通常可以避免含过敏成分的食物,关注健康膳食的人会倾向选择鸡、鸭、鹅等脂肪含量低的家禽肉类代替牛、羊、猪等脂肪含量高的畜肉类,患有某些疾病的患者也需要遵循医嘱忌食或少食某些种类的食物,各种宗教信仰人士会不吃某些特殊的肉类,如牛肉或猪肉,而素食者会禁食任何含肉的食品。食品掺假严重的甚至会威胁到人的生命,如安徽阜阳“大头娃娃事件”中的涉案乳粉,以植物淀粉代替乳粉原料掺假,不仅造成婴儿严重营养不良,而且导致许多婴儿死亡[7]。因此保证食品标签的真实性对于消费者非常重要,而食品掺假的鉴定及检测技术是保障食品标签真实性的关键。
我国新出台的《食品安全法(2015版)》明文规定:禁止生产经营掺假掺杂食品。随着食品质量安全监管部门检测技术的不断完善和更新,食品掺假的手段也在不断提高。如牛乳掺假从最初掺水、尿、淀粉、三聚氰胺到最近的皮革水解蛋白,又如从向肉制品中注水到用猪肉、鸡肉、鸭肉通过浸泡、加香精等方式伪装成牛肉、羊肉。这种用同类物质部分掺假的方法由于其隐蔽性、复杂性,而检测技术、监管体系又相对薄弱,导致被较多使用。Chen等[8]用酶联免疫吸附测定(enzyme linked immunosorbent assay,ELISA)方法从902 种食品中分别检测到了15.9%的生肉类食品及22.9%的熟肉类食品中含有未标明的动物肉类成分。Ayaz等[9]在2006年对100 种肉类食品进行调查研究,研究发现,有22.0%含有标签上未标明的肉源成分,它们中大部分是用禽肉代替牛肉进行掺假。为了维护市场的公平竞争,需要加强食品标签管理及行之有效的食品成分检测方法,建立快速、高效、定量的新的检测体系是食品监管的当务之急。
目前从物理技术、化学技术、免疫学技术、电泳技术以及DNA技术等方面均有对食品成分掺假鉴别的研究。物理技术可以针对肉类结构的不同关键信息进行检测,通过对检测结果的比对,对肉类的品种和品质进行鉴定,特别适合用来评估肉的品质。樊玉霞[10]通过可见-近红外光谱对猪肉肉糜中粗脂肪、肌内脂肪、蛋白质、水分和14 种脂肪酸进行了研究,结合化学计量学方法对猪肉肉糜品质进行研究。化学方法有气相色谱(gas chromatography,GC)、气相色谱质谱联用(gas chromatography-mass spectrometer,GC-MS)、高效液相色谱(high performance liquid chromatography,HPLC)等,GC、GC-MS通过检测甘油三酯C2位置上的饱和脂肪酸和不饱和脂肪酸的比值或某些特定脂肪酸的含量来进行检测鉴别。然而,由于肉制品中脂肪含量的变化范围很宽,因此脂肪分析只能应用于动物来源的脂肪粗略检测。HPLC[11-12]以及免疫学方法中的ELISA法[13]、蛋白质等电聚焦方法[14]等对物种特有的蛋白质或抗原决定簇进行辨别,可有效鉴定未加工生食物原料,但对混合样品或者用香精、香料等处理后的样品则无法准确分析出其中的物种,并且蛋白质热稳定性差,经过加工处理可能会改变其结构,从而破坏物种辨别依据——特有的蛋白质或抗原决定簇,因此不适于加工熟肉制品;而且不同种属之间蛋白质也存在一定的种属交叉反应,尤其是近缘物种间的交叉反应更为严重(例如绵羊和山羊),因此也不适于近缘物种的鉴别检测[15-16]。大量以DNA为检测对象的方法已经用于食品成分造假检测及种类鉴定,如PCR[17-18]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[19-20]、随机扩增多态性DNA标记(random ampli?ed polymorphic DNA,RAPD)[21]、基因芯片技术[22]。基于DNA的种源鉴定检测方法目前最受欢迎,源于DNA分子能耐受食品加工过程中的高温高压等极端条件而保持相对稳定,并被有效扩增[23]。目前,实时探针荧光PCR技术运用荧光探针,利用荧光信号积累实时监测整个过程,具备特异性高、样本需求低(少量受检样品即可检测)、耗时短、操作过程简单同时具有无污染(闭管操作)、无毒性、高通量、多重扩增、可定量分析的优点[24]。因此,荧光PCR技术体系应用于食品掺假的监督和管理将是未来发展趋势。目前国内外已有关于利用实时荧光PCR技术进行肉种来源鉴定的文献报道[25-27],但涉及双重荧光PCR的研究相对较少。
该实验的目的在于建立一种荧光PCR方法,同时检测动物源性食品中猪源性和鸡源性成分,该研究的关键在于物种特异基因的正确选择,测试选定的细胞色素b基因位于线粒体,线粒体DNA是遗传检测特定序列的理想目标,主要原因在于:1)含量高[28]:在所有的组织细胞中均存在大量的线粒体,可以获得大量的线粒体DNA,即使是由于剧烈的加工条件而使得DNA严重破碎的情况下也能获取;2)特异性高[29]:线粒体DNA在不同物种间具有高度的特异性;3)研究成熟:国内外成分鉴定的研究中选择Cytb的较多,技术比较成熟[30-34];4)获取途径便捷:在Genbank上,已经有很丰富的动物Cytb基因序列登录上传,这些在一定程度上给实验的实施提供了简便前提条件。
参考文献:
[1] 史竞男, 邹伟. 公安部公布肉制品犯罪典型案例[N]. 中国食品安全报, 2013-05-04(03).
[2] 王海鹏. 17.5 吨假牛肉全是用猪肉做的[N]. 西安晚报, 2013-9-12(08).
[3] 马志英. 常见的食品掺假方式有哪些[J]. 生命与灾害, 2013(3): 26-29.
[4] WOOLFE M, PRIMROSE S. Food forensics: using DNA technology to combat isdescription and fraud[J]. Trends in Biotechnology, 2004, 22: 222-226. DOI:10.1016/j.tibtech.2004.03.010.
[5] 金萍, 丁洪流, 李培, 等. 2013年苏州地区肉及其制品掺假情况调查[J]. 中国食品卫生杂志, 2014, 26(2): 168-172.
[6] 陈文斌. 微生物发酵碳同位素分馏机理的研究及其应用[D]. 南宁: 广西大学, 2013.
[7] 佟树广. 阜阳“大头娃娃”回访录[J]. 记者观察(上半月), 2008(11): 44-46.
[8] CHEN F C, HSIEH Y H. Detection of pork in heat-processed meat products by monoclonal antibody-based ELISA[J]. Journal of AOAC International, 2000, 83: 79-85.
[9] AYAZ Y, AYAZ N D, EROL I. Detection of species in meat and meat products using enzyme-linked immunosorbent assay[J]. Journal of Muscle Foods, 2006, 17(2): 214-220.
[10] 樊玉霞. 猪肉肉糜品质与安全可见/近红外光谱快速检测方法的实验研究[D]. 杭州: 浙江大学, 2011.
[11] SCH?NHERR J. Analysis of products of animal origin in feeds by detection of carnosine and related dipeptides by high-performance liquid chromatography[J]. Journal of Agricultural and Food Chemistry, 2002, 50(7): 1945-1950. DOI:10.1021/jf0112116.
[12] ESPINOZA E O, KIEMS M A, FILIPEK M S. Identification and quantitation of source from hemoglobin of blood and blood mixtures by high performance liquid chromatography[J]. Journal of Forensic Science, 1996, 41: 804-811.
[13] GRIFFITH N M, BILLINGTON M J. Evaluation of an ELISA for beaf blood serumto determine indirectly the apparent beaf content of beaf joints and model mixtures[J]. Journal of the Science of Food and Agriculture, 1984, 35: 909-914.
[14] SKARPEID H J, KVAAL K, HILDRUM K I. Identification of animal species in ground meat mixtures by multivariate analysis of isoelectric focusing protein profiles[J]. Electrophoresis, 1998, 19: 3103-3109.
[15] 朱雨薇. 肉类掺假检测鉴定技术的研究进展[J]. 食品工业, 2014, 35(11): 242-248.
[16] 卜登攀, 王加启, 贺云霞, 等. 动物源性饲料检测技术研究进展[J]. 中国畜牧兽医, 2008, 35(2): 54-59.
[17] SPYCHAJ A, MOZDZIAK P E, POSPIECH E. PCR methods in meat species identification as a tool for the verification of regional and traditional meat products[J]. Acta Scientiarum Polonorum Technologia Alimentaria, 2009, 8(2): 5-20.
[18] 孙艳华, 张智禹, 牛晋阳, 等. PCR法快速检测熟肉制品中肉类来源[J]. 食品研究与开发, 2010, 31(5): 139-142.
[19] HOPWOOD A J, FAIRBROTHER K S, LOCKLEY A K, et al. An actin gene-related polymerase chain reaction (PCR) test for identification of chicken in meat mixtures[J]. Meat Science, 1999, 53: 227-231.
[20] CHIKUNI K, TABATA T, KOSUGIYAMA M, et al. Polymerase chain reaction assay for detection of sheep and goat meats[J]. Meat Science, 1994, 37: 337-345. DOI:10.1016/0309-1740(94)90051-5.
[21] CALVO J H, ZARAGOZA P, OSTA R. A quick and more sensitive method to identify pork in processed and unprocessed food by PCR amplification of a new specific DNA fragment[J]. Journal of Animal Science, 2001, 79: 2108-2112.
[22] CAWTHORN D M, STEINMAN H A, HOFFMAN L C. A high incidence of species substitution and mislabelling detected in meat products sold in South Africa[J]. Food Control, 2013, 32: 440-449. DOI:10.1016/j.foodcont.2013.01.008.
[23] ARSLAN A, ILHAK O I, CALICIOGLU M. Effect of method of cooking on identification of heat processed beef using polymerase chain reaction (PCR) technique[J]. Meat Science, 2006, 72(2): 326-330. DOI:10.1016/j.meatsci.2005.08.001.
[24] 李富威, 高琴, 张舒亚, 等. 实时荧光PCR方法在食品真伪辨别中的应用[J]. 食品工业科技, 2012, 33(14): 367-370.
[25] DOOLEY J J, PAINE K E, GARRETT S D, et al. Detection of meat species using TaqMan real-time PCR assays[J]. Meat Science, 2004, 68(3): 431-438.
[26] RODR?GUEZ M A, GARC?A T, GONZ?LEZ T, et al. TaqMan real-time PCR for the detection and quantitation of pork in meat mixtures[J]. Meat Science, 2005, 70: 113-120. DOI:10.1016/j.meatsci.2004.04.010.
[27] RODR?GUEZ D, AGOSTINO D, PLA M, et al. Construction strategy for an internal ampli?cation control for real-time diagnostic assays using nucleic acid sequence-based ampli?cation: development and clinical application[J]. Journal of Clinical Microbiology, 2004, 42(12): 5832-5836.
[28] BELLAGAMBA F, MORETI V M, COMINICINI S, et al. Identification of species in animal feedstuffs by polymerase chain reaction-restriction fragment length polymorphism analysis of mitochondrial DNA[J]. Journal of Agricultural and Food Chemistry, 2001, 49: 3775-3781. DOI:10.1021/jf0010329.
[29] UNSELD M, BEYERMANN B, BRANDT P, et al. Identification of the species origin of highly processed meat products by mitochondrial DNA sequences[J]. PCR Methods and Applications, 1995, 4: 241-243.
[30] 张娟, 宗卉, 张利平. PCR-mtDNA技术鉴别检测不同动物肌肉组织和饲料中鸭源性成分[J]. 生物工程学报, 2008, 24(10): 1832-1836.
[31] 冯海永, 刘丑生, 何建文, 等. 利用线粒体DNA Cytb基因PCR-RFLP分析方法鉴别羊肉和鸭肉[J]. 食品工业科技, 2012, 33(13): 319-321.
[32] TARTAGLIA M, SAULLE E, PESTALOZZA S, et al. Detection of bovine mitochondrial DNA in ruminant feeds: a molecular approach to test for the presence of bovine-derived materials[J]. Journal of Food Protection, 1998, 61: 513-518.
[33] WALLACE D C. Mitochondrial DNA variation in human evolution, degenerative disease, and aging[J]. American Journal of Human Genetics, 1995, 57(2): 201-223.
[34] XU X, GULLBERG A, ARNASON U. The complete mitochondrial DNA (mtDNA) of the donkey and mtDNA comparisons among four closely related mammalian species pairs[J]. Journal of Molecular Evolution, 1996, 43(5): 438-446. DOI:10.1007/BF02337515.

