丝状真菌纤维素酶合成诱导及转录调控
张飞,白凤武,,赵心清
1 大连理工大学 生命科学与技术学院,辽宁 大连 116024 2 微生物代谢国家重点实验室 上海交通大学 生命科学技术学院,上海 200240
丝状真菌纤维素酶合成诱导及转录调控
张飞1,白凤武1,2,赵心清2
1 大连理工大学 生命科学与技术学院,辽宁 大连 116024 2 微生物代谢国家重点实验室 上海交通大学 生命科学技术学院,上海 200240
张飞, 白凤武, 赵心清. 丝状真菌纤维素酶合成诱导及转录调控. 生物工程学报, 2016, 32(11): 1481-1495.
Zhang F, Bai FW, Zhao XQ. Induction and regulation of cellulase expression in filamentous fungi: a review. Chin J Biotech, 2016, 32(11): 1481-1495.
利用廉价可再生木质纤维素资源水解产生的可发酵糖生产生物能源和生物基化学品是近年来国内外研究的热点。纤维素酶酶解是木质纤维素原料生物降解的重要手段,但目前纤维素酶生产成本过高,限制了纤维素生物转化和生物炼制的工业化应用。对丝状真菌纤维素酶基因表达和调控进行研究,有利于进一步选育纤维素酶高产菌株,降低纤维素酶生产成本。随着高通量测序及丝状真菌遗传操作等技术的进步,对丝状真菌纤维素酶诱导和基因表达调控机理有了更深入的认识。本文综述了近年来丝状真菌纤维素酶诱导和纤维素酶基因表达调控的最新进展,重点论述糖转运蛋白、转录因子和染色质重塑对纤维素酶表达调控的影响,并对利用人工锌指蛋白进行丝状真菌纤维素酶诱导调控研究进行了展望。
纤维素酶,基因表达调控,转录因子,里氏木霉
利用可再生生物质资源生产生物能源和生物基化学品对实现经济可持续发展具有重要意义。由于化石能源的不可再生,以及其燃烧利用后产生的全球变暖等环境效应,发展生物能源作为化石能源的替代品受到普遍关注。木质纤维素生物质是地球上储量丰富的廉价可再生生物质资源,通过其降解及发酵产生燃料乙醇,是国内外学者关注的热点[1]。木质纤维素主要由纤维素、半纤维素和多酚类的木质素组成,这些成分构成紧密交联的天然屏障,这种紧密的结构使生物质具有极强的抗逆性,因此需要利用木质纤维素降解酶进行水解,释放可发酵糖。目前限制木质纤维素原料高效生物转化的瓶颈之一是纤维素降解酶的生产成本较高,因此,研究如何提高纤维素酶的高效生产成为当前生物技术研究的重要内容。
对纤维素的高效分解和利用的微生物几乎分布在整个真菌界中,因此真菌被认为是自然界中纤维素物质的主要降解微生物。丝状真菌分泌的纤维素酶是一类聚合酶,可以通过酶催化模式与结构特征将其酶系分为3类,分别是内切葡聚糖酶,或称1,4-β-D-葡聚糖-葡聚糖水解酶 (EC3.2.1.4),简称EG (Endo-glucanase);外切葡聚糖酶,或称1,4-β-D-葡聚糖-纤维二糖水解酶 (EC3.2.1.91),简称CBH (Cellobiohydrolase)和β-葡萄糖苷酶 (EC3.2.1.21),简称BGL (β-glucosidase),上述主要3类纤维素酶通过协同作用来对纤维素进行降解[2]。纤维素降解除了纤维素酶起主要作用,一些辅助蛋白对纤维素的辅助作用近年来也受到普遍关注,这些辅助蛋白包括膨胀素 (Swollenin)[3]和溶解性多糖单加氧酶 (Lytic polysaccharide monooxygenases, LPMOs)[4]。当前研究较深入的产纤维素酶真菌主要是里氏木霉Trichoderma reesei、草酸青霉Penicillium oxalicum、黑曲霉Aspergillus niger和粗糙脉孢菌Neurospora crassa等[5],其中里氏木霉是工业上的主要纤维素酶产生菌[6]。过去几十年,对丝状真菌纤维素酶的诱导及调控有了初步的了解,国内学者近年来也对丝状真菌纤维素酶诱导物及调控机理进行了综述[7-9],但目前对2013年以来报道的结果涉及很少,而近两年来,大量最新研究结果进一步揭示了丝状真菌诱导产生纤维素酶的新机理。本文对纤维素酶的诱导及表达调控的最新进展进行了全面综述,将碳信号与纤维素酶诱导和转录调控等过程整合,系统整理了纤维素酶诱导表达调控的分子机理。
1 纤维素酶初始诱导
木质纤维素底物常以不可溶形式存在,影响了其直接被吸收利用。因此真菌需要在纤维素底物存在时形成基本的纤维素酶来完成底物的初步降解和胞内纤维素酶基因的大量诱导,这个过程是真菌在自然界中生存的基础。目前实现初始诱导有几种不同假说。第一种假说是少量的特定纤维素酶本底组成型表达,并初步对纤维素进行降解,释放少量的寡糖,这些寡糖进入细胞内诱导后续的纤维素酶合成。该假说的依据是当有抗纤维素酶抗体存在时,可以阻止细胞内纤维素酶诱导形成[10];另外,里氏木霉在非诱导条件下,可以检测到少量纤维二糖水解酶Cel7A和Cel7B[11]。第二种假说是分子孢子表面存在一系列不同的纤维素降解酶可参与纤维素底物的最初降解,这些酶的去除能严重影响孢子在纤维素条件下的萌发过程[12],在没有外界诱导物存在下,里氏木霉孢子初始萌发阶段也检测到了大量糖苷水解酶基因的转录[13]。其次,在碳源耗尽20−30 h后,从菌丝体中可以检测到纤维素酶的转录[14],但这些转录不是简单因为碳代谢阻遏效应去除的结果,可能是由于真菌细胞壁中释放的多糖诱导纤维素酶产生。
通过初始纤维素降解会产生寡糖片段、纤维二糖和葡萄糖,其中寡糖和纤维二糖可作为特异诱导物来激活后续大量纤维素酶的表达分泌。葡萄糖会产生较强的碳代谢阻遏效应,无法有效激活纤维素酶表达。纤维二糖可诱导里氏木霉纤维素酶的产生,在纤维素培养基中释放的纤维二糖被胞外β葡萄糖苷酶降解的效率不如进入细胞内转化为诱导物效率高,更容易作为诱导物被利用[15]。里氏木霉中主要纤维素酶可以通过纤维素和一系列二糖衍生物,包括乳糖、纤维二糖和槐糖进行诱导,尤其槐糖对里氏木霉纤维素酶的诱导能力最显著,相反果糖、葡萄糖和甘油却没有诱导效果[14]。里氏木霉基因组中至少有11个β葡萄糖苷酶,主要分布在糖苷水解酶1和3家族[16],其中胞内的β葡萄糖苷酶转糖苷作用对诱导物的形成很关键[17],最新研究结果显示,β葡萄糖苷酶BGLII的V409F突变体β葡萄糖苷酶活性降低,反之转糖苷活性明显提高,将该突变体转入里氏木霉后,纤维素酶活性也相应提高[18]。当培养基中加入β葡萄苷酶抑制剂野艽霉素 (Nojirimycin)后,槐糖的诱导能力消失,推测最终的诱导过程可能还有其他转糖苷产物参与[19]。因此,丝状真菌产纤维素酶的真正诱导物,以及如何诱导还需要进一步深入研究。
2 糖转运蛋白
纤维二糖和其转糖苷产物寡糖都可以作为里氏木霉的纤维素酶诱导物,而寡糖和纤维二糖进入细胞时,糖转运蛋白对这些诱导物的运输和功能行使起到关键的作用。糖转运蛋白在纤维素酶诱导过程中的作用早有报道[20]。在草酸青霉中分别过表达CdtC、CdtD和CdtG,可以明显提高纤维素酶产量[21]。通过对粗糙脉孢菌在纤维素条件下的转录组数据进行分析,发现了2个重要的糖转运蛋白CDT-1和CDT-2,其缺失突变体在微晶纤维素上无法诱导产生纤维素酶。除了功能性转运蛋白之外,还有一类膜蛋白具有与糖转运蛋白相同的蛋白结构,但是这类蛋白不具有糖转运的功能,机能上倾向于感受纤维素诱导信号,为了验证这2个糖转运蛋白的碳信号感应功能,研究人员突变了CDT-1和CDT-2中糖转运功能关键氨基酸残基,发现二者的突变体对纤维二糖下的纤维素酶诱导能力没有影响,说明这2种糖转运蛋白不但具有转运寡糖功能,对纤维素酶诱导信号的介导也具有重要作用[22]。最近在粗糙脉孢菌中发现的CLP1蛋白,其与CDT-2蛋白序列高度同源,结构预测也是一种糖转运蛋白。通过实验分析发现CLP1没有糖转运蛋白功能,其更多参与到了纤维素酶诱导信号转导过程中。同时敲除CDT-2、CLP1和胞内三类β葡萄糖苷酶基因的突变体菌株,可使纤维二糖诱导能力提高到出发菌株的6.9倍,CLP1和CDT-2共同作用来识别和传递纤维二糖信号,并控制纤维素酶诱导途径下游基因的表达[23]。
以上这种碳信号识别效应在里氏木霉中也有存在,在里氏木霉中发现的糖转运蛋白Stp1和Crt1在诱导物进入细胞和纤维素酶诱导过程中发挥不同的作用。stp1基因的敲除会大量激活在纤维素培养基中的纤维素酶诱导产生,在纤维二糖培养基中表现出了相反的效果,但在槐糖条件下却没有影响,说明Stp1的转运在纤维素酶诱导信号的传递过程中没有起到积极的作用[24]。而另外一个糖转运蛋白Crt1尽管也定位在细胞膜上,但其表现出了不同的趋势,Crt1对纤维二糖与槐糖的转运与消耗并没有影响,但对诱导物乳糖的转运起到了关键的作用,Crt1的缺失使细胞无法正常利用乳糖来生长,而且同时也会使乳糖和槐糖的纤维素酶诱导效果消失[25],说明糖转运蛋白在纤维素酶诱导过程中不仅仅起到了转运寡糖的作用,而且在碳信号识别、传递功能上也会发挥很关键的作用。
3 转录调控因子
丝状真菌内纤维素酶基因的表达多数受转录因子调控,这些转录因子响应来自外界的碳源诱导物传递的信号,从而对纤维素酶产生激活或者抑制的效应。当前研究较深入的里氏木霉中至少存在3个正调控转录因子Xyr1、Ace2、Ace3和2个负调控转录因子Cre1和Ace1参与纤维素酶的调控。
里氏木霉中Xyr1是纤维素和半纤维素酶最主要的转录调控因子,Xyr1过表达可以提高纤维素酶转录表达,其缺失导致所有已知诱导物失去诱导效果[26]。通过对Xyr1敲除转录组分析发现,在不同碳源诱导条件下,Xyr1转录因子的变化可以影响部分转录因子、转运蛋白家族蛋白和大部分糖苷水解酶转录水平,并影响纤维素酶的诱导产生[27],而且Xyr1进入细胞核发挥作用必须要有诱导物存在[28]。Xyr1是Zn2Cys6型锌双核簇转录因子,可结合特定核苷酸5′-GGC (A/T)4并行使调控功能,而在FTFRMH区域中的点突变 (A824V),不但会显著提高菌株纤维素酶的诱导能力,还会消除木聚糖酶的诱导效应[29]。在槐糖诱导物存在时,Xyr1进入细胞核内,诱导停止后Xyr1蛋白迅速降解[28],但目前还不清楚转录激活因子Xyr1如何从不同的诱导物中接受诱导信号。对黑曲霉、构巢曲霉、里氏木霉、禾谷镰刀菌和稻瘟病菌中的Xyr1基因功能分析发现,在木聚糖诱导物中Xyr1转录因子对糖苷水解酶的诱导调控具有实质性差异,在里氏木霉和黑曲霉中XlnR/Xyr1对纤维素酶调控有主要影响,构巢曲霉和稻瘟病菌中纤维素酶基因调控不受XlnR的影响,而禾谷镰刀菌中XlnR的缺失会提高其纤维素酶基因的转录和表达[30]。研究表明曲霉中的Xln1/R不同程度的磷酸化决定其功能,Noguchi等研究表明米曲霉中Xyr1转录因子同源基因Xln1,没有木糖时Xln1具有不同的磷酸化程度,当培养基中木糖增加时,Xln1的磷酸化程度增加[31];黑曲霉中的XlnR也通过木糖诱导,被磷酸化后可进入细胞核,发挥转录调控功能[32]。
里氏木霉中除了Xyr1,锌双核簇转录因子Ace2对纤维素酶起到正调控作用。体外实验表明Ace2可以结合Cel7A启动的GGCTAATAA核酸序列,Xyr1也可结合这个靶位点,同时在Xyr1基因的启动子区域发现Ace2的潜在结合位点,推测可能是Ace2可以微调Xyr1的表达来响应不同的诱导信号。Ace2基因被敲除后会降低不同纤维素酶的转录量,并且在纤维素诱导条件下总酶活会下降30%−70%,但在槐糖诱导时,Ace2的缺失对纤维素酶酶活没有影响[33]。另外Ace2需要经过磷酸化并以二聚体形式发挥调控功能[34],而在其他丝状真菌中没有发现Ace2的同源基因,说明了里氏木霉调控纤维素酶表达的特异性。对里氏木霉基因组分析发现,其与其他丝状真菌不同之处是纤维素酶和其他糖苷水解酶定位在一些分散的基因簇中,转录调控因子基因经常与β葡萄糖苷酶和转运蛋白基因聚集在一起[35]。Häkkinen等利用这个特点鉴定出了几个新的纤维素酶转录激活因子,其中Ace3在乳糖培养基中可显著诱导纤维素酶基因表达,敲除该基因会同时抑制纤维素酶、半纤维素酶的诱导表达[36]。
粗糙脉孢菌产纤维素酶诱导机制和里氏木霉大不相同,尽管XlnR/Xyr1同源基因转录因子XLR-1对粗糙脉孢菌的木聚糖酶的调控很关键,但其敲除对纤维素酶的诱导调控影响甚微[37]。通过对粗糙脉孢菌转录因子敲除文库筛选分析,获得了粗糙脉孢菌中锌双核簇转录因子Clr-1和Clr-2,在纤维素条件下纤维素酶诱导产生作用非常显著,尽管在木聚糖条件下对木聚糖酶诱导表达影响不大。当Clr-1缺失时,菌株无法在纤维素和纤维二糖上生长,而且也无法产生clr-2转录体[38]。Clr-2在纤维素酶调控过程中发挥更重要的作用,其组成型表达也会像里氏木霉中Xyr1突变体一样,使纤维素酶的产生不受诱导物影响[38]。通过纤维素诱导条件下的组学分析发现,受到Clr-1和Clr-2调控的基因包括主要的糖苷水解酶基因,蛋白分泌途径相关的基因,以及内质网蛋白加工和修饰的相关基因,说明Clr-1和Clr-2对粗糙脉孢菌纤维素酶表达的重要作用[39]。研究表明,粗糙脉孢菌中Clr-1和Clr-2都分别以同源二聚体形式发挥功能,通过免疫共沉淀和转录组结合分析找出了Clr-1/-2和Xlr-1的直接靶点基因,Clr-1和Clr-2直接作用调控大量的纤维素酶和糖转运蛋白;而Xlr-1虽然也直接调控大量胞外分泌蛋白,但并不直接作用在纤维素酶启动子区域,主要结合基因是木聚糖降解酶基因,并且其缺失只对纤维素酶的产生有微弱影响[37,40]。丝状真菌中广泛存在clr-1和clr-2的同源基因来调控纤维素酶的转录和表达,如构巢曲霉中的同源基因clrA和clrB。构巢曲霉中调控纤维素酶表达的是ClrB,其对纤维素酶基因表达至关重要,但ClrA的缺失也同Clr-1缺失一样会影响纤维二糖的利用[39]。相同现象也在草酸青霉中发现,在草酸青霉中,ClrB是纤维素酶基因主要调控因子,XlnR更多的调节半纤维素酶基因发挥重要作用[41]。最近对黑曲霉研究发现ClrA和ClrB的作用功能与粗糙脉孢菌中的同源基因有相似趋势,ClrB对糖苷水解酶的调控有一定作用,但黑曲霉胞内的转录因子XlnR在纤维素酶调控过程中却十分重要而显著,同时XlnR还正调控着ClrA/ClrB的转录[42]。这些结果都说明了XlnR/Xyr1和ClrB两组转录调控因子在丝状真菌纤维素酶诱导中的关键作用,二者相互关联,但在不同的真菌中这两类转录因子对纤维素酶诱导调控机制不同。
除了以上所论述的转录激活因子,在里氏木霉中也发现纤维素酶诱导相关的转录抑制因子。Ace1是一个C2H2型锌指蛋白抑制因子,其特异结合序列是5′-AGGGA。敲除ace1的突变体,可以显著增加在纤维素和槐糖诱导条件下的主要纤维素酶和半纤维素酶[43]。研究表明,ace1基因敲除突变株在木糖条件下xyr1基因的表达显著提高,促进纤维素酶合成。在构巢曲霉中,ace1基因的同源基因stzA编码对氨基酸代谢相关基因的压力响应因子,其可潜在结合在cpcA基因的启动子区域,相应地在stzA/ace1的启动子区域也具有CpcA的潜在结合位点[44];同时在里氏木霉培养基中加入蛋氨酸后可增强纤维素酶的表达[45]。以上结果也说明了培养基中氨基酸种类和纤维素酶基因表达有着密切的联系。
里氏木霉纤维素酶诱导培养基中含有高浓度葡萄糖时 (葡萄糖浓度大于0.5%),葡萄糖会激活碳代谢阻遏 (CCR),不会有纤维素酶产生,当培养基中过量葡萄糖耗尽时,诱导物会继续发挥作用使得纤维素酶产生[14]。葡萄糖的碳代谢阻遏效应表现在两个方面,一方面,葡萄糖可抑制糖转运蛋白对二糖诱导物的转运能力,降低细胞对诱导物的摄入[15];另一方面,葡萄糖可抑制里氏木霉中主要的纤维素酶基因的转录[14]。葡萄糖碳代谢阻遏和纤维素酶诱导条件下比较发现,至少在糖转运蛋白和转录因子在转录水平上存在显著差距[46]。
研究表明里氏木霉C2H2型转录因子Cre1在CCR过程中起重要调控作用[47]。在里氏木霉、粗糙脉孢菌中大量的纤维素酶、半纤维素酶转录都受到CreA/Cre1因子的抑制作用,其也会对转录激活因子Xyr1产生负效应,最终会抑制大量纤维素酶的转录表达[48-49]。Cre1结合位点是两个临近的5'-SYGGRG核酸序列,通过这个核酸序列可以直接激活Cre1的抑制效应。Cre1转录因子以双重调节形式存在,Cre1除了可以直接作用在相应糖苷水解酶基因的启动子区域来进行调控,其还间接作用于其他功能基因来抑制纤维素酶的产生。如里氏木霉中cre1基因的缺失或者在其结构基因结合位点 (5′-SYGGRG)的突变会导致主要纤维素酶基因cbh1和半纤维素酶基因xyn1的去阻遏效应[50];同时通过cre1基因缺失的转录组结果表明,Cre1转录因子可以间接调控纤维素酶转录激活因子、转运蛋白家族蛋白和部分糖苷水解酶,从而抑制纤维素酶的诱导和产生[51-52]。在构巢曲霉中也存在上述双重调节现象,CreA不但抑制正调控转录因子XlnR的转录,而且CreA还直接结合抑制木质纤维素降解酶xlnA基因的表达[53-54]。工业菌株T. reesei Rut-C30中的cre1基因缺失了一个锌指蛋白结构,因此其体内的纤维素酶转录表达具有一定能力的葡萄糖耐受性[55],不过单独的由于cre1缺失产生的碳代谢阻遏消除并不能有效的产纤维素酶,细胞生长环境中诱导物的存在是产纤维素酶所必需的[56]。
纤维二糖普遍被认为作为一个诱导物前体存在,里氏木霉在纤维二糖培养基中,诱导初期转录因子BglR可以特异上调胞内β葡萄糖苷酶基因bgl1转录来诱导纤维素酶的初步产生,但随着降解的葡萄糖量增多会产生碳代谢阻遏效应,因此在纤维二糖培养基中,转录因子BglR的缺失会提高纤维素酶产量[57]。里氏木霉中葡萄糖木糖-脱氢酶蛋白Grd1对纤维素酶表达具有正向的效应,同时其也具有降解胞内纤维二糖的作用,但效率很低,不足以影响胞内纤维二糖作为基础产生下一步的纤维素酶诱导信号[58]。最新研究结果表明,锌指蛋白转录因子Crz1可特异地响应环境中的Ca2+信号,对纤维素酶基因转录有激活作用,体外实验证明Crz1可直接作用在xyr1和cbh1基因的启动子区域,并且与纤维素酶转录抑制因子Cre1竞争相同的作用靶点[59]。在棘孢曲霉中转录因子ClbR可以诱导一些不受XlnR调控的基因来响应纤维二糖和纤维素信号[60]。粗糙脉孢菌中转录因子Vib-1介导细胞程序性死亡,在碳饥饿条件下其参与到胞外蛋白水解酶分泌。vib-1基因缺失的突变菌株会增强细胞内的CCR效应,从而抑制粗糙脉孢菌中主要转录因子Clr-2的转录表达,后者影响胞外主要纤维素酶的转录和表达[61]。
糖转运蛋白及转录因子对丝状真菌纤维素酶的诱导见图1。
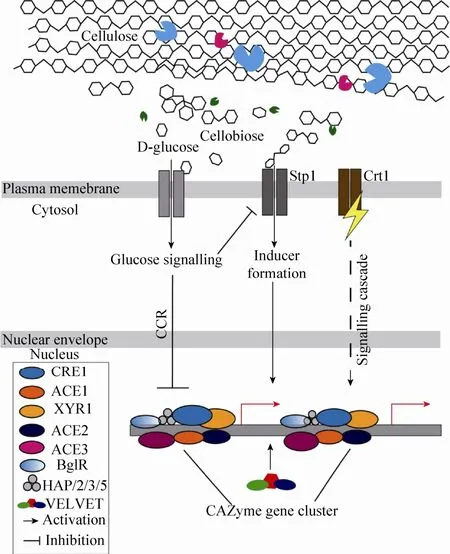
图1 里氏木霉纤维素酶调控示意图[62]Fig. 1 Schematic diagram for the regulation of cellulase genes in Trichoderma reesei (Modified from the reference[62]).
除了以上纤维素酶转录调控因子之外,近年来还发现了其他转录调控因子对纤维素酶的产生起到了重要的作用。米曲霉中转录因子ManR最初被认为是甘露聚糖基因相关的调控因子,研究表明至少参与5种主要纤维素酶的调控,和XlnR转录因子共同对主要纤维素酶的调控表达[63]。草酸青霉中AmyR是受到Clr-2和CreA调控的纤维素酶调控抑制因子,研究表明其主要功能是控制着淀粉和纤维素碳源利用的平衡。草酸青霉中AmyR的缺失会显著提高纤维素酶的产生但同时会抑制淀粉酶基因的转录表达[41]。构巢曲霉中的McmA是SRF型MADS box 调控蛋白,这一类调控蛋白时常与胞内其他调控因子共同作用来发挥功能,McmA主要参与介导纤维二糖条件下的2个内切酶和1个外切酶基因的转录调控[64]。
很多纤维素酶转录调控蛋白,包括Xyr1、Cre1、Ace1、Ace2、PacC、Clr-1及Clr-2等都是锌指蛋白。锌指蛋白广泛存在于多种微生物,对微生物的生长和代谢具有重要调控作用[65]。C2H2型锌指蛋白是最简单的转录因子调控模块,可特异识别核酸序列中3个相邻的核苷酸序列,根据锌指蛋白独特的模块结构,将来源和结合位点不同的C2H2型锌指模块通过基因合成,得到三/四锌指混合的锌指文库,锌指蛋白后加上激活/抑制功能域,便可得到人工锌指蛋白转录因子文库[65]。将此文库转化微生物后,人工转录因子可以随机对细胞体内不同基因的转录进行调节,因此可获得特异表型提高的突变体。韩国学者使用人工锌指蛋白文库转化大肠杆菌和酵母,获得了耐温性及耐药性提高的突变体,证明人工转录因子对细胞代谢存在有效控制[66]。本课题组利用人工锌指蛋白文库转化酿酒酵母,获得了乙醇和乙酸耐性显著提高的酿酒酵母突变体,并发现了人工锌指蛋白可能调控的基因QDR3与酿酒酵母乙酸耐性相关[67]。近期本课题组也成功构建了可在丝状真菌中表达的人工锌指蛋白文库,并转化里氏木霉Rut-C30,获得了多个纤维素酶产量提高的突变体[68]。通过纤维素酶活性显著提高转化子的转录组分析,进一步确定了和纤维素酶转录调控相关的调控通路 (未发表资料)。人工转录因子作为新的工具可用于丝状真菌纤维素酶的转录调控,并有希望挖掘新的纤维素酶的调控机制。
除了转录因子之外,泛素化的相关蛋白也存在于构巢曲霉和里氏木霉的碳代谢抑制途径中[69]。里氏木霉中泛素羧基末端水解酶Cre2的敲除使不同诱导碳源条件下纤维素酶表达都有提高,尽管其诱导效果没有Cre1敲除显著[70]。而在构巢曲霉中的F-box泛素化蛋白FbxA参与半纤维素酶的转录调控[71]。表1为近年来发现的不同丝状真菌中纤维素酶和半纤维素酶转录调控因子的总结。
4 染色质调控及其他相关调控方式
近期丝状真菌纤维素酶诱导过程中染色质重塑作用也受到了关注。研究表明,在cbh1基因启动子和编码区区域内,诱导和阻遏条件下该基因的核小体定位不同。里氏木霉在槐糖诱导物存在时核小体只出现在启动子区域,此时核小体重新定位的区域使转录激活因子有效作用,同时抑制因子作用的区域被封闭;野生型里氏木霉在葡萄糖条件下,核小体定位在cbh1启动子和编码区域,核小体在cbh1基因启动子上结合区域有两段,一段是在-840 bp到-1 140 bp,其包括Ace1结合位点,另一段是在-340 bp到-500 bp,与激活转录因子Xyr1和Ace2结合位点相邻,核小体的存在阻碍了其靶结合序列。值得注意的是,此时截短或缺失的Cre1转录因子起不到与完整Cre1转录因子相同的效果,导致在Cbh1编码区的核小体消失,说明Cre1不但可以直接作用在被抑制基因的启动子区域,也可以调节核小体重塑来调控转录[73]。整个过程中是否存在Cre1调控的染色质重塑因子来参与有待于进一步揭示。另外,里氏木霉中纤维素酶基因cel6A启动子区域的CCAAT盒与HAP2/3/5复合物和转录抑制因子Cre1结合,继而会影响核小体的定位,从而对cel6A的转录起到了严格而特异的调节作用[74]。最近研究表明Xyr1对纤维素酶基因具有直接调控之外,还通过影响纤维素酶基因的染色质状态来影响基因的转录和表达。研究表明,即使在有槐糖诱导物条件下,xyr1基因的缺失也会使主要纤维素酶基因cbh1、cbh2的染色质压缩,以至于阻遏了有效地展开和转录,通过分析找出了10个受Xyr1调控并与染色质重塑相关的基因,Xyr1直接作用还是受其调控的下游基因发挥作用有待于进一步揭示[75]。
在构巢曲霉中,CreA的功能依赖于组蛋白乙酰化状态,组蛋白酰基转移酶GceE可以看作是CreA的共调控因子,激活或者抑制靶基因的转录[76]。研究表明,里氏木霉体内gcn5基因编码组氨酸乙酰化转移酶,其对纤维素酶表达是必需的,该基因的缺失使在cbh1启动子区域核小体的H3K9和H3K14乙酰基化和CBH1的表达大大降低[77]。过表达另外一个CGN5乙酰基转移酶,可以使纤维素酶表达增加一倍[36]。同时发现这个CGN5酰基转移酶受到Lae1的调控,Lae1并不直接作用在纤维素酶的组蛋白上,而是间接调控纤维素酶的转录和表达[78]。

表1 不同真菌中新发现的纤维素酶和半纤维素酶调控子Table 1 Novel candidate regulators for cellulase and hemicellulase genes of different fungi
甲基转移酶是具有对核蛋白甲基化的蛋白,在许多丝状真菌中甲基转移酶作为全局调控子影响次级代谢产物的表达。里氏木霉中的甲基转移酶Lae1可调节细胞内大量基因转录的变化,其中包括调控主要纤维素酶基因表达,在缺失Lae1的突变体中,无法表达纤维素酶和其糖苷水解酶;Lae1过表达时,这些糖苷水解酶会急剧增加,说明Lae1在纤维素酶表达中起重要作用,并且其功能和转录因子Xyr1相互依赖。研究表明,尽管Lae1在曲霉中的等位基因LaeA参与组蛋白甲基化修饰,在染色质水平调控次级代谢产物的转录水平[79],但是里氏木霉中Lae1不直接对主要纤维素酶基因的组蛋白进行甲基化修饰,其主要和vel1等基因产物形成VELVET蛋白复合物[80],这个复合物调节丝状真菌中的次级代谢和糖苷水解酶基因[81]。在里氏木霉中VELVET复合物中的Lae1和Vel1缺失不诱导纤维素酶产生,并且VELVET的激活方式不受光影响,而其他真菌中光可抑制VELVET的形成和进入细胞核[72]。
蛋白磷酸化的转录后修饰可以调节蛋白功能、蛋白质转换、蛋白质之间相互作用和胞内细胞信号转导等作用。通过对槐糖诱导条件下对里氏木霉蛋白磷酸化分析,发现共1 721个磷酸化位点。被磷酸化的蛋白主要参与信号转导和转录调控[82]。诱导条件下糖转运蛋白的磷酸化使细胞获取胞外纤维素酶碳信号分子,同时也发现中心碳代谢的一些蛋白有磷酸化现象。另外转录因子Ace2、Cre1等的磷酸化对其在调控纤维素酶基因转录过程功能有重要影响[34,83]。最近研究表明,Hap2在诱导条件下磷酸化程度很高,但磷酸化对其功能的影响还需要进一步研究[82]。除此之外,一些信号途径中的蛋白酶具有明显的磷酸化现象,其中包括腺苷酸环化酶(Acy1) 和光信号感应蛋白 (VeA),RAS信号途径组件和MAPK信号途径组件中的蛋白。这些蛋白在胞外信号到胞内传递过程中起到了关键的作用。在纤维素培养基中RAS信号组件对纤维素酶的诱导存在重要的调节作用,里氏木霉的Ras2蛋白可感应和调节来自胞外的信号,通过下游途径介导纤维素酶基因的主要调控因子的表达和调控,继而对纤维素酶产生进行调节[84]。最近发现里氏木霉中促分裂原激活蛋白激酶 (MAPK) 途径也参与纤维素酶的调控和表达。MAP激酶TMK3对纤维素酶产生有正调节效应[85],而TMK2则对纤维素酶生产起抑制效果[86]。里氏木霉中另外一个来自MAPK家族的Ime2蛋白激酶的敲除显著提高纤维素酶产生,并发现cre1和xyr1的转录量同时降低,推测可能受Cre1的磷酸化缺失影响[87]。
5 展望
近年来,越来越广泛的系统生物学方法 (基因、转录、蛋白、代谢工具) 对揭示丝状真菌感应纤维素信号和纤维素酶诱导产生起到巨大推进作用,在纤维素酶产生的转录和响应调控方面的研究有了更多进展,然而纤维素酶诱导机制、信号传导途径等过程的分子基础还有许多不明之处有待揭示。同时基因组、转录组、代谢组和蛋白组等海量数据将会提供更详尽的关于纤维素酶诱导调控的生化途径和调控信息。这种整合不但有助于进一步揭示纤维素酶产生的分子基础,也为科研工作者提供更多的改造靶点和通路,从而促进纤维素酶高效生产,并推进生物质的有效降解。本课题组的最新研究工作证明了人工转录因子可调控丝状真菌产纤维素酶,通过人工转录因子技术的应用,可以定向设计纤维素酶的全局调控方式,有助于更加深入理解各种环境信号和信号传递通路在纤维素酶诱导调控过程中起到的作用。此外,利用人工锌指蛋白有希望发现新的纤维素酶基因表达调控的位点,深入对丝状真菌纤维素酶基因表达调控的机理认识,并在此基础上实现对纤维素酶表达调控的定向控制。未来可以结合丝状真菌内源的调控元件及人工调控元件,设计出更高效的纤维素酶调控系统,提高纤维素酶合成和分泌的效率,并结合发酵控制技术,提高纤维素酶的产量,降低生产成本,促进纤维素酶在木质纤维素原料生物炼制中的应用。
REFERENCES
[1] Zhao XQ, Zi LH, Bai FW, et al. Biotechnology in China III: Biofuels and Bioenergy. Springer-Verlag Berlin Heidelberg, 2011: 25-51.
[2] Mansfield SD, Mooney C, Saddler JN. Substrate and enzyme characteristics that limit cellulose hydrolysis. Biotechnol Prog, 1999, 15(5): 804-816.
[3] Andberg M, Penttila M, Saloheimo M. Swollenin from Trichoderma reesei exhibits hydrolytic activity against cellulosic substrates with features of both endoglucanases and cellobiohydrolases. Bioresour Technol, 2015, 181: 105-13.
[4] Hemsworth GR, Johnston EM, Davies GJ, et al. Lytic polysaccharide monooxygenases in biomass conversion. Trends Biotechnol, 2015, 33(12): 747-761.
[5] Glass NL, Schmoll M, Cate JHD, et al. Plant cell wall deconstruction by ascomycete fungi. Annu Rev Microbiol, 2013, 67: 477-98.
[6] Seiboth B, Ivanova C, Seidl-Seiboth V. Biofuel production-recent developments and prospects. InTech, 2011: 310-339.
[7] Xin Q, Xu JT, Wang TH, et al. Transcriptional regulation of cellulases and hemicellulases gene in Hypocrea jecorina-a review. Acta Microbiol Sin, 2010, 50(11): 1431-1437 (in Chinese).辛琪, 徐金涛, 汪天虹, 等. 丝状真菌红褐肉座菌 (Hypocrea jecorina) 纤维素酶基因的转录调控研究进展. 微生物学报, 2010, 50(11): 1431-1437.
[8] Yang F, Wang J. Progress of zinc finger protein in filamentous fungi's cellulase gene expression regulation. J Cellulose Sci Technol, 2014, 12(4): 75-81 (in Chinese).杨帆, 王娟. 锌指蛋白在丝状真菌纤维素酶基因表达调控中的研究进展. 纤维素科学与技术, 2014, 12(4): 75-81.
[9] Wang FZ, Jiang Y, Liu KM, et al. Regulation of cellulase and hemicellulase production in filamentous fungi. Chin J Bioprocess Eng, 2014, 12(1): 72-79 (in Chinese).王方忠, 蒋艺, 刘奎美, 等. 丝状真菌中纤维素酶与半纤维素酶的合成调控. 生物加工过程, 2014, 12(1): 72-79.
[10] El-Gogary S, Leite A, Crivellaro O, et al. Mechanism by which cellulose triggers cellobiohydrolase I gene expression in Trichoderma reesei. Proc Natl Acad Sci USA, 1989, 86(16): 6138-6141.
[11] Carle-Urioste JC, Escobar-Vera J, El-Gogary S, et al. Cellulase induction in Trichoderma reesei by cellulose requires its own basal expression. J Biol Chem, 1997, 272(15): 10169-10174.
[12] Kubicek CP, Muhlbauer G, Klotz M, et al. Properties of a conidial-bound cellulase enzyme system from Trichoderma reesei. J Gen Microbiol, 1988, 134(5): 1215-1222.
[13] Metz B, Seidl-Seiboth V, Haarmann T, et al. Expression of biomass-degrading enzymes is a major event during conidium development in Trichoderma reesei. Eukaryotic Cell, 2011, 10(11): 1527-1535.
[14] Ilmen M, Saloheimo ANU, Onnela M. Regulation of cellulase gene expression in the filamentous fungus Trichoderma reesei. Appl Environ Microbiol, 1997, 63(4): 1298-1306.
[15] Kubicek CP, Messner R, Gruber F, et al. Triggering of cellulase biosynthesis by cellulose inTrichoderma reesei. Involvement of a constitutive, sophorose-inducible, glucose-inhibited beta-diglucoside permease. J Biol Chem, 1993, 268(26): 19364-19368.
[16] Hakkinen M, Arvas M, Oja M, et al. Re-annotation of the CAZy genes of Trichoderma reesei and transcription in the presence of lignocellulosic substrates. Microb Cell Fact, 2012, 11(1): 134.
[17] Vaheri M, Leisola M, Kauppinen V. Transglycosylation products of cellulase system of Trichoderma reesei. Biotechnol Lett, 1979, 1(1): 41-46.
[18] Shida Y, Yamaguchi K, Nitta M, et al. The impact of a single-nucleotide mutation of bgl2 on cellulase induction in a Trichoderma reesei mutant. Biotechnol Biofuels, 2015, 8: 230.
[19] Mach RL, Seiboth B, Myasnikov A, et al. The bgl1 gene of Trichoderma reesei QM 9414 encodes an extracellular, cellulose-inducible β-glucosidase involved in cellulase induction by sophorose. Mol Microbiol, 1995, 16(4): 687-697.
[20] Fritscher C, Messner R, Kubicek CP. Cellobiose metabolism and cellobiohydrolase I biosynthesis by Trichoderma reesei. Exp Mycol, 1990, 14(4): 405-415.
[21] Li J, Liu GD, Chen M, et al. Cellodextrin transporters play important roles in cellulase induction in the cellulolytic fungus Penicillium oxalicum. Appl Microbiol Biotechnol, 2013, 97(24): 10479-10488.
[22] Znameroski EA, Li X, Tsai JC, et al. Evidence for transceptor function of cellodextrin transporters in Neurospora crassa. J Biol Chem, 2013, 289(5): 2610-2619.
[23] Cai PL, Wang B, Ji JX, et al. The putative cellodextrin transporter-like protein CLP1 is involved in cellulase induction in Neurospora crassa. J Biol Chem, 2015, 290(2): 788-796.
[24] Zhang WX, Kou YB, Xu JT, et al. Two major facilitator superfamily sugar transporters from Trichoderma reesei and their roles in induction of cellulase biosynthesis. J Biol Chem, 2013, 288(46): 32861-32872.
[25] Ivanova C, Baath JA, Seiboth B, et al. Systems analysis of lactose metabolism in Trichoderma reesei identifies a lactose permease that is essential for cellulase induction. PLoS ONE, 2013, 8(5): e62631.
[26] Stricker AR, Grosstessner-Hain K, Wurleitner E, et al. Xyr1 (xylanase regulator 1) regulates both the hydrolytic enzyme system and D-xylose metabolism in Hypocrea jecorina. Eukaryot Cell, 2006, 5(12): 2128-2137.
[27] Dos Santos Castro L, de Paula RG, Antonieto AC, et al. Understanding the role of the master regulator XYR1 in Trichoderma reesei by global transcriptional analysis. Front Microbiol, 2016, 7: 175.
[28] Lichius A, Seidl-Seiboth V, Seiboth B, et al. Nucleo-cytoplasmic shuttling dynamics of the transcriptional regulators XYR1 and CRE1 under conditions of cellulase and xylanase gene expression in Trichoderma reesei. Mol Microbiol, 2014, 94(5): 1162-1178.
[29] Derntl C, Gudynaite-Savitch L, Calixte S, et al. Mutation of the Xylanase regulator 1 causes a glucose blind hydrolase expressing phenotype in industrially used Trichoderma strains. Biotechnol Biofuels, 2013, 6(1): 62-72.
[30] Klaubauf S, Narang HM, Post H, et al. Similar is not the same: differences in the function of the (hemi-)cellulolytic regulator XlnR (Xlr1/Xyr1) in filamentous fungi. Fungal Genet Biol, 2014, 72: 73-81.
[31] Noguchi Y, Tanaka H, Kanamaru K, et al. Xylose triggers reversible phosphorylation of XlnR, the fungal transcriptional activator of xylanolytic and cellulolytic genes in Aspergillus oryzae. Biosci Biotechnol Biochem, 2011, 75(5): 953-959.
[32] Hasper AA, Trindade LM, van der Veen D, et al. Functional analysis of the transcriptional activator XlnR from Aspergillus niger. Microbiology, 2004, 150(5): 1367-1375.
[33] Aro N, Saloheimo A, Ilmen M, et al. ACEII, a novel transcriptional activator involved in regulation of cellulase and xylanase genes of Trichoderma reesei. J Biol Chem, 2001, 276(26): 24309-24314.
[34] Stricker AR, Trefflinger P, Aro N, et al. Role of Ace2 (Activator of Cellulases 2) within the xyn2transcriptosome of Hypocrea jecorina. Fungal Genet Biol, 2008, 45(4): 436-445.
[35] Martinez D, Berka RM, Henrissat B, et al. Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei (syn. Hypocrea jecorina). Nat Biotechnol, 2008, 26(5): 553-560.
[36] Häkkinen M, Valkonen MJ, Westerholm-Parvinen A, et al. Screening of candidate regulators for cellulase and hemicellulase production in Trichoderma reesei and identification of a factor essential for cellulase production. Biotechnol Biofuels, 2014, 7(1): 14.
[37] Sun J, Tian C, Diamond S, et al. Deciphering transcriptional regulatory mechanisms associated with hemicellulose degradation in Neurospora crassa. Eukaryot Cell, 2012, 11(4): 482-493.
[38] Coradetti ST, Xiong Y, Glass NL. Analysis of a conserved cellulase transcriptional regulator reveals inducer-independent production of cellulolytic enzymes in Neurospora crassa. Microbiology Open, 2013, 2(4): 595-609.
[39] Coradetti ST, Craig JP, Xiong Y, et al. Conserved and essential transcription factors for cellulase gene expression in ascomycete fungi. Proc Natl Acad Sci USA, 2012, 109(19): 7397-402.
[40] Craig JP, Coradetti ST, Starr TL, et al. Direct target network of the Neurospora crassa plant cell wall deconstruction regulators CLR-1, CLR-2, and XLR-1. mBio, 2015, 6(5): e01452-15.
[41] Li ZH, Yao GS, Wu RM, et al. Synergistic and dose-controlled regulation of cellulase gene expression in Penicillium oxalicum. PLoS Genet, 2015, 11(9): e1005509.
[42] Raulo R, Kokolski M, Archer DB. The roles of the zinc finger transcription factors XlnR, ClrA and ClrB in the breakdown of lignocellulose by Aspergillus niger. AMB Express, 2016, 6(1): 1-12.
[43] Aro N, Ilmen M, Saloheimo A, et al. ACEI of Trichoderma reesei is a repressor of cellulase and xylanase expression. Appl Environ Microbiol, 2003, 69(1): 56-65.
[44] Chilton IJ, Delaney CE, Barham-Morris J, et al. The Aspergillus nidulans stress response transcription factor StzA is ascomycete-specific and shows species-specific polymorphisms in the C-terminal region. Mycol Res, 2008, 112(Pt 12): 1435-1446.
[45] Gremel G, Dorrer M, Schmoll M. Sulphur metabolism and cellulase gene expression are connected processes in the filamentous fungus Hypocrea jecorina (anamorph Trichoderma reesei). BMC Microbiol, 2008, 8(1): 174.
[46] Dos Santos Castro L, Pedersoli WR, Antonieto AC, et al. Comparative metabolism of cellulose, sophorose and glucose in Trichoderma reesei using high-throughput genomic and proteomic analyses. Biotechnol Biofuels, 2014, 7(1): 41.
[47] Portnoy T, Margeot A, Linke R, et al. The CRE1 carbon catabolite repressor of the fungus Trichoderma reesei: a master regulator of carbon assimilation. BMC Genomics, 2011, 12(1): 269.
[48] Aro N, Pakula T, Penttila M. Transcriptional regulation of plant cell wall degradation by filamentous fungi. FEMS Microbiol Rev, 2005, 29(4): 719-739.
[49] Znameroski EA, Coradetti ST, Roche CM, et al. Induction of lignocellulose-degrading enzymes in Neurospora crassa by cellodextrins. Proc Natl Acad Sci USA, 2012, 109(16): 6012-6017.
[50] Kubicek CP, Mikus M, Schuster A, et al. Metabolic engineering strategies for the improvement of cellulase production by Hypocrea jecorina. Biotechnol Biofuels, 2009, 2: 19.
[51] Antonieto AC, Dos Santos Castro L, Silva-Rocha R, et al. Defining the genome-wide role of CRE1 during carbon catabolite repression in Trichoderma reesei using RNA-Seq analysis. Fungal Genet Biol, 2014, 73c: 93-103.
[52] Antoniêto ACC, Paula RGd, Castro LdS, et al. Trichoderma reesei CRE1-mediated carbon catabolite repression in response to sophorose through RNA sequencing analysis. Current Genomics, 2016, 17:119-131.
[53] Orejas M, MacCabe AP, Perez Gonzalez JA, et al. Carbon catabolite repression of the Aspergillus nidulans xlnA gene. Mol Microbiol, 1999, 31(1): 177-184.
[54] Tamayo EN, Villanueva A, Hasper AA, et al. CreAmediates repression of the regulatory gene xlnR which controls the production of xylanolytic enzymes in Aspergillus nidulans. Fungal Genet Biol, 2008, 45(6): 984-993.
[55] Seidl V, Gamauf C, Druzhinina IS, et al. The Hypocrea jecorina (Trichoderma reesei) hypercellulolytic mutant RUT C30 lacks a 85 kb (29 gene-encoding) region of the wild-type genome. BMC Genomics, 2008, 9: 327.
[56] Nakari-Setala T, Paloheimo M, Kallio J, et al. Genetic modification of carbon catabolite repression in Trichoderma reesei for improved protein production. Appl Environ Microbiol, 2009, 75(14): 4853-4860.
[57] Nitta M, Furukawa T, Shida Y, et al. A new Zn(II)(2)Cys(6)-type transcription factor BglR regulates beta-glucosidase expression in Trichoderma reesei. Fungal Genet Biol, 2012, 49(5): 388-397.
[58] Schuster A, Kubicek CP, Schmoll M. Dehydrogenase GRD1 represents a novel component of the cellulase regulon in Trichoderma reesei (Hypocrea jecorina). Appl Environ Microbiol, 2011, 77(13): 4553-4563.
[59] Chen L, Zou G, Wang JZ, et al. Characterization of the Ca2+-responsive signaling pathway in regulating the expression and secretion of cellulases in Trichoderma reesei Rut-C30. Mol Microbiol, 2016, DOI: 10.1111/mmi.13334.
[60] Kunitake E, Tani S, Sumitani J, et al. A novel transcriptional regulator, ClbR, controls the cellobiose-and cellulose-responsive induction of cellulase and xylanase genes regulated by two distinct signaling pathways in Aspergillus aculeatus. Appl Microbiol Biotechnol, 2013, 97(5): 2017-2028.
[61] Xiong Y, Sun JP, Glass NL. VIB1, a link between glucose signaling and carbon catabolite repression, is essential for plant cell wall degradation by Neurospora crassa. PLoS Genet, 2014, 10(8): e1004500.
[62] Seiboth B, Herold S, Kubicek CP. Metabolic engineering of inducer formation for cellulase and hemicellulase gene expression in Trichoderma reesei. In: Wang XY, Chen J, Quinn PJ (eds). Reprogramming microbial metabolic pathways, p. 367-390. Springer Netherlands, 2012.
[63] Ogawa M, Kobayashi T, Koyama Y. ManR, a transcriptional regulator of the beta-mannan utilization system, controls the cellulose utilization system in Aspergillus oryzae. Biosci Biotechnol Biochem, 2013, 77(2): 426-429.
[64] Yamakawa Y, Endo Y, Li N, et al. Regulation of cellulolytic genes by McmA, the SRF-MADS box protein in Aspergillus nidulans. Biochem Biophys Res Commun, 2013, 431(4): 777-782.
[65] Liu Z, Zhang F, Zhao XQ, et al. Effects of zinc-finger proteins and artificial zinc-finger proteins on microbial metabolisms-a review. Chin J Biotech, 2014, 30(3): 331-340 (in Chinese).刘卓, 张飞, 赵心清, 等. 锌指蛋白及人工锌指蛋白对微生物代谢影响的研究进展. 生物工程学报, 2014, 30(3): 331-340.
[66] Park KS, Lee DK, Lee H, et al. Phenotypic alteration of eukaryotic cells using randomized libraries of artificial transcription factors. Nat Biotechnol, 2003, 21(10): 1208-1214.
[67] Ma C, Wei XW, Sun CW, et al. Improvement of acetic acid tolerance of Saccharomyces cerevisiae using a zinc-finger-based artificial transcription factor and identification of novel genes involved in acetic acid tolerance. Appl Microbiol Biotechnol, 2015, 99(5): 2441-2449.
[68] Zhang F, Zhao XQ, Bai FW. Enhanced cellulase production from Trichoderma reesei Rut-C30 by engineering with an artificial zinc finger protein library. Biotech J, 2016, 11(10): 1282-1290.
[69] Lockington RA, Kelly JM. Carbon catabolite repression in Aspergillus nidulans involves deubiquitination. Mol Microbiol, 2001, 40(6): 1311-1321.
[70] Denton JA, Kelly JM. Disruption of Trichoderma reesei cre2, encoding an ubiquitin C-terminal hydrolase, results in increased cellulase activity. BMC Biotechnol, 2011, 11: 103.
[71] Colabardini AC, Humanes AC, Gouvea PF, et al. Molecular characterization of the Aspergillus nidulans fbxA encoding an F-box protein involved in xylanase induction. Fungal Genet Biol, 2012,49(2): 130-140.
[72] Aghcheh RK, Németh Z, Atanasova L, et al. The VELVET A orthologue VEL1 of Trichoderma reesei regulates fungal development and is essential for cellulase gene expression. PLoS ONE, 2014, 9(11): e112799.
[73] Ries L, Belshaw N, Ilmén M, et al. The role of CRE1 in nucleosome positioning within the cbh1 promoter and coding regions of Trichoderma reesei. Appl Microbiol Biotechnol, 2014, 98(2): 749-762.
[74] Zeilinger S, Schmoll M, Pail M, et al. Nucleosome transactions on the Hypocrea jecorina (Trichoderma reesei) cellulase promoter cbh2 associated with cellulase induction. Mol Genet Genomics, 2003, 270(1): 46-55.
[75] Mello-de-Sousa TM, Rassinger A, Pucher ME, et al. The impact of chromatin remodelling on cellulase expression in Trichoderma reesei. BMC Genomics, 2015, 16: 588.
[76] Strauss J, Reyes-Dominguez Y. Regulation of secondary metabolism by chromatin structure and epigenetic codes. Fungal Genet Biol, 2011, 48(1): 62-69.
[77] Xin Q, Gong YJ, Lv XX, et al. Trichoderma reesei histone acetyltransferase Gcn5 regulates fungal growth, conidiation, and cellulase gene expression. Curr Microbiol, 2013, 67(5): 580-589.
[78] Fekete E, Karaffa L, Aghcheh RK, et al. The transcriptome of lae1 mutants of Trichoderma reesei cultivated at constant growth rates reveals new targets of LAE1 function. BMC Genomics, 2014, 15(1): 447.
[79] Reyes Dominguez Y, Bok JW, Berger H, et al. Heterochromatic marks are associated with the repression of secondary metabolism clusters in Aspergillus nidulans. Mol Microbiol, 2010, 76(6): 1376-1386.
[80] Bayram Ö, Krappmann S, Ni M, et al. VelB/VeA/LaeA complex coordinates light signal with fungal development and secondary metabolism. Science, 2008, 320(5882): 1504-1506.
[81] Seiboth B, Karimi RA, Phatale PA, et al. The putative protein methyltransferase LAE1 controls cellulase gene expression in Trichoderma reesei. Mol Microbiol, 2012, 84(6): 1150-1164.
[82] Nguyen EV, Imanishi SY, Haapaniemi P, et al. Quantitative site-specific phosphoproteomics of Trichoderma reesei signaling pathways upon induction of hydrolytic enzyme production. J Proteome Res, 2016, 15(2): 457-467.
[83] Cziferszky A, Mach RL, Kubicek CP. Phosphorylation positively regulates DNA binding of the carbon catabolite repressor Cre1 of Hypocrea jecorina (Trichoderma reesei). J Biol Chem, 2002, 277(17): 14688-14694.
[84] Zhang JW, Zhang YM, Zhong YH, et al. Ras GTPases modulate morphogenesis, sporulation and cellulase gene expression in the cellulolytic fungus Trichoderma reesei. PLoS ONE, 2012, 7(11): e48786.
[85] Wang MY, Zhao QS, Yang JH, et al. A mitogen-activated protein kinase Tmk3 participates in high osmolarity resistance, cell wall integrity maintenance and cellulase production regulation in Trichoderma reesei. PLoS ONE, 2013, 8(8): e72189.
[86] Wang MY, Dong YM, Zhao QS, et al. Identification of the role of a MAP kinase Tmk2 in Hypocrea jecorina (Trichoderma reesei). Sci Rep, 2014, 4: 6732.
[87] Chen F, Chen XZ, Su XY, et al. An Ime2-like mitogen-activated protein kinase is involved in cellulase expression in the filamentous fungus Trichoderma reesei. Biotechnol Lett, 2015, 37(10): 2055-2062.
(本文责编 陈宏宇)
Induction and regulation of cellulase expression in filamentous fungi: a review
Fei Zhang1, Fengwu Bai1,2, and Xinqing Zhao2
1 School of Life Science and Biotechnology, Dalian University of Technology, Dalian 116024, Liaoning, China 2 State Key Laboratory of Microbial Metabolism, School of Life Science and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China
Production of bioenergy and bio-based chemicals by using fermentable sugars released from low-costrenewable lignocellulosic biomass has
great attention. Efficient cellulolytic enzymes are crucial for lignocellulose bioconversion, but high cellulase production cost is limiting the bioconversion efficiency of cellulosic biomass and industrial applications of lignocellulose biorefinery. Studies on induction and regulation of cellulase in filamentous fungi will help to further develop superior fungal strains for efficient cellulase production and reduce cellulase production cost. With the advances in high-throughput sequencing and gene manipulation technology using fungal strains, an in-depth understanding of cellulase induction and regulation mechanisms of enzyme expression has been achieved. We reviewed recent progresses in the induction and regulation of cellulase expression in several model filamentous fungi, emphasizing sugar transporters, transcription factors and chromatin remodeling. Future prospects in application of artificial zinc finger proteins for cellulase induction and regulation in filamentous fungi were discussed.
March 15, 2016; Accepted: April 20, 2016
Xinqing Zhao. Tel: +86-21-34206673; Fax: +86-21-34208028; E-mail: xqzhao@sjtu.edu.cn
cellulase, gene expression and regulation, transcription factor, Trichoderma reesei
Supported by: National Natural Science Foundation of China (Nos. 31461143029, 51561145014), National High Technology Research and Development Program of China (863 Program) (No. 2012AA021205).
国家自然科学基金 (Nos. 31461143029, 51561145014),国家高技术研究发展计划 (863计划) (No. 2012AA021205) 资助。

