生物胁迫相关NAC1转录因子的生物信息学分析
朱峰++陈孝仁++钱坤++纪兆林++孔维文++杨益众


摘要:植物NAC1转录因子在调控植物的抗生物胁迫反应中起着重要的作用。为探究生物逆境相关NAC1转录因子的功能,通过生物信息学的方法对8个生物逆境胁迫相关NAC1蛋白氨基酸序列一致性、氨基酸组成、理化性质、亲/疏水性、保守结构域、磷酸化位点、亚细胞定位、二级结构及三级结构等进行了预测和分析。结果表明,8个生物逆境胁迫相关NAC1蛋白N-端保守性较强,包括5个保守的亚结构域,共同组成NAC1结构域。C-端含有多个保守的氨基酸,具有转录激活功能。同时蛋白中含有多个丝氨酸(S)、苏氨酸(T)和酪氨酸(Y)磷酸化位点。8个NAC1蛋白都为亲水性蛋白,大多定位于细胞核,个别定位于细胞质或叶绿体。二级结构则以α-螺旋和β-折叠为主。8个NAC1蛋白三维结构上的相似性暗示了功能上存在相似。本研究结果为进一步挖掘生物逆境相关NAC1转录因子的功能和改良植物抗生物逆境特性提供理论依据。
关键词:生物胁迫;NAC1转录因子;生物信息学;抗生物逆境特性
中图分类号: Q78文献标志码: A
文章编号:1002-1302(2016)10-0043-06
收稿日期:2016-04-14
基金项目:国家自然科学基金(编号:31500209);江苏省高校自然科学研究面上项目(编号:15KJB210007);江苏省扬州市自然科学基金-青年科技人才项目(编号:YZ2015106)。
作者简介:朱峰(1985—),男,江西九江人,博士,讲师,主要从事植物病理与分子生物学研究。E-mail:zhufeng@yzu.edu.cn。
[ZK)]
NAC(NAM/ATAF/CUC)转录因子家族是目前发现的最大的一类植物特有的转录因子,在其他真核生物中尚未发现该家族成员[1-2]。NAC家族的命名源于矮牵牛(Petunia hybrida)NAM(no apical meristem)和拟南芥(Arabidopsis thaliana)[WTBX][STBX]ATAF1、ATAF2[WTBZ][STBZ]以及[WTBX][STBX]CUC2[WTBZ][STBZ](cup-shaped cotyledon)基因[3-4]。研究表明,NAC转录因子具有多种功能,在植物的生长发育、器官建成、激素信号应答及逆境胁迫中均发挥重要的作用,已成为当前植物基因功能及表达网络调控研究中的热点[2,5-6],如枝顶端分生组织发育[7]、植物的衰老[8-10]、细胞分裂[11]、植物激素调控与信号转导[12-13]和参与生物胁迫中植物的防御反应[14-16]等。
NAC转录因子是一个庞大的家族,大量的NAC基因被识别,其中在拟南芥中已发现117个NAC基因,水稻中发现151个NAC基因,葡萄中发现79个NAC基因,烟草中发现152个NAC基因[17]。越来越多的研究表明了NAC1基因在调控植物的抗生物胁迫反应起着重要的作用。辣椒[WTBX][STBX]CaNAC1[WTBZ][STBZ]在细菌性斑点病菌侵染后快速诱导,而且非寄主病菌侵染和抗病信号分子SA和ET处理后也能诱导[WTBX][STBX]CaNAC1[WTBZ][STBZ]强烈的表达[18]。先前的研究表明假单胞杆菌可以诱导番茄[WTBX][STBX]SlNAC1[WTBZ][STBZ]基因的表达[19],进一步研究表明番茄[WTBX][STBX]SlNAC1[WTBZ][STBZ]在调控植物抗性抵抗假单胞杆菌侵染起着重要的作用,并且番茄[WTBX][STBX]SlNAC1[WTBZ][STBZ]和本生烟[WTBX][STBX]NbNAC1[WTBZ][STBZ]有高度的同源性,抑制[WTBX][STBX]NbNAC1[WTBZ][STBZ],削弱植物的抗性,增加本生烟对假单胞杆菌的敏感性[20]。葡萄[WTBX][STBX]VvNAC1[WTBZ][STBZ]基因调控葡萄的防御反应应答坏死型和活体营养型病原菌的入侵,在拟南芥中过量表达[WTBX][STBX]VvNAC1[WTBZ][STBZ]基因可以增强植物对坏死型和活体营养型病原菌的入侵,并且防御相关的植物激素(如SA、MeJA、ABA和乙烯)可以诱导葡萄[WTBX][STBX]VvNAC1[WTBZ][STBZ]基因的表达[21]。在大麦中过量表达[WTBX][STBX]HvSNAC1[WTBZ][STBZ]基因能够增强大麦对镰刀菌的抗性,减轻叶斑病症状[22]。一些研究表明,NAC转录因子在应答病毒入侵时也起着一定的作用。番茄曲叶病毒(tomato leaf curl virus,TLCV)能够诱导番茄[WTBX][STBX]SlNAC1[WTBZ][STBZ]基因的表达,并且该病毒编码的Ren(geminiviral replication enhancer)蛋白能与[WTBX][STBX]SlNAC1[WTBZ][STBZ]蛋白相互作用,过量表达SlNAC1后可以加速TLCV的DNA积累[23]。
因此本研究将通过生物信息学的方法对生物逆境胁迫相关的普通烟[WTBX][STBX]NtNAC1[WTBZ][STBZ]、番茄[WTBX][STBX]SlNAC1[WTBZ][STBZ]、拟南芥[WTBX][STBX]AtATAF1[WTBZ][STBZ]、辣椒[WTBX][STBX]CaNAC1[WTBZ][STBZ]、大麦[WTBX][STBX]HvSNAC1[WTBZ][STBZ]、小麦[WTBX][STBX]TaNAC1[WTBZ][STBZ]、水稻[WTBX][STBX]OsNAC1[WTBZ][STBZ]和葡萄[WTBX][STBX]VvNAC1[WTBZ][STBZ]转录因子的理化性质、等电点、亲/疏水性、序列一致性、磷酸化位点、保守结构域、亚细胞定位、二级结构和蛋白三维结构等进行预测和比较分析,旨在为进一步挖掘生物逆境相关NAC1转录因子的功能和改良植物抗生物逆境特性提供理论支持。
1材料与方法
1.1材料
从NCBI(http://www.ncbi.nlm.nih.gov/)的GenBank上下载普通烟[WTBX][STBX]NtNAC1[WTBZ][STBZ]、番茄[WTBX][STBX]SlNAC1[WTBZ][STBZ]、拟南芥[WTBX][STBX]AtATAF1[WTBZ][STBZ]、辣椒[WTBX][STBX]CaNAC1[WTBZ][STBZ]、大麦[WTBX][STBX]HvSNAC1[WTBZ][STBZ]、小麦[WTBX][STBX]TaNAC1[WTBZ][STBZ]、水稻[WTBX][STBX]OsNAC1[WTBZ][STBZ]和葡萄[WTBX][STBX]VvNAC1[WTBZ][STBZ]转录因子的氨基酸序列。
1.2NAC1蛋白氨基酸序列比对及系统发生树构建
利用DNAMAN软件进行氨基酸序列一致性分析。利用在线程序Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/)进行氨基酸多重序列比对。利用MEGA 5.0软件[24]构建系统发生树,采取遗传距离建树法的相邻连接法(neighbor-joining,NJ)建树,对构建的树进行自检(bootstrap),重复设定为1 000。
1.3NAC1蛋白一级结构及理化特性分析
利用在线程序ProtParam(http://web.expasy.org/protparam/)对蛋白理化性质进行分析[25]。利用在线程序ProtScale(http://web.expasy.org/protscale/)进行氨基酸疏水性分析,利用NetPhos 2.0 Server (http://www.cbs.dtu.dk/services/NetPhos/)进行蛋白磷酸化位点修饰分析[26]。
1.4NAC1蛋白二级结构预测及亚细胞定位分析
[JP2]利用在线程序SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测α-螺旋(alpha helix)、β-折叠(extended strand)、β-转角(beta turn)、无规卷曲(random coil)等蛋白二级结构。利用在线程序WoLF PSORT(http://www.genscript.com/wolf-psort.html)进行蛋白亚细胞定位[27]。
1.5NAC1蛋白三级结构预测
利用SWISS-MODEL workspace (http://swissmodel.expasy.org/workspace/) 对蛋白的三维结构进行同源模拟[28]。其中[WTBX][STBX]SlNAC1、CaNAC1、AtATAF1 TaNAC1、NtNAC1、OsNAC1和HvSNAC1[WTBZ][STBZ]的模板PDB号都是3ulxA,VvNAC1的模板PDB号是1ut7B。然后通过UCSF Chimera软件将蛋白三维结构读取出来[29]。
2结果与分析
2.1NAC1蛋白氨基酸序列比对及系统发生树构建
通过DNAMAN软件对这8个生物逆境胁迫相关NAC1蛋白氨基酸序列的一致性进行分析,发现它们的氨基酸序列一致性为46.52%。进一步通过在线程序Clustal Omega对它们进行多重序列比对,寻找NAC1中的保守区域。如图1所示,8个生物逆境胁迫相关NAC1转录因子在氨基酸序列N-端保守性较强,其结构域由高度保守的约150个氨基酸残基组成。C-端氨基酸则具有高度的多样性,但C-端仍有几个较为保守的氨基酸,如酪氨酸(Y)、亮氨酸(L)、缬氨酸(V)、丝氨酸(S)。利用MEGA 5.0软件对这8个生物逆境胁迫相关NAC1蛋白构建系统进化树。如图2所示,SlNAC1和CaNAC1亲缘关系最近,和AtATAF1亲缘关系稍近,与 NtNAC1 的亲缘关系最远。[FL)]
[FK(W33][TPZF1.tif][FK)]
[FK(W8][TPZF2.tif][FK)]
2.2NAC1蛋白理化性质及亲疏水性分析
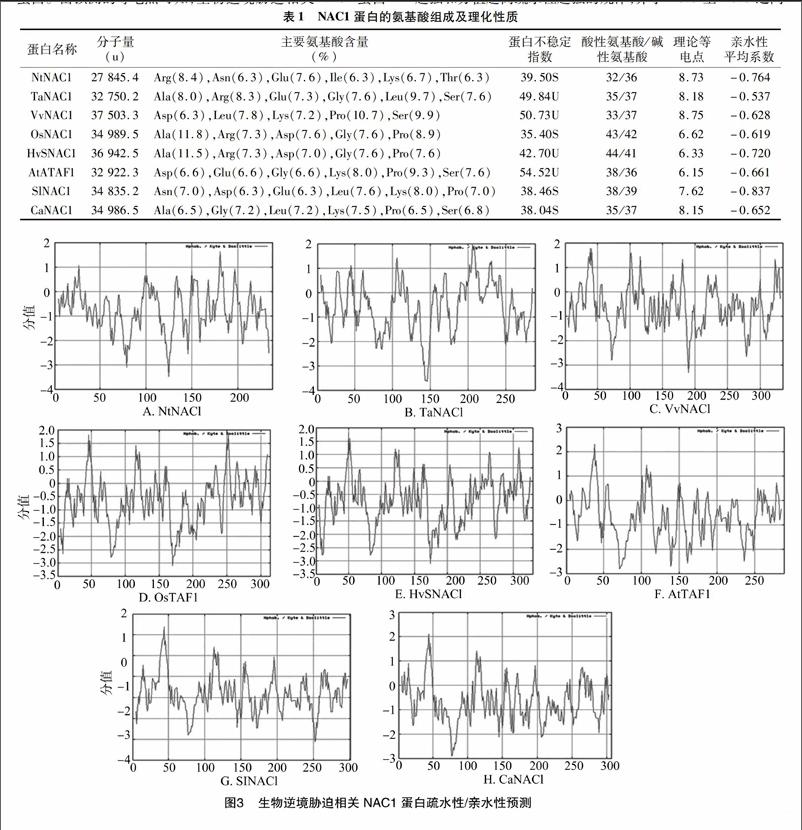
利用在线程序ProtParam预测NAC1蛋白的理化性质(表1),结果表明,NtNAC1、OsNAC1、SlNAC1和CaNAC1为稳定蛋白,而TaNAC1、VvNAC1、HvSNAC1和AtATAF1为不稳定蛋白。由预测的等电点可知,生物逆境胁迫相关NAC1蛋白中碱性等电点居多,如NtNAC1、SlNAC1、CaNAC1、TaNAC1和VvNAC1,也有酸性等电点,如OsNAC1、HvSNAC1、AtATAF1。从蛋白组成可以看出,NtNAC1中精氨酸(Arg)含量最高,TaNAC1中亮氨酸(Leu)含量最高,OsNAC1和HvSNAC1中丙氨酸(Ala)含量最高,VvNAC1和AtATAF1中脯氨酸(Pro)含量最高,SlNAC1和CaNAC1中赖氨酸(Lys)含量最高。
另外由表1可以看出,8个生物逆境胁迫相关NAC1蛋白的亲水性平均系数都为负值,说明它们都是亲水性蛋白,其中SlNAC1的亲水性平均系数最小,说明SlNAC1蛋白的亲水性最强。为了进一步证实NAC1蛋白都属于亲水性蛋白,利用在线程序ProtScale对8个生物逆境胁迫相关NAC1蛋白进行疏水性/亲水性预测(图3)。根据氨基酸分值越低亲水性越强和分值越高疏水性越强的规律,介于+0.5至-0.5之间
的主要为两性氨基酸,从整体来看,这8个生物逆境胁迫相关NAC1蛋白氨基酸的分值为负值,也就说明了它们都为亲水性蛋白。从图3还可以看出,SlNAC1蛋白氨基酸整体上的分值是最低的,说明了SlNAC1蛋白的亲水性最好。这也证实了在线程序ProtParam预测8个生物逆境胁迫相关NAC1蛋白都为亲水性蛋白的结果。其中在SlNAC1多肽链中第252位的谷氨酸(Glu)具有最低分值为-3.122,亲水性最强,第44位的异亮氨酸(Ile)具有最高分值2.389,疏水性最强。
2.3NAC1蛋白磷酸化分析
蛋白质合成后,化学修饰是一种对其活性进行调节的重要形式。因此对氨基酸序列翻译后修饰的预测和分析可以为认识蛋白质的亚细胞定位以及推测蛋白功能提供重要的帮助。蛋白质磷酸化(protein phosphorylation)是生物界最普遍也是最重要的一种蛋白质翻译后修饰。蛋白质磷酸化和去磷酸化是原核和真核生物细胞表达调控的关键环节,对许多生物的细胞功能起开关调控作用。如蛋白激酶C的磷酸化对于基因表达、细胞分化、机体代谢和增殖起着重要的作用。其中蛋白磷酸化主要有氨基酸序列中苏氨酸(T)、酪氨酸(Y) 和丝氨酸(S)的磷酸化。因此利用NetPhos 2.0 Server在线程序对这8个NAC1蛋白氨基酸磷酸化位点进行了预测。生物逆境胁迫相关NAC1蛋白的磷酸化位点数量如表2所示。结果显示,这8个生物逆境胁迫相关NAC1蛋白中丝氨酸磷酸化位点最多。
2.4NAC1蛋白二级结构预测及亚细胞定位分析
蛋白质的二级结构主要有α-螺旋、 β-折叠、β-转角和无规卷曲等。如表3所示,通过在线程序SOPMA的预测,结果表明,生物逆境胁迫相关NAC1蛋白中无规卷曲的比例较高(38.66%~54.33%),它具有连接其他二级结构元件的作用;此外的主要二级结构元件有α-螺旋(18.69%~32.80%)和β-折叠(14.63%~24.57%)。生物逆境胁迫相关NAC1蛋白二级结构预测见图4,它们在二级结构上也具有较高的相似性。尤其是SlNAC1和CaNAC1中的二级结构(如α-螺旋和β-折叠)出现的氨基酸残基的位置比较相近,同样[WTBX][STBX]OsNAC1和HvSNAC1[WTBZ][STBZ]中的二级结构出现的氨基酸残基的位置也很相近。
利用在线程序WOLF PSORT分别对8个NAC1成员蛋白质的亚细胞定位进行分析,结果以得分形式表现。结果(表4)表明,TaNAC1定位于细胞质的可能性较大,CaNAC1定位于叶绿体的可能性较大,其余蛋白定位于细胞核,这也从侧面表明了这些转录因子的存在及其发挥功能的场所主要是细胞核。
2.5NAC1蛋白三维结构分析
蛋白质三级结构的预测和分析对理解蛋白质结构和功能
3讨论与结论
植物转录因子的研究不仅是当前生物科学研究领域的热点,而且是功能基因组学研究的重要内容之一。大量研究表明,NAC转录因子在植物生长发育中具有重要的调控作用[2],而且也参与植物对干旱、高盐、低温等非生物和病原菌侵染等生物胁迫的抗逆反应、激素信号途径的转导、机体的凋亡等方面[2,5-6]。越来越多的研究表明,[WTBX][STBX]NAC1[WTBZ][STBZ]基因在调控植物的抗生物胁迫(如细菌、真菌、病毒的侵染)方面起着重要的作用。因此本研究重点针对[WTBX][STBX]NAC1[WTBZ][STBZ]基因进行了生物信息学分析。
氨基酸序列比对结果表明NAC1转录因子在N-端保守性非常强,其结构域可以分为5个亚结构域。研究表明,NAC转录因子C-端富含酸性氨基酸、谷氨酰胺和脯氨酸等氨基酸时,一般是转录激活区域[30-32]。本研究比对结果显示,生物逆境胁迫相关NAC1转录因子的C-端氨基酸组成比较符合这个特征,推测参与转录激活。近年来,关于核定位信号 (nuclear localization signal,NLS)的研究已经越来越深入,大致可分为以下3类[33]:(1)单一型(monopartite)NLS。这种NLS最初发现存在于猿猴病毒40的大T抗原中,包含7个氨基酸(PKKKRKV)的短肽[34]。单一型NLS一般是由4~8个氨基酸组成的短肽,富含带正电荷的Lys-和Arg-,通常还含有Pro。(2)双分型(bipartite)NLS。这种NLS是由2簇碱性氨基酸组成,中间是由10~12个非保守性氨基酸分隔而形成的序列。研究表明双向的信号可能是标准的NLS[35]。Nogueira等发现SsNAC23包含1个双向的核定位信号[36-37]。(3)其他类型NLS。除上述2类典型的NLS外,还有一些没有特定序列特征的NLS,它们主要存在于可在细胞核和细胞质间穿梭的蛋白质中。本研究中8个生物逆境胁迫NAC1转录因子大多定位于细胞核,由亚细胞定位的结果可以推断,具有相近生物学功能的NAC1蛋白可能定位于相同的亚细胞结构。
对这些生物逆境胁迫NAC1蛋白的二级结构进行分析进一步得知这些蛋白均属于亲水蛋白,但他们在亲水程度方面存在差异。SlNAC1蛋白的亲水性最强,表明SlNAC1蛋白更易水解。蛋白三维结构与其生物学功能息息相关[38]。通过SWISS-MODEL workspace 对NAC1蛋白三维结构进行同源模拟,结果发现,SlNAC1、CaNAC1和AtATAF1结构相似,包含A链和B链,各自形成1个对称的同源二聚体。目前,已有研究通过晶体衍射的方法将拟南芥ANAC蛋白和水稻胁迫响应NAC1蛋白的结构解析出来。结果证实它们的晶体结构为1个对称的同源二聚体,并且每个单体包含6个反向平行的β-折叠和3个α-螺旋[39-40]。总体来看,8种成员的二级结构中的α-螺旋、β-折叠和无规卷曲具有较高的保守性。这也从某种程度上反映了它们在三级结构上的相似性以及生物学功能的相似性。
综上所述,NAC1转录因子保守结构域的序列将直接影响到NAC1蛋白的亲水性、疏水性、亚细胞定位、二级结构以及三级结构的形成等,进而影响NAC1蛋白的生理功能。因此本研究应用生物信息学的方法对已知蛋白序列NAC1进行序列比对、分析、推断和预测其结构和功能,为进一步挖掘生物逆境相关NAC1转录因子的功能和改良植物抗生物逆境特性提供理论依据。
参考文献:
[1]Riechmann J L,Heard J,Martin G,et al. Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes[J]. Science,2000,290(5499):2105-2110.
[2]Olsen A N,Ernst H A,Leggio L L,et al. NAC transcription factors:structurally distinct,functionally diverse[J]. Trends in Plant Science,2005,10(2):79-87.
[3]Souer E,Vanhouwelingen A,Kloos D,et al. The no apical meristem gene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell,1996,85(2):159-170.
[4]Aida M,Ishida T,Fukaki H,et al. Genes involved in organ separation in Arabidopsis:an analysis of the cup-shaped cotyledon mutant[J]. The Plant Cell,1997,9(6):841-857.
[5]Zheng X,Chen B,Lu G,et al. Overexpression of a NAC transcription factor enhances rice drought and salt tolerance[J]. Biochemical and Biophysical Research Communications,2009,379(4):985-989.
[6]Nuruzzaman M,Sharoni A M,Satoh K,et al. Comprehensive gene expression analysis of the NAC gene family under normal growth conditions,hormone treatment,and drought stress conditions in rice using near-isogenic lines (NILs) generated from crossing Aday Selection (drought tolerant) and IR64[J]. Molecular Genetics and Genomics,2012,287(5):389-410.
[7]Nikovics K,Blein T,Peaucelle A,et al. The balance between the MIR164A and CUC2 genes controls leaf margin serration in Arabidopsis[J]. The Plant Cell,2006,18(11):2929-2945.
[8]Guo Y,Gan S. AtNAP,a NAC family transcription factor,has an important role in leaf senescence[J]. The Plant Journal,2006,46(4):601-612.
[9]Uauy C,Distelfeld A,Fahima T,et al. A NAC gene regulating senescence improves grain protein,zinc,and iron content in wheat[J]. Science,2006,314(583):1298-1301.[ZK)]
[10]Balazadeh S,Siddiqui H,Allu A D,et al. A gene regulatory network controlled by the NAC transcription factor ANAC092/AtNAC2/ORE1 during salt-promoted senescence[J]. The Plant Journal:for Cell and Molecular Biology,2010,62(2):250-264.
[11]Kim S Y,Kim S G,Kim Y S,et al. Exploring membrane-associated NAC transcription factors in Arabidopsis:implications for membrane biology in genome regulation[J]. Nucleic Acids Research,2007,35(1):203-213.
[12]He X J,Mu R L,Cao W H,et al. AtNAC2,a transcription factor downstream of ethylene and auxin signaling pathways,is involved in salt stress response and lateral root development[J]. The Plant Journal,2005,44(6):903-916.
[13]Kim S G,Lee A K,Yoon H K,et al. A membrane-bound NAC transcription factor NTL8 regulates gibberellic acid-mediated salt signaling in Arabidopsis seed germination[J]. The Plant Journal,2008,55(1):77-88.
[14]Delessert C,Kazan K,Wilson I W,et al. The transcription factor ATAF2 represses the expression of pathogenesis-related genes in Arabidopsis[J]. The Plant Journal,2005,43(5):745-757.
[15]Lin R M,Zhao W S,Meng X B,et al. Rice gene OsNAC19 encodes a novel NAC-domain transcription factor and responds to infection [JP2]by Magnaporthe grisea[J]. Plant Science,2007,172(1):120-130.
[16]Donze T,Qu F,Twigg P,et al. Turnip crinkle virus coat protein inhibits the basal immune response to virus invasion in Arabidopsis by binding to the NAC transcription factor TIP[J]. Virology,2014,449(20):207-214.
[17]Nuruzzaman M,Sharoni A M,Kikuchi S. Roles of NAC transcription factors in the regulation of biotic and abiotic stress responses in plants[J]. Frontiers in Microbiology,2013,4(4):248.
[18]Oh S K,Lee S,Yu S H,et al. Expression of a novel NAC domain-containing transcription factor (CaNAC1) is preferentially associated with incompatible interactions between chili pepper and pathogens[J]. Planta,2005,222(5):876-887.
[19]Mysore K S,Crasta O R,Tuori R P,et al. Comprehensive transcript profiling of Pto-and Prf-mediated host defense responses to infection by Pseudomonas syringae pv. tomato[J]. The Plant Journal,2002,32(3):299-315.
[20]Huang W,Miao M,Kud J,et al. SlNAC1,a stress-related transcription factor,is fine-tuned on both the transcriptional and the post-translational level[J]. The New Phytologist,2013,197(4):1214-1224.
[21]Le Hénanff G,Profizi C,Courteaux B,et al. Grapevine NAC1 transcription factor as a convergent node in developmental processes,abiotic stresses,and necrotrophic/biotrophic pathogen tolerance[J]. Journal of Experimental Botany,2013,64(16):4877-4893.
[22]Mcgrann G R,Steed A,Burt C A,et al. Contribution of the drought tolerance-related Stress-responsive NAC1 transcription factor to resistance of barley to Ramularia leaf spot[J]. Molecular Plant Pathology,2015,16(2):201-209.
[23]Selth L A,Dogra S C,Rasheed M S,et al. A NAC domain protein interacts with tomato leaf curl virus replication accessory protein and [JP3]enhances viral replication[J]. The Plant Cell,2005,17(1):311-325.
[24]Tamura K,Peterson D,Peterson N,et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,2011,28(10):2731-2739.
[25]Gasteiger E,Hoogland C,Gattiker A,et al. Protein identification and analysis tools on the ExPASy server [M]. The proteomics protocols handbook. New York:Humana Press,2005:571-607.
[26]Blom N,Gammeltoft S,Brunak S. Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J]. Journal of Molecular Biology,1999,294(5):1351-1362.
[27]Horton P,Park K J,Obayashi T,et al. WoLF PSORT:protein localization predictor[J]. Nucleic Acids Research,2007,35(Web Server issue):W585-W587.
[28]Arnold K,Bordoli L,Kopp J,et al. The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J]. Bioinformatics,2006,22(2):195-201.
[29]Zhu F,Xu M,Wang S,et al. Prokaryotic expression of pathogenesis related protein 1 gene from Nicotiana benthamiana:antifungal activity and preparation of its polyclonal antibody[J]. Biotechnology Letters,2012,34(5):919-924.
[30]Sadowski I,Ma J,Triezenberg S,et al. GAL4-VP16 is an unusually [JP3]potent transcriptional activator[J]. Nature,1988,335(6190):563-564.
[31]Courey A J,Holtzman D A,Jackson S P,et al. Synergistic activation by the glutamine-rich domains of human transcription factor Sp1[J]. Cell,1989,59(5):827-836.
[32]Mermod N,Oneill E A,Kelly T J,et al. The proline-rich transcriptional activator of CTF/NF-I is distinct from the replication and DNA binding domain[J]. Cell,1989,58(4):741-753.[ZK)][HT][HJ][HT][FL)]
[KH*4D]
[HT8.]
[33]陶华平. 核定位信号研究进展[J]. 生物学通报,2014,49(8):5-10.
[34]Kalderon D,Richardson W D,Markham A F,et al. Sequence requirements for nuclear location of simian virus 40 large-T antigen[J]. Nature,1984,311(5981):33-38.
[35][JP3]Dingwall C,Laskey R A. Nuclear targeting sequences—a consensus?[J]. Trends in Biochemical Sciences,1991,16(12):478-481.
[36]Nogueira F S,Schlogl P S,Camargo S R,et al. SsNAC23,a member of the NAC domain protein family,is associated with cold,herbivory and water stress in sugarcane[J]. Plant Science,2005,169(1):93-106.
[37]李伟,韩蕾,钱永强,等. 非生物逆境胁迫相关NAC转录因子的生物信息学分析[J]. 西北植物学报,2012,32(3):454-464.
[38]康美玲,周振华,田忠景,等. 抗逆性转录因子NAC的生物信息学分析[J]. 湖北农业科学,2014,53(17):4199-4204.
[39]Ernst H A,Olsen A N,Larsen S,et al. Structure of the conserved domain of ANAC,a member of the NAC family of transcription factors[J]. EMBO Reports,2004,5(3):297-303.
[40]Chen Q,Wang Q,Xiong L,et al. A structural view of the conserved domain of rice stress-responsive NAC1[J]. Protein & Cell,2011,2(1):55-63.

