基于SRAP分子标记的黄颡鱼属遗传多样性分析
龚瑞玥++方春林++万正义++舒汉鼎++胡成钰++毛慧玲
doi:10.15889/j.issn.1002-1302.2016.10.021
摘要:选用64对SRAP引物对黄颡鱼属进行遗传多样性研究。结果显示,有46对引物能成功扩增,其中19对条带清晰、显示较高多态性。检测其PCR产物共获得179条,每对引物扩增条带5~15条不等,扩增片段介于90~510 bp 之间,物种间遗传相似系数范围为0.500~1.000,黄颡鱼属各物种间具有较大的遗传差异性。SRAP聚类结果显示,瓦氏黄颡鱼与其他物种差异最大,普通黄颡鱼、长须黄颡鱼和中间黄颡鱼亲缘关系较近。遗传多样性分析结果显示,5种黄颡鱼遗传多样性处于较高水平,有助于后续深入对黄颡鱼属各物种亲缘关系和系统演化进行分析。
关键词:黄颡鱼属;SRAP;遗传多样性;遗传相似系数
中图分类号: S961.2文献标志码: A文章编号:1002-1302(2016)10-0092-03
收稿日期:2015-08-15
基金项目:江西省科技支撑资助项目(编号:20112BBF60011);江西省教育厅科技资助项目 (编号:GJJ14152)。
作者简介:龚瑞玥(1990—),女,江西南昌人,硕士研究生,主要从事生物化学与分子生物学研究。 E-mail:enidpace713@gmail.com。
通信作者:毛慧玲,硕士,教授,主要从事鱼类遗传与育种研究。E-mail:huilinm6@163.com。黄颡鱼俗称黄丫头,是备受我国老百姓喜爱的主要经济鱼类之一。据中国动物志中报道,黄颡鱼属共有普通黄颡鱼(P.eupogon)、长须黄颡鱼(P.fulvidraco)、瓦氏黄颡鱼(P.vachelli)、光泽黄颡鱼(P.nitidus)和中间黄颡鱼(P.intermedius)等5种,中间黄颡鱼仅分布于珠江水系及海南岛,其他4种均广泛分布于长江水系[1-2]。随着市场对黄颡鱼需求的增大,其养殖规模不断扩大,随之出现鱼种混杂,品种退化现象,主要表现在生长速度变慢、抗病力及抗逆力降低等方面。从良种选育、养殖和种质资源保护角度考虑,亟须提供一种有效的方法来鉴别这些物种,并研究其遗传多样性。
序列相关扩增多态性(SRAP,sequence-related amplified polymorphism)分子标记是由美国加州大学蔬菜系Li等于2001年提出[3],该标记具有简单、高效、高共显、高重复、易测序等优点[4-5],广泛运用于物种遗传多样性、品种鉴定和亲缘关系的研究当中。本研究从分子水平角度,应用SRAP分子标记技术来鉴别黄颡鱼属5个种,并探讨它们的遗传多样性及其亲缘关系,为黄颡鱼属种质资源保护和开发利用提供理论资料[6-7],对黄颡鱼渔业资源的可持续发展具有十分重要的科学指导意义。
1材料与方法
1.1样品采集
2011年6月至2012年9月,从江西鄱阳湖和广东西江野外采得鱼类(表1),并应用传统形态学方法将它们逐一进行分类。
1.2基因组DNA
每个物种随机选取4尾,共20尾样品。取背部肌肉,采用苯酚-氯仿法提取基因组DNA[8]。
1.3SRAP引物来源
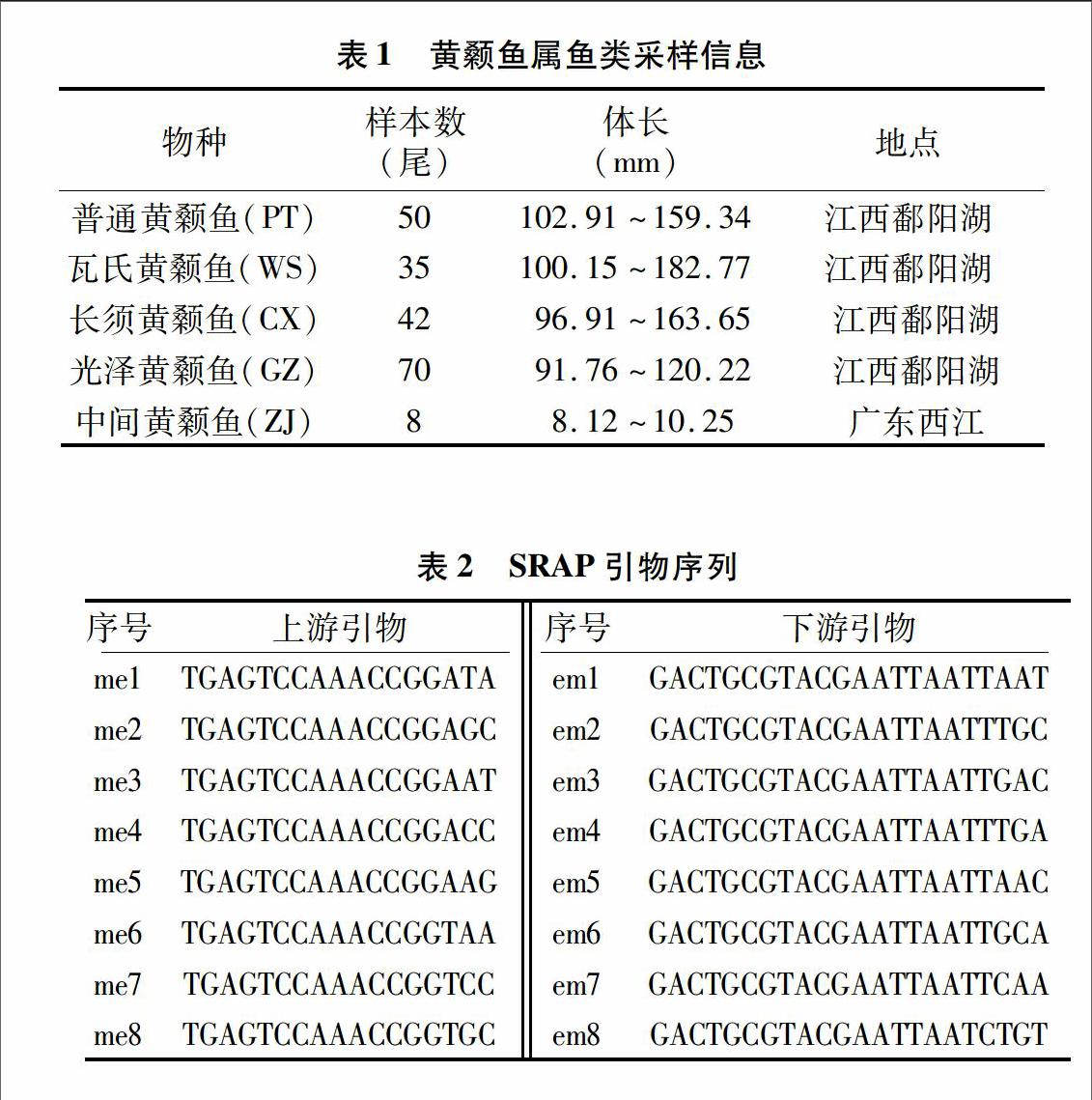
由表2可知,SRAP引物序列,将上游引物与下游引物交叉组合得到64对SRAP引物,按照简写上下游序号原则进行命名,如上游2号引物me与下游3号引物em组合而成的SRAP引物命为m2e3。
1.4PCR扩增和电泳检测
20 μL SRAP的PCR扩增体系含:100 ng/μL模板DNA 1.0 μL,50 μmol/L上下游引物各0.06 μL,10×PCR buffer (Mg2+) 2.0 μL,200 μmol/L dNTPs 1.6 μL,5.0 U/μL Taq DNA聚合酶0.2 μL,ddH2O 15.08 μL;PCR反应条件为:94 ℃ 预变性5 min;94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸2 min,5个循环;94 ℃变性1 min,53 ℃退火1 min,72 ℃延伸2 min,35个循环;72 ℃延伸7 min。采用6%的聚丙烯酰胺凝胶电泳检测PCR产物多态性。
1.5数据统计分析
对着SRAP的PCR扩增图谱,自下而上按迁移率不同排列计数,无带则赋值为“0”,有带则赋值为“1”,建立不同SRAP引物“0-1”矩阵表。应用软件包括POPGENE32、POWERMARKER V3.25等不同软件处理、分析“0-1”矩阵表数据,获得等位基因数(Na)、有效等位基因数(Ne)、等位基因有效率(A)、多态信息含量(PIC)等遗传多样性指标。分别应用NTSYS-pc2.0软件、UPGMA程序计算遗传相似系数[9]、聚类分析[10]。
2结果与分析
2.1SRAP扩增结果
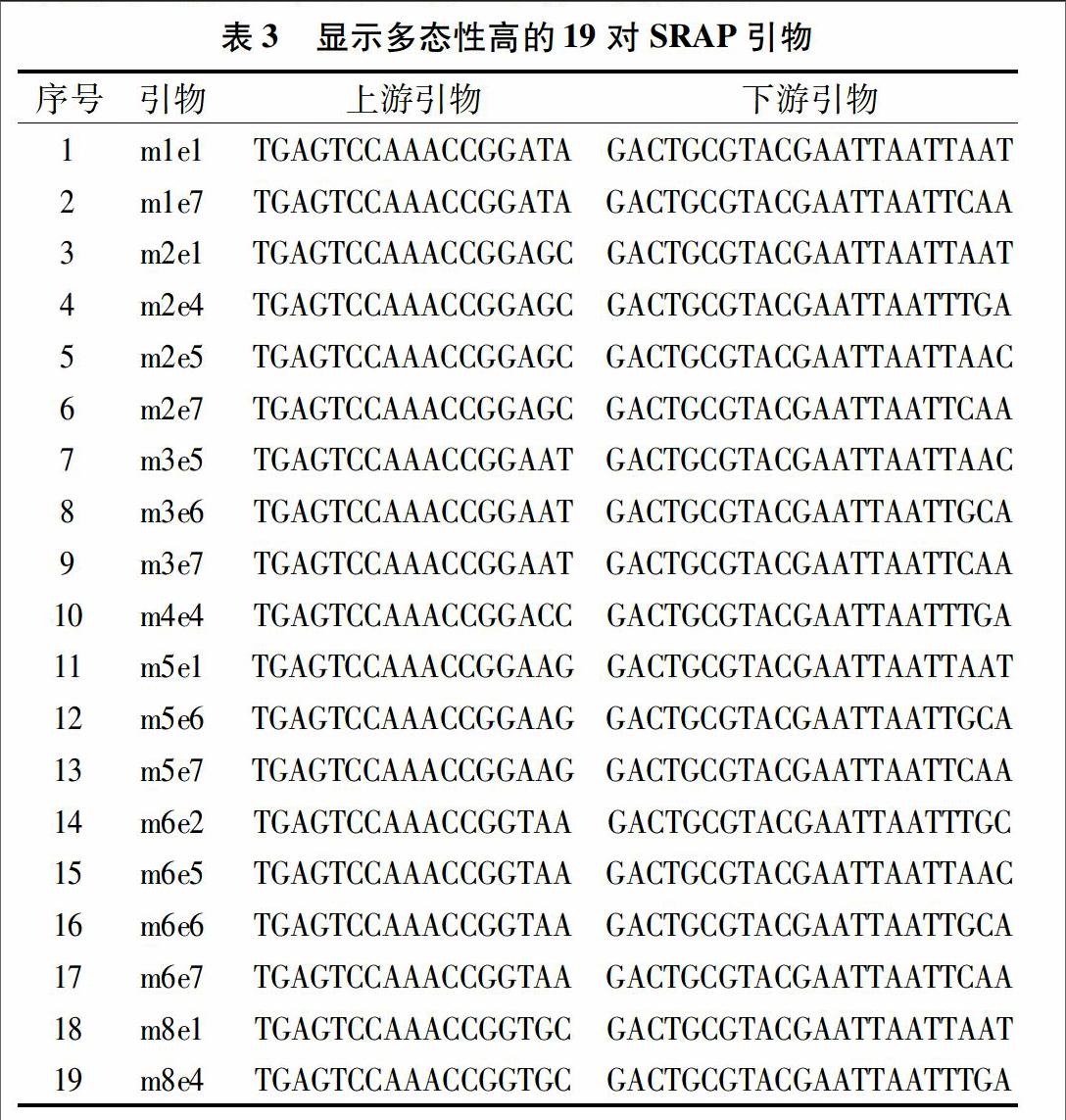
由64对SRAP引物扩增结果获悉,46对引物能成功扩增,占引物总数的71.9%。检测引物扩增的PCR产物,共获得179条,每对引物扩增条带5~15条不等,扩增片段介于 90~510 bp之间。由此可确定46对引物是黄颡鱼属SRAP标记的有效引物。
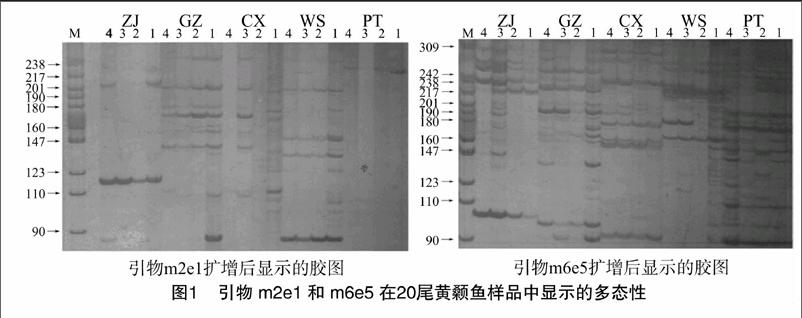
选取其中条带清晰、多态性较高的19对引物(表3)用于分析。DNA图谱(图1)清楚地显示了引物m2e1、m6e5在20尾黄颡鱼样品特异性扩增产物所具有的多态性,相同引物下扩增出的同种不同个体均能显示明显的相同特征性条带,体现出种内差异较小;而同属不同种间条带差异明显,多态性高,清晰地体现出黄颡鱼属5个种之间的差异。
2.2遗传多样性指标分析
19对SRAP引物对黄颡鱼属5个物种20尾样品进行扩增,检测到118个等位基因,每个位点可扩增出的等位基因数为4~8个不等,平均数为6.2个,大小在90~510 bp之间,计算出有效等位基因数。由表4可知,有效等位基因数等位基因有效率、多态信息含量等平均数值分别为5.348 6、0.834 2、0.766 7。由表5可见,除中间黄颡鱼外的4个物种Shannon信息指数均较高,特别是长须黄颡鱼高达0.683 9。上述数据都是反映遗传多样性的指标,其数值越大,说明基因丰富度越高。
2.3黄颡鱼属各种聚类分析
由图2可知,根据PCR产物的多态性和聚类分析结果,5种黄颡鱼间遗传相似系数范围为0.500~1.000,这说明黄颡鱼属各物种间具有较大的遗传差异性。其中,不同物种间遗传相似系数低,同一物种间遗传相似系数高,说明亲缘关系近。根据材料间的遗传相似系数对黄颡鱼属进行聚类分析,在遗传相似系数为0.50水平时,可将瓦氏黄颡鱼与其他物种区分开;在遗传相似系数为0.56水平时,可明确地将光泽黄颡鱼与其他3个物种区分开来;在遗传相似系数为 0.64 的水平下,可将普通黄颡鱼与长须黄颡鱼、中间黄颡鱼分开;随后在0.72水平时,长须黄颡鱼和光泽黄颡鱼分为2类。综合来说,供试的黄颡鱼属5个物种相似系数不高,遗传距离相对较远,分属于不同种。19对SRAP引物所构成的各物种的特异性谱系,能够为黄颡鱼属各物种提供更准确的分子水平上的鉴定。
3讨论
SRAP作为一种新型的分子标记技术,不需要像RAPD、SSR标记那样花费大量人力和时间进行引物设计开发,重复试验结果稳定,PCR扩增所需求的DNA质量要求低,最为重要的一点是SRAP标记稳定且多态性可与AFLP标记相媲美[11-12]。本试验利用了19对SRAP引物对黄颡鱼属进行遗传多样性研究,结果显示5种黄颡鱼遗传多样性处于较高水平。相对于其他几个物种而言,中间黄颡鱼的多样性指数偏低,原因可能是广州西江采样点为经济高度发达区域,环境污染、过度捕捞等人为干扰比较严重,而其余4个物种都采于生态环境较好的鄱阳湖,物种受到破坏程度相对较小[13]。
对196个扩增条带构建的各物种特异性谱系,虽然能够用于对黄颡鱼属幼苗和幼鱼的鉴别,但是由于SRAP引物扩增出的条带较多,需通过多对引物扩增检测才能对黄颡鱼属进行区分,这将浪费大量人力、物力,如果将SRAP转化为SCAR标记能够克服这一缺点[14-17]。丁炜东等通过SRAP方法对3种草鱼基因组进行分析,设计了3对SCAR引物,其中SCAR1能够区分人工养殖和野生草鱼种群[18]。由此可见,将SRAP标记转化为SCAR标记后,其特异性和稳定性均大幅度提高,可以更方便快捷地应用于相似物种的鉴定。
鲿科鱼类的分类系统一直比较混乱,在属的划分上还存在许多争议,黄颡鱼属亦如此。肖调义等对洞庭湖4种黄颡鱼的生态学特征和遗传多样性的RAPD分析结果表明,长须黄颡鱼与瓦氏黄颡鱼具有较近的亲缘关系,而光泽黄颡鱼与普通黄颡鱼关系较近[19],与本研究结果相反。SRAP聚类结
果表明普通黄颡鱼和长须黄颡鱼关系相近,光泽黄颡鱼与瓦氏黄颡鱼关系相近,这与丁言伟对黄颡鱼属分子系统发育研究结果[20]相近;与赵哲霞等的SSR分析结果[9]略有不同,主要原因可能在于普通黄颡鱼和瓦氏黄颡鱼之间的位置调换了,各物种种内遗传差异与SSR分析相比较小。其可能的原因在于SSR引物设计是根据普通黄颡鱼相关序列设计的,扩增出来的多态性条带相对于其他物种较多;而SRAP分子标记是通过独特的引物设计对ORFs进行扩增。
基因组DNA不同区域进化速率相差很大,而分子标记是根据特定序列设计引物进行扩增检测来解决所研究物种的亲缘关系和系统发生,结果肯定存在差别。至于选取那种方法得出的结论最准确、具有更高的可信性都还没有定论,需要之后进一步研究比较。
参考文献:
[1]褚新洛,郑葆珊,戴定远,等. 中国动物志:硬骨鱼纲鲇形目[M]. 北京:科学出版社,1999:1-43.
[2]刘世平. 鄱阳湖黄颡鱼生物学研究[J]. 动物学杂志,1997,32(4):10-15.
[3]Li G,Quiros C F. Genetic analysis,expression and molecular characterization of BoGSL-ELONG,a major gene involved in the aliphatic glucosinolate pathway of brassica species[J]. Genetics,2002,162(4):1937-1943.
[4]Budak H,Shearman R C,Parmaksiz I,et al. Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationship using ISSRs,SSRs,RAPDs,and SRAPs[J]. Theoretical and Applied Genetics,2004,109(2):280-288.
[5]Budak H,Shearman R C,Parmaksiz I,et al. Molecular characterization of buffalograss germplasm using sequence-ralated amplified polymorphism markers[J]. Theoretical and Applied Genetics,2004,108(2):328-334.
[6]Sekino M,Hara M. Inheritance characteristics of microsatellite DNA loci in experimental families of Japanese flounder Paralichthys olivaceus[J]. Marine Biotechnology,2001,3(4):310-315.
[7]Nakabo T. The third international symposium on the marine sciences
of the yellow sea[C]. Qingdao:Ocean Press,1994:81.
[8]赵哲霞,蒋珊,王滨花,等. 黄颡鱼属SSR分子鉴定及其遗传多样性[J]. 南昌大学学报:理科版,2014,38(5):498-501.
[9]Yu H T,Lee Y J,Huang S W,et al. Genetic analysis of the populations of Japanese anchovy (Engraulidae:Engraulis japonicus) using microsatellite DNA[J]. Marine Biotechnology,2002,4(5):471-479.
[10]何琳,王群. 基于PCR的SSR标记分离方法综述基因组学与应用生物学[J]. 遗传组学与应用生物学,2010,29(4):775-782.
[11]Ferriol M,Belén Picó M,Nuez F. Genetic diversity of some accessions of Cucurbita maxima from Spain using RAPD and SBAP markers[J]. Genetic Resources and Crop Evolution,2003,50(3):227-238.
[12]Lin X Y,Kaul S,Rounsley S,et al. Sequence and analysis of chromosome 2 of the plant Arabidopsis thaliana[J]. Nature,1999,402:761-768.
[13]冉玮,张桂蓉,王卫民,等. 利用SRAP标记分析3个团头鲂群体的遗传多样性[J]. 华中农业大学学报,2010,29(5):601-606.
[14]谢传晓,朱苏文,李培金,等. 玉米对生性状两个显性基因SCAR分子标记[J]. 高技术通讯,2002,12(8):38-41.
[15]张增艳,辛志勇,陈孝,等. 源于L1的小麦抗黄矮病基因的特异PCR标记及辅助育种的研究[J]. 作物学报,2002,28(4):486-491.
[16]石金锋,贾建航,金德敏,等. 紫菜无性系特异SCAR标记的获得[J]. 海洋学报,2003,25(1):128-131.
[17]La Rosa R,Angiolillo A,Guerrero C,et al. A first linkage map of olive (Olea europaea L.) cultivars using RAPD,AFLP,RFLP and SSR markers[J]. Theoretical and Applied Genetics,2003,106(7):1273-1282.
[18]丁炜东,曹丽萍,曹哲明. 草鱼种质相关SRAP及SCAR的分子标记[J]. 动物学报,2008,54(3):475-481.
[19]肖调义,张学文,章怀云,等. 洞庭湖四种黄颡鱼基因组DNA遗传多样性的RAPD分析[J]. 中国生物工程杂志,2004,24(3):84-89.
[20]丁言伟. 黄颡鱼属(硬骨鱼纲,鲿科)鱼类分子系统发育及种群遗传结构的研究[D]. 武汉:华中农业大学,2005:1-58.张仕林,许玉超,帅强,等. 洋葱不同组织RNA提取方法比较分析[J]. 江苏农业科学,2016,44(10):95-97.

