厌氧氨氧化功能微生物PCR—DGGE分析方法优化
毕玮++李祥++刘恒蔚++黄勇++袁怡++刘忻

doi:10.15889/j.issn.1002-1302.2016.10.026
摘要:基于厌氧氨氧化(anaerobic ammonium oxidation,Anammox)开发的相关工艺在污水脱氮处理中具有重要作用;但Anammox微生物是一类目前尚不能分离和纯培养的微生物,分析其功能微生物的类型,对于研究其生物学机理并改良其工艺具有重要意义。本研究对变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)在Anammox微生物应用中的分析条件进行了优化,以提高PCR-DGGE的分析效果。结果表明,采用针对16S rDNA V3区的 GC-F341/R518引物组合,PCR最佳退火温度为64 ℃;DGGE电泳中,最佳上样量为5~7 μL,最佳胶浓度为8%,最佳电压/时间为80 V/16 h。优化后的PCR-DGGE具有准确性高和灵敏度好的特点。
关键词:厌氧氨氧化;PCR-DGGE;微生物类型;条件优化
中图分类号: S188文献标志码: A文章编号:1002-1302(2016)10-0110-03
收稿日期:2015-09-30
基金项目:国家自然科学基金(编号:51478284);国家自然科学基金青年科学基金(编号:51408387)。
作者简介:毕玮(1990—),男,硕士研究生,主要从事环境生物催化机理研究。E-mail:1012296010@qq.com。
通信作者:刘恒蔚,博士,副教授。E-mail:liuhw@mail.usts.edu.cn。厌氧氨氧化(anaerobic ammonium oxidation,Anammox)是指厌氧氨氧化细菌在厌氧条件下以羟胺和肼为中间产物,以NH+4 为电子供体、NO2-为电子受体,将NH+4 和NO-2 转变为N2 的过程[1]。该反应颠覆了人们对自然界氮素循环的传统认识。此前普遍认为通过异养反硝化即硝酸盐还原生成N2,是地表氮素生成N2的唯一自然途径,而目前却发现Anammox反应的广泛存在。据估计由AAOB完成了海洋生态系统氮转化的30%~50%[2]。Anammox过程在地表氮素循环,包括农业水体氮素循环中起着重要作用。基于Anammox过程研究者开发了多种污水处理工艺[3-6]。与传统的硝化-反硝化工艺相比,优点在于不需要额外电子供体,同时也可使剩余污泥产量降至最低,从而节省大量污泥处置费用。厌氧氨氧化细菌(即Anammox菌)是一类不可培养的微生物类型,难以使用传统手段进行研究。目前共发现了包括Candidates Kuenen stuttgartiensis、Candidatus Brocadia anammoxidans、Candidatus Anammoxoglobus sulfate在内的5个属12个种之多,而且不同的研究得到的优势种群不同[7]。分析其功能微生物的类型,对于研究其生物学机理并改良其工艺具有重要意义。
PCR结合变性梯度凝胶电泳(deatring gradient gel electrophoresis,DGGE)技术不但克服了传统的微生物培养方法的局限性,而且可以对微生物种群进行定性和定量,以及预测微生物种群的系统发育关系,成为研究环境微生物多样性的常用手段之一。其主要原理是序列不同的DNA双链解链所需的变性剂(尿素和甲酰胺)浓度也不同,在含梯度变性剂的凝胶电泳中,不同的DNA序列在不同的浓度处发生解链,导致空间构型改变,电泳速率急剧下降,从而停留在不同的位置,形成条带分开的指纹图谱。为了提高DGGE的分辨率,可在引物的一端加上1个含30~50个碱基的“GC夹板”(GC clamp)[8]。理论上,DGGE可以检测出只有1个碱基差异的DNA序列。但是DGGE效果受多种因素影响,特别是对于不同的引物和不同的样品,PCR-DGGE分析的参数差别很大。例如使用不同引物在对厌氧污泥样本PCR-DGGE分析中[5-6,9-15],聚丙烯酰胺凝胶浓度从5%~10%、电泳电压从75~200 V均有使用。DGGE的分辨率直接影响结果的准确性和可靠性,而分辨率又会受到电泳中的变性剂梯度范围、时间、电压以及上样量等因素影响;因此针对具体的样品类型和引物,需对DGGE试验各个关键参数进行优化,才能达到预期的效果。16S rDNA是细菌系统分类研究中最常用的分子指纹,分子大小适中,在结构与功能上具有高度的保守性。基于16S rDNA的V3区是微生物种群分析最常采用的目标序列之一,在分子生态研究中有广泛的应用。
本研究基于Anammox微生物16S rDNA的V3区的 GC-F341/R518引物组合,对PCR的退火温度和DGGE的时间/电压、胶的浓度以及上样量进行了优化,该结果对于 Anammox 反应相关微生物的分析具有参考意义。
1材料与方法
1.1材料
1.1.1试验材料样品来自苏州科技学院环境生物技术研究所亚硝酸盐型Anammox污泥。
1.1.2主要仪器与试剂DNA快速提取试剂盒购自MPBIO公司;NanoDrop2000仪;ExTaq酶购自TaKaRa公司;DGGE电泳仪(AD0412LS-C50)、DGGE电泳槽(DCode型)、凝胶成像系统(Gel DocTM XR+)购自美国伯乐公司;引物由上海生工生物工程有限公司合成。
1.2方法
1.2.1核酸提取与PCR-DGGE分析污泥取样后立即使用液氮研磨成干粉,-80 ℃保存备用。使用DNA快速提取试剂盒提取DNA,提取的DNA经1%琼脂糖电泳检测和NanoDrop2000仪定量并检测质量后使用。使用针对细菌16S rRNA基因V3可变区通用引物进行PCR,引物序列为:CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTA
CGGGAGGCAGCAG(即GC-F341);ATTACCGCGGCTGCTGG(即R518)。
PCR扩增体系总体积为30 μL,包括15 ng模板DNA,ExTaq酶1.5 U,10 μmol/L引物各1.5 μL,dNTPs Mixture(各2.5 mmol/L)3 μL,ExTaq酶buffer 3 μL,ddH2O补足至 30 μL。PCR扩增采用98 ℃预变性30 s;98 ℃变性10 s,根据设置的温度退火30 s,72 ℃延伸5 s,共29个循环;72 ℃延伸5 min。扩增产物经2%琼脂糖电泳检测后,按不同上样量均加入15 μL上样缓冲液,并使用ddH2O补足至25 μL,利用DGGE电泳仪和DGGE电泳槽在变性梯度为45%~65%的聚丙烯酰胺凝胶中电泳(100%的变性剂浓度为体积分数40% 甲酰胺和7 mol/L 尿素)。参考乐晓萍等的银染方法[16]进行染色。银染时凝胶在固定液(含体积分数10%乙醇和0.5%的乙酸)轻摇固定10 min,超纯水漂洗1 min后放入染色液(含体积分数2%硝酸银)中染色15 min,超纯水快速冲洗2次后放入显影液(含体积分数1.5%氢氧化钠、体积分数02%甲醛)中至显影清晰,用超纯水冲去残余的显影液。银染结果通过凝胶成像系统照相分析。
1.2.2PCR-DGGE优化(1)PCR退火温度的优化:退火温度从61~66 ℃之间设置6个梯度,梯度为1 ℃,PCR产物通过2.0%琼脂糖电泳,选择最合适的退火温度用于PCR-DGGE分析以及后续优化过程。(2)电泳时间和电压的优化:制备8%聚丙烯酰胺凝胶,设置60、80、100、120 V共4个电压梯度;每隔2 h上样1次,共上样5次,即各设置5个时间梯度;上样量为4.0 μL。在60 ℃条件下对不同电压下分别进行不同时间进程的电泳试验,以确定最佳的电压/时间组合。(3)电泳上样量的优化:配制总体积相同、但PCR扩增产物不同的上样体系,上样量(PCR扩增产物)为1~10 μL,梯度1 μL。使用最适变性剂梯度和已确定的最佳电压/时间条件,在8%聚丙烯酰胺凝胶中进行电泳。(4)凝胶浓度优化:使用上述已确定的上样量、最佳电泳时间和电压组合,分别配制浓度为6%、8%和10%的聚丙烯酰胺凝胶,根据电泳效果来确定最适合的胶浓度。
2结果与分析
2.1土壤基因组DNA的提取
提取的Anammox样品DNA经1.0%的琼脂糖凝胶电泳,结果(图1)表明,DNA样品质量较好,适合进行PCR-DGGE分析。
2.2PCR扩增的退火温度优化
图2是61~66 ℃不同退火温度下的PCR电泳结果。图2表明,退火温度为64 ℃时,扩增效率高,效果最好,是本研究的最佳退火温度。电泳结果显示PCR产物大小与预计结果(230 bp)基本一致。
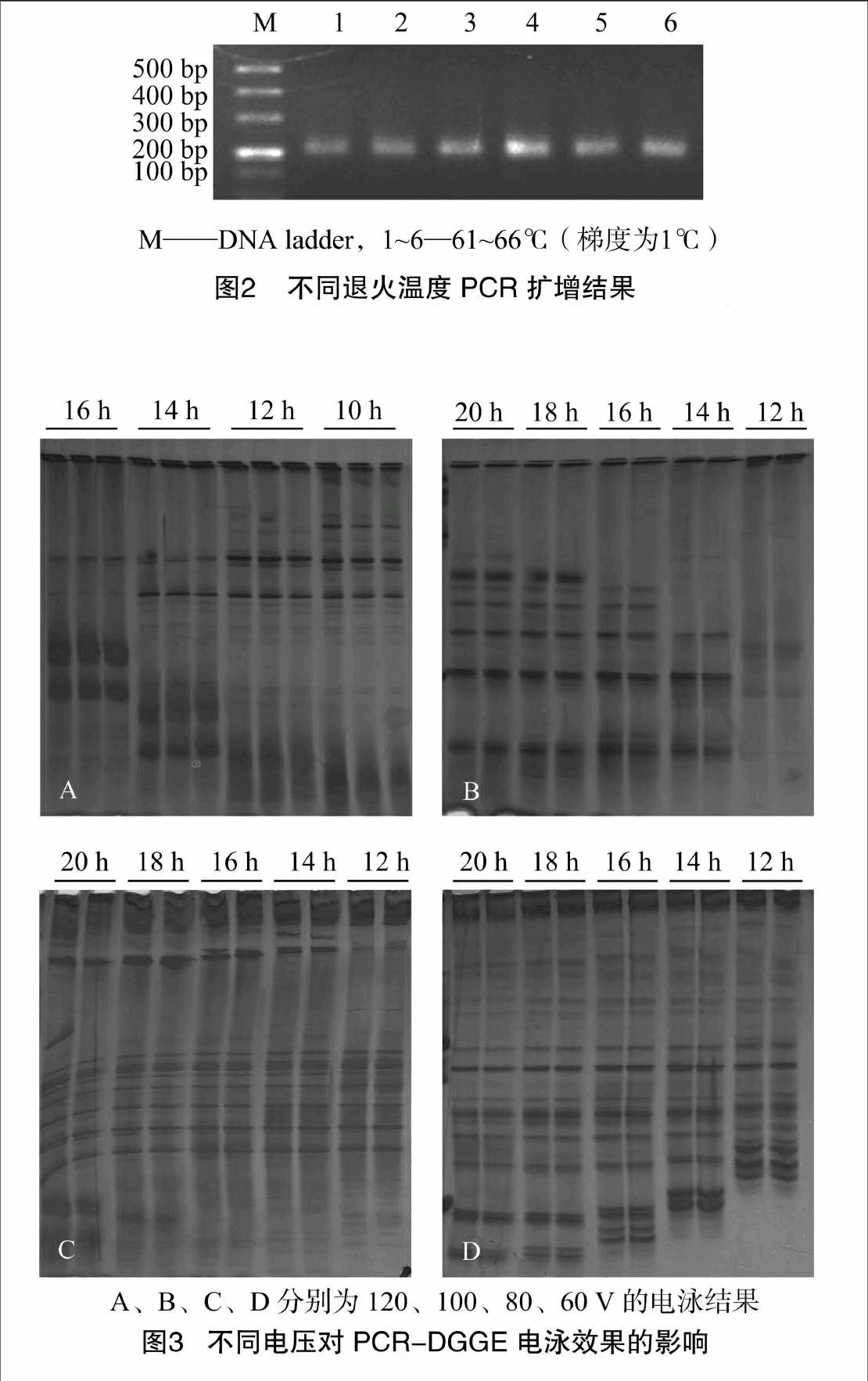
2.3电泳时间和电压的优化
本试验在预试中使用了200 V电压进行DGGE电泳,结果发现,该电压下条带有弥散严重,分离效果较差,因此选择在低电压区进行优化。通过比较120 V(图3-A)、100 V(图3-B)、80 V(图3-C)和60 V(图3-D)的电泳结果说明,电压高低对条带的分离效果影响较为明显,随着电压降低,弥散减弱,且条带数增加,条带更为清晰锐利。在80 V(图3-C)和60 V(图3-D)之间差异不大,均能得到数目较多且清晰锐利的条带。随着电泳时间延长,条带分离更加明显,易于区分,在80 V电压时,多数条带在16 h时分离效果最好,延长电泳时间变化不明显,与60 V的电压时18 h的电泳效果基本一致。鉴于此,选择80 V/16 h作为最佳的电压/时间组合。
2.4电泳上样量的优化
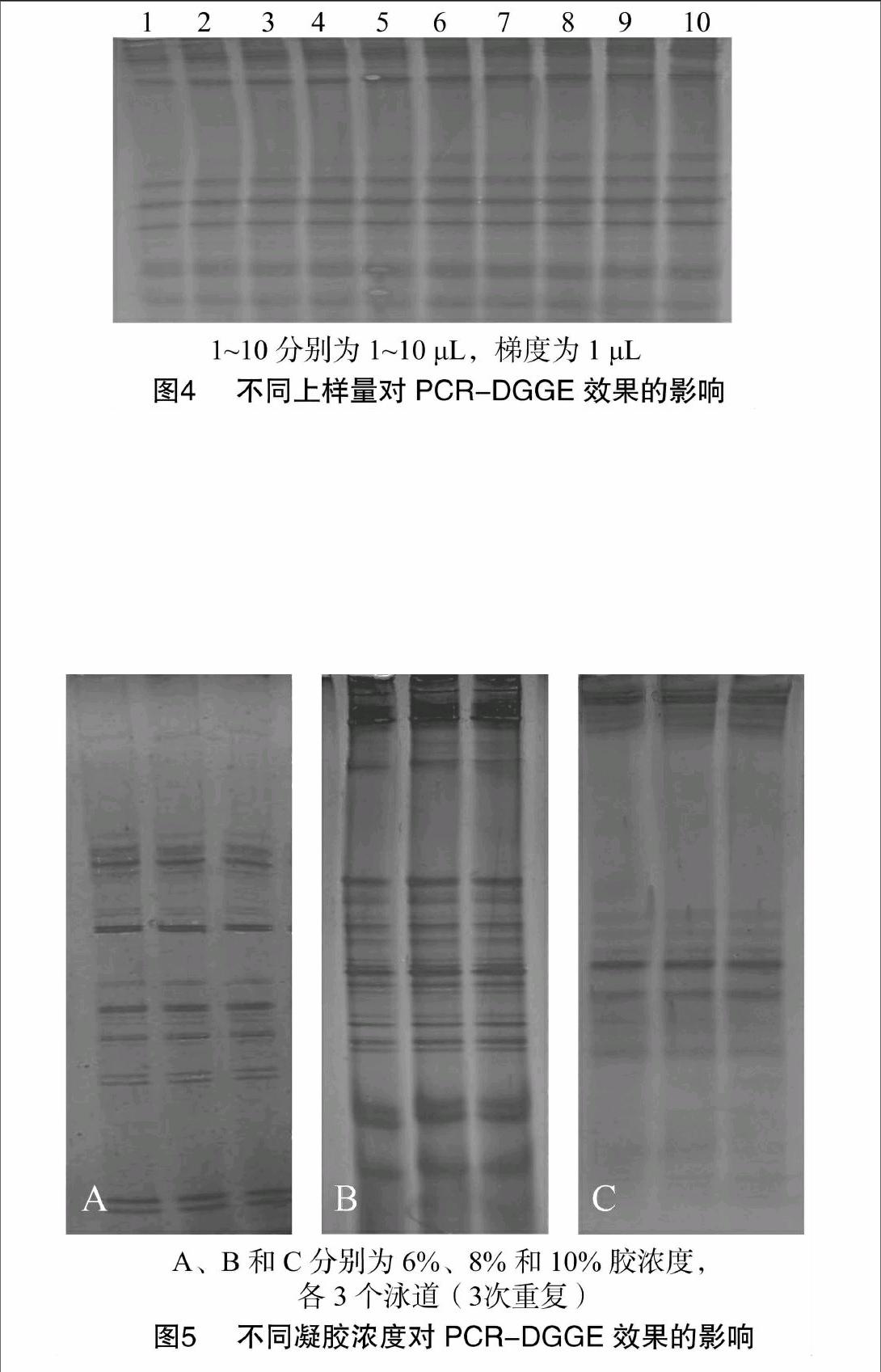
从图4可以看出上样量对DGGE图谱也有着重要的影响。上样量过少,部分条带不清晰,染色后可见的条带数偏少;上样量过多,则图谱的背景过重,不利于后续的分析。考虑到二者的平衡,在Anammox微生物的PCR-DGGE分析中选择5~7 μL的上样量效果最好。
2.5凝胶浓度优化
本试验通过3种不同浓度的凝胶[分别是6%(图5-A)、8%(图5-B)、10%(图5-C)]来判断最佳的凝胶浓度。从图5的3个浓度对比可以看出,8%凝胶的分离度最好,条带清晰锐利,为最佳凝胶浓度选择。
3讨论
DGGE技术被广泛用于分析环境样本微生物群落的生物多样性,适合调查种群的时空变化,并可以通过对目的条带进行序列分析来鉴定群落组成。理想的PCR引物和DGGE分析条件应适合探测更多的微生物类型。例如王宁等对厌氧颗粒污泥PCR-DGGE分析进行优化,发现古菌和细菌在80 V/10 h的电泳条件下均能得到分离效果较好的DGGE 图谱[17]。而本研究结果说明对于Anammox活性污泥,采用GC-F341/R518通用引物,PCR最佳退火温度为64 ℃;变性剂浓度为45%~65%的聚丙烯酰胺凝胶中进行DGGE电泳的最佳上样量为5~7 μL,胶浓度为8%,最佳电压/时间为80 V/16 h。由于本研究采用针对16S rDNA的V3区的特异引物,对于环境样本的不同微生物种类将具有更广泛的适用性。
PCR和电泳是PCR-DGGE分析的2个最关键环节,本试验研究表明适当提高退火温度有利于减少非特异性扩增,上样量的多少与胶浓度的大小也对DGGE的分辨率有重要影响。此外,与一般的PCR对模板DNA要求低不同,本研究中还发现DNA质量对PCR影响较大,且PCR扩增质量显著影响DGGE的分辨率。
参考文献:
[1]Strous M,Fuerst J A,Kramer E H,et al. Missing lithotroph identified as new planctomycete[J]. Nature,1999,400(6743):446-449.
[2]Dalsgaard T,Canfield DE,Petersen J,et al. N2 production by the anammox reaction in the anoxic water column of Golfo Dulce,Costa Rica[J]. Nature,2003,422(6932):606-608.
[3]Van Dongen U,Jetten M S,van Loosdrecht M C. The SHARON-Anammox process for treatment of ammonium rich wastewater[J]. Water Science and Technology,2001,44(1):153-160.
[4]Despland L M,Clark M W,Vancov T,et al. Nutrient removal and microbial communities development in a young unplanted constructed wetland using BauxsolTM pellets to treat wastewater[J]. Science of the Total Environment,2014,484:167-175.
[5]Liang Y,Li D,Zhang X,et al. Stability and nitrite-oxidizing bacteria community structure in different high-rate CANON reactors[J]. Bioresource Technology,2015,175:189-194.
[6]Zhong Y M,Jia X S. Simultaneous ANAMMOX and denitrification (SAD) process in batch tests[J]. World Journal of Microbiology and Biotechnology,2013,29(1):51-61.
[7]Ding S,Zheng P,Lu H,et al. Ecological characteristics of anaerobic ammonia oxidizing bacteria[J]. Applied Microbiology and Biotechnology,2013,97(5):1841-1849.
[8]Strathdee F,Free A. Denaturing gradient gel electrophoresis (DGGE):methods and protocols[M]//Makovet S.DNA Electrophoresis. New York:Humana Press,2013:145-157.
[9]Vipindas P V,Anas A,Jasmin C,et al. Bacterial domination over Archaea in ammonia oxidation in a monsoon-driven tropical estuary[J]. Microb Ecol,2015,69(3):544-553.
[10]Kawagoshi Y,Fujisaki K,Tomoshige Y,et al. Temperature effect on nitrogen removal performance and bacterial community in culture of marine anammox bacteria derived from sea-based waste disposal site[J]. Journal of Bioscience and Bioengineering,2012,113(4):515-520.
[11]Sonthiphand P,Neufeld J D. Evaluating primers for profiling anaerobic ammonia oxidizing bacteria within freshwater environments[J]. PLoS One,2013,8(3):e57242.
[12]Zekker I,Rikmann E,Tenno T,et al. Anammox enrichment from reject water on blank biofilm carriers and carriers containing nitrifying biomass:operation of two moving bed biofilm reactors (MBBR)[J]. Biodegradation,2012,23(4):547-560.
[13]Xiao Y,Zeng G M,Yang Z H,et al. Coexistence of nitrifiers,denitrifiers and Anammox bacteria in a sequencing batch biofilm reactor as revealed by PCR‐DGGE[J]. Journal of Applied Microbiology,2009,106(2):496-505.
[14]Gabarró J,Ganigué R,Gich F,et al. Effect of temperature on AOB activity of a partial nitritation SBR treating landfill leachate with extremely high nitrogen concentration[J]. Bioresource Technology,2012,126:283-289.
[15]于洁. 可降解纤维素的微生物菌群筛选及其性质初探[D]. 天津:天津理工大学,2010.
[16]乐晓萍,杜鹏,张钦宪,等. 聚丙烯酰胺凝胶银染技术改良[J]. 河南医科大学学报,2001,36(4):395-396.
[17]王宁,刘永红,李佳凤,等. 厌氧颗粒污泥PCR-DGGE图谱的优化研究[J]. 中国沼气,2014(3):8-13.李静婷,赵旭耀,刘超凡,等. 热胁迫对转TasHSP16.9拟南芥幼苗生长生理特性的影响[J]. 江苏农业科学,2016,44(10):113-116.

