子宫内膜异位症相关基因和microRNA的挖掘及生物信息学分析
王蕾++欧阳玲


[摘要] 目的 应用生物信息学方法分析和预测子宫内膜异位症(EMs)患者相关基因的表达,为进一步在基因水平上揭示EMs的本质和发现药物治疗靶点、开发新型的治疗药物提供新的依据。 方法 在公共基因芯片数据库(GEO)中下载EMs相关基因数据,应用protparam、MotiScan、SignalP 4.0、NetPhos 2.0、TMHMM、GO、KEGG、STRING、BRB-Array Tools软件等生物信息学技术进行数据挖掘和生物信息学分析研究。 结果 共筛选出91个EMs相关基因以及54个microRNA。这些EMs基因主要与细胞增殖调控、细胞凋亡调控、趋化作用等过程有关。蛋白质网络互作预测得到了19个重要的EMs相关蛋白质靶标,联合靶基因的数据挖掘,发现了134个EMs相关的靶基因。 结论 用生物信息学方法分析基因芯片数据可以获取生物的内在信息,为子EMs的早期诊断提供了新的诊断标志物和思路。
[关键词] 基因芯片;子宫内膜异位症;生物信息学分析;靶基因;microRNA
[中图分类号] R711.710.46 [文献标识码] A [文章编号] 1673-7210(2017)04(a)-0012-05
[Abstract] Objective To analyze and predict the expression of endometriosis (EMs) genes by bioinformatics methods, in order to provide a new basis for revealing the essence of EMs at the gene level and developing new treatment drugs. Methods Download gene dates which were related to EMs in Gene Expression Omnibus(GEO), were mined and analyzed by a series of bioinformatics tools, such as protparam, MotiScan, SignalP 4.0, NetPhos 2.0, TMHMM, GO, KEGG, STRING, BRB-Array Tools. Results 91 EMs related genes and 54 microRNA had been found in this study. These genes mainly involved in the process of cell proliferation regulation, cell apoptosis regulation and chemotaxis. Protein interaction network predicted 19 important EMs-related protein targets. Combined with target gene data mining, 134 EMs-related target genes were found. Conclusion Using bioinformatics method to analyze gene microarray data can acquire inner information of organisms, and provide new diagnostic markers and diagnostic thoughts for the early diagnosis of EMs.
[Key words] Microarray; Endometriosis; Bioinformatics; Target gene; MicroRNA
子宮内膜异位症(endometriosis,EMs)是一种常见的慢性妇科疾病,在女性人群中,发病率为10%~15%[1],其临床表现为不孕、痛经、慢性盆腔痛、性交痛等[2],给年轻的女性带来巨大的痛苦和经济负担。EMs是在子宫腔外部出现经过增殖、出血和再生的子宫内膜样组织,其发病机制尚不清楚[3-4]。由于EMs病因复杂,目前主要治疗手段是手术和激素治疗,但该病的复发率高,达40%~50%[5]。因此,亟需新的有效的EMs治疗方法。
基因调控在EMs的发展中起重要作用[6]。研究EMs患者的基因特征是开发新疗法的有效步骤[7-8],基因芯片数据能够大规模地揭示基因遗传背景。根据基因芯片数据可以发现,免疫内分泌的功能障碍是影响子宫内膜异位的重要因素[9]。生物信息学被应用于整理基因表达、基因功能、基因产物以及细胞功能相关的大量信息,来鉴定发病过程中的关键因子,预测合适的治疗靶标[10]。目前这种方法已被用于改进肝细胞癌[11]、淋巴瘤[12]和口腔癌的诊断[13]。基因芯片技术与生物信息学分析的结合能够为疾病的分子生物学研究提供新的研究视角。
本研究应用基因芯片分析软件BRB-Array Tools对基因芯片公共数据库的EMs相关基因和microRNA进行数据挖掘,并进行microRNA的靶基因预测。用生物信息学的方法对EMs的相关基因进行通路和功能的分析,找出EMs相关蛋白质相互作用的网络调控的关键靶标,研究EMs的发病机制,为进一步在基因水平上揭示EMs的本质和发现药物治疗靶点、开发治疗新药提供新的依据。
1 材料与方法
1.1 材料
从美国国立生物信息技术中心(National Center for Biotechnology Information,NCBI)的公共基因芯片数据库(Gene Expression Omnibus,GEO)[14]下载与EMs相关的基因和microRNA。
1.2 方法
①把EMs相关基因上传到String(Search Tool for the Retrieval of Interacting Genes)数据库在线分析工具(http://string-db.org)[15-16]可获得EMs相关基因蛋白-蛋白相互作用的网络,筛选节点(Hub)蛋白。
②把EMs相关基因上传到DAVID(Database for Annotation,Visualization,and Integrated Discovery)[17],用功能注释工具(Functional Annotation Tool),研究EMs相关基因参与FOTERM_MF_5以及GOTERM_BP_5基因本体数据库(Gene Ontology,GO)[18]的分子功能和生物过程,分析EMs相关基因参与的PANTHER-PATHWAY和KEGG-PATHWAY数据库中的生物学通路。
③应用PicTar2005[19]、TargetScan 5.1[20]、miRanda V5[21]3种软件预测靶基因,有两种或两种以上的软件同时预测到的结果则认为可靠。
2 结果
2.1 EMs相关基因的筛选
从公共基因芯片数据库(Gene Expression Omnibus,GEO)下载与EMs相关的基因,共得到91个相关基因,结果见表1。
2.2 EMs相关基因的分析
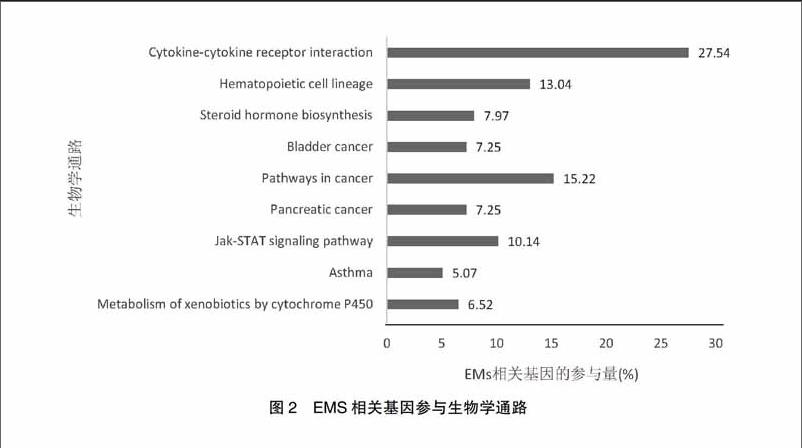
对91个EMs相关基因编码的蛋白进行蛋白-蛋白的相互作用网络分析显示,处于网络节点的蛋白质有19个基因与之对应,分别是EGF、RELA、VEGFA、PCNA、PTEN、PIKCA、MDM2、MMP9、MMP1、NGF、PGR、PTGS2、IL11B、IL6、IL10、CD44、TP53、TNF、FOXO1,說明它们可能在致病中发挥着重要作用。GO富集分析结果显示,EMs相关基因主要涉及细胞增殖、细胞凋亡、细胞代谢、信号转导、趋化作用等反应过程(图1)。生物学通路分析表明,EMs相关基因主要参与细胞因子受体互作、肿瘤通路、造血细胞系、Jak-STAT信号等生物学的通路(图2)。
2.3 microRNA的靶基因预测
在PubMed数据库中检测到54个EMs相关的microRNA,联合靶基因的数据挖掘和预测,共得到134个EMs的相关基因。
3 讨论
EMs给社会和妇女带来了严重的临床上和经济上的负担,因此,需要将研究和资源的效用最大化来提高对疾病的了解,以便发展新的有效的治疗方法。随着近几年生物信息学技术的兴起,基因芯片技术已经成为生物医学研究的基本方法。基因芯片是一种大规模高效率获取生物信息的新型技术,能够检测分析各个组织内的表达基因的差异,随着计算机技术的快速提高和生物数据的急剧增长,生物信息学这一刚刚兴起的学科得到了前所未有的迅速发展[22],尤其是应用生物信息学方法发现新基因和基因芯片,利用已知的核酸序列作为探针,与互补的靶核苷酸序列相互杂交,再进行信号的监测,最终完成定量或者定性的分析,在预防和新药开发、辅助诊断疾病方面有广阔的前景。生物信息学是涉及应用物理学、数学、生物学、化学、计算机等交叉学科的一门新兴学科,应用现有的分析软件和公共数据库,可以探索生物分子结构和功能特性,为后续研究提供新的研究思路和方向。EMs的生物学过程复杂,决定了从基因组水平筛选与转移相关表型的功能基因成为EMs治疗研究的重要方向[23]。
本研究发现,EGF、RELA、VEGFA、PCNA、PTEN、PRKCA、MDM2、MMP9、MMP1、NGF、PGR、PTGS2、IL11B、IL6、IL10、CD44、TP53、TNF、FOXO1在EMs相关基因编码蛋白-蛋白的作用网络中起到节点蛋白的作用,推测这些基因对EMs的发病起重要作用。本研究通过GO富集分析和通路分析发现,EMs相关基因主要与细胞增殖调控、细胞凋亡调控、趋化作用有关。
综上所述,本文应用生物信息学的方法对基于基因芯片数据库挖掘的EMs基因及蛋白进行分析,为揭示EMs相关基因及microRNA的结构、功能、蛋白的相互作用提供了重要依据,发现了关键基因在EMs发生发展过程中可能起到重要的作用,为日后进一步研究EMs的发病机制、发现药物治疗的靶点,及为临床治疗和预防提供了新的切入点。
[参考文献]
[1] Sourial S,Tempest N,Hapangama DK. Theories on the path?鄄ogenesis of Endometriosis [J]. International Journal of Reproductive Medicine,2014,2014:9.
[2] Giudice LC. Clinical pracice. Endometriosis [J]. N Engl J Med,2010,362(25):2389-2398.
[3] Mathew D,Drury JA,Valentijn AJ,et al. In silico,in vitro and in vivo analysis identifies a potential role for steroid hormone regulation of FOXD3 in endometriosis-associated genes [J]. Human Reproduction,2016,31(2):345-354.
[4] Aznaurova YB. Molecular aspects of development and regul?鄄ation of endometriosis [J]. Reproductive Biology and Endo?鄄crinology,2014,12(1):50.
[5] Guo SW. Recurrence of endometriosis and its control [J]. Human Reproduction Update,2009,15(4):441.
[6] Kobayashi H,Imanaka S,Nakamura H,et al. Understanding the role of epigenomic,genomic and genetic alterations in the development of endometriosis(review)[J].Molecular Medi?鄄cine Reports,2014,9(5):1483-1505.
[7] Burney RO,Talbi S,Hamilton AE,et al. Gene expression analysis of endometrium reveals progesterone resistance and candidate susceptibility genes in women with endome?鄄triosis [J]. Endocrinology,2007,148(8):3814-3826.
[8] Baranov VS,Ivaschenko TE,Liehr T,et al. Systems genetics view of endometriosis:a common complex disorder [J]. Euro?鄄pean Journal of Obstetrics & Gynecology & Reproductive Biology,2015,185:59-65.
[9] Khan MA. Genome-wide expressions in autologous euto?鄄pic and ectopic endometrium of fertile women with endom?鄄etriosis [J]. Reproductive Biology and Endocrinology,2012, 10(1):84.
[10] Ping S,Ma C,Liu P,et al. Molecular mechanisms underl?鄄ying endometriosis pathogenesis revealed by bioinformatics analysis of microarray data [J]. Archives of Gynecology and Obstetrics,2016,293(4):1-8.
[11] Zhang Y,Wang S,Dong L,et al. A Systems Biology-Based Classifier for Hepatocellular Carcinoma Diagnosis [J]. Plos One,2011,6(7):e22426.
[12] O'Neill MC,Li S. Neural network analysis of lymphoma microarray data:prognosis and diagnosis near-perfect [J]. BMC Bioinformatics,2003,4(1):13.
[13] Singaraju S,Prasad H,Singaraju M. Evolution of dental informatics as a major research tool in oral pathology [J]. Journal of Oral & Maxillofacial Pathology,2012,16(1):83-87.
[14] Barrett T,Wilhite SE,Ledoux P,et al. NCBI GEO:Archive for functional genomics data sets—update [J]. Nucleic Acids Res,2013,41(Database issue):D991-D995.
[15] Szklarczyk D,Franceschini A,Kuhn M,et al. The STRING database in 2011:functional interaction networks of prot?鄄eins,globally integrated and scored [J]. Nucleic Acids Res,2011,39(Database issue):D561-D568.
[16] Franceschini A,Szklarczyk D,Frankild S,et al. STRING v9.1:protein-protein interaction networks,with increased coverage and integration [J]. Nucleic Acids Res,2013,41(Database issue):D808-D815.
[17] Huang DW,Sherman BT,Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinf?鄄ormatics resources [J]. Nature Protocol,2009,4(1):44-57.
[18] Ashburner M,Ball CA,Blake JA,et al. Gene ontology:tool for the unification of biology. The Gene Ontology Consortium [J]. Nat Genet,2000,25(1):25-29.
[19] Kerk A,Grun D,Poy MN,et al. Combinatorial microRNA target predictions [J]. Nat Genet,2005,37(5):495-500.
[20] Lewis BP,Burge CB,Bartel DP. Conserved seed pairing,often flanked by adenosines,indicates that thousands of human genes are microRNA targets [J]. Cell,2005,120(1):15-20.
[21] Griffiths-Jones S,Saini HK,Dongen SV,et al. Tools for microRNA genomics [J]. Nucleic Acids Research,2008, 36(Database issue):D154-D158.
[22] GuC,ShenT. cDNA microarray and bioinformatic analysis for the identification of key genes in Alzheimer's disease [J]. International Journal of Molecular Medicine,2014,33(2):457-461.
[23] 戴淑真,劉海霞,王斌,等.检测子宫内膜异位症患者血清中差异表达蛋白的研究[J].中国误诊学杂志,2006,6(21):4099-4101.
(收稿日期:2016-12-10 本文编辑:程 铭)

