象草基因的克隆及生物信息学分析
吴娟子 钱晨 张建丽


摘要:从象草中克隆了基因,并对其进行了初步的生物信息学分析。利用转录组测序获得的表达序列信息,设计1对特异性的PCR引物,应用RT-PCR技术从象草中扩增出CpCCoAOMT2基因。对获得的基因进行序列同源性搜索、结构域搜索及3D结构预测、分子系统进化树构建等生物信息学分析。测序结果表明,CCoAOMT2基因全长926 bp,含110 bp的5′-UTR、741 bp的CDS和75 bp的3′-UTR,共编码246个氨基酸,GenBank登录号为BankIt1975804;Blastn和BlastT分析结果显示,[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因及其编码的蛋白在核苷酸水平和氨基酸水平上与多种植物的CCoAOMT基因和CCoAOMT蛋白具同源性;预测CpCCoAOMT2蛋白相对分子质量为27 253,等电点为508,是一个不含跨膜结构域、不含信号肽分子的亲水性、稳定蛋白。分子进化树分析结果表明,CpCCoAOMT2蛋白与禾本科植物的CCoAOMT蛋白亲缘关系最近,与裸子植物和蕨类植物关系较近,而与双子叶植物的关系较远。
关键词:象草;咖啡酰酸辅酶A-O-甲基转移酶;基因克隆;生物信息学分析
中图分类号: Q785文献标志码:
文章编号:1002-1302(2017)16-0033-04
收稿日期:2016-12-14
基金项目:国家自然科学基金(编号:31302025);江苏省农业科技自主创新资金[编号:CX(14)5035];国家牧草产业技术体系盐城综合试验站項目(编号:CARS-35);浙江省宁波市科技计划(编号:2016C10018)。
作者简介:吴娟子(1977—),女,湖北京山人,博士,副研究员,主要从事牧草育种和产业化关键技术研究。E-mail:jzwu2014@jaasaccn。
象草(Cenchrus purpureus Schum)是一种多年生禾本科C4植物,具有高生物量,高光合效率、多用途、不占用耕地的优点。它是世界上生物量最高的草本,年生物量可达45 t/hm2。它可作为草食畜禽的优质青饲料,理想的水土保持作物,高质量人造板、纸浆和木质纤维素类能源的优质原料[1-7]。大量研究表明,植物细胞壁组成和特征对木质纤维素类的高效利用具有重要意义,例如,提高其细胞壁的纤维素含量,降低其木质素含量,可以改善其饲料品质和木质纤维素转化利用效率[8-10];提高其木质素含量,有利于其作为板材的性能。因此,木质素是限制细胞壁降解利用的关键因素之一,如何合理改变象草木质素组成和含量对于有效利用象草具有重要意义。
木质素是一种复杂的酚类聚合物,木质素单体合成是木质素合成的关键环节,研究表明,主要有10种酶参与了木质素单体的合成。目前,国内外通过改变这10种酶基因的表达,调控植物木质素含量与组分的研究进展迅速,已相继从多种不同的植物中分离克隆了多个木质素生物合成代谢中的关键酶基因[10-11]。咖啡酰辅酶A甲基转移酶(caffeoyl CoA O-methyltransferase,CCoAOMT)是植物木质素生物合成过程中一类重要的甲基转移酶,催化咖啡酰CoA甲基化为阿魏酰CoA[12-14]。国内外的研究表明,抑制杨树和辐射松[14]等植物的CCoAOMT基因表达,植物木质素含量下降,同时伴随S/G比值增大,木质素结构变得疏松,易于去除,且不影响植物的正常生长。迄今为止,已经从玉米、小麦、大麦、水稻、绿竹、南荻和柳枝稷等禾本科植物中克隆了CCoAOMT基因。NCBI中已有一个象草CCoAOMT基因(核酸登录号:KJ9957361)的记录,但未见其他研究和相关报道。本研究从象草中分离克隆了另1个CCoAOMT基因,命名为[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ],并对其进行了生物信息学分析,以期为研究象草CCoAOMT基因及其编码蛋白在象草木质素代谢中的功能提供理论依据,为象草的木质素改良提供基础。
1材料和方法
11试验材料
象草品系为eg7,种植于江苏省农业科学院试验田,在自然条件下生长,待植株生长5个月后取植株中部的茎段,迅速置于液氮中,带回实验室-80 ℃保存备用。
12试验方法
121引物设计
基于笔者所在课题组完成的象草转录组测序数据,利用Primer50软件设计1对特异性PCR扩增引物F:5′-GAGTAGTGGGCGACCTGTGA-3′和R:5′-GAAGGGGAAGGAAAACTTATTG-3′,引物序列由通用生物公司合成。
122象草总RNA的提取、纯化及cDNA的合成
采用TRIzol(Invitrogen)试剂提取象草茎总RNA,用RNase-free DNase Ⅰ(Fermentas)除去象草总RNA样品中的DNA污染,12% 琼脂糖凝胶电泳检测总RNA纯化效果,NanoDrop2000检测浓度。参照RevertAid First Strand cDNA Synthesis Kit(Fermentas)说明书进行cDNA的合成。
123象草基因的RT-PCR扩增
25 μL PCR反应体系为:10×LA PCR Buffer 25 μL,MgCl2 25 μL,dNTP Mixture 4 μL,正、反向引物各05 μL,LA Taq DNA酶025 μL,ddH2O 1325 μL,模板cDNA 15 μL。PCR反应使用降落PCR,PCR反应程序为:94 ℃ 5 min;94 ℃ 30 s,退火(1~2循环70 ℃,3~4循环69 ℃,5~6循环68 ℃,7~8循环67 ℃,9~10循环66 ℃,11~12循环65 ℃,13~14循环64 ℃,15~16循环63 ℃,17~18循环62 ℃,19~20循环 61 ℃,21~22循环60 ℃,23~24循环59 ℃,25~26循环 58 ℃,27~28循环57 ℃,29~30循环56 ℃)每个循环40 s,72 ℃ 90 s;72 ℃ 10 min,4 ℃ 10 min。1%琼脂糖凝胶电泳检测PCR扩增产物。采用凝胶回收试剂盒回收纯化目的条带,扩增产物经PCR验证与目标条带一致后送通用生物公司测序。[JP]endprint
124合成序列拼接、比对和生物信息学分析
所得的序列用Sequencher 48进行拼接并根据峰图进行手工校正。测序完成后,3次测序结果中的2次共同核苷酸合并为基因片段最终序列。利用DNAMAN V6和BioXM26软件进行序列比对和氨基酸序列的推导,并通过NCBI的BLAST工具与GenBank中的已知序列比较,进行蛋白序列的相似性分析。利用ProtParam软件在线分析蛋白质的分子结构和理化性质;采用TMHMM在线分析软件预测蛋白质的跨膜结构域;通过SignalIP分析软件对信号肽进行预测;蛋白質的二级结构和三级结构预测分别使用在线分析软件NPSA和SWISS-MODEL完成。利用MEGA606软件对氨基酸序列进行系统进化树分析。
2结果与分析
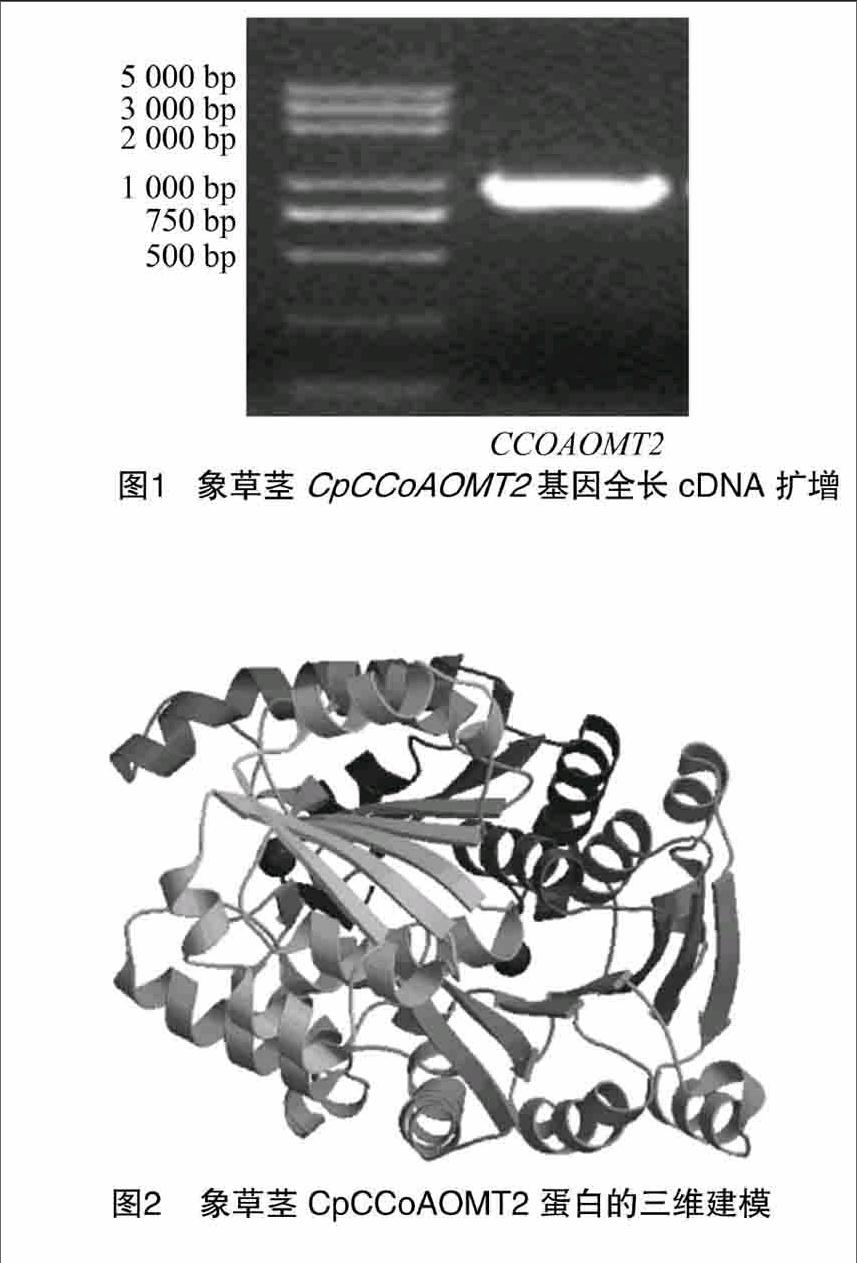
21象草茎基因全长cDNA克隆
以象草茎中RNA为模板,利用F/R引物进行RT-PCR扩增,获得了预期大小的cDNA片段(图1)。将目的片段进行回收、纯化、验证并测序,获得的CpCCoAOMT2基因全长为926 bp,含110 bp的5′-UTR、75 bp的3′-UTR和1个 741 bp(111~851 bp)的开放阅读框,起始密码子为ATG,终止密码子为TGA,G+C含量为649%,共编码246个氨基酸,GenBank登录号为BankIt1975804。
22象草CpCCoAOMT2蛋白的生物信息学分析
用在线网站(http://webExpasyorg/protparam)预测编码蛋白质的分子式为C1 212H1 926N328O365S10,总原子数为3 841个,相对分子质量为27 253。Leu、Ala和Asp在氨基酸组成中出现频率较高,分别占总氨基酸的122%、102%和93%,而Trp、Cys和His出现的频率较低,仅占 08%、12%和24%;理论pI为508,其中带正电荷(Arg+Lys)和负电荷(Asp+Glu)残基总数分别为27个和37个;不稳定系数为2605,是不稳定蛋白;脂肪族系数为9793;亲水性平均系数为-0100,属亲水性蛋白;半衰期为30 h。
TMHMM软件(http://wwwcbsdtudk/services/TMHMM/)预测CpCCoAOMT2蛋白不存在跨膜结构域。采用分析软件SignalIP(http://wwwcbsdtudk/services/SignalP/)对获得的CpCCoAOMT2氨基酸序列的信号肽序列进行预测,预测其没有信号肽序列。通过NPS(https://npsa-prabiibcpfr/cgi-bin/secpred_hnnpl)预测CpCCoAOMT2氨基酸序列的二级结构,发现其多肽链中主要结构元件是α螺旋和无规则卷曲,分别占4797%和3862%,延伸链占1341%;运用SWISSMODEL工具(https://swissmodelexpasyorg)对CpCCoAOMT2的氨基酸序列进行同源三维建模(图2),模型以Automated mode方式构建,建模范围是从第19个到第245个氨基酸,其建模参考模板为高粱(Sorghum bicolor)咖啡酰辅酶A甲基转移酶(登陆号:5kva1A),目标蛋白与参考蛋白的序列比对一致性为6344%。
23象草[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因推导酶蛋白氨基酸序列比较与分子进化分析
在Pfam(http://pfamxfamorg)和NCBI上对获得的[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]编码的氨基酸序列进行保守结构域分析,发现CpCCoAOMT2具有Methyltransf_3(O-methyltransferase,PfamPF01596)和AdoMet_MTases(S-adenosylmethionine-dependent methyltransferases,cd02440)保守区,是一种甲基转移酶(图3)。将[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]编码的氨基酸序列与柳枝稷(Panicum virgatum,BAO208821)、玉米(Zea mays,NP_0011525111)和水稻(Oryza sativa,BAG925181)编码的CCoAOMT氨基酸序列进行多重比对分析,其与柳枝稷和玉米的CCoAOMT酶蛋白序列同源性高达93%、90%,具有高度的保守性,且具有植物甲基酶所共有的A、B、C保守序列元件和CCoAOMT基因家族中所特有的标签序列D、E、F、G、H等5个保守序列元件[15](图4)。
象草[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因推导的酶蛋白氨基酸序列与其他植物已报道的相应序列进行多序列比较,并构建系统树(图5)。结果显示,象草CpCCoAOMT2酶蛋白氨基酸序列与禾本科的柳枝稷(Panicum virgatum:BAO208821)、玉米(Zea mays:NP_0011525111)的同源性相对较高;与禾本科的水稻(Oryza sativa:BAG925181、BAG984141)、小麦(Triticum aestivum:BAD063211)的同源性次之;象草CpCCoAOMT2酶蛋白氨基酸序列与NCBI中已有记录的象草CpCCoAOMT(Cenchrus purpureus:AIN395351)的同源性相对较低;而CpCCoAOMT与禾本科中的美洲狼尾草(Cenchrus americanus:ALL436671)、 柳枝稷(Panicum virgatum:BAO208811) 和芒草(Miscanthus lutarioriparius:AEJ830601)的同源性相对较高。
在系统进化树分析中可见,本研究所克隆的象草[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因与禾本科植物的同类基因亲缘关系最近,与裸子植物(台湾杉,Taiwania cryptomerioides;台湾扁柏,Chamaecyparis formosensis)和蕨类植物(江南卷柏,Selaginella moellendorffii)关系较近,而与双子叶植物关系的较远(拟南芥,Arabidopsis thaliana;油菜Brassica napus;亚麻Camelina sativa)(图5、表1),本结论与李雪平等在禾本科植物绿竹和南荻中的研究结果[16-17]类似,也符合象草分类学中的地位。endprint
3讨论
本研究从象草品系eg7中克隆了1个[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]全长基因,[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]编码的氨基酸序列保守结构域分析发现,其具有Methyltransf_3和AdoMet_MTases保守结构域,是一种甲基转移酶。对[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]编码的氨基酸序列进行多重比对发现,CpCCoAOMT2不仅包含了植物甲基转移酶中普遍存在的A、B和C 3个保守序列元件,还包含CCoAOMT基因家族中所特有的标签序列,即D、E、F、G和H 5个保守序列元件[CM(25],說明该基因具有CCoAOMT类基因所有的标志性特征元
件[15],因此推测CpCCoAOMT2是CCoAOMT基因家族的一个成员,但其具体功能还有待于进一步研究。在系统发育树分析中,CpCCoAOMT2基因与禾本科植物的同类基因亲缘关系最近,与裸子植物和蕨类植物关系较近,而与双子叶植物较远,本结论与李雪平等在禾本科植物绿竹和南荻的研究结果[16-17]类似,也与象草分类学中的位置一致。
参考文献:
[ZK(#]del Río J C,Prinsen P,Rencoret J,et al Structural characterization of the lignin in the cortex and pith of elephant grass (Pennisetum purpureum) stems[J] Journal of Agricultural and Food Chemistry,2012,60(14):3619-3634
Bhandari A P,Sukanya D H,Ramesh C R Application of isozyme data in fingerprinting Napier grass (Pennisetum purpureum Schum) for germplasm management[J] Genetic Resources & Crop Evolution,2006,53(2):253-264
[3]Strezov V,Evans T J,Hayman C Thermal conversion of elephant grass (Pennisetum purpureum Schum) to bio-gas,bio-oil and charcoal[J] Bioresource Technology,2008,99(17):8394-8399
[4]Harris K Genetic relationships among napiergrass (Pennisetum purpureum Schum) nursery accessions using AFLP markers[J] Plant Genetic Resources,2010,8(1):63-70
[5]Jakob K,Zhou F S,Paterson A H,et al Genetic improvement of C4 grasses as cellulosic biofuel feedstocks[J] Vitro Cellular & Developmental Biology Plant,2009,45(3):291-305
[6]Liu X H,Shen Y X,Lou L Q,et al Copper tolerance of the biomass crops elephant grass (Pennisetum purpureum Schumach),vetiver grass (Vetiveria zizanioides) and the upland reed (Phragmites australis) in soil culture[J] Biotechnology Advances,2009,27(5):633-640[HT][HJ][HT][FL)][LM]
[KH4D]
[HT8]
[7][ZK(#]Somerville C,Youngs H,Taylor C,et al Feedstocks for lignocellulosic biofuels[J] Science,2010,329(5993):790-792
[8]Himmel M E,Ding S Y,Johnson D K,et al Biomass recalcitrance:engineering plants and enzymes for biofuels production[J] Science,2007,315(5813):804-807
[9]Li X,Ximenes E,Kim Y,et al Lignin monomer composition affects Arabidopsis cell-wall degradability after liquid hot water pretreatment[J] Biotechnology for Biofuels,2010(3):27
[10][ZK(#]Simmons B A,Loqué D,Ralph J Advances in modifying lignin for enhanced biofuel production[J] Current Opinion in Plant Biology,2010,13(3):313-320
[11]Bonawitz N D,Chapple C The genetics of lignin biosynthesis:connecting genotype to phenotype[J] Genetics,2010,44(44):337-363
[12]Zhong R,Iii W H,Negrel J,et al Dual methylation pathways in lignin biosynthesis[J] Plant Cell,1998,10(12):2033-2046
[13]Zhong R,Morrison W H,Himmelsbach D S,et al Essential role of caffeoyl coenzyme A O-methyltransferase in lignin biosynthesis in woody poplar plants[J] Plant Physiology,2000,124(2):563-578
[14]Wagner A,Tobimatsu Y,Phillips L,et al CCoAOMT suppression modifies lignin composition in Pinus radiata[J] The Plant Journal,2011,67(1):119-129
[15]王华美,于延冲,付春祥,等 木质素合成关键酶咖啡酰辅酶A氧甲基转移酶的研究进展[J] 基因组学与应用生物学,2014,32(2):458-466
[16][JP2]李雪平,高志民,彭镇华,等 绿竹咖啡酰辅酶A-O-甲基转移酶基因的克隆与分析[J] 分子植物育种,2008,6(3):587-592[JP]
[17]洪艳云,黄志刚,易图永 南荻CCoAOMT基因的克隆与生物信息学分析[J] 中南林业科技大学学报,2011,31(10):94-99endprint

