玉米sbe1和bt2基因SNPs位点的筛选
郭新梅,董 冰,葛兆鹏,裴玉贺,赵美爱,宋希云*
(1.青岛农业大学,山东 青岛 266019;2.青岛市主要农作物种质资源创新与应用重点实验室,山东 青岛 266019)
【研究意义】单核苷酸多态性(single nucleotide polymorphisms SNPs)指在基因组水平上由单核苷酸的变异所引起的一种DNA 序列多态性,这种变异是由单碱基的转换、颠换及单碱基的插入或缺失所引起的[1]。【前人研究进展】1994年,SNPs这个术语首次在人类分子遗传杂志上出现,Lander正式提出SNPs为新一代分子标记[2]。1997年,作为第三代分子标记的SNPs的提出与应用,在生物界引起了DNA多态性研究的新高潮。在1999年的第二次“SNPs与复杂基因组”国际会议中,新增加了群体遗传学与信息学的领域[3]。随着SNPs的飞速发展,其在动植物中的研究开发也越来越受到关注,各种研究迅速开展,尤其是在重要农作物和模式动物中已经取得了可喜的成绩[4-6]。SNPs分子标记得到了较好的开发[7],由于SNPs密度高,片段短,遗传稳定,易于高通量、自动化分析[8],广泛用于绘制EST图谱、群体遗传学和连锁不平衡、遗传标记。而SNPs的检验方法有很多,测序和单链构象多态性(SSCP)检测技术已经非常的成熟,除此之外,有很多新的检测技术频频出现,并逐渐成为主要的检测手段。SNPs为基于测序的高密度遗传图谱的构建提供了有力工具[9]。直接测序法是目前最简单的SNPs检测方法,检出率基本可达到100 %。除此之外,采用直接测序的方法还能得到SNPs的类型以及准确位置等SNPs分型所需的重要参数。由于DNA测序趋于自动化以及测序成本的降低,使得直接测序法越来越多的应用于SNPs的检测和分型,出现了很多生物类型的全基因组分型SNPs芯片,然而这种芯片对大群体的试验来说,费用高,大部分实验室无法完成这类全基因组SNPs分型扫描。【本研究切入点】本试验利用候选基因直接测序比对后获得SNPs位点,并通过HRM筛选和验证SNPs,分析、总结直接测序结合HRM法进行SNPs位点的筛选和验证的优缺点,【拟解决的关键问题】以期为直接测序法进行大规模SNPs筛选提供参考。
1 材料与方法
1.1 实验材料
玉米自交系H21,糯玉米紫糯96-619及171个以H21为轮回亲本、紫糯96-619为非轮回亲本回交5代自交2代的H21BC5F2∶3。材料由青岛农业大学分子育种实验室提供。
1.2 DNA的提取
取适量玉米种子播种于培养盆中,培养盆中沙土湿度适中,将培养盆放置28 ℃培养箱中进行暗培养,待玉米苗长至2~3片叶片时剪取叶片, CTAB法提取DNA,将其稀释到100 ng/μl,-80 ℃冰箱中冻存备用。
1.3 候选基因的测序
候选基因sbe1(AC211441)全长序列由上海生工生物工程技术服务有限公司完成。根据候选基因AGPS1a-1(bt2)(AF334959)全长序列,利用Primer 5进行引物设计,共设计了8对分段引物,引物由上海生工生物工程技术服务有限公司合成,具体的引物设计情况如表1所示。
引物合成后进行PCR扩增,PCR反应体系(50 μl)为:dNTP 5 μl、5×buffer 10 μl、DNA模板 4 μl、上游引物 2 μl、下游引物 2 μl、高保真酶 1 μl、ddH2O 26 μl。PCR反应程序为:①95 ℃预变性1 min;②95 ℃变性20 s;③根据不同引物退火温度(50到55 ℃不等),退火20 s;④72 ℃延伸30 s;⑤2~4重复35个循环;⑥72 ℃后延伸5 min。每一个PCR反应重复3次,产物送上海生工生物工程技术服务有限公司测序部进行测序。测序结果使用DNAMAN软件进行比对分析。再利用ContigExpress软件将核实后的测序结果进行基因片段序列的拼接,确定AGPS1a-1(bt2)的全长序列。
1.4 SNPs位点的查找
将获得的sbe1和bt2候选基因全长核酸序列用SoftBerry软件翻译成氨基酸序列,比对氨基酸序列的差异性。用Antheprot 5.0软件进行蛋白质二级结构预测;用SWISS MODEL网站进行蛋白质三级结构预测和功能域分析。根据亲本的基因序列及氨基酸序列比对结果,在SNPs位点设计引物,引物设计情况如表2所示。
1.5 SNPs位点在群体中的检测
利用表2中的引物对亲本进行普通PCR扩增之后,确定引物的最适退火温度。按照确定好的每对引物的最佳退火温度用于Rotor Gene Q(QIAGEN)的HRM(高分辨率熔解曲线) PCR扩增,通过与亲本间曲线的比对情况检测后代基因型的分型情况。
2 结果与分析
2.1 基因组DNA的提取
亲本及部分子代DNA提取结果如图1所示,提取的DNA的纯度和质量较好,没有RNA和其他杂质污染,条带较为清晰,可以进行后续的试验研究。
2.2 bt2目的片段的扩增
引物合成后进行目的片段PCR扩增,经0.8 %的琼脂糖凝胶电泳检测,部分结果如图2所示。

表1 bt2引物序列Table 1 Information of primers for bt2
表2 SNPs引物设计情况统计Table 2 Information of SNPs primers for bt2 and sbe1
2.3 bt2基因氨基酸序列比对与蛋白结构预测
从图3(封三)可以看出,bt2基因的氨基酸序列在第322处不同,H21在此处的氨基酸为甲硫氨酸,而紫糯96-619的为异亮氨酸。根据以上的氨基酸序列进行的蛋白质二级结构预测结果如图4所示。
从图4(封三)可以看出,bt2基因编码的蛋白质二级结构绝大多数都是α-螺旋和β-折叠,在亲本中的表现很相似,但是又存在不同(图4中箭头所示),主要是折叠的位置不同或者区域大小有差别,如左侧箭头所示位置的β-折叠,H21比紫糯96-619明显小;如右侧箭头所示的位置H21比紫糯96-619明显少了一段β-折叠,说明bt2基因在亲本间的差异引起了蛋白二级结构的变化。

A:M.15000 DNA marker;1.紫糯96-619;2.H21;B:M.15 000 DNA marker;1~12:部分子代图1 亲本(A)及部分BC5 F2∶3(B)的DNA提取Fig.1 DNA extraction of parents(A)and some BC5F2∶3(B) individuals
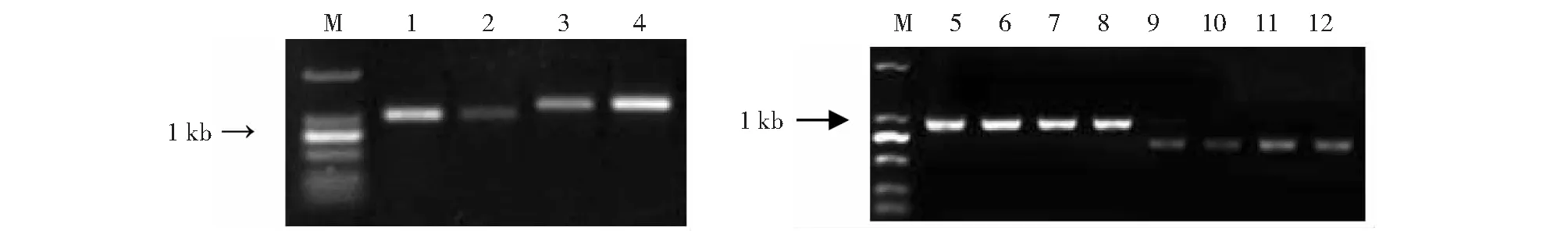
M. 2000 bp DNA marker; 1. bt2-5 H21; 2. bt2-5 紫糯; 3. bt2-6 H21; 4. bt2-6 紫糯;5. bt2-2 H21; 6. bt2-2 H21; 7. bt2-2 紫糯; 8. bt2-2 紫糯; 9. bt2-7 H21; 10. bt2-7 H21; 11. bt2-7 紫糯; 12. bt2-7 紫糯图2 bt2部分引物对H21和紫糯96-619的扩增Fig.2 Partial results of H21 and ZN96-619 PCR amplification using primers of bt2
根据氨基酸序列的变化,在SWISS MODEL网站上应用同源建模法对蛋白质三级结构预测的结果如图5所示,bt2基因编码的两亲本之间的蛋白质空间结构的预测模型几乎是一样的,只在箭头所示的位置有所不同。说明bt2基因在亲本间的差异影响了蛋白三级结构。
本文利用AIS数据,构建船舶领域统计方法模型,并根据目标船周围最近船舶的相对位置分布情况,采用最小二乘法确定领域边界;利用荆州AIS数据对模型进行验证,并对比分析横驶船舶与直航船舶的船舶领域,得出横驶船舶领域与直航船舶领域形状特征的差异;对比不同尺度的上行和下行船舶的船舶领域,得到船舶尺度、航速对船舶领域大小的影响,为船舶在水上航行时的避碰和风险研究提供一定的理论依据。
2.4 sbe1基因氨基酸序列比对与蛋白结构预测
sbe1基因的氨基酸序列比对结果如图6所示,H21在第448个氨基酸为丙氨酸,紫糯96-619为天冬氨酸。根据以上的氨基酸序列进行的蛋白质二级结构预测结果如图7所示。
图7(封三)显示,sbe1基因编码的蛋白质二级结构绝大多数都是α-螺旋和β-折叠,折叠和无规则卷曲的位置及区域大小有差别,如箭头所示的位置H21与紫糯96-619一段β-折叠大小及位置明显不同,说明sbe1基因在亲本间的差异引起了蛋白二级结构的变化。
根据氨基酸序列的变化,在SWISS MODEL网站上应用同源建模法对蛋白质三级结构预测的结果如图8所示,sbe1基因编码的两亲本之间的蛋白质空间结构的预测模型几乎是一样的。
上述结果显示bt2和sbe1基因编码的各自的亲本蛋白质在空间结构上一样的,只是在螺旋的长度和折叠的数量上有细微的差别,其编码的蛋白质的具体功能有待进一步研究。

图8 sbe1基因编码的紫糯96-619和H21的蛋白质空间结构预测Fig.8 Spatial structure prediction of proteins encoded by gene sbe1 in ZN 96-619 and H21
2.5 bt2和sbe1基因中SNPs位点的筛选
根据sbe1和bt2候选基因全长核酸序列比对结果,在bt2基因找到17个SNPs位点,在sbe1基因找到3个SNPs位点(图略),结合亲本氨基酸序列比对结果,共设计20对 SNPs引物,引物情况如表2所示。
从图9可以看出,纵轴显示的是荧光值,横轴表示的是温度,由于DNA双链中有SNPs存在,因此亲本之间在双链解开时候的温度有细微差异,以此将亲本间的SNPs检测出来。如图所示,引物bt2-5129对亲本紫糯96-619扩增出的DNA熔解峰值在76.7 ℃,对H21扩增出的DNA熔解峰值在77.2 ℃,有较为明显的峰值差别,在对171个子代进行分析的过程中,绝大多数后代的基因型与亲本紫糯96-619相同,只有5个与亲本H21相同。而部分引物如bt2-1918对亲本扩增的基因片段熔解最适温度差异过小(0.08 ℃),无法进行基因分型。引物bt26271、引物bt26408扩增出的亲本基因片段熔解的最适温度差值分别为0.18、0.16 ℃,差异较小,也不宜用于后代基因分型。
如图10所示,引物sbe1-2240对亲本紫糯96-619扩增出的DNA熔解峰值在77.42 ℃,对H21扩增出的DNA熔解峰值在77.02 ℃,有较为明显的峰值差别,在对171个子代进行分析的过程中,绝大多数后代的基因型与亲本紫糯96-619相同,只有16个与亲本H21相同。
如图11所示,引物sbe1-2361对亲本紫糯96-619扩增出的DNA熔解峰值在78.33 ℃,对H21扩增出的DNA熔解峰值在78.85 ℃,有较为明显的峰值差别,在对171个子代进行分析的过程中,123个后代的基因型与亲本H21相同,只有48个与亲本紫糯96-619相同。

图9 引物bt2-5129对后代扩增的高分辨率熔解曲线和峰值Fig.9 HRM curves and peaks of some amplication results by primers bt2-5129
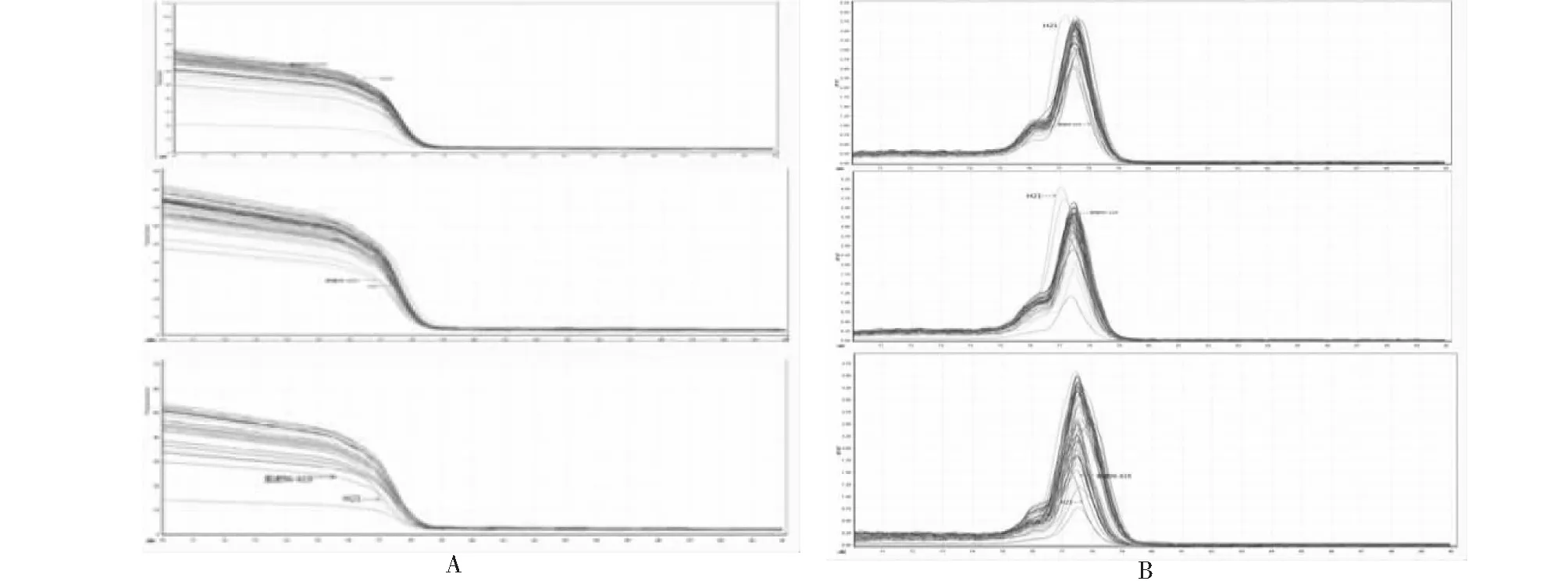
图10 引物sbe1-2240对后代扩增的高分辨率熔解曲线和峰值Fig.10 HRM curves and peaks of some amplication results by primers sbe1-2240
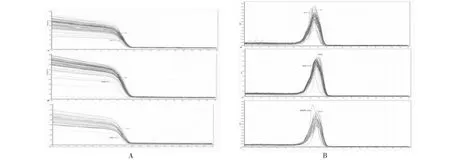
图11 引物sbe1-2361对后代扩增的高分辨率熔解曲线和峰值Fig.11 HRM curves and peaks of some amplication results by primers sbe1-2361
3 结 论
本文首先利用直接测序法获得bt2和sbe1基因的全长序列(基因登录号分别为KR337996、KR706462、KR706463和KR337995),通过亲本间序列比对,找到了20个SNPs位点,通过高分辨率熔解曲线(HRM),成功对171个高世代回交群体的SNPs位点分型。
4 讨 论
直接测序法是目前最简单的SNPs检测方法。其原理是:通过对不同个体的同一基因或者基因片段进行测序以及序列的比较,确定我们所研究的碱基是否变异,检出率可达到100 %。除此之外,采用直接测序的方法还能得到SNPs的类型以及准确位置等SNPs分型所需的重要参数。变性高效液相色谱(DHPLC)技术是在SSCP基础上发展起来的新技术[10],这种方法的灵敏度和自动化程度很高,在未知SNPs位点的检测方面具有明显的优势,并且适合较大片段DNA的分析。其缺点是只能检测样本有无SNPs,不能确定SNPs的类型和位置,若想获得准确的SNPs类型与位置还是要通过测序的方法才能得到[11-12]。
目前检测SNPs的方法还有很多,如焦磷酸测序(pyrosequencing),微测序(SNaPshot)、基于杂交原理的Taqman等。这些方法各有优势,但也存在如适宜位点数量少的样本,或者准确率低、费用较高等的缺点[13-14]。高分辨熔解曲线(High Resolution Melting, HRM)于2003 年由美国Wittwer 实验室第一次提出[15]。这种检测方法操作简便、快速,使用成本低,结果准确,并且实现了真正的闭管操作而受到普遍的关注,被 广泛用于特定突变位点SNPs(包括缺失、重复)的筛查、基因突变扫描、等位基因频率分析 、物种鉴定、品种鉴定 、甲基化研究 、动植物品质相关多态性位点的研究等[16-20]。
本文首先利用直接测序法获得bt2和sbe1基因的全长序列(基因登录号分别为KR337996、KR706462、KR706463和KR337995),通过序列比对获得详细地SNPs位点信息,同时通过HRM技术快速、准确地对群体后代的基因分型。通过这2种方法的结合能高效完成大群体SNPs位点的筛选和分型。但在自主设计引物进行全基因扩增获得全序列的过程中,遇到了以下几个问题:一是参考基因序列显示某些区段GC含量较高,这会导致PCR扩增不出目的片段或所得的目的片段,目的条带不够亮,从而影响测序的结果;二是引物除扩增出特异性片段之外还有非特异性的片段,这对测序结果的准确性有一定的影响。因此,为了获得准确的测序结果,本实验中每个样品测序均进行了3次重复,以上所得结果是3次重复比对之后的结果,准确性较高。同时利用在通过HRM进行后代群体分型的过程中,部分引物如bt2-1918、bt2-6271、bt2-6408由于亲本基因片段熔解的最适温度差值较小,无法进行基因分型,需要通过重新测序或重新设计引物或可以解决,而有些位点受基因序列本身的影响很难设计出符合HRM分析的引物,因此,HRM技术还需要进一步完善与发展,使其更加有效地分析突变位点。
[1]王 会,刘 佳,付 丽,等. 单核苷酸多态性在多倍体作物油菜中的研究进展[J]. 中国油料作物学报,2014,36(3):422-429.
[2]Lander E S. The new genomics:global views of biology[J]. Science, 1996, 274:536-539.
[3]Nebert D W. Pharmacogenetics:Why is this relevant to the clinical geneticist[J]. Clin Genet, 1999, 56(4):247-258.
[4]杜春芳, 刘惠民, 李润植,等. 单核苷酸多态性在作物遗传及改良中的应用[J]. 遗传, 2003, 25(6):735-739.
[5]郝岗平, 杨 清, 吴忠义,等. 植物的单核苷酸多态性及其在作物遗传育种中的应用[J]. 植物学通报, 2004,21(5):618-624.
[6]Huang J M, Zhang Y, Li J, et al. Development of SNPs markers in Crassostrea hongkongensis based on the next-generation sequencing and high resolution melting analysis[J]. Conservation Genetics Resources,2014(6):559-562.
[7]Qi J, Liu X, Shen D, et al. A genomic variation map provides insights into the genetic basis of cucumber domestication and diversity[J]. Nature Genetics, 2013, 45 (12):1510-1515.
[8]Landegren U, Nilsson M, Kwork P Y. Reading bits of genetic information:methods for single nucleotide poly-morphism analysis[J]. Genome Res, 1998, 8(8):769-776.
[9]袁金红,李俊华,黄小城,等. 基于全基因组重测序的SNPs分析在作物基因定位中的研究进展[J]. 植物生理学报,2015,51(9):1400-1404.
[10]仪军玲,李彩霞,胡 兰. 单核苷酸多态性及其检测方法[J]. 证据科学,2008,16(6):757-762.
[11]刘雅诚,郝金萍,严江伟,等. 用dHPLC技术检测线粒体DNA编码区单核苷酸多态性[J]. 中国法医学杂志,2006,21(3):142-146.
[12]秦效英,李国选,江 滨,等. 应用变性高效液相色谱检测CD31563位点单核普酸多态性[J]. 中华检验医学杂志,2006,29(7):627-630.
[13]张素华,李 莉,李成涛,等. TaqMan 探针技术用于X-SNPs 位点的分型[J]. 法医学杂志,2010(1):22-25.
[14]王冬梅,李 俊,杨红敏,等. 高分辨率熔解曲线HRM 在SNPs 检测中的应用[J]. 基因组学与应用生物学,2015,34(4):892-895.
[15]Vossen R H, Van D M, Daha M R, et al. High-throughput genotyping of mannose-binding lectin variants using high-resolution DNA-melting analysis[J]. Human Mutation, 2010, 31(4):1286-1293.
[16]Chen X, Kong F, Wang Q, et al.Rapid detection of isoniazid, rifampin, and ofloxacin resistance in mycobacterium tuberculosis clinical isolates using high-resolution melting analysis[J]. Journal of Clinical Microbiology, 2011,49(10):3450-3457.
[17]李 琛,许岸高,甘爱华,等. 高分辨率溶解曲线分析与基因测序检测粪便DNA 对大肠癌筛查作用的比较[J].临床消化病杂志,2011(6):356-358.
[18]吴 波,杨润婷,朱世平,等.宽皮柑橘单核苷酸多态性的高分辨率熔解曲线分型[J]. 园艺学报, 2012(4):777-782.
[19]武 林,王 艳,孙梦阳,等. 大豆脂肪酸Fad3a 基因HRM 检测体系优化研究[J]. 大豆科学,2012(6):882-886.
[20]肖正龙,张龚炜,杨 宇,等. 家兔ATG16L1基因多态性与非特异性消化道紊乱易感性的研究[J]. 华北农学报,2013(4):234-238.

