云南主要贸易类干巴菌遗传多样性分析*
周 汐,冯云利,陈正启,余金凤,吴素蕊,罗 瑞,桂明英
(中华全国供销合作总社昆明食用菌研究所,云南 昆明 650223)
干巴菌是属于担子菌门(Basidiomycota) 蘑菇纲(Agaricomycetes) 革菌目 (Thelephorales) 革菌科(Thelephoraceae) 革菌属(Thelephora) 的一类可食用真菌[1]。作为云南珍贵的野生食用菌,干巴菌凭借其独特的风味和丰富的营养成为云南贸易类野生菌市场中不可或缺的一员[2]。
在云南地区,传统广义上的干巴菌包括革菌属的6个种,莲座革菌(Thelephora vialisSchwein)、干巴菌(Thelephora ganbajunM.Zang)、橙黄革菌(Thelephora aurantiotinctaCorner)、淡褐革菌 [Thelephora fuscella(Ces.)Lloyd]、日本革菌(Thelephora japonicaYasuda) 和掌状革菌 [Thelephora palmate(Scop.)Fr.]。常见的主要有4种,莲座革菌(T.vialis)、干巴菌 (T.ganbajun)、橙黄革菌 (T.aurantiotincta) 和掌状革菌(T.palmate),主要产地为昆明、玉溪、曲靖、楚雄、普洱、丽江、保山和大理,分布区域大约位于北纬 22°~27°,东经 99°~106°,海拔800m~2200m的范围内[3-4]。干巴菌仅在滇中及邻近地区有食用习惯,且作为一种共生真菌,其生长条件复杂,导致干巴菌的人工栽培难以实现,因此,国内外对此类真菌的研究成果不如其他菌类丰富,主要涉及分类鉴定、分离纯化、营养成分测定、生态学研究几个方面,在分子生物学方面有部分研究但相对较少[5]。
本文通过对2015年至2016年,采集于野生环境中的34份贸易类干巴菌样品进行遗传多样性分析,以期了解贸易类干巴菌的种类、分布以及数量,从分子水平为云南贸易类干巴菌资源的开发与保护提供一定的理论依据。
1 材料和方法
1.1 试验材料
自2015年至2016年,从云南贸易类干巴菌8个主产地中的普洱市、曲靖市马龙县、保山市、玉溪市元江县、玉溪市峨山县、玉溪市易门县、玉溪市新平县、大理州洱源县、大理州祥云县、楚雄市、楚雄州武定县、昆明市富民县、昆明市嵩明县、昆明市宜良县和昆明市寻甸县采集贸易类干巴菌样品40份。采集的样本用硅胶干燥后备用。
1.2 试剂与仪器
1.2.1主要试剂
CTAB,TaKaRa Taq,ITS1/ITS4 引物,RV-M/M13-47引物,TaKaRa胶回收试剂盒,pMD18-T载体,大肠杆菌DH5α感受态细胞,Amp,IPTG,X-gal。
1.2.2主要仪器
凝胶成像仪,Gensnap公司;常温高速离心机,SCILOGEX公司;PCR仪,ABI公司;电泳仪,六一生物科技有限公司;紫外透射仪,Benchtop公司;微型分光光度计,Nano Drop公司。测序服务由北京擎科新业生物技术有限公司提供。
1.3 试验方法
1.3.1形态学鉴定
通过观察形态特征和显微结构,按照传统分类方法,参考黄年来、卯晓岗[6-7]对于革菌属各个种的形态描述,初步确定每份样品的属种名。
1.3.2DNA提取
去除样品表面的泥土等杂质后用无菌去离子水和75%的酒精对样品进行清洗并粗略切割,吸干样品表面液体后将样品置于经过高温消毒的研钵中,加入液氮将样品研磨至粉末,采用CTAB法提取样品的DNA保存备用。
1.3.3PCR
利用真菌ITS通用引物对ITS1/ITS4对样品进行PCR扩增,设置反应体系为Buffer 5μL,dNTP 4 μL,引物各1μL,Taq酶0.5μL,DNA模板1μL,无菌去离子水补足至50μL。设置PCR仪扩增程序为95℃预变性5 min;94℃变性30 s,55℃退火40 s,72℃延伸1min,35个循环;72℃延伸7min。
1.3.4克隆
使用1%的琼脂糖凝胶电泳对样品的PCR扩增产物进行检测,割胶后使用胶回收试剂盒对含有目的DNA片段的回收胶条进行纯化。将纯化产物连接到pMD18-T载体上,转化入大肠杆菌DH5α感受态细胞,使用含有Amp/IPTG/X-gal的LB平板筛选阳性单克隆,挑取单克隆于0.8mL含Amp的LB液体培养基中,37℃、200 r·min-1振荡培养 14 h~16 h。
1.3.5测序
使用pMD18-T载体上的引物对RV-M/M13-47对菌液进行菌落PCR验证,使用1%的琼脂糖凝胶电泳对PCR产物进行检测,选择含有目的条带的菌液保存。将PCR验证有目的条带的阳性克隆送公司进行测序。
1.3.6序列分析
使用DNASTAR软件包中的SeqMan程序,对照测序峰图对序列进行手动校对,去除序列两端多余的碱基,以确保序列的准确性。通过Blast在线工具(http://www.ncbi.nlm.nih.gov/Blast.cgi)进行比对,将测序获得的序列同GenBank中已录入的干巴菌(T.ganbajun) 以及革菌科(Thelephoraceae) 其他种序列进行比对。
1.3.7系统进化树的构建
以银耳(Tremella fucifomis) 作为外群,使用MAGA5.05软件中的系统分析法(phylogeny)对序列进行系统关系分析,采用邻接法(neighbor-joining method,NJ) 构建系统发育树。系统树的每个分枝的统计学显著性分析以Boot strap方法进行检验,重复次数为1000次,显著性低于50%时,数值不在进化树中显示。
1.3.8遗传距离的计算
将测得的序列和从GenBank中通过BLAST检索获得的参考序列进行多重对位排列,用MAGA5.05软件中的Kimura-2-parameter(K2P) 模型计算遗传距离,构建距离矩阵。
1.3.9遗传多样性分析
使用软件DnaSP 5.10对来自5个地理群的25个同种样品进行变异位点、单倍型数量、单倍型多样性指数和核苷酸多样性指数的统计。
2 结果和分析
2.1 ITS序列鉴定结果
对经形态学鉴定为贸易类干巴菌的34个样品的ITS序列进行BLAST比对,结果列于表1。
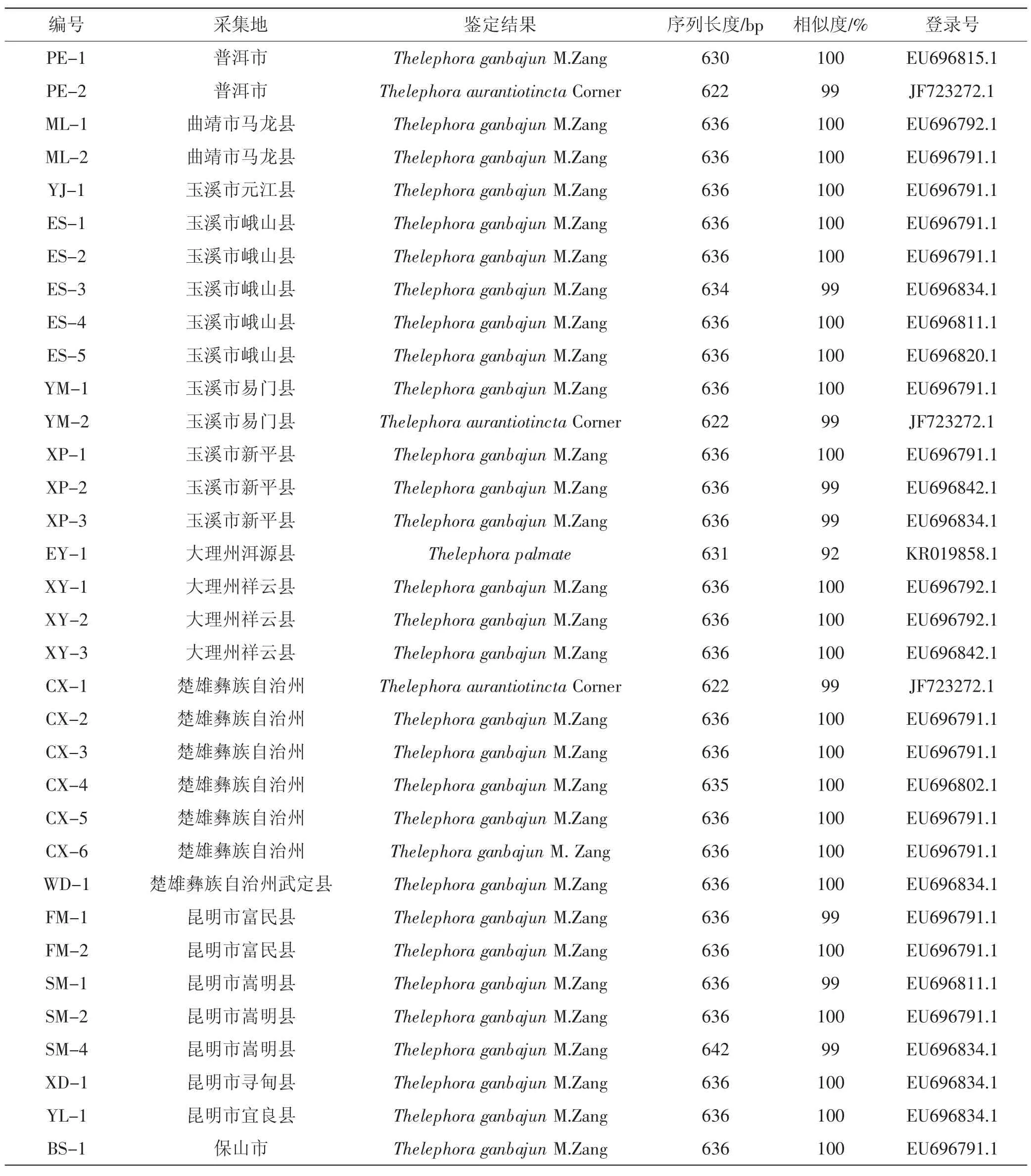
表1 ITS鉴定结果Tab.1 ITS identification result
在对测定序列进行BLAST比对的过程中,发现3个样品在GenBank数据库中只能比对到属名(Thelephorasp.),为了确定其种名,根据文献溯源,发现该种最初由Yagame等[8]在对小舌唇兰与其伴生真菌的混合营养模式的研究中被测序并登录,登录号为JF723272.1。同时,由于食用菌数量广且形态多样,利用单一的手段对某个菌种进行鉴定会造成假阳性[9-11],影响后续研究,因此在本文的研究中,通过对采集的样品进行宏观形态鉴定和显微结构观察(图1),将形态鉴定与ITS序列鉴定相结合,确定PE-2为橙黄革菌(T.aurantiotincta)。
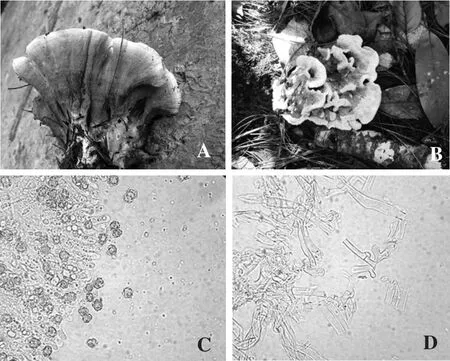
图1 PE-2形态鉴定Fig.1 Morphological identification of PE-2
在上述结论的基础上,由表1可知,34份样品的ITS序列长度在640 bp左右,其中PE-2、YM-2和CX-1为橙黄革菌(T.aurantiotincta),EY-1以92%的相似率比对到掌状革菌(T.palmate),其余30份样品均为干巴菌(T.ganbajun)。
从地域分布看,昆明市周围县级地区8份样品均为干巴菌(T.ganbajun);玉溪市11份样品以干巴菌(T.ganbajun) 为主,且于易门县采集到1份橙黄革菌(T.aurantiotincta);大理市的4份样品除采集于洱源县的1份掌状革菌(T.palmate) 外,其他均为干巴菌(T.ganbajun);楚雄州7份样品中发现一份橙黄革菌(T.aurantiotincta) 其余6份为干巴菌(T.ganbajun)。而普洱市的两份样品中1份为干巴菌(T.ganbajun),另一份为橙黄革菌(T.aurantiotincta)。曲靖市的2份样品均为干巴菌(T.ganbajun)。
2.2 系统进化树分析
样品的系统进化树分析见图2。
由图2可知,EY-1与登录号为KR019858.1的掌状革菌聚为一支且与其他样品亲缘关系较远;其次CX-1和YM-2与登录号为JF723272.1的1个革菌属序列亲缘关系较近且与其他样品亲缘关系相对较远;其余样品基本聚为一支,但仍呈现出一定的个体差异。
2.3 干巴菌遗传多样性分析
干巴菌(T.ganbajun)样品的遗传多样性分析如表2所示。

图2 基于ITS的云南主要贸易类干巴菌样品间的NJ系统树Fig.2 NJphylogenetic tree ofmain trading Thelephora samples in Yunnan based on ITS

表2 干巴菌遗传多样性分析Tab.2 Genetic diversity analysis of Thelephora ganbajun M.Zang
表2数据显示,5个地区的干巴菌均显示出一定的遗传多样性,尤其玉溪的样品最为明显,单倍型数、多态位点数最多,以9个样品达到了0.722的单倍型多样性。
2.4 贸易类干巴菌遗传距离分析
主要贸易类干巴菌样品间的遗传距离见表3。

从表3可以看出,通过遗传距离的计算得知,云南地区贸易类干巴菌的总体遗传距离范围为0.000~0.165;样品EY-1和YM-2之间的遗传距离与EY-1和CX-1之间的遗传距离均达到了0.165,表明橙黄革菌(T.aurantiotincta) 与掌状革菌(T.palmate)之间存在明显的种间差异;EY-1与其他干巴菌(T.ganbajun) 样品的遗传距离分别能达到0.126、0.114、0.116、0.110、0.136 和 0.130,说明掌状革菌(T.palmate) 与干巴菌(T.ganbajun) 之间存在较大的种间差异;YM-2/CX-1和其他干巴菌(Thelephora ganbajun) 样品之间的遗传距离范围在0.091~0.111,说明掌状革菌(T.palmate) 与干巴菌(T.ganbajun) 之间存在种间差异;YM-2与CX-2的遗传距离有0.005,说明橙黄革菌(T.aurantiotincta) 种内存在一定差异;其他干巴菌(T.ganbajun) 样品相互之间的遗传距离范围在 0.000~0.047,0.047的遗传距离存在于SM-1和CX-4之间,说明云南各地干巴菌(T.ganbajun)种内也有显著差异。
3 结论与讨论
本文从云南8个贸易类干巴菌主产地采集了34份样品,覆盖了15个区县。通过ITS序列分析,确认34份样品中有30份样品为干巴菌(T.ganbajun),说明云南地区贸易类干巴菌以干巴菌(T.ganbajung)为主要种。除干巴菌外,掌状革菌(T.palmate) 和橙黄革菌(T.aurantiotincta)亦有分布但资源较为稀缺。说明云南地区贸易类干巴菌具有一定的多样性,但本次研究中体现出多样性不丰富。
系统发育树及遗传距离分析显示,干巴菌(T.ganbajun)、掌状革菌(T.palmate) 和橙黄革菌(T.aurantiotincta)分散为3支,且干巴菌的一个大支分为了3个小支,但与地域差异关联不明显,种间有明显差距,种内有一定差距。
在本次研究中,掌状革菌(T.palmate) 样品EY-1采集于云南省大理州洱源县的针阔混交林,片段长度为631 bp,以92%的相似性比对到NCBI数据库中登录号为KR019858.1的序列。另外3份橙黄革菌(T.aurantiotincta) 样品中,PE-2采集自云南省普洱市倚象镇石膏箐的混交林,片段长度为622,以99%的相似率比对到JF723272.1;YM-2采集自云南省玉溪市易门县的松纯林,片段长度为622,以99%的相似率比对到JF723272.1;CX-1采集自楚雄彝族自治州的针阔混交林,片段长度为622,同样以99%的相似率比对到JF723272.1。参考前人的研究[12-14],本文认为从生物多样性角度出发,这两种菌种产量较小且分布零星,应加大保护力度并尝试进行原生境菌种栽培方面的探索。莲座革菌(T.vialis)、淡褐革菌(T.fuscella) 和日本革菌(T.japonica) 3个种在本次研究中并未发现,期望在今后的试验中通过扩大采样区域、采样密度,增加样品数量得以发现。
参考文献:
[1]冯云利,郭相,杨珍福,等.革菌属干巴菌研究进展[J].食用菌,2016(4):1-3.
[2]周彤燊,任玮.云南可食的革菌属真菌的分类研究[J].云南植物研究,1986,8(3):295-297.
[3]桂明英,刘蓓,朱萍,等.干巴菌生态学初步研究[J].西南农业学报,2005,8(3):325-327.
[4]郭安,胡慧蓉,赵凤喜,等.云南优特野生食用菌-干巴菌的开发利用[J].西南林学院学报,1998,18(2):106-108.
[5]Corner EJH.A monograph ofThelephora(Basidiomycetes)[M].Cambridge:Verlag Von J.Cramer,1968.
[6]黄年来.中国大型真菌原色图鉴[M].北京:中国农业出版社,1998.
[7]卯晓岚.中国大型真菌[M].郑州:河南科学技术出版社,2000.
[8]Takahiro Y,TakamichiO,Marc-Andre S,et al.Mixotrophy ofPlatantheraminor,an orchid associated with ectomycorrhizalforming Ceratobasidiaceae fungi[J].New Phytologist,2012,193(1):178-187.
[9]朱洪庆,王盼盼,张萌,等.rDNA-ITS序列分析法与传统分类法相结合在真菌鉴定中的应用[J].2016,西华师范大学学报:自然科学版,37(3):264-268.
[10]燕勇,李卫平,高雯洁,等.rDNA-ITS序列分析在真菌鉴定中的应用[J].中国卫生检验杂志,2008,18(10):1958-1961.
[11]陈剑山,郑服丛.ITS序列分析在真菌分类鉴定中的应用[J].安徽农业科学,2007,35(13):3785-3786,3792.
[12]Sha T,Xu JP,Malliya.Genetic diversity of the endemic gourmetmushroomThelephora ganbajunfrom south-western China[J].Microbiology,2008,154(11):3460-3468.
[13]Wang PF,Zhang Y,Sha TS,et al.The completemitochondrial genome of the edible Basidiomycete mushroomThelephora ganbajun[J].Mitochondrial DNA Part B,2017,2(1):103-105.
[14]Wang PF,Sha T,Zhang YR,et al.Frequent heteroplasmy and recombination in themitochondrial genomes of the basidiomycetemushroomThelephora ganbajun[J].Scientific R-eports,2017,7(1):1626.

