基于线粒体基因ND1、COX1和Cytb的双壳贝类分子系统进化研究
李瑶瑶, 刘云国, 刘凌霄, 马 超, 李 倩
(1.临沂大学生命科学学院,山东 临沂 276005;2.新疆大学生命科学与技术学院,新疆 乌鲁木齐 830046;3.烟台大学生命科学学院,山东 烟台 264005;4.临沂市农业科学院,山东 临沂 276012)
双壳贝类是无脊椎动物中生活领域最广的门类之一,是仅次于腹足类的第二大纲[1],约有2万种,分布广泛,大部分生活在海水里,也有少数生活在淡水里,还有极少数为寄生(内寄蛤Entovalva、恋蛤Peregrinamor等).任建峰将双壳纲分为古列齿亚纲(Palaeotaxodonta)又称原鳃亚纲(Protobranchia)、翼形亚纲(Pteriomorphia)、古异齿亚纲(Palaeoheterodonta)、异齿亚纲(Heterodonta)和异韧带亚纲(Anomalodesmata) 5亚纲10目[2],但经查阅,还有一亚纲:隐齿亚纲(Cryptodonta),只有一科一目.中国海共记录有1 104种,分属于77个科393个属[3].双壳贝类也是软体动物中最具经济价值的一个纲,本纲全部种类皆可食用,肉味鲜美,营养丰富,是名贵的海珍品.许多种可直接入药或提取各种药物;有些种能产价值昂贵的珍珠;还有些种可供玩赏或作高级装饰品.中国很早就对牡蛎、蚶、扇贝、贻贝进行人工养殖,已有约20种在水产养殖中占有重要地位.
线粒体DNA具有结构简单、在细胞中大量存在、多为母系遗传、缺少重组和内含子以及较高的进化速率等特点[4,5],因此作为一种分子标记被广泛用于隐存种确定[6]、分子进化、种群遗传、物种鉴定和不同分类水平上的系统发生关系研究[7,8]及双单亲遗传研究[9,10].线粒体基因组是一种十分有力的系统发生研究工具.双壳贝类的分类学和系统发育学一直存在着众多争议[11],形态学并不能很好的解决分类学问题,而分子生物学技术的应用为确定双壳类的系统进化与分类关系提供了新的手段,同时,它也成为重要经济种类种质培育和鉴定以及遗传资源保护的突破口,分子水平上的系统发生研究可以较准确的进行分类,因此研究双壳贝类分子系统进化关系具有重要意义.
双壳类线粒体DNA全基因组测序工作进展很快[12-15],迄今为止,在GenBank和MitoZoa可检索到的双壳类线粒体全基因组有77个,覆盖7个目[16].本研究利用生物信息学方法对66种(分属于6亚纲11目20科)双壳贝类的线粒体ND1、COX1和Cytb基因序列进行比较,探讨序列特征及变异,并在分子水平上分析双壳贝类的系统发生关系,从而为双壳类系统发生研究提供宏观资料,对中国双壳类的管理提供参考依据.
1 材料与方法
1.1 样品的采集
2016年8月份在山东威海乳山采集魁蚶样品20只,均为自然群体,样品采集后即保存在-20 ℃冰箱中备用,鉴定后取闭壳肌肌肉组织提取DNA.
在NCBI的GenBank数据库(http://www.ncbi.nlm.nih.gov/)中输关键词Bivalvia+genome进行搜索,下载得到双壳纲11个目20个科65个种类的双壳贝类线粒体ND1、COX1和Cytb全/部分基因序列.所有物种的命名和分类地位采用Nelson提出的分类标准[17].所属目、科、种名及序列GenBank登录号等相关信息见表1.
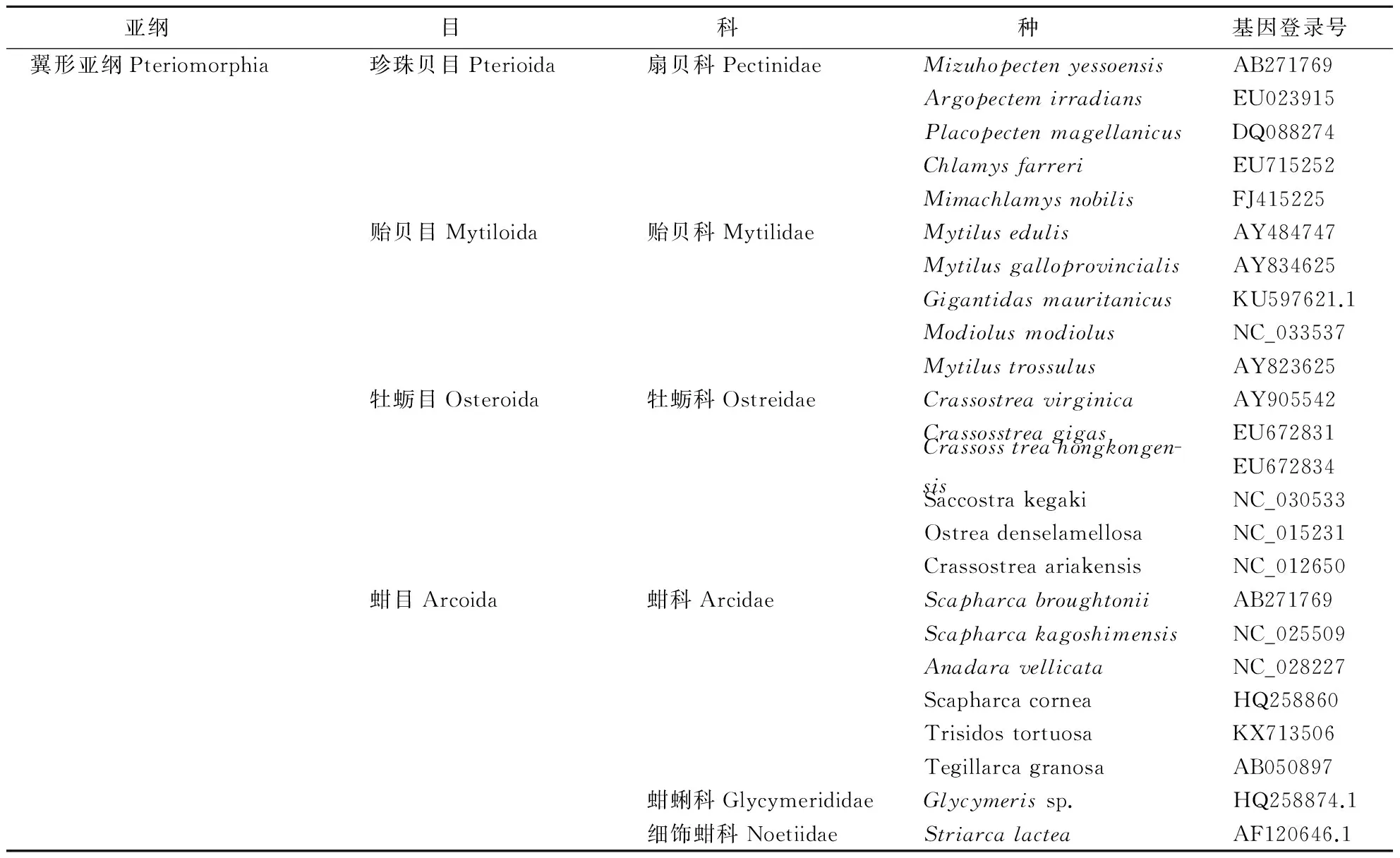
表1 双壳贝类线粒体基因组信息Table 1 Mitochondrial genome information of bivalve
续表1

亚纲目科种基因登录号异齿亚纲Heterodonta帘蛤目Veneroida帘蛤科VeneridaeMeretrixpetechialisEU145977RuditapesphilippinarumAB065375AnomalocardiaauberianaDQ184834.1MercenariamercenariaDQ184836.1LeukomastamineaKF643722.1ChioneelevataKC429136.1ChameleastriatulaKF808159.1竹蛏科SolenidaeSinonovaculaconstrictaEU880278蛤蜊科MactridaeCoelomactraantiquataJN692486MactraveneriformisJN674622.1MactrachinensisNC_025510MactromerispolynymaKF643868.1TresuscapaxKF643926.1SpisulasolidissimaKF644238.1球蚬科SphaeriidaePisidiumobtusaleKF483387.1MusculiumlacustreKF483391.1SphaeriumstriatinumAF120667.1鸟蛤科CardiidaeAcanthocardiatubercula-taNC_008452满月蛤科LucinaLoripeslacteusNC_013271LucinelladivaricataNC_013275海螂目Myoida缝栖蛤科HiatellidaeHiatellaarcticaNC_008451PanopeaabruptaNC_033538海笋科PholadidaeBarneacandidaKC429152.1PholasorientalisKJ125423.1PholasdactylusAY070141.1古异齿亚纲Palaeoheterodon-ta蚌目Unionoida蚌科UnionidaeLampsilisornataNC_005335HyriopsiscumingiiNC_011763QuadrulaquadrulaNC_013658PyganodongrandisNC_013661CristariaplicataNC_012716HyriopsisschlegeliiNC_015110UniopictorumNC_015310MargaritiferidaeMargaritiferafalcataNC_015476古列齿亚纲Palaeotaxodonta胡桃蛤目Nuculoida胡桃蛤科NuculoidaeNuculanucleusEF211991NuculasulcataAF207654.1AcilacastrensisKC429087.1NuculaproximaAF120641.1异韧带亚纲Anomalodesmata笋螂目Pholadomyoida里昂司蛤科LyonsiidaeLyonsiafloridanaAF120654.1隔鳃目Septibranchida杓蛤科CuspidariidaeCardiomyacostellataKR084474.1隐齿亚纲Cryptodonta蛏螂目Solemyoida蛏螂科SolemyidaeSolemyavelesianaKY244081SolemyapervernicosaKY244080SolemyaelarraichensisKY244079
1.2 线粒体基因组DNA提取
20个样品中各取闭壳肌约100 mg充分剪碎,用血液/组织/细胞基因组提取试剂盒(DP304)(天根生化科技北京有限公司)提取线粒体基因组,0.8%的琼脂糖凝胶电泳检测DNA质量.
1.3 PCR扩增和测序
COX1基因引物设计参考孔晓瑜等[18],扩展序列为魁蚶线粒体基因组的1 490~2 198的片段(AB729113).引物序列为:COIL1490:5′-GGTCAACAAATCATAAAGATATTGG-3′;COIH2198:5′-TAAACTTCAGGGTGACCAAAAAATCA-3′.ND1和Cytb基因的引物设计是在魁蚶日本群体ND1和Cytb基因序列的基础上,用引物设计软件Primer Premier 5.0[19,20]设计的.ND1基因的引物序列为:F1:5′-TGTGTGGTTCTGTCAGTAGC-3′;R1:5′-ACTCTTTGTCCGCTAGGGTTG-3′.Cytb基因的引物序列为:F2:5′-TAGCTTGCCACTACAACCC-3′;R2:5′-TGGGTTGGAAGAATGGATGTAG-3′.
PCR反应体系为40 μL,包含20 μL 2×Utaq PCR MasterMix,14 μL ddH2O,上、下游引物各2.0 μL(10 μM)和2.0 μL的DNA模板.PCR反应条件是:94 ℃预变性5 min,94 ℃条件下变性45 s,52 ℃退火40 s,72 ℃延伸1 min,共循环30次,最后72 ℃充分延伸5 min.将PCR扩增产物经1%琼脂糖凝胶电泳检测后,PCR扩增产物送上海生工生物工程有限公司测序,为保证序列准确性,序列经过正反2次重复测定.
1.4 核苷酸序列分析
利用Clustal X[21]软件以默认参数进行DNA序列的多重序列比对分析,生成.aln文件保存分析结果.用MEGA 5.05[22]软件将.aln转换成MEGA格式,统计分析序列的长度、碱基组成、保守位点及变异位点等信息,依据现行形态学的分类将所有研究类群按目和科划分成组,基于Kimura双参数模型[23]计算各目以及科之间的遗传距离.
1.5 系统发育分析
利用软件Clustal X[21]对66种双壳贝类线粒体基因组序列进行多重比对并人工校正,通过MEGA 5.05[22]软件进行整理和分类,用邻接法(Neighbor Joining-NJ法)[24]和最大似然法(Maximum likelihood-ML法)[22]构建了66种双壳贝类(图1-图3)的系统进化树[25],进行系统进化分析,其各分支的置信度经Bootstrap法检验,共1000次循环,其他参数为软件默认值[26].

图1 根据ND1基因序列构建的ML系统发育树Fig.1 ML phylogenetic tree constructed according to the sequence of ND1 gene

图2 根据COX1基因序列构建的ML系统发育树Fig.2 ML phylogenetic tree constructed according to the sequence of COX1 gene
2 结果与分析
2.1 双壳贝类线粒体基因序列分析
分析了双壳纲6个亚纲11个目21科66个物种的ND1、COX1和Cytb序列,其中有翼形亚纲(Pteriomorphia)4个目7个科24个种,异齿亚纲(Heterodonta)2个目8个科25个种,古异齿亚纲(Palaeoheterodonta)1个目2个科8个种,古列齿亚纲(Palaeotaxodonta)1个目1个科4个种,异韧带亚纲(Anomalodesmata)2个目2个科2个种,隐齿亚纲(Cryptodonta)1个目1个科3个种.
本研究所报道的11个目的ND1、COX1和Cytb序列存在明显长度多态性:ND1的全序列长度在570~993 bp之间,其中本研究所测得的魁蚶的ND1基因全长为924 bp.用ClustalX[21]软件对40个ND1序列进行完全比对后,将5′端和3′端剪辑取齐.用MEGA 5.05[22]进行对位排列分析,共有945个比对位点,碱基组成平均为T:37.3%,C:16.3%,A:24.5%,G:21.9%,A+T(61.8%)含量明显高于G+C含量,有明显的AT偏好.其中保守位点58 bp,变异位点867 bp,简约信息位点841 bp.序列中有许多插人/缺失位点,其中最长的连续插入序列约46 bp.用Kimura 2-parameter参数计算遗传距离,40个个体的平均遗传距离为0.964.
40个个体的COX1全序列长度为1 455~1 709 bp,26个个体的COX1的部分序列长度在597~906 bp,其中本研究所测得的魁蚶的COX1基因全长为1 584 bp.用ClustalX[21]软件对66个COX1序列进行完全比对后,将5′端和3′端剪辑取齐.用MEGA 5.05[22]进行对位排列分析,共有1 519个比对位点,碱基组成平均为T:35.7%,C:17.6%,A:25.7%,G:20.9%,A+T(61.4%)含量明显高于G+C含量,具有明显的AT偏好.其中保守位点119 bp,变异位点1394 bp,简约信息位点1369 bp.

图3 根据Cytb基因序列构建的ML系统发育树Fig.3 ML phylogenetic tree constructed according to the sequence of Cytb gene
40个个体的Cytb全序列长度1 077~1 308 bp,本研究所测得的魁蚶的Cytb基因全长为1 278 bp.用ClustalX[21]软件对66个Cytb序列进行完全比对后,将5′端和3′端剪辑取齐.用MEGA 5.05[22]进行对位排列分析,共有1 286个比对位点,碱基组成平均为T:37.1%,C:17.4%,A:25.3%,G:20.1%,A+T(62.4%)含量明显高于G+C含量,具有明显的AT偏好.其中保守位点70 bp,变异位点1 088 bp,简约信息位点1 286 bp.用Kimura 2-parameter参数计算遗传距离,40个个体的平均遗传距离为0.799.核苷酸的替换以颠换为主,转换(Ts)/颠换(Tv)比值平均为0.66.
2.2 遗传距离
用Kimura 2-parameter参数计算66个个体的COX1基因的遗传距离.目内不同种间的平均遗传距离为:蚌目(2个科)0.178;帘蛤目(6个科)0.399;海螂目(2个科)0.468;蛏螂目(1个科)0.169;蚶目(3个科)0.311;珍珠贝目(1个科)0.288;贻贝目0.375(1个科);牡蛎目(1个科)0.263;胡桃蛤目(1个科)0.272.由表2可见,11个目间遗传距离0.323~2.557.珍珠贝目与蚌目间的遗传距离最大(2.557),胡桃蛤目与蛏螂目间的遗传距离最小(0.323).

表2 双壳类11目间遗传距离Table 2 Genetic distances among 11 orders of bivalve
除贻贝科(5个种)遗传距离相差较大外,在0.011~0.534之间,其它几科的遗传距离都相差较小:蚌科(7个种)遗传距离0.071~0.209,牡蛎科(6个种)为0.136~0.335,扇贝科(5个种)为0.244~0.311,蚶科(6个种)为0.111~0.399,帘蛤科(7个种)为0.205~0.559,蛤蜊科(6个种)为0.174~0.394,蛏螂科(3个种)为0.163~0.175,胡桃蛤科(4个种)为0.222~0.302.
2.3 系统发育关系分析
ND1、COX1和Cytb是双壳贝类线粒体内具有较好的系统发育信息的基因,通过对其进行PCR扩增和序列测定,分析比较双壳贝类的种间变异程度和亲缘关系.本研究检测了双壳贝类6个亚纲11个目20科66个物种的线粒体基因ND1、COX1和Cytb,运用MEGA 5.05软件中的邻接法和最大似然法构建了系统进化树,结果如图1~图3所示.NJ树的拓扑结构与ML树相似,图片不再列出.将获得的拓扑结构与传统划分的亚纲、目、科和属以及已发表过的分子学研究一起对比分析.
由图中可见,不同基因用不同方法得到的系统进化树是有差异的.基于ND1基因构建的ML系统发育树中,古异齿亚纲和古列齿亚纲聚为一支,隐齿亚纲位于拓扑结构的底部,翼形亚纲为单系发生,其中珍珠贝目与异齿亚纲的海螂目聚为一支,除了帘蛤目中的鸟蛤科单独为一支外,其他异齿纲各目聚为一支.基于COX1基因构建的ML系统发育树中,古列齿亚纲和隐齿亚纲聚为一支,异韧带亚纲形成两个独立进化的分支,异齿亚纲为单系发生,海螂目和帘蛤目没有形成两个独立的进化分支,其中海螂目的海笋科和缝栖蛤科各形成4小支,翼形亚纲为多系发生,其中贻贝科与隔鳃目的杓蛤科聚为一支,古列齿亚纲位于拓扑结构底部,单独形成一支.基于Cytb基因构建的ML系统发育树中,古列齿亚纲和古异齿亚纲聚为一支,异齿亚纲中帘蛤目单独聚为一支,海螂目的一个种单独成一支,翼形亚纲为多系发生,各目单独形成一支.
通过系统发育树可得知,所有双壳纲的种类构成了单系发生的进化枝;双壳纲主要分为异齿亚纲和翼形亚纲两大类群,异齿亚纲是单系发生,翼形亚纲的系统发生关系则随方法的不同有所区别,贻贝目的位置是造成此差异的主要因素.
异齿亚纲中,海螂目和帘蛤目的物种没有完全聚在所属的目内,帘蛤目是并系发生的.关于海螂目的系统发生关系目前并没有统一的结论,Morton[27]根据形态学的标准认为海螂目是多系发生的,各科的系统发生位置分散于帘蛤目的各科间,海螂目不应该划分为独立的目,该假说得到了许多基于分子研究的证明[28-30].另外一种得到广泛认同并为现有分类系统所采纳的假说是,海螂目是单系发生的,异齿亚纲可向下划分为帘蛤目和海螂目[31].与其他基于分子的结果不同,本研究中海螂目的多系发生得到了很好的支持,各科的系统发生位置分散于帘蛤目的各超科间,该结果支持第一种假说,即海螂目不应该划分为独立的目.蛏螂目位于进化树的基部,与其他种类构成单系群,显示了该目在双壳纲内较为原始的进化地位.双壳纲各科都构成了单系群,缝栖蛤科和海笋科除外,二者为多系发生.古异齿亚纲和古列齿亚纲聚为一支,二者可能具有较近的亲缘关系.
3 讨论
3.1 双壳纲种类的分类地位
传统的双壳贝类系统发生研究以形态学性状,即表型为基础.相对于传统意义形态学的同源性状,基因序列信息可从本质上反应物种进化过程中的分歧,从而尽可能完整地还原生物的进化过程[32].随着分子生物学的发展,以生物大分子为研究对象的分子进化得到迅速发展,并成为进化生物学的重要领域.
本研究比较分析了双壳纲66种贝类的线粒体ND1、COX1和Cytb基因序列的碱基组成、变异位点以及遗传距离等信息,并用邻近法和最大似然法构建了分子系统发育树,结果显示所有双壳纲的种类构成了单系发生的进化枝,不同基因构建的进化树中翼形亚纲的系统发生关系不一致,其拓扑结构随着建树方法的不同有所差异,与形态学结果相似.本研究确认了异齿亚纲的单系发生,与Canapa等人[29]的结果一致.其他双壳纲分子研究中,很少有关于蛏螂目的报道,本研究中利用ND1和Cytb基因构建的进化树中,蛏螂目都位于进化树的底端,单独成一支系,基于COX1基因的进化树中,该目与古列齿目聚为一支,位于异齿亚纲各科间.
3.2 不同线粒体基因用于双壳纲分子系统进化的功效
目前可用于系统发育分析的线粒体基因主要有12SrRNA, 16SrRNA,COI,COⅡ,ND1,ND2和ND5等,其中以16S,COI以及核基因18S作为DNA条形码对物种系统发生关系的研究尤为普遍[33].国内外已有很多研究以18S、16S或者COI为目的DNA片段对一些双壳类进行了序列分析[28,34],并对扇贝科[35]、帘蛤科[36,37]、蛏类[38]等双壳类的系统发生与进化进行了探讨.
本研究中,以ND1构建的ML树中,翼形亚纲为单系发生,与Canapa[29]以及Steiner[39]等人18SrRNA的研究结果一致;异齿亚纲中鸟蛤科单独聚为一支,不支持异齿亚纲的单系发生.以COX1构建的ML树中,翼形亚纲的贻贝目单独成一支,此结果不支持翼形亚纲的单系发生,与其他分子研究的结果一致[40,41].异齿亚纲为单系发生,异韧带亚纲为并系发生,隐齿亚纲和古列齿亚纲聚为一支.以Cytb构建的ML树中,翼形亚纲的牡蛎目单独成一支,此结果不支持翼形亚纲的单系发生.异齿亚纲为单系发生,隐齿亚纲位于进化树底端.应用不同分子标记构建的系统发育树存在较大差异,这可能是因为线粒体基因组的不同区域所发挥的功能不同,在进化中受到的选择压力强度也不一样,所以使其进化速率有所差异,因此不同区域所包含的系统发育信息也不尽相同[42].
总体而言,本研究结果与传统形态学、古生物学分析的结果基本一致,Cytb基因比COX1和ND1可以更好的解析双壳纲的系统发生关系.双壳纲贝类的养殖在我国水产养殖业中占有重要地位,对其进行分子系统进化的研究,不仅对于揭示其遗传变异和繁殖发育规律有重要意义,还为重要养殖种类的杂交和育种提供理论依据,具有重要的生产应用意义.
[1] 徐凤山,张素萍,王少青.中国海产双壳类图志[M].科学出版社,2008.
[2] 任建峰.软体动物线粒体基因组学及分子系统发生研究[D].青岛:中国科学院海洋研究所,2008.
[3] 徐凤山,张均龙.中国海典型生境双壳类软体动物多样性特点[J].生物多样性,2011,19(6):716-722.
[4] 张源真,王伟,姜志强,等.基于16SrRNA部分序列探讨部分鳚亚目鱼类的分子系统进化关系[J].海洋科学,2012,36(9):89-95.
[5] 远洋.九种异齿亚纲贝类线粒体基因组研究[D].中国海洋大学,2013.
[6] IANNELLI F, PESOLE G, SORDINO P, et al. Mitogenomics reveals two cryptic species in Ciona intestinalis.[J]. Trends in Genetics Tig, 2007,23(9):419-422.
[7] BOORE J L, FUERSTENBERG S I. Beyond linear sequence comparisons: the use of genome-level characters for phylogenetic reconstruction.[J]. Philos Trans R Soc Lond B Biol Sci, 2004,363(1496):1 445-1 451.
[8] XU K, KANNO M, YU H, et al. Complete mitochondrial DNA sequence and phylogenetic analysis of Zhikong scallopChlamysfarreri(Bivalvia: Pectinidae)[J]. Molecular Biology Reports, 2011,38(5):3 067-3 074.
[9] BRETON S, BEAUPRÉ H D, STEWART D T, et al. Comparative mitochondrial genomics of freshwater mussels (Bivalvia: Unionoida) with doubly uniparental inheritance of mtDNA: gender-specific open reading frames and putative origins of replication[J]. Genetics, 2009,183(4):1 575-1 589.

[11] 程汉良,彭永兴,王芳,等.6种帘蛤科贝类18S rRNA基因全序列比较分析[J].中国水产科学,2008,15(4):559-567.
[12] REN J, SHEN X, JIANG F, et al. The mitochondrial genomes of two scallops, Argopecten irradians and Chlamys farreri (Mollusca: Bivalvia): the most highly rearranged gene order in the family Pectinidae.[J]. Journal of Molecular Evolution, 2010,70(1):57-68.
[13] DANICTCHALEU G, HEURTEBISE S, MORGA B, et al. Complete mitochondrial DNA sequence of the European flat oysterOstreaedulisconfirms Ostreidae classification[J]. BMC Research Notes, 2011,4(1):1-10.
[14] XU X, WU X, YU Z. Comparative studies of the complete mitochondrial genomes of fourPaphiaclams and reconsideration of subgenusNeotapes(Bivalvia: Veneridae).[J]. Gene, 2012,494(1):17-23.
[15] WANG H X, ZHANG S P, LI Y, et al. Complete mtDNA ofMeretrixlusoria(Bivalvia: Veneridae) reveals the presence of anatp8 gene, length variation and heteroplasmy in the control region.[J]. Comparative Biochemistry & Physiology Part D: Genomics & Proteomics, 2010,5(4):256-264.
[16] 孟学平,申欣,赵娜娜,等.双壳类线粒体基因组结构分析[J].水产科学,2013,32(12):721-729.
[17] SEKINE, Y., YAMAKAWA, H., TAKAZAWA, S. et al. Geographic Variation of the COX1 Gene of the Short-neck Clam Ruditapes philippinarum in Coastal Regions of Japan and China[J]. Venus Journal of the Malacological Society of Japan, 2006,65:229-240.
[18] 孔晓瑜,姜艳艳,相建海,等.魁蚶线粒体16S rRNA和COI基因片段序列测定及其应用前景[J].海洋科学,2001,25(12):46-48.
[19] 翟中会,陈希南,王娟.利用Primer Premier 5.0进行引物设计[J].西北医学教育,2008,16(4):695-698.
[20] 任亮,朱宝芹,张轶博,等.利用软件Primer Premier 5.0进行PCR引物设计的研究[J].锦州医学院学报,2004,25(6):43-46.
[21] THOMPSON JD, GIBSON TJ, PLEWNIAK F, et al. The CLUSTAL_X windows interface flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucl Acids Res, 1997,25:4 876-4 882.
[22] TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011,28(10):2 731-2 739.
[23] KIMURA M. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980,16(2):111-120.
[24] SAITOU N, NEI M. The Neighbor-joining method: A new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987,4(4):406-425.
[25] COAN E V, SCOTT P V, BERNARD F R, et al. Bivalve Seashells of Western North America. Marine Mollusks from Artic Alaska to Baja California[M]. Bivalve Seashells of Western North America: Marine Bivalve Mollusks from Arctic Alaska to Baja California, 2000.
[26] 金逍逍,孙悦娜,王日昕,等.虾虎鱼类线粒体全基因组序列结构特征分析及系统发育关系探讨[J].遗传,2013,12:1 391-1 402.
[27] MORTON B. The biology and functional morphology ofCorbulacrassa(Bivalvia: Corbulidae) with special reference to shell structure and formation[J]. The Marine Flora and Fauna of Hong Kong and Southern China Ⅱ, 1990,3:1 055-1 073.
[28] ADAMKEWICZ S L, HARASEWYCH M G, BLAKE J, et al. A molecular phylogeny of the bivalve mollusks.[J]. Molecular Biology & Evolution, 1997,14(6):619-629.
[29] CANAPA A, BARUCCA M, MARINELLI A, et al. A molecular phylogeny of Heterodonta (Bivalvia) based on small ribosomal subunit RNA sequences.[J]. Molecular Phylogenetics & Evolution, 2001,21(1):156-161.
[30] DOLEZEL J, BARTOS J, VOGLMAYR H, et al. Nuclear DNA content and genome size of trout and human. CytometryA, 2003,51(2):127-128.
[31] PURCHON R D, BROWN D. Phylogenetic interrelationships among families of bivalve molluscs. Malacologia, 1969,9:163-171.
[32] 高凌云.基于双壳纲18S rDNA序列的系统发生分析及核DNA含量与其进化地位关系的初步探讨[D].中国海洋大学,2007.
[33] 崔爱明,黄原.利用线粒体16S rRNA基因全序列分析直翅目主要类群的系统发生关系[J].遗传,2012,34(5):597-608.
[34] KIM S C, KIM J J, HONG H C. Molecular phylogeny of veneroidea (Bivalvia: Heterodonta) on the basis of partial sequences of 28S rRNA gene[J].The Korean Journal of Malacology, 2005,21(2):147-161.
[35] CANAPA A, BARUCCA M, MARINELLI A, et al. Molecular data from the 16S rRNA gene for the phylogeny of Pectinidae (Mollusca: Bivalvia)[J]. Journal of Molecular Evolution, 2000,50(1):93-97.
[36] CANAPA A, ROLLO F E, MAROTA I. The small-subunit rRNA gene sequences of venerids and the phylogeny of bivalvia.[J]. Journal of Molecular Evolution, 1999,48(4):463-468.
[37] CANAPA A, SCHIAPARELLI I, MAROTA M. Molecular data from the 16S rRNA gene for the phylogeny of Veneridae[J]. Marine Biology, 2003,142:1 125-1 130.
[38] 陈丽梅,孔晓瑜,喻子牛,等.3种蛏类线粒体16S rRNA和COI 基因片段的序列比较及其系统学初步研究[J].海洋科学,2005(8):29-34.
[39] GERHARD S, HERMANN D. Molecular phylogeny of Scaphopoda (Mollusca) inferred from 18S rDNA sequences: support for a Scaphopoda-Cephalopoda clade[J]. Zoologica Scripta, 2010,32(4):343-356.
[40] STEINER G, HAMMER S. Molecular phylogeny of the Bivalvia inferred from 18S rDNA sequences with particular reference to the Pteriomorphia[J]. Geological Society London Special Publications, 2000,177(1):11-29.
[41] WINNEPENNINCKX B, BACKELJAU T, DE WACHTER R. Investigation of molluscan phylogeny on the basis of 18S rRNA sequences[J]. Mol Biol Evol, 1996,13:1 306-1 317.
[42] 赵凯.鱼类线粒体DNA(mtDNA)及其在分子系统学中的应用[J].青海大学学报(自然科学版),2006(2):49-53.

