基于线粒体控制区的中国近海 绿鳍鱼遗传多样性分析
卢丽锋, 章 群, 杨喜书, 黄镇宇, 唐楚林, 周 琪
(暨南大学生态系,广州 510632)
绿鳍鱼(Chelidonichthyskumu)隶属于鲉形目(Scorpaeniformes)鲂鮄科(Triglidae)绿鳍鱼属,俗称绿翅鱼、角鱼、国公鱼等,小眼绿鳍鱼(Chelidonichthysspinosus)为其同种异名[1]。绿鳍鱼是广泛分布于印度洋-西太平洋河口至大陆架边缘泥沙质海区的暖水性中小型底层鱼类,能以胸鳍游离鳍条在海底匍匐爬行,春夏之交游近沿岸河口海湾产漂浮性卵,秋末冬初游向外海越冬[1]。绿鳍鱼的背脊具有一定毒性,主要摄食虾类、软体动物和小鱼,同时也是其它经济鱼类的饵料,在沿海生态系统中具有重要作用[2]。绿鳍鱼在日本、韩国与中国近海均有分布,其肉质鲜美,蛋白质和脂肪含量高,有较高的营养价值和食用价值,适合养殖开发,是中国、日本、东南亚等地的经济鱼类之一。近年来,随着大黄鱼(Pseudosciaenacrocea)[3]、真鲷(Pagrusmajor)和黑鲷(Acanthopagrusschlegelii)[4]等大中型经济鱼类资源衰退,绿鳍鱼已成为中国沿海流刺网和底层拖网渔业的主要捕捞种类之一,也是游钓渔业重要的对象[5]。海州湾鱼类资源调查[6]表明绿鳍鱼是海州湾底栖生物食性类群中秋季优势度最高的鱼种,占秋季总生物量的28.61%;但长江口绿鳍鱼资源有明显波动[7]。
目前,国内外对绿鳍鱼的研究局限于生理习性、种群数量及生长环境等方面[6],研究表明[8],在东亚,绿鳍鱼有秋冬从渤海、黄海北部南下至济州岛西部海域越冬的黄渤海群,冬季在舟山外海越冬、翌年春夏至长江口北部后分散活动的东海群,以及周年栖息于九州近海、对马南部和五岛西南部鱼群,但并未述及南海绿鳍鱼种群,且迄今未见研究这些群体间是否存在遗传差异的报道。动物线粒体DNA为母系遗传,结构简单无重组,进化速率快,是动物种群遗传学研究的首选分子标记[9];其中控制区的遗传变异速率为3%~10%/百万年[10],是线粒体基因组中进化速率最快的区段,常用于研究动物遗传多样性和种群结构。本研究测定并分析了辽宁金石滩、辽宁东港、山东青岛、江苏吕四、浙江舟山、广东南澳等中国沿海5省6个地理群体的线粒体控制区序列,旨在更全面的了解中国近海绿鳍鱼的遗传背景,为种质资源的管理保护和合理利用提供科学依据。
1 材料与方法
1.1 实验材料与方法
实验所用野生绿鳍鱼样品由近海作业的渔民处直接购买所得,分别采集于:黄海辽宁丹东东港(LDG1~13)、辽宁大连金石滩(LJST1~18)、山东青岛(LQD1~27)、江苏吕四港(SLS1~12);东海浙江舟山(ZZS1~28)和南海广东南澳(YNA1~14),采样点见图1。提取样品肌肉保存于95%乙醇备用。PCR扩增引物为本实验室自行设计的CrL: 5′-TCGGTCTTGTAATCCGAA-3′与CrR: 5′-AGTCAGGACCAAGCCTTT-3′。DNA提取和PCR扩增参照乐小亮等[11]方法,将电泳检测为条带清晰明亮的PCR扩增产物送至华大基因有限公司切胶纯化并测序。
1.2 数据处理
利用MEGA 6.0[13]对测定序列进行校对与对位排列,计算碱基组成、多态位点、简约信息位点、转换与颠换比以及基于Kimura-2-Parameter模型的遗传距离,构建邻接树。通过DnaSP 5.1[14]计算单倍型数、单倍型多样性(h)、核苷酸多样性(π),以及遗传分化系数(Fst)和基因流(Nm)值。利用Network 5.0[15]构建单倍型网络图。使用Arlequin 3.5[16]进行分子方差分析(AMOVA),检验群体遗传变异;进行Tajima’sD和Fu’sFs中性检验获得SSD值、Raggedness值,通过核苷酸不配对分析检验种群历史动态[17],根据公式τ=2ut和T=t×(代时),估算种群扩张时间,其中τ是时间扩张参数;u=μk,μ为序列变异速率,k为序列长度;t表示种群自然扩张以来的时间。
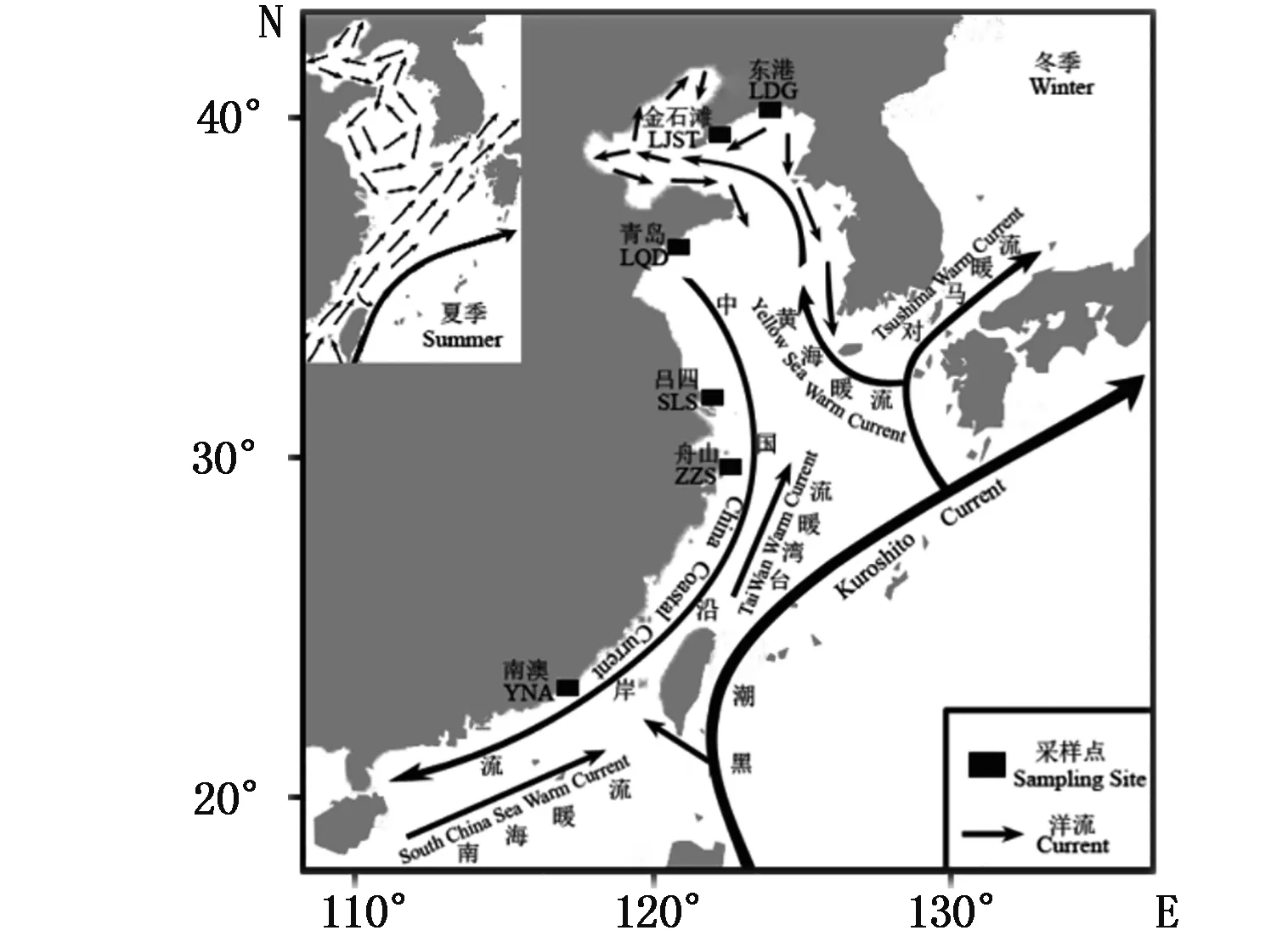
图1 绿鳍鱼采样地点分布图(根据孙海平等[12]修改)Fig.1 Sampling sites of Chelidonichthys kumu(Modified from SUN et al.[12])注:LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’aoNote:LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
2 结果与分析
2.1 绿鳍鱼线粒体控制区序列特征、遗传多样性和遗传分化
在6个群体112条绿鳍鱼线粒体控制区838 bp全序列中,A、T、C、G的平均含量分别是30.5%、30.3%、21.2%、18.0%,A+T的含量(60.8%)明显高于G+C的含量(39.2%),表现出典型的反G偏倚,吻合脊椎动物线粒体DNA特征[18]。共检测到15个变异位点,10个简约信息位点;转换与颠换比1.97,表明序列变异未饱和,可用于种群遗传分析。在22个单倍型中,共享单倍型11个。绿鳍鱼整体呈现高单倍型多样性(h=0.849±0.019)与低核苷酸多样性(π=0.001 85±0.000 12)特点,其中江苏吕四群体(h=0.818±0.096,π=0.002 33±0.000 59)遗传多样性最高;浙江舟山群体(h=0.794±0.042,π=0.001 60±0.000 23)遗传多样性最低,详见表1。
K-2-P模型构建的邻接树和单倍型网络图(图2)均显示,不同地理来源的个体混杂分布,没有明显的地理聚群。群体间和群体内的遗传距离(表2)为0.001 6~0.002 3;群体间的遗传分化系数Fst(表2)为-0.034 61~0.038 28(P>0.05);按照6个地理群体间、样品采集地所属海域及台湾海峡南北两侧(即东海-黄海群组与南海群组)分组进行AMOVA分析,发现群体内的变异比例达到99%(表3)以上,6个群体间(Fst=0.000 5,P=0.45)、不同海域间(Fct=-0.013 2,P=0.79)及台湾海峡南北两侧(Fct=0.011 9,P=0.49)均不存在显著的遗传结构,表明遗传变异主要存在群体内的个体之间,不同地理群体间和不同海区间都不存在遗传分化。
2.2 绿鳍鱼的种群历史动态
中性检验结果见表4:除东港群体Tajima’sD值为正值(0.009 2)外,其它群体(-0.945 1~-0.613 6)与中国近海绿鳍鱼总体(-1.234 3)的Tajima’sD值均为负值,但都不显著(P>0.05)。除吕四群体Fu’sFs值为负值(-1.176 6)且不显著(P=0.125)外,其它群体(-5.798 1~-1.391 5)与中国近海绿鳍鱼总体(-16.099 6)都为负值且显著(P<0.05)。核酸不配对分析的SSD值均较小且不存在显著差异,表明并未显著偏离种群扩张模型。由于Fu’sFs检验比Tajima’sD检验对群体近期扩张更敏感[19],东港、金石滩、青岛、舟山、南澳等5个群体及绿鳍鱼整体Fu’sFs值(表4)为显著负值;核苷酸不配对分析峰图(图3)为明显单峰分布;表明上述5个群体和绿鳍鱼整体近期历史上经历了明显的快速扩张事件[20],根据整体扩张参数τ值为1.68,以线粒体控制区的遗传变异速率(3%~10%)/百万年[10],由此估算出整体种群扩张发生在3.00万年~10.00万年前的晚更新世时期。
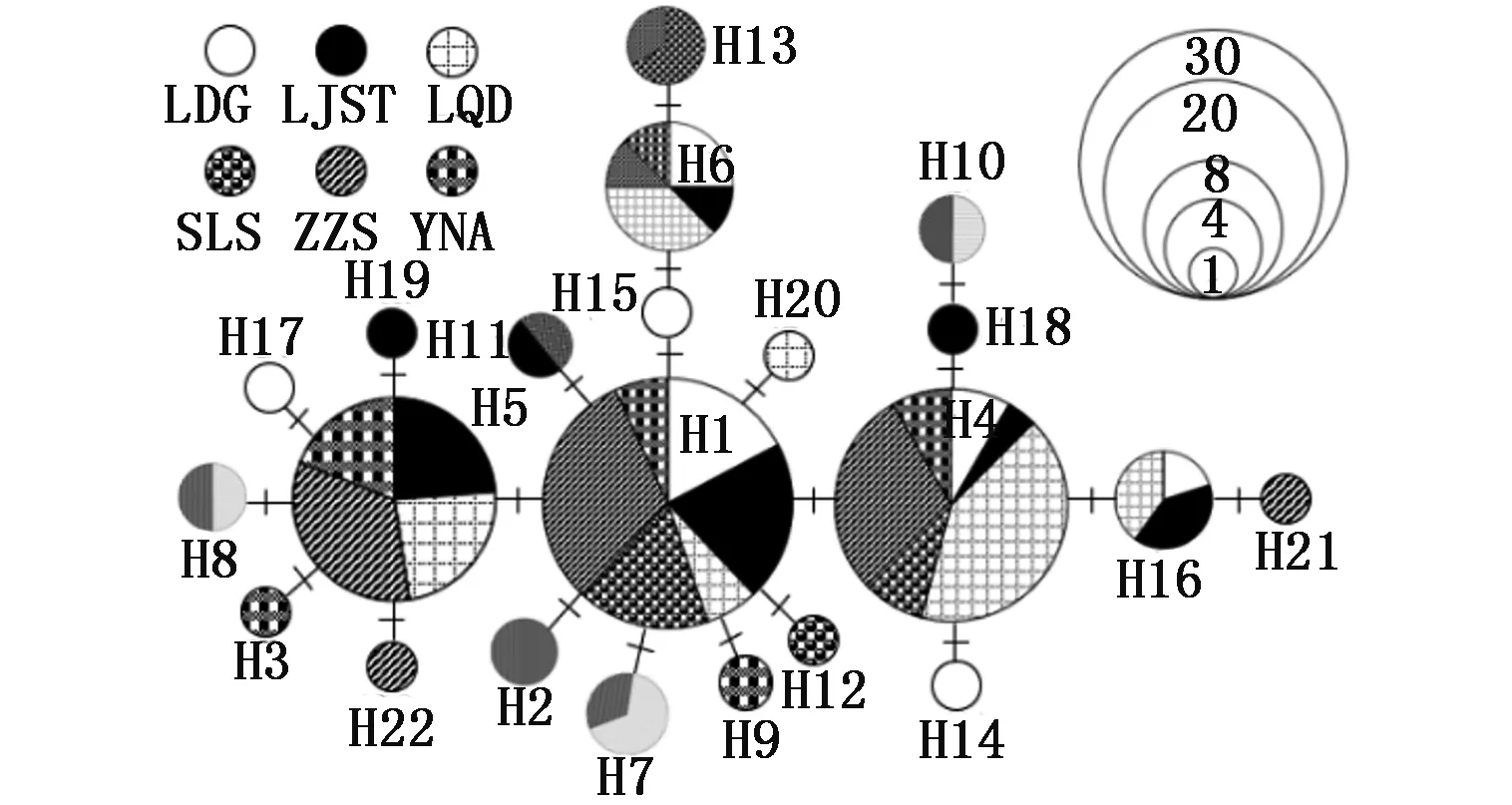
图2 线粒体CR基因序列单倍型网络图Fig.2 Haplotype network using mtDNA CR sequence data showing geographic distribution of haplotypes注:图中每个图圈代表一个单倍型且其面积大小与所包含的个体数量成正比; 枝上短线表示突变步骤Note:Circles are unique haplotypes proportional to frequencies,short lines represent mutation steps
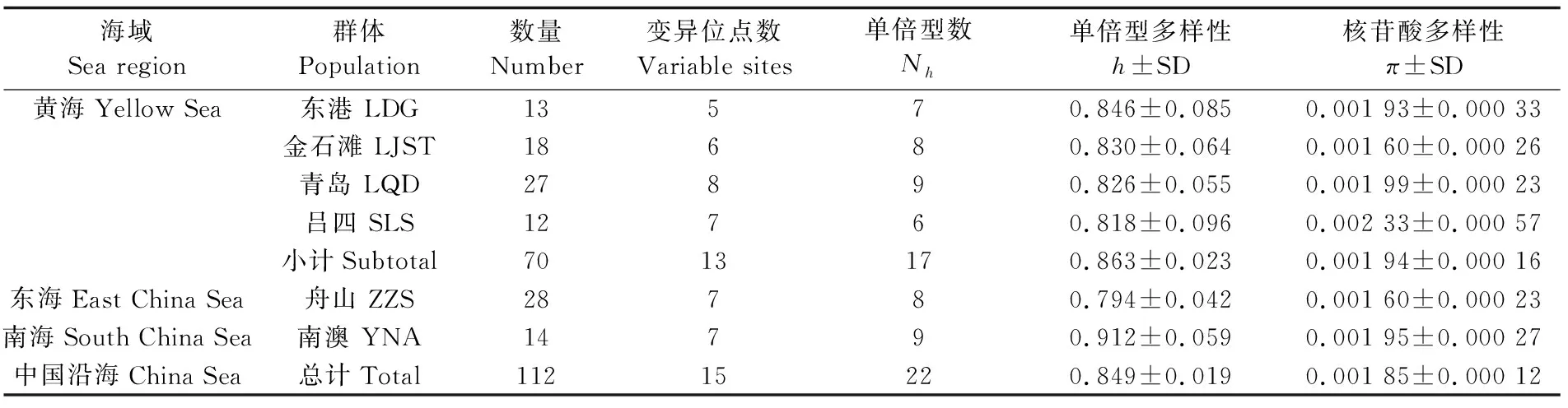
海域Sea region群体Population数量Number变异位点数Variable sites单倍型数Nh单倍型多样性h±SD核苷酸多样性π±SD黄海Yellow Sea东港 LDG13570.846±0.0850.001 93±0.000 33金石滩 LJST18680.830±0.0640.001 60±0.000 26青岛 LQD27890.826±0.0550.001 99±0.000 23吕四 SLS12760.818±0.0960.002 33±0.000 57小计Subtotal7013170.863±0.0230.001 94±0.000 16东海East China Sea舟山 ZZS28780.794±0.0420.001 60±0.000 23南海South China Sea南澳 YNA14790.912±0.0590.001 95±0.000 27中国沿海China Sea总计Total11215220.849±0.0190.001 85±0.000 12
注:LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
Note:LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
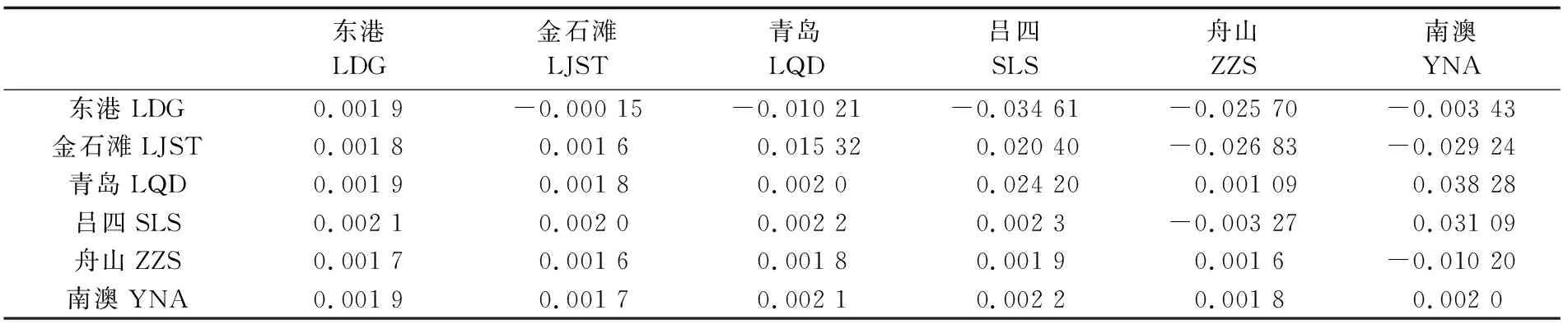
表2 绿鳍鱼群体内(对角线)和群体间遗传距离(对角线下)及群体间 Fst(对角线上)Tab.2 Genetic distances within (along diagonal)and among populations (below diagonal), and Fst among populations (above diagonal) of Chelidonichthys kumu populations
注:表中所有Fst值均无显著性差异(P>0.05)。LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
Note: AllFstvalues in the table have no significant difference(P>0.05).LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
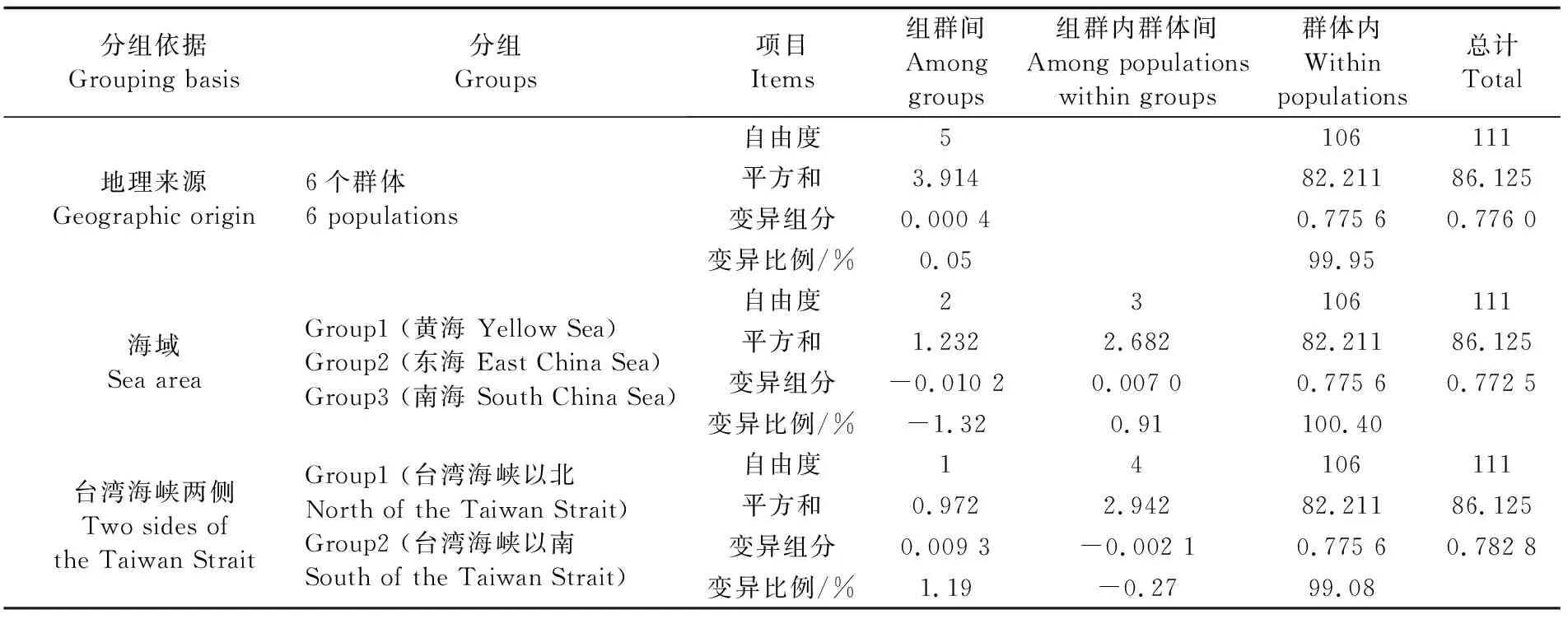
表3 中国近海绿鳍鱼种群结构的分子方差分析Tab.3 AMOVA analysis based on mtDNA differentiation of Chelidonichthys kumu in the coastal waters of China
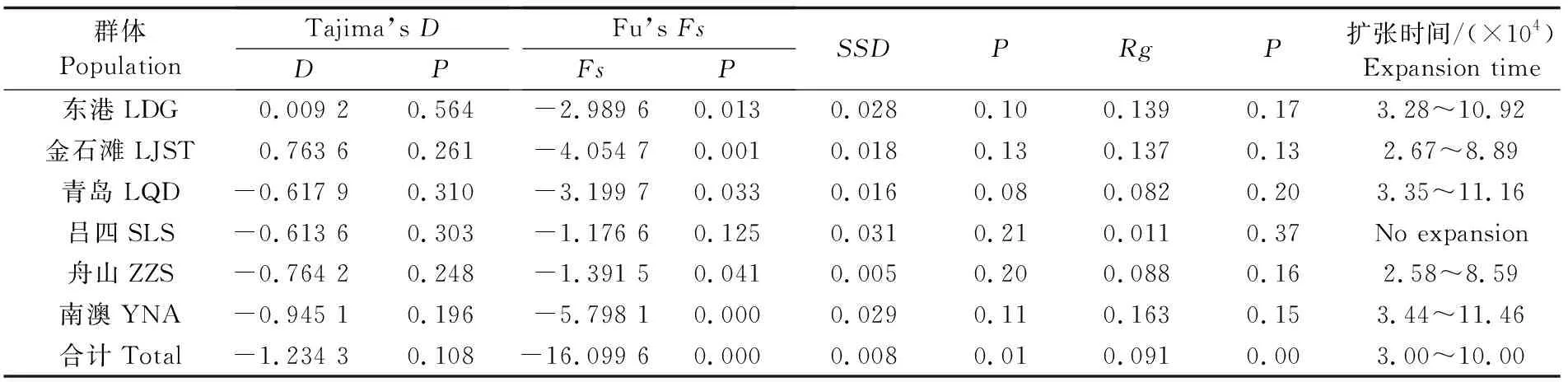
表4 中国近海绿鳍鱼中性检验、不配对分布Tab.4 Neutrality tests and mismatch distribution of Chelidonichthys kumu
注:LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
Note:LDG:Donggang; LJST:Jinshitan; LQD:Qingdao; SLS:Lvsi; ZZS:Zhoushan; YAN:Nan’ao
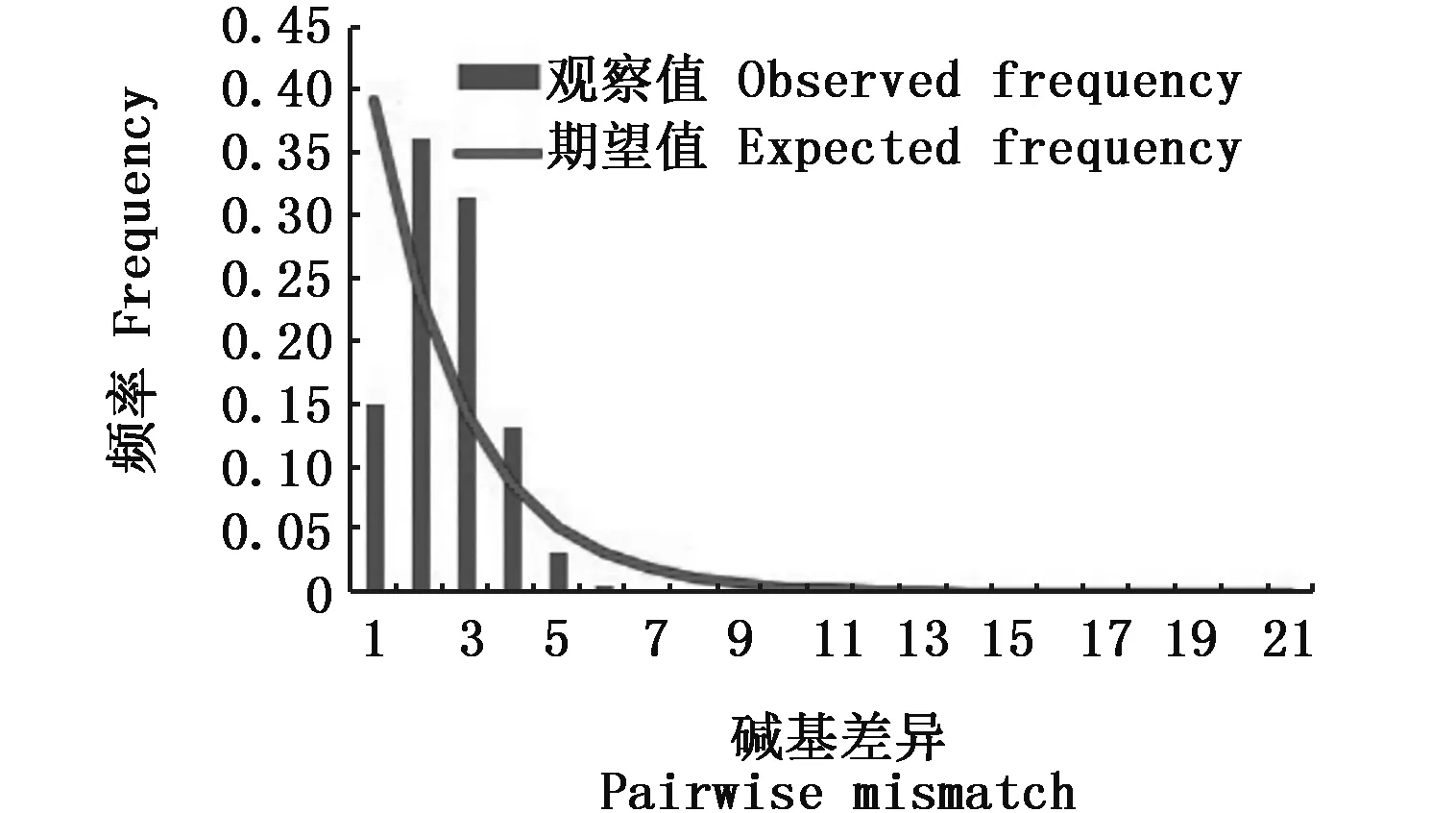
图3 绿鳍鱼线粒体控制区序列核酸不配对分析图Fig.3 Nucleotion mismatch distribution of Chelidonichthys kumu mtDNA control region sequences
3 讨论
3.1 绿鳍鱼的遗传多样性、种群结构和历史动态
中国近海绿鳍鱼整体的线粒体控制区单倍型多样性(h=0.849)和核苷酸多样性(π=0.001 85),与西北太平洋同域分布的黑鲷(h=0.994,π=0.007 4) 、平鲷(Rhabdosargussarba) (h=0.999,π=0.007 3)[4]、花鲈(Lateolabraxmaculatus) (h=0.96,π=0.003)[21]等一样具有高单倍型多样性和低核苷酸多样性分布模式[22]。推测是线粒体序列在短时间内出现的少量核苷酸变异即可提高单倍型多样性,但不能明显增加核酸多样性[23]。但绿鳍鱼遗传多样性远低于黑鲷、平鲷、花鲈等鱼类[4,21],表明中国近海绿鳍鱼的遗传多样性相当低,推测可能经历过严重的瓶颈效应,这个推论与曹艳[4]对中国近海黑鲷的研究结果相似。
绿鳍鱼6个地理群体间的遗传分化系数Fst值为-0.034 61~0.038 28(P>0.05),遗传距离为0.001 6~0.002 3,表明群体间不存在遗传分化;AMOVA分子方差分析结果也显示,不同地理群体间、不同海域群组间、台湾海峡南北两侧群组内的遗传变异比例达99%以上,这与吕金磊等[24]对中国南海卵形鲳鲹(Trachinotusovatus)的研究结果相似,表明绿鳍鱼遗传分化主要在群体内的个体之间;在单倍型网络图中,不同地理来源个体相互混杂,不能形成明显地理聚群,表明没有明显的地理结构,推测原因如下:绿鳍鱼能进行大范围的越冬洄游[1];卵具有漂浮性,受向北的南海暖流和黑潮以及沿岸流的影响,可进行较远距离的传播,导致不同地理群体间频繁的基因交流,不能形成明显的地理结构。这个推论与SONG等[25]对西北太平洋斑尾复虾虎(Synechogobiusommaturus)等研究结果相似。黄渤海群、东海群以及南海绿鳍鱼群体之间几乎没有遗传分化,这与大黄鱼情况相似。依据大黄鱼形态学和生态学特征的差别,中国沿海大黄鱼由北至南分成岱衢族、闽-粤东族和硇洲族 3 个地理种群,但线粒体序列分析同样未能检测到明显遗传分化[3]。
中国近海绿鳍鱼核苷酸不配对峰图为单峰分布;除吕四群体之外,其它5个地理群体中性检验Fu’sFs为显著至极显著负值(表4),均表明这些群体在近期历史上出现过种群快速扩张事件,推算出中国近海绿鳍鱼整体扩张时间发生在3.00万年~10.00万年前,即中国近海绿鳍鱼可能在受晚更新世的倒数第二次盛冰期海平面下降导致大陆架出露而急剧减少了分布范围导致的种群瓶颈后,在随后的间冰期至末次盛冰期前的相对高海平面时期因分布范围扩大而出现种群扩张。中国近海绿鳍鱼在末次盛冰期间前发生种群扩张,这一推论与银鲳(Pampusargenteus)等结果相似[26],即更新世晚期冰期与间冰期循环导致的海平面升降变化是影响大部分中国近海鱼类的种群快速扩张的关键因素。
3.2 绿鳍鱼种群资源保护
中国近海绿鳍鱼不同地理群体间与不同组群间几乎不存在母系遗传分化,故可作为一个管理保护单位。中国绿鳍鱼相较其它同域分布的鱼类遗传多样性明显偏低,需要引起渔业管理部门重视。江苏吕四群体遗传多样性高于其它群体,建议优先保护[27];浙江舟山群体多样性最低,应采取措施避免遗传多样性的下降造成种群适应性下降而衰退。
由于线粒体仅为母系遗传,不能反映双亲的遗传信息,以线粒体控制区为分子标记并不不一定能完整地反映绿鳍鱼的遗传分化,在将来的研究中,可在本研究的基础上,运用微卫星和AFLP等分子标记技术从核基因遗传多样性角度进行研究,并结合其生理习性、生态环境以及形态特征进行更全面的分析,同时扩大绿鳍鱼采样群体的地理范围,以更好地揭示中国近海绿鳍鱼的遗传结构,为其种质资源的保护、合理利用提供科学依据。
[1] 陈明茹, 杨圣云. 台湾海峡及其邻近海域鱼类图鉴[M]. 北京: 中国科学技术出版社, 2013: 95.
CHEN M R, YANG S Y. Illustrations of fish in the Taiwan Strait and its adjacent waters[M]. Beijing: China Science and Technology Press, 2013: 95.
[2] PARK J M, COBURN E, PLATELL M E,etal. Diet and resource partitioning among three sympatric gurnards in northeastern Tasmanian waters, Australia[J]. Marine and Coastal Fisheries: Dynamics, Manangement and Ecosystem Science, 2017(9): 305-319.
[3] 毛 勇, 蒋秋芬, 曾华嵩, 等. 大黄鱼线粒体DNA控制区遗传多样性分析[J]. 厦门大学学报(自然科学版), 2010, 49(03): 440-444.
MAO Y, JIANG Q F, ZENG H S,etal. Genetic diversity ofPseudosciaenacrocea(Richardson)based on mitochondrial DNA control region sequences[J]. Journal of Xiamen University (Natural Science), 2010, 49(3): 440-444.
[4] 曹 艳. 基因线粒体控制区序列的中国沿海3种鲷科鱼类遗传多样性分析[D]. 广州: 暨南大学, 2016: 22-60.
CAO Y. Genetic diversity of 3 sparid species in coastal waters of China based on mtDNA control region sequences[D]. Guangzhou: Jinnan University, 2016: 22-60.
[5] ClEARWATER S J, PANHURST N W. Reproductive biology and endocrinology of female red gurnard, Chelidonichthys kumu (Lesson and Garnot) (Family Triglidae), from the Hauraki Gulf, New Zealand[J]. Marine & Freshwater Research, 1994(45):131-139.
[6] 隋昊志, 薛 莹, 任一平, 等. 海州湾鱼类生态类群的研究[J]. 中国海洋大学学报, 2017, 47(12): 59-71.
SUI H Z, XUE Y, REN Y P,etal. Studies on the ecological groups of fish communities in Hai Zhou Bay, China[J]. Periodical of Ocean University of China, 2017, 47(12): 59-71.
[7] 张迎秋. 长江口近海鱼类群落环境影响分析[D]. 青岛: 中国科学院研究生院(海洋研究所), 2012.
ZHANG Y Q. Environmental impact on the fish assemblage structure in adjacent sea area of the Yangtze River Estuary[D]. Qingdao: Graduate University of Chinese Academy of Sciences (Institute of Oceanology), 2012.
[8] 庄 平, 王幼槐, 李圣法, 等. 长江口鱼类[M]. 上海: 上海科学技术出版社, 2016.
ZHUANG P, WANG Y H, LI S F,etal. Fishes of the Yangtze Estuary[M]. Shanghai: Shanghai Scientific and Technical Publishers, 2016.
[9] CANINO M F, SPIES I B, LOWE S A,etal. Highly discordant nuclear and mitochondrial DNA diversity in Atka Mackerel[J]. Marine and Coastal Fishers: Dynamics, Management, and Ecosystem Science, 2010(2): 375-387.
[10] HOCHACHKA P W, MOMMSEN T. Biochemistry and molecular biology of fishes: Environmental and ecological biochemistry[J]. Science, 1993(2): 1-38.
[11] 乐小亮, 章 群, 赵 爽, 等. 一种高速快效的鱼类标本基因组DNA提取方法[J]. 生物技术通报, 2010(2): 202-204.
YUE X L , ZHANG Q, ZHAO S,etal. A fast and efficient method for isolation of genomic DNA from fish specimens[J]. Biotechnology Bulletin, 2010(2): 202-204.
[12] 孙海平. 中国近海区域海洋[M]. 北京: 海洋出版社, 2006: 97-98.
SUN H P. China offshore area ocean[M]. Beijing: China Ocean Press, 2006: 97-98.
[13] TAMURA K, CTECHER G, PETERSON D,etal. MEGA 6:Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729.
[14] LIBRADO P, ROZAS J. DnaSP v5 a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452.
[15] BANDELT H J, FORSTER P, ROHL A. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biolgy and Evolution, 1999, 16(1): 37-48.
[16] EXCOFFIER L, LISCHER H E L. Arelquin suite ver 3.5:a new series of program to perform population genetic analyses under Linux and Windows[J]. Molecular Biolgy Resources, 2010, 10(3): 564-567.
[17] NEI M, TAJIMA F. DNA polymorphism detectable by restriction endonucleases[J]. Genetics, 1981, 97(1): 145-163.
[18] KNIGHT A, MINDELL D P. Substations bias, weighting of DNA sequence evolution, and the phylogenetic positions offea’sviper[J]. Systematic Biology, 1993, 42(1): 18-31.
[19] FU Y X. Statistical tests of neutrality of mutation against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915-925.
[20] ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biolgy and Evolution, 1992, 9(3): 552-569.
[21] LIU J X, GAO T X, YOKOGAWA K,etal. Differential population structuring and demographic history of two closely related fish species, Japanese sea bass (Lateolabraxjaponicus) and spotted sea bass (Lateolabraxmaculatus) in Northwestern Pacific[J]. Molecular Phylogenetics and Evolution, 2006,39(3):799-811.
[22] GRANT W, BOWEN B. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Heredity, 1998, 89(5): 415-426.
[23] BOWEN B W, BASS A L, ROCHA L A,etal. Phylogeography of the trumpetfishes (Aulostomus): ring species complex on a global scale[J]. Evolution, 2001, 55(5): 1029-1039.
[24] 吕金磊, 章 群, 杨喜书, 等. 基于线粒体控制区的中国南海海域卵形鲳鲹遗传多样性[J]. 海洋渔业, 2017, 39(2): 241-248.
LV J L, ZHANG Q, YANG X S,etal. Genetic diversity ofTrachinotusovatusin the South China Sea inferred from mitochondrial DNA control region sequences[J]. Marine Fisheries, 2017, 39(2): 241-248.
[25] SONG N, ZHANG X M, SUN X F,etal. Population genetic structure and larval dispersal potential of spottedtail gobySynechogobiusommaturusin the north-west Pacific[J]. Journal of Fish Biology, 2010, 77(2): 388-402.
[26] 赵 峰, 庄 平, 章龙珍, 等. 基于线粒体Cytb基因的黄海南部和东海银鲳群体遗传结构分析[J]. 水生生物学报, 2011(35): 745-752.
ZHAO F, ZHUANG P, ZHANG L Z,etal. Population genetic structure ofPampusargenteusin the south Yellow Sea and East China Sea based on the mitochondrialCytbsequence[J]. Acta Hydrobiologica Sinica, 2011(35): 745-752.
[27] 曹 艳, 章 群, 宫亚运, 等. 基于线粒体COI序列的中国沿海蓝点马鲛遗传多样性[J]. 海洋渔业, 2015, 37(6): 485-493.
CAO Y, ZHANG Q, GONG Y Y,etal. Genetic variation ofScomberomorusniphoniusin the coastal waters of China based on mtDNACOIsequences[J]. Marine Fisheries, 2015, 37(6): 485-493.

