棉花重组自交系铃重性状的QTL定位
曲朝阳,贾晓昀,马启峰,王寒涛,魏恒玲,范术丽*
(1.中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南 安阳455000;2.河北省农林科学院粮油作物研究所,石家庄050035)
铃重是构成棉花产量的基本因子之一[1]。在稳定衣分和单位面积总铃数的基础上,若能稳步提高铃重,可进一步提高棉花产量[2]。
铃重既受品种遗传控制,也受栽培技术、环境条件等因素的影响。仅依靠传统育种定向提高铃重,不但具有一定的盲目性而且育种周期较长[3],严重制约选育高铃重材料的进程。近年来,随着生物技术的快速发展,分子标记辅助选择技术在作物育种上的应用研究也日益深入[4-6]。开发与铃重相关的分子标记需要寻找有效的数量性状位点(Quantitative trait loci,QTL)。李成奇等[7]利用F2:3群体定位到2个铃重QTL,分布于A10和A12染色体,解释的表型变异率为6.1%~7%。王婷[8]利用F2群体和F2:3群体分别定位到4个和1个铃重QTL,解释的表型变异率为2.7%~10.6%;Wu等[9]利用重组自交系(Recombinant inbred lines,RILs)群体定位到3个铃重QTL,解释的表型变异率为7.3%~11.7%;Wang等[10]利用180个家系组成的RILS群体定位到5个铃重QTL,解释的表型变异率为4.83%~9.66%;Zhang等[11]利用115个家系组成的BC3F2回交群体,在4个环境下共定位到51个铃重QTL,解释的表型变异率为1.02%~13.23%;Fan等[12]利用143个家系组成的RILS群体,在3个环境下共定位到10个铃重QTL,解释的表型变异率为0.97%~19.77%;Yu等[13]利用146个家系组成的BC2F4回交群体,在4个环境下共定位到10个铃重QTL,解释的表型变异率为8.48%~25.27%;Zhang等[14]利用196个家系组成的RILS群体,在11个环境下共定位到146个铃重QTL,分布于25条染色体,最高可解释的表型变异率为16.70%。这些QTL为分析铃重遗传机理提供了依据。同时,利用与铃重QTL紧密连锁的遗传标记,并结合田间表型数据进行分子标记辅助选择育种,能有效减少连锁累赘,提高对铃重选择的准确性,这对提高棉花产量育种的效率意义重大。
本研究以分辨率高、重复性好的RILs为作图群体,构建以SNP(Single nucleotide polymorphisms)为主要标记的遗传连锁图谱,结合田间多年的铃重数据,检测与铃重相关的主效QTL,以期为棉花高产育种提供一定的理论依据。
1 材料和方法
1.1 亲本及作图群体
本研究以陆地棉品种中棉所36为母本、海岛棉染色体片段渐渗系材料G2005为父本配制杂交组合,2006年于河南安阳(AY)经去雄、授粉,收获F1种子,当年冬天于海南加代、自交;2007年于安阳种植F2,通过连续多代自交,于2010年构建成包含137个株系的F2:9RILs群体[15]。2011―2015年连续5年在安阳种植,采用完全随机区组设计,设3次重复,单行区,行长4 m,行距0.8 m,株距20 cm,田间种植均按照农场标准化管理。
1.2 遗传连锁图谱的构建
采集双亲及137个株系幼嫩叶片,采用CTAB(Hexadecyl trimethyl ammonium bromide)法[16]提取DNA,送至深圳华大基因,采用RAD-seq(Restriction-site associated DNA sequencing)测序技术对亲本及群体进行100 bp的双末端测序[17-18];筛选亲本间的SNP,结合杨继龙[15]筛选的SSR标记,采用HighMap策略进行遗传图谱构建[19]。
1.3 数据分析及QTL定位
吐絮盛期,在取样行采摘50个正常吐絮的中上部棉铃,晒干称重,3个重复的平均值为当年铃重数据。用Excel 2007对群体的铃重进行基本统计分析,包括均值、最大值、最小值、方差、变异系数、峰度、偏度等。利用WinQTLCart 2.5软件和复合区间作图法[20]进行QTL定位及多环境均值联合分析,窗口大小为5 cM,步长为1 cM,背景标记为10个,LOD值为2.5。以下模式为QTL命名的标准[21]:q+性状名称英文缩写+染色体序号+QTL编号。如qBW-A3-1表示位于A3染色体的第1个控制铃重的QTL。
2 结果与分析
2.1 亲本及群体性状的表型分析
5个环境下分别选取亲本及RILs群体株系的优质棉铃,称重并统计。结果表明5个环境下两亲本的铃重差异均显著或极显著(表1),为铃重QTL的分子标记筛选提供较好的遗传基础。不同年份群体平均值表现不同的分离模式,高于或低于低值亲本。5个环境下,137个株系中所获得的铃重数据有较大波动,除2014年安阳铃重的峰度外,偏度及峰度的绝对值都小于1,基本符合正态分布(图1),这表明铃重受微效多基因控制,符合数量性状遗传模型,可以用来进行QTL定位研究。在RILs群体中,铃重表现明显的双向超亲分离现象,最大值均高于高值亲本,说明分离群体中含有高铃重材料,可以通过聚合育种或分子标记辅助选择育种选育高铃重品种。
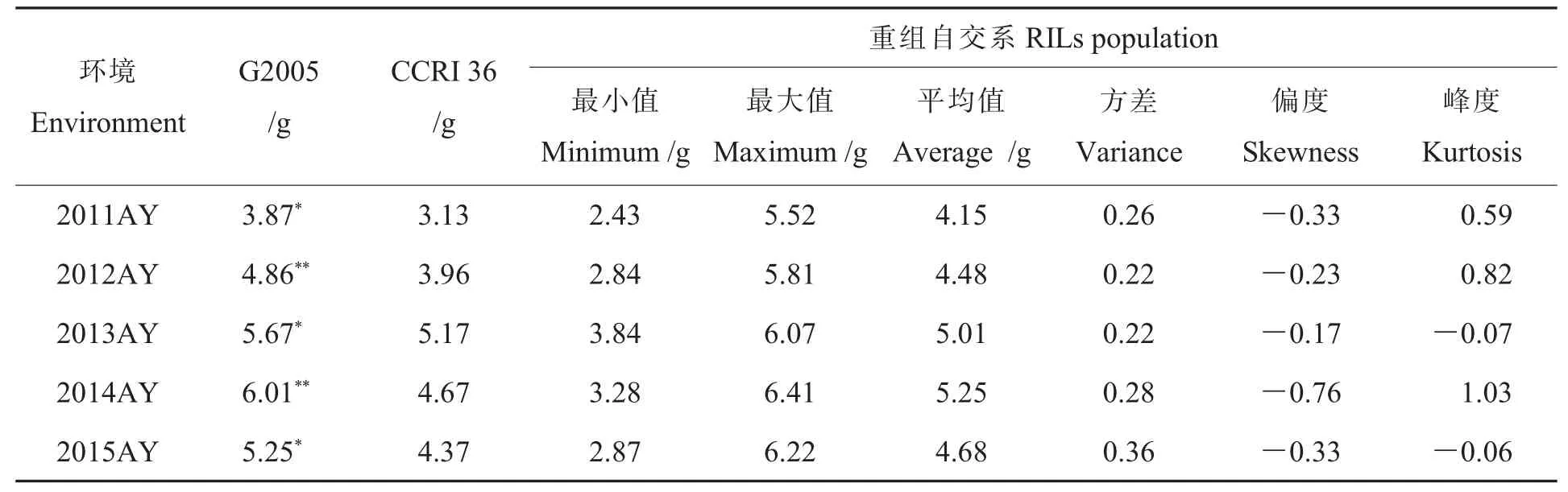
表1 5个环境下重组自交系群体及亲本的铃重表现Table 1 Boll weight of RILs population and their parents in five environments
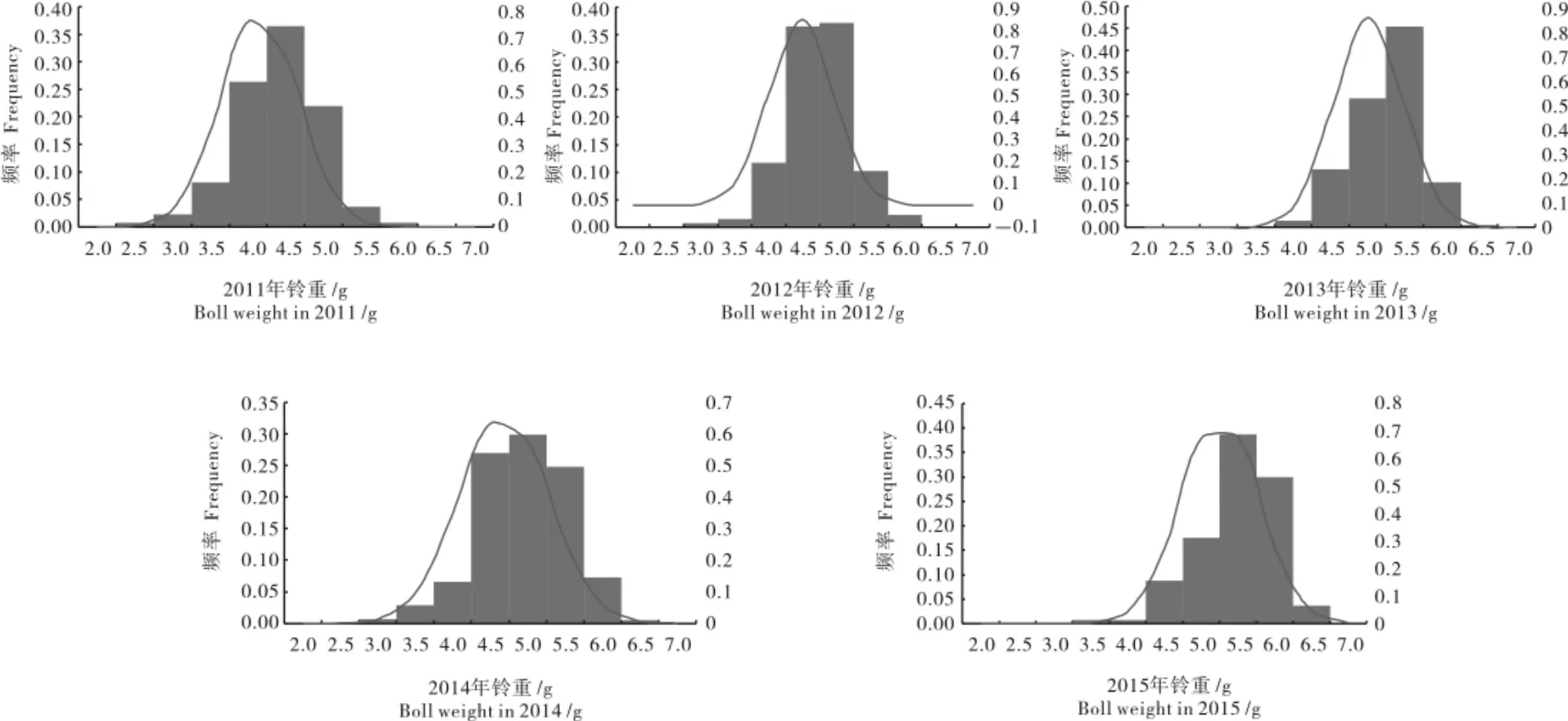
图1 2011―2015年RILs群体铃重分布图Fig.1 Distribution of boll weight of RILs population in five environments
2.2 遗传图谱构建
亲本中棉所36和G2005测序深度分别为6.43×和10.57×,Q30值分别达到89.75%和90.95%;重组自交系群体平均每个株系测序深度约4.59×,Q30值为86.38%。通过与陆地棉参考基因组比对[22],共得到369 223个SNP,其中亲本间的多态性SNP有78 127个,多态性比例达到21.16%[23-24]。结合多态性SSR(Simple sequence repeat)标记[15],构建1张包含6 434个位点的高密度遗传图谱(表2),图谱中包含6 295个SNP标记和139个SSR标记,总遗传图距为4 071.98 cM,标记间平均距离为0.63 cM,标记间最大距离为18.24 cM,是目前陆地棉种内标记密度最大的图谱之一[25-26]。
2.3 铃重性状的QTL定位
5个环境共检测到32铃重QTL(表3,图2)。其中,能在不同年份被检测到的有5个,分别为qBW-A4-1、qBW-A4-2、qBW-A5-2、qBW-D9-1和qBW-D9-2。qBW-A4-1在2013年和2015年均被检测到,加性效应值为-0.14~-0.15,解释的表型变异率为5.07%~9.79%。qBW-A5-2在2011年和2012年均被检测到,加性效应值为-0.12~-0.15,解释的表型变异率为6.32%~7.73 %;qBW-A4-2在2011年和2012年均被检测到,加性效应值为-0.20~-0.29,解释的表型变异率均达到15.8%,增效基因都来自高值亲本G2005,可能为多环境下较为稳定的主效QTL;qBW-D9-1和qBW-D9-2在2013年和2014年均被检测到,加性效应值分别为-0.13~-0.14和-0.13,解释的表型变异率依次为6.73%~7.35%和5.52%~6.78%。多数检测到的QTL为主效QTL,说明铃重可能受主基因和微效多基因共同控制。在所检测的QTL中,26个来自父本G2005,占81.3%;6个来自母本中棉所36,占18.7%。由于高代RILs群体后代趋于纯合,显性及与显性相关的上位性随着同质性的增加逐渐消失,遗传效应主要表现为加性效应[27]。

表2 遗传图谱详细信息统计Table 2 Detail information of the genetic map
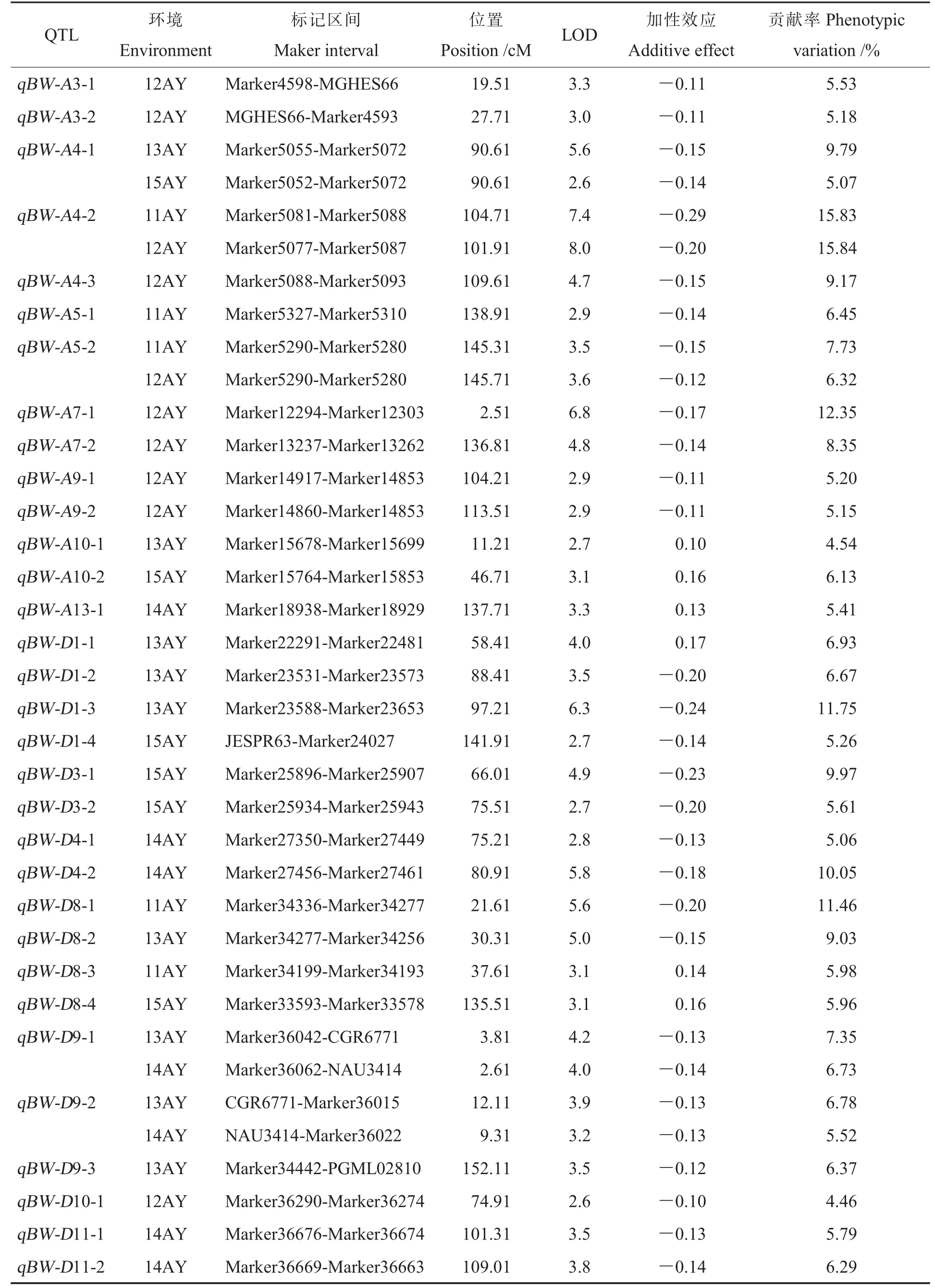
表3 5个环境中检测到的铃重QTLsTable 2 Detection of QTLs associated with boll weight in five different environments
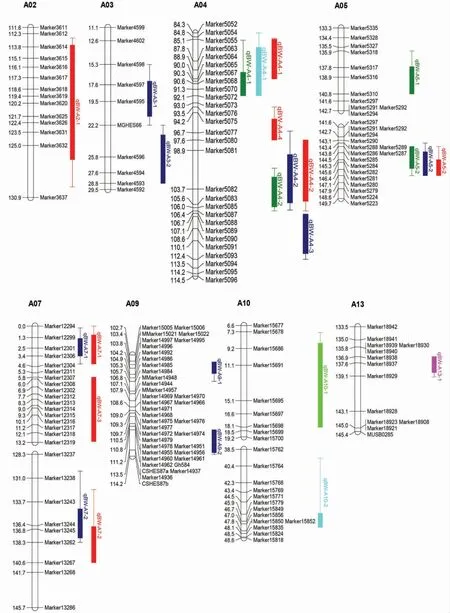
图2 铃重QTL定位结果Fig.2 The results of QTL mapping for boll weigh

图2 (续)Fig.2(Continued)
利用5个环境下的数据平均值进行分析,共检测到9个铃重相关QTL(表4,图2)。qBW-A4-1、qBW-A4-2、qBW-A5-2加性效应值分别为-0.12、-0.14和-0.09,解释的表型变异率分别为9.27%、11.92%和5.04%。这3个QTL在单环境联合分析中也被检测到,说明这些位点稳定性较好。qBW-A7-1和qBW-A7-2在单环境联合分析中也被检测到,加性效应值分别为-0.10和-0.13,解释的表型变异率分别为5.08%和9.62%。在A4和A7两条染色体上各检测到3个QTL,推测这两条染色体与铃重关系较为密切。
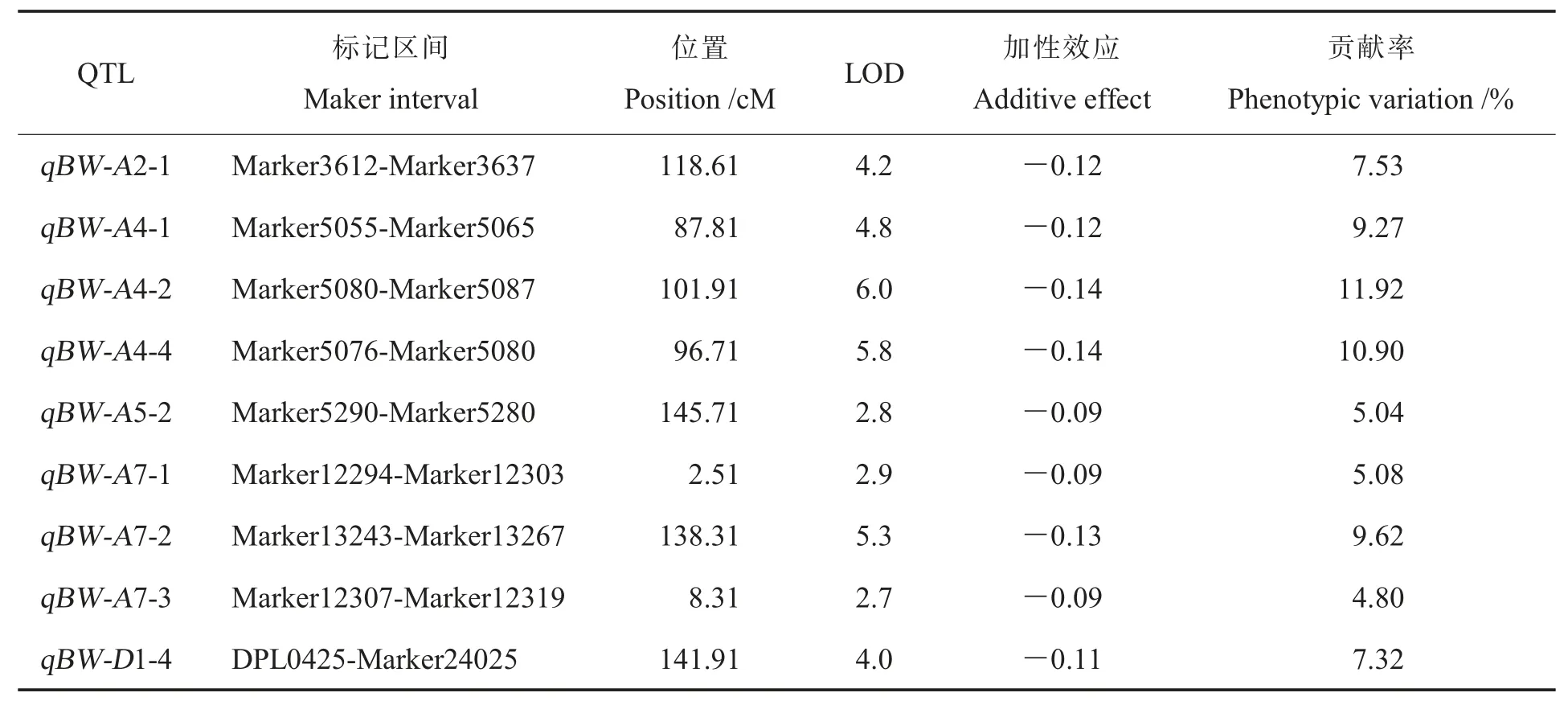
表4 利用5个环境中铃重数据平均值检测到的QTLsTable 4 Detection of QTLs associated with boll weight basing on averages of the five environments
3 讨论
3.1 海陆杂交的RILs群体适合QTL定位
由于陆陆杂交群体遗传背景单一,同质性强,后代遗传变异范围较小[28],因此该研究采用陆地棉中棉所36和海岛棉渐渗系G2005构建RILs群体,从而增加了群体的多态性。每个家系由F2个体经过多年连续自交得到,家系间基因型各异,群体内铃重数据近似正态分布,符合数量性状遗传的特点。RILs群体重组程度高于F2群体,其遗传图谱比F2群体有着更高的解析度,提高了作图的精确度。每个家系基因型纯合稳定,可在多个环境下进行表型鉴定,这些对获得稳定准确的QTL意义重大。
3.2 SNP标记构建的遗传图谱利于QTL定位
陆地棉基因组大,遗传背景复杂,重复序列多,标记开发较困难。SNP标记是基因组中数量最多、分布最均匀、多态性最高的分子标记[29];相对于SSR标记,它的遗传稳定性更好,易实现分析自动化,是构建遗传图谱理想的分子标记[30]。随着测序成本的不断降低,SNP标记将在分子标记辅助选择育种中发挥更大的作用。本研究总共得到369 223个SNP,亲本间具有多态性的有78 127个,多态性标记的比例达到21.16%,远大于常规SSR标记的多态性标记比例。利用SNP数据和SSR标记相结合构建的遗传图谱,不仅提高了图谱的密度和覆盖度,也间接比较了图谱的准确性[31-32],因此定位的QTL结果更可信。中棉所36和G2005的测序深度分别为6.43×和10.57×,可能是两者在遗传背景上存在部分差异。2个材料的生育期、开花期、吐絮期、果枝始节等性状也存在差异[23],进一步说明两者基因组成有一定差异。RAD-seq是1种对酶切产生的基因组标签序列进行高通量测序的技术,测序深度相差较大可能是由于不同材料基因组酶切位点分布及数量不同,但是本研究中的测序深度可以满足后续的分析。
3.3 环境对铃重的影响
铃重属于数量性状,易受外界环境和栽培条件的影响,5年间的表现略有差异。2011、2013、和2014年铃重波动较大,主要原因是:2011年安阳气候条件总体是前期旱,中后期雨涝。具体表现为7月中旬之前干旱无雨,棉铃发育受影响;8~9月份阴雨天较多,对棉铃的充实和吐絮极为不利。2013年和2014年,安阳的气候条件总体是前中期雨水多,后期天气晴好。具体表现为7月中旬到8月初阴雨较多,不利于成铃;9月份秋高气爽,气温较高,有利于上部棉铃的充实,虽然单株铃数较少,但铃重较高。
3.4 增效基因来源于双亲
中棉所36和G2005均含有与铃重相关的QTL增效位点,说明增效基因并非总是来源于高值亲本[33],低值亲本对铃重提高也有一定贡献。增效基因来自低值亲本的原因如下:高值亲本中含有激活有利基因的调控基因,杂交后代获得其调控基因后可激活低值亲本有利基因的表达;由于隐蔽效应导致增效基因在低值亲本中不表达,当发生重组时才被检测到;两者相互作用的结果[34]。
3.5 QTL数据库比对
将棉花QTL数据库(http://www.cottonqtldb.org)铃重相关的QTL转化成对应染色体的物理图距[35-36],并与本研究结果进行比对,结果发现13个QTL位点(qBW-A4-1、qBW-A4-2、qBW-A4-3、qBW-A4-4、qBW-A5-1、qBW-A5-2、qBW-A9-1、qBW-A9-2、qBW-A10-2、qBW-A13-1、qBW-D4-1、qBW-D4-2、qBW-D8-2)与前人总结的结果相一致,说明这些位点对铃重有一定的影响。qBW-A4-1、qBW-A4-2和qBW-A5-2在本研究的多个环境下被检测到,且贡献率较大,最大达15.83%,可能是较为稳定的QTL。qBW-D4-1和qBW-D8-2在数据库中分别对应的QTLBW11.g和qBW-07A-c24-1,贡献率分别为38.8%和13.54%,为 主 效QTL。所 以qBW-A4-1、qBW-A4-2、qBW-A5-2、qBW-D4-1和qBW-D8-2这5个QTL可为铃重相关候选基因的筛选和验证提供参考。
4 结论
本研究构建的遗传图谱丰富了棉花遗传图谱 信 息。qBW-A4-1、qBW-A4-2、qBW-A5-2、qBW-D4-1和qBW-D8-2为分析铃重遗传机理提了依据。

