端粒酶与体细胞重编程的最新研究进展
贺小英 荆乾鸽 姜欣颖 吴宪 兰宗宝 马利兵
摘要:端粒酶与体细胞重编程是当今生物界最热门的研究领域之一。研究端粒酶能揭示生物体胚胎早期发育、衰老和癌症发生的机制;细胞重编程则有望从根本上解决体细胞克隆技术问题,从而开启生命再造新时代。文章通过对端粒酶与体细胞重编程及二者互作机制的最新研究进展进行详细阐述,发现细胞重编程面临的主要问题是效率低下,其根本原因是对其作用机制尚未完全了解;而最新研究证实,生殖细胞的端粒酶活性发生时序性变化可能在胚胎发育及细胞重编程过程中发挥重要调控作用。因此,揭示细胞重编程机制是今后研究工作的重心,尤其是揭示端粒酶在胚胎发育过程的分子调控机理及端粒酶调控细胞重编程可能开启的信号通路,对提高细胞重编程效率,推进克隆动物的生产实践并开启生命再造,以及为治疗癌症等人类重大疾病具有重要意义。
关键词: 端粒酶;体细胞重编程;表观修饰;调控作用;胚胎发育
中图分类号: S814.8 文献标志码: A 文章编号:2095-1191(2019)05-1133-08
0 引言
端粒酶(Telomerase)是一种核糖核酸蛋白复合体,由端粒酶RNA(TER)、端粒酶催化亚基(TERT)及相关结合蛋白组成。其中,TERT是端粒酶蛋白组分中的催化亚单位,能直接刺激体外细胞增殖并延长细胞寿命(Blasco,2005)。端粒(Telomeres)是由简单的DNA高度重复序列组成,且沿5'到3'方向富含GT,其中人类端粒由大量重复的TTAGGG组成。人们通常认为,端粒酶有延长端粒、保护端粒DNA完整性的功能,但越来越多研究证实其功能更强大和丰富。Park等(2009)研究表明,端粒酶通过与染色体重建蛋白BRG1相互作用而直接激活Wnt通路。Tsai等(2013)研究发现,过表达端粒酶活性的神经母细胞瘤经RNA干扰手段灭活端粒酶后,细胞内的DNA出现浓缩及细胞周期抑制现象。近十年来,关于端粒酶又有新的分子功能发现,即端粒酶调控细胞重编程。Zvereva等(2010)研究发现,人类TERT能进行细胞的转录调控和代谢重编程;Kinoshita等(2014)研究证实,当灭活供体细胞端粒酶活性时,许多与转录和表观修饰相关的基因表达均发生一定程度的上调或下调。可见,端粒酶在体细胞重编程过程中发挥着极其重要的作用。關于端粒,曾认为其只是不编码的一个简单重复序列(TTAGGG)n,但现在越来越多研究认为端粒除了维持染色体的结构完整和功能稳定外,还在调控细胞生长、发育分化乃至细胞重编程等一系列生命活动中扮演着重要角色(Mason et al.,2011)。《Science》在2007年刊登了进一步的重大发现:瑞士实验癌症研究所的Joachim Linger及其同事发现端粒能充当RNA的合成模板,产生许多RNA分子(Azzalin et al.,2007),但至今尚未明确这些合成RNA的具体作用。Wang等(2012)研究报道,与生殖细胞相比,体细胞端粒异质性极可能是引起核重编程不彻底的关键因素,最终导致核移植效率低下或失败。端粒维持生殖细胞染色体的完整性,而端粒酶通过延长端粒使其在胚胎发育中实现完全重编程,与此不同的是,大多克隆动物重编程效率低下,其主要原因是端粒并未被完全重编程,即端粒酶和端粒可能是影响重编程效率的关键因素。为此,本文就端粒酶与体细胞重编程及二者互作机制的最新研究进展进行详细阐述,为今后开展克隆动物生产及再生医学等领域的相关研究提供参考。
1 端粒酶的调控作用
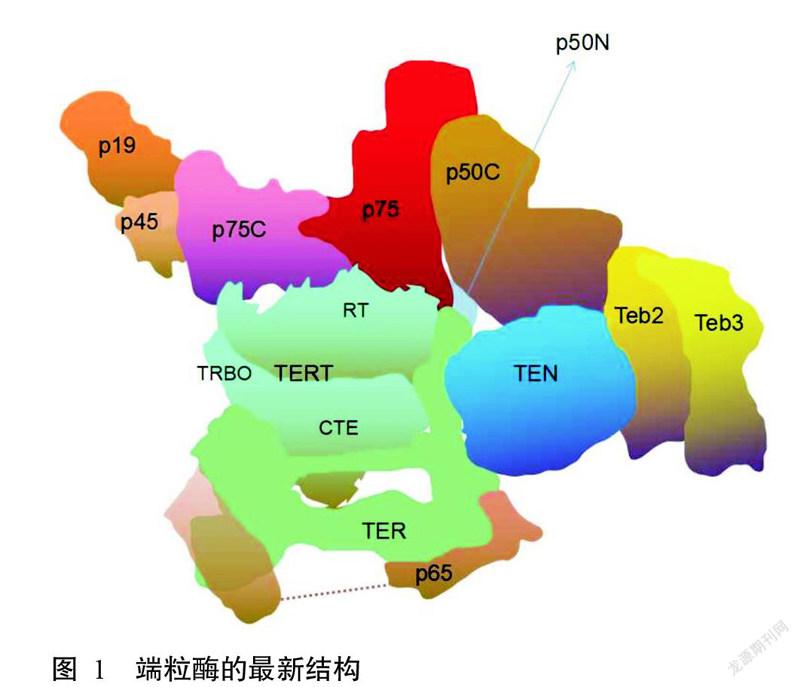
端粒酶是一种特殊的DNA逆转录酶。美国加州大学伯克利分校的研究人员对端粒酶结构进行了更精细的绘制(Jiang et al.,2015,2018),根据结构图(图1)发现了两个新蛋白Teb2和Teb3,二者与单链端粒DNA结合蛋白Teb1形成复合物(TEB),以增加端粒酶的活性。这两个新蛋白的发现更正了人们认为端粒酶由7个蛋白亚基和1个RNA链组成的观点。此外,端粒酶的催化核心由RNA链及其互作蛋白TERT和p65组成。其中,RNA链形成一个环裹住TERT蛋白;端粒酶3个结构蛋白p75、p45和p19较保守,四膜虫与人类的结构类似,且关键蛋白p50与TERT、Teb1和p75协同互作参与端粒酶的功能(Upton et al.,2017)。TER作为RNA模板,具有模板的核心和TER活化结构域(CR4/5)两部分结构域,但其激活端粒酶的机制尚不清楚。最新研究发现,全酶蛋白TCAB1能控制CR4/5的构象,从而调控端粒酶复合体的组装及其活性功能(Chen et al.,2018)。在人类胚胎早期发育阶段,端粒酶在多种组织中具有活性,尤其在体外培养的绝大多数癌细胞和永生化细胞中均能检测到端粒酶活性,但在成体细胞中缺乏端粒酶活性。
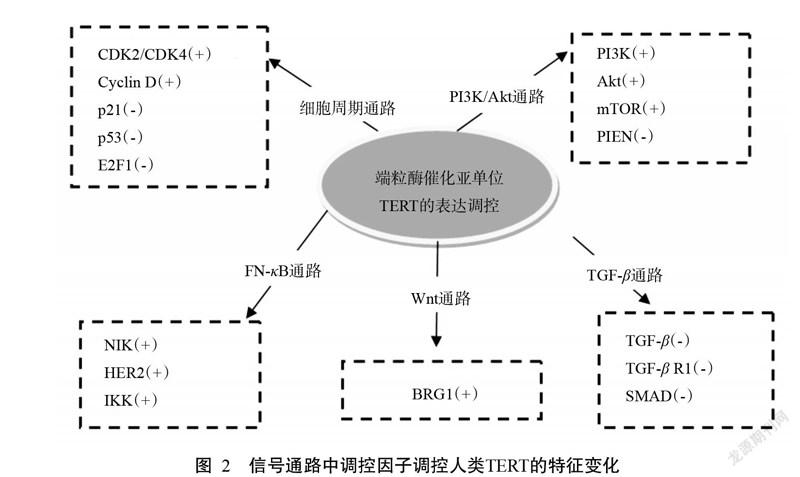
人类TERT的表达调控在胚胎早期发育过程中发挥重要作用,主要发生在转录水平上,且其表达量的调控对许多转录因子结合位点均有影响,如锌指转录因子Sp1、癌基因(c-Myc)和抑癌基因(p53)在调控端粒酶活性中发挥重要作用。Sp1结合位点的任何突变均能显著降低人类TERT启动子转录表达(Su et al.,2018),通常Sp1需要与c-Myc协同作用才能激活端粒酶的表达(Zheng et al.,2018),而端粒酶活性的时序性调控不断维持早期胚胎染色体的完整性和稳定性。此外,端粒酶在胚胎发育过程中扮演着除延长端粒长度之外的其他重要调控作用。Lewinska等(2017)也研究证明,敲除人类TERT可导致p53转录升高,进而减少细胞增殖,进一步说明p53在人类TERT表达过程中发挥负反馈调控作用。近几年的研究发现,TERT可直接与转录因子结合,以辅助激活子的形式调控多个重要信号通路的下游靶基因表达(图2)。信号通路TGF-β在胚胎发育和器官形成过程中发挥重要作用,其中,活化蛋白1(AP-1)能抑制人类TERT的转录,而AP1、TGF-β R1和SMAD3能抑制TGF-β活性途径(Cherlet and Murphy,2007;Peek and Tollefsbol,2016;Yang et al.,2019)。也有研究认为,ERα与上游人类TERT调控区结合而激活雌激素,是通过抑制TGF-β受体或AP1干扰TGF-β途径来实现(Peek and Tollefsbol,2016)。Yano等(2016)、Lewinska等(2017)、Qin等(2018)研究报道,p21、p53和E2F1能抑制人类TERT活性,CDK2/CDK4和Cyclin D却激活人类TERT表达而影响细胞增殖,从而改变细胞周期信号途径。PI3K/Akt与TERT间存在反馈调节,TERT表达可激活PI3K/Akt信号通路,而PI3K/Akt信号通路又可通过多种机制反作用增加TERT表达。PI3K/Akt和MAP激酶通路上调人类TERT转录是通过MAD1磷酸化,导致泛素介导的蛋白水解(Hein et al.,2011)。PI3K/Akt的通路作用可能导致人类TERT失调,最终导致细胞增殖甚至发生永生化(Peek and Tollefsbol,2016)。现有研究证实,在PI3k/Akt信号通路中,转录调控因子PI3K、Akt和mTOR参与正调控TERT(Blasco,2005;Hein et al.,2011;Dogan and Biray Avci,2018),而PTEN参与负调控TERT(Yang et al.,2019)。NF-κB和c-Myc诱导的人表皮生长因子受体2(HER2)在照射后能激活人类TERT的NF-κB通路,进而破坏细胞周期调控进程(Tilborghs et al.,2017)。此外,炎症因子IKK通过激活端粒酶的活性而调控NF-κB通路,从而刺激细胞增殖(An et al.,2016)。最新研究发现,表皮生长因子受体(EGFR)可上调TERT的表达从而触发MEK信号通路(Lin et al.,2018)。
2 端粒酶与体细胞重编程的互作机制
2. 1 体细胞重编程研究进展
细胞重编程是抹去基因组中原本存在的体细胞或生殖细胞类型特异的表观遗传修饰模式,建立起具有胚胎干细胞特异的表观遗传修饰模式,主要包括DNA甲基化修饰、组蛋白修饰、印记基因修饰及X染色体失活等表观遗传修饰的改变。
2. 1. 1 DNA甲基化修饰 当前的研究表明,克隆胚和克隆动物存在异常的表观遗传修饰,如较高的DNA甲基化和较低的组蛋白乙酰化水平(Silveira et al.,2018)。在克隆牛早期胚胎中可观察到不完全的去甲基化现象(Bonk et al.,2008),而这种不完全的修饰会导致个体发育异常,如大型胎儿综合症等(Giraldo et al.,2008)。Kremenskoy等(2006)通过调查克隆牛胎儿全基因组范围内的DNA甲基化模式,发现克隆牛胎儿的脑部和胎盘存在异常DNA甲基化。Yang等(2007)研究显示,正常牛胚胎重新甲基化发生在8-细胞期到16-细胞期,但克隆牛胚胎在4-细胞期提前发生重新甲基化,且滋养层细胞存在超甲基化的异常修饰。还有研究认为,甲基化程度较低的体细胞克隆胚重编程效率相对较高(Blelloch et al.,2006)。因此,克隆动物发育异常及克隆动物失败均会发生DNA甲基修饰的异常,而选择易于重编程的供体细胞类型至关重要(Solter,2000)。此外,Sahakyan等(2017)通过检测克隆和非克隆公牛精子发育调节基因(PEG3、XIST、OCT4和NANOG)的甲基化状态,发现二者并无明显差异,且囊胚中的甲基化状态也相似。可见,克隆公牛的精子在其发生过程中需要充分重新编程,才能产生表观遗传正常的胚胎,提示端粒酶在精子细胞重编程过程可能发挥重要作用。
2. 1. 2 组蛋白修饰 组蛋白修饰在细胞重编程过程中也发挥着重要作用,是由于组蛋白单一位点的修饰即可改变染色体构象,进而调控基因表达(Marx,2006;Park et al.,2018)。目前的研究表明,与体外受精(IVF)胚胎相比,核移植胚胎的組蛋白修饰存在不同程度的变异,从牛核移植胚胎原核期到8-细胞期的H3K9、H3K18、H4K5和H4K8乙酰化水平均显著高于IVF胚胎,且在两种胚胎中H3K9和H3K18的乙酰化均呈动态变化:在8-细胞期减弱,在囊胚和桑椹胚期又明显增强(Wu et al.,2011)。此外,卵子对供体细胞不完全重编程可能也与组蛋白修饰异常有关。Marión等(2011)研究发现,体细胞的端粒DNA大部分呈甲基化修饰状态,且富含组蛋白H3第9位赖氨酸三甲基化(H3K9me3)和H4第20位赖氨酸三甲基赖氨酸(H4K20me3)修饰,凡是能诱导成为iPS细胞的体细胞同样伴随着端粒重编程及其组蛋白的去甲基化修饰。
2. 1. 3 印记基因修饰 印记基因是指仅来自亲本一方的等位基因表达,印记控制区(Imprinting control region,ICR)调控主要通过DNA甲基化的方式来实现。由于基因印记相关疾病的症状与体细胞克隆动物发育异常出现的症状相似,故推测重编程过程中的印记紊乱是导致体细胞克隆动物发育异常的原因之一(Shen et al.,2012)。有关发育异常和死亡的体细胞克隆动物研究发现,印记基因的表达和甲基化模式异常在体细胞克隆动物各组织上均有存在。Deng等(2017)研究发现,死亡的克隆羊与存活的克隆羊相比,前者各组织中H19基因甲基化程度更高,但二者在脑组织中的差异不显著。Curchoe等(2009)发现体细胞克隆牛两个印记基因(H19和IGF2)间的区域表现出异常超低甲基化修饰。此外,有研究发现印记基因XIST可能与雌性克隆牛的存活率有关,IGF2基因可能参与雄性克隆牛的存活率,也就是说印记基因在克隆动物性别发育过程中发挥重要作用(Ruan et al.,2018)。
2. 1. 4 X染色体失活 X染色体失活一般发生在哺乳动物的早期发育阶段。X染色体失活是为了实现具有两个X染色体的哺乳动物雌性和具有单个X染色体的雄性与性别决定性Y染色体间的剂量等效(Sahakyan et al.,2017);在体细胞克隆过程中重编程不完全也会引起X染色体失活的异常。正常情况下,XIST基因仅作用于失活的X染色体(Inactive X chromosome,Xi),另一条有活性的X染色体(Active X chromosome,Xa)则不受其影响。已有研究表明,在雄性体外受精囊胚期的胚胎中检测不到XIST基因mRNA,但在体细胞囊胚期发现有XIST基因 mRNA(Inoue et al.,2010)。在失活起始阶段,X染色体被XIST基因包裹后,其组蛋白抑制性修饰如H3K27me3大量增加,而活性修饰如H3K4的甲基化被去除,在Xa染色体上则恰好相反,即H3K27me3相对较低而H3K4甲基化水平相对较高(Sado and Brockdorff,2013)。
2. 2 端粒酶调控体细胞重编程
2. 2. 1 端粒酶调控端粒重编程 端粒酶的首要生物学功能是维持端粒长度。体细胞中缺乏端粒酶的活性,端粒长度则随DNA复制逐渐缩短;在生殖细胞中,同一精子产生的不同细胞端粒长度存在明显差别。Achi等(2000)通过对小鼠未成熟精原细胞、不同阶段生精细胞及成熟具有受精能力的精子进行端粒长度检测,结果发现未成熟精原细胞的端粒较其他生精细胞的短,且在生精细胞到精子成熟的过程中端粒长度不断增加。由于存在末端复制问题,尽管体细胞端粒长度随年龄的增加而缩短,但精子端粒长度因端粒酶活性的存在保持不变,其目的是为了保护精子染色体遗传物质的完整性,最终实现亲代精子在胚胎发育中端粒的完全重编程(Yatsenko et al.,2012)。关于克隆动物中端粒的重编程问题,Shiels等(1999)认为克隆羊多莉的端粒长度并未完全重编程,因此导致其表现出早衰现象;Betts和King(1999)在克隆牛的所有组织和细胞中也未发现端粒长度延长现象。但Lanza等(2000)研究得出完全不同的结论,即克隆牛的端粒长度显著长于其供体成纤维细胞;Dean等(2001)同样发现克隆猪的端粒发生了重编程现象。关于核移植后端粒是否发生重编程的报道各不相同,其原因可能是体细胞中端粒的长度变化是一个动态过程,且克隆动物的数量有限,不具有统计学意义。因此,很难从克隆前、后动物的体细胞探究端粒的长度变化规律。目前,对于端粒酶和端粒在体细胞重编程过程中的作用机制尚不清楚,但可以肯定的是在胚胎形成期通过端粒酶重新合成端粒DNA具有重要意义(Schaetzlein and Rudolph,2005)。Wang等(2012)研究报道,在诱导iPS细胞重编程过程中端粒酶的活性起关键调控作用,端粒长度发生动态变化,且这种动态变化可能在指导胚胎不断发育和分化。
端粒重编程发生不仅包括端粒的长度变化,还包括许多参与端粒结合及与端粒调控有关的蛋白变化。至今已发现的端粒相关蛋白有TRF1、TRF2、TP1、TERT、Tankyrase和POT1等,其中,TRF1和TRF2是两种端粒DNA结合蛋白(Telomere-specific DNA binding protein),且有学者认为TRF1可能是通过抑制端粒酶来调控端粒长度,TRF2则可能具有保护染色体末端的作用(Smogorzewska et al.,2000)。TP1是端粒酶蛋白组分中的亚单位,从构成上来看是一种多功能蛋白(Blasco,2005);Tankyrase是一种人类端粒相关蛋白,推测其是通过失活TRF1的途径来调控端粒酶活性;而人类端粒保护蛋白1(Human protection of telomeres 1,hPOT1)能与端粒单链重复序列TTAGGG特异性结合,并与其他端粒结合蛋白及端粒酶等共同完成端粒的保护和长度调节(Hockemeyer et al.,2006)。此外,Huang等(2012)研究发现,人类的Stn1蛋白在保护端粒完整性上发挥重要作用;Mersaoui和Wellinger(2019)研究发现,酵母端粒长度调控蛋白Cdc13特殊区域的突变会引起DNA分子破坏。因此,在端粒重编程过程中这些关键调控端粒的相关蛋白是否会发生变化,尚有待进一步探究。
2. 2. 2 端粒酶调控体细胞重编程 目前,针对端粒酶的研究主要集中在衰老和癌症方面,细胞重编程的研究也以表观修饰和调控为主,有关端粒酶和体细胞重编程互作方面的报道很少,但近期有研究表明端粒酶活性在细胞重编程过程中发挥着极其重要的作用。Marión等(2009a)研究认为,只有供体细胞中具有端粒酶活性才能有效诱导产生iPS细胞,此时供体细胞的端粒发生重编程,获得的iPS细胞与胚胎干细胞(ES细胞)特征一致。此后,Blasco及其研究团队在2009年的《Nature》杂志上报道,当供体细胞缺失端粒酶活性就会导致端粒很短或DNA发生损失,此时供体细胞会阻碍细胞重编程的发生(Marión et al.,2009b)。Huang等(2011)也研究证实,供体细胞端粒长度是否被重编程决定了ESCs/iPSCs最终能否进行多能性发育。可见,体细胞作为供核细胞时,其端粒与端粒酶活性在诱导iPS细胞重编程过程中发挥着重要作用,因此确定端粒酶在体细胞核移植后核重编程过程中同样具有极其重要的作用。Wang等(2012)研究表明,与生殖细胞相比,体细胞端粒的异质性极可能是引起核重编程不彻底的关键因素,最终导致核移植效率低下或失败。此外,有报道称端粒酶在体细胞核移植中除了延长端粒长度外,还发挥着其他作用。当供体细胞敲除TERT或突变TERT致使端粒酶失活后,在细胞重编程中发现有75个基因表达上调和1571个基因表达下调,而且这些基因大多数与转录和表观修饰相关(Kinoshita et al.,2014)。最新研究还发现关于TERT基因DNA甲基化在细胞重编程相关基因表达中的一个新功能:在人类诱导多能干细胞与亲代细胞的远端区域TERT启动子存在差异甲基化区域(DMR),在诱导多能干细胞TERT基因高度甲基化后,供体细胞呈低水平甲基化,即甲基化TERT-DMR能上调IPSC的启动子活性,从而调控细胞重编程进程(Takasawa et al.,2018)。
端粒酶活性在哺乳动物生殖细胞和早期胚胎发育过程还具有时序性变化规律。早期腔前和排卵前卵母细胞的端粒酶活性较高,但成熟卵母细胞的端粒酶活性很低(Kosebent et al.,2018)。对于雄性的性源细胞来说,精母细胞和精子细胞具有端粒酶活性,而在成熟精子中检测不到端粒酶活性(Ravindranath et al.,1997;Pech et al.,2015)。值得注意的是,卵母细胞受精后端粒酶活性明显升高,但其发育成正常体细胞后端粒酶活性几乎完全消失(Kosebent et al.,2018)。受精卵在胚胎发育的不同阶段其端粒酶活性也会发生时序性变化,Betts和King(1999)研究發现在不同物种包括人类和牛受精胚在4-细胞期端粒酶活性再次出现,且囊胚期活性表现相当高。早期胚胎中的端粒延长呈端粒酶依赖性,Schaetzlein等(2004)研究表明,在小鼠和牛的桑椹胚到胚泡过渡期发生端粒重编程,并在胚胎发生过程中建立了特定的端粒长度,无论供体细胞核的端粒长度如何,该过程都能恢复克隆胚中的端粒长度。胚胎发育过程中端粒的延伸对于确保从亲本到子代的正常端粒长度分离至关重要,且可能对子代的再生、衰老和致癌等具有直接影响。Xu和Yang(2001)通过检查来自孤雌激活和体细胞克隆牛胚胎中的端粒酶活性,并在胚胎发育不同阶段获取从受精卵到胚泡的端粒酶活性,结果显示孤雌激活和体细胞克隆牛胚胎中的端粒酶活性与IVF胚胎具有相似的动态特征。此外,在鼠和牛的胚胎发育期间,端粒酶在桑椹胚/胚泡转变中被强烈激活,端粒明显延长。在胚胎发育的不同阶段,无论胚胎获得方法如何,检测到的端粒酶活性均在胚泡阶段达最高水平。
3 展望
细胞重编程在《Science》2008年评出的“十大科学进展”中位列第一,日本科学家山中伸弥和英国科学家约翰·伯特兰·格登也因研究细胞重编程取得重大突破而获得2012年度诺贝尔生理学或医学奖。至此,细胞重编程成为当今生物界许多学者热衷研究的领域之一。由于细胞重编程有望从根本上解决体细胞克隆技术问题及攻克再生医学领域面临的技术难题,因此是未来动物改良育种、人类不孕不育乃至癌症等重大疾病治疗中最具应用价值的一项前沿技术。目前,细胞重编程面临的共同问题是效率低下,而根本原因是对其作用机制尚未完全了解,例如生殖细胞的端粒酶活性不断发生时序性变化在胚胎发育乃至细胞重编程过程中究竟发挥着怎样的作用,是否在有序调控细胞重编程或开启某些信号通路的变化等。因此,阐明细胞重编程机制,尤其是揭示端粒酶在胚胎发育过程中的分子调控机理及端粒酶调控细胞重编程可能开启的信号通路,对提高细胞重编程效率,推进克隆动物的生产实践并开启生命再造,以及治疗癌症等人类重大疾病具有重要意义。
參考文献:
Achi M V,Ravindranath N,Dym M. 2000. Telomere length in male germ cells is inversely correlated with telomerase activity[J]. Biology of Reproduction,63(2):591-598.
An J,Wu M,Xin X,Lin Z,Li X,Zheng Q,Gui X,Li T,Pu H,Li H,Lu D. 2016. Inflammatory related gene IKKα,IKKβ,IKKγ cooperates to determine liver cancer stem cells progression by altering telomere via heterochromatin protein 1-HOTAIR axis[J]. Oncotarget,7(31):50131-50149.
Azzalin C M,Reichenbach P,Khoriauli L,Giulotto E,Ling-ner J. 2007. Telomeric,repeat containing RNA and RNA surveillance factors at mammalian chromosome ends[J]. Science,318(5851):798-801.
Betts D H,King W A. 1999. Telomerase activity and telomere detection during early bovine development[J]. Developmental Genetics,25(4):397-403.
Blasco M A. 2005. Telomeres and human disease:Ageing,cancer and beyond[J]. Nature Reviews Genetics,6(8):611-622.
Blelloch R,Wang Z,Meissner A,Pollard S,Smith A,Jaenisch R. 2006. Reprogramming efficiency following somatic cell nuclear transfer is influenced by the differentiation and methylation state of the donor nucleus[J]. Stem Cells,24(9):2007-2013.
Bonk A J,Li R,Lai L,Hao Y,Liu Z,Samuel M,Fergason E A,Whitworth K M,Murphy C N,Antoniou E,Prather R S. 2008. Aberrant DNA methylation in porcine in vitro- parthenogenetic-,and somatic cell nuclear transfer-produced blastocysts[J]. Molecular Reproduction Development,75(2):250-264.
Chen L,Roake C M,Freund A,Batista P J,Tian S,Yin Y A,Gajera C R,Lin S,Lee B,Pech M F,Venteicher A S,Das R,Chang H Y,Artandi S E. 2018. An activity switch in human telomerase based on RNA conformation and shaped by TCAB1[J]. Cell,174(1):218-230.
Cherlet T,Murphy L C. 2007. Estrogen receptors inhibit Smad3 transcriptional activity through Ap-1 transcription factors[J]. Molecular and Cellular Biochemistry,306(1-2):33-42.
Curchoe C L,Zhang S Q,Yang L,Page R,Tian X C. 2009. Hypomethylation trends in the intergenic region of the imprinted IGF2 and H19 genes in cloned cattle[J]. Animal Reproduction Science,116(3-4):213-225.
Dean W,Santos F,Stojkovic M,Zakhartchenko V,Walter J,Wolf E,Reik W. 2001. Conservation of methylation reprogramming in mammalian development:Aberrant reprogramming in cloned embryos[J]. Proceedings of the National Academy of Sciences of the United States of Ame-rica,98(24):13734-13738.
Deng M,Ren C,Liu Z,Zhang G,Wang F,Wan Y. 2017. Epigenetic status of H19-Igf2 imprinted genes and loss of 5-Hydroxymethylcytosine in the brain of cloned goats[J]. Cellular Reprogramming,19(3):199-207.
Dogan F,Biray Avci C. 2018. Correlation between telomerase and mTOR pathway in cancer stem cells[J]. Gene,641:235-239.
Giraldo A M,Hylan D A,Ballard C B,Purpera M N,Vaught T D,Lynn J W,Godke R A,Bondioli K R. 2008. Effect of epigenetic modifications of donor somatic cells on the subsequent chromatin remodeling of cloned bovine embryos[J]. Biology of Reproduction,78(5):832-840.
Hein N,Jiang K,Cornelissen C,Lüscher B. 2011. TGFβ1 enhances MAD1 expression and stimulates promoter-bound Pol II phosphorylation:Basic functions of C/EBP,SP and SMAD3 transcription factors[J]. BMC Molecular Biology,12:9. doi:10.1186/1471-2199-12-9.
Hockemeyer D,Daniela J P,Takai H,de Lange T. 2006. Recent expansion of the telomeric complex in rodents:Two distinct POT1 proteins protect mouse telomeres[J]. Cell,126(1):63-77.
Huang C H,Dai X Y,Chai W H. 2012. Human Stn1 protects telomere integrity by promoting efficient lagging-strand synthesis at telomeres and mediating C-strand fill-in[J]. Cell Research,22(12):1681-1695.
Huang J J,Wang F,Okuka M,Liu N,Ji G,Ye X,Zuo B,Li M,Liang P,Ge W W,Tsibris J C,Keefe D L,Liu L. 2011. Association of telomere length with authentic pluripotency of ES/iPS cells[J]. Cell Research,21(5):779-792.
Inoue K,Kohda T,Sugimoto M,Sado T,Odonuki N,Matoba S,Shiura H,Ikeda R,Mochida K,Fujii T,Sawai K,Otte A P,Tian X C,Yang X,Ishino F,Abe K,Ogura A. 2010. Impeding Xist expression from the active X chromosome improves mouse somatic cell nuclear transfer[J]. Science,330(6003):496-499.
Jiang J,Chan H,Cash D D,Miracco E J,Ogorzalek Loo R R,Upton H E,Cascio D,O’Brien Johnson R,Collins K,Loo J A,Zhou Z H,Feigon J. 2015. Structure of Tetrahymena telomerase reveals previously unknown subunits,functions,and interactions[J]. Science,350(6260):aab4070. doi:10.1126/science.aab4070.
Jiang J,Wang Y,Sušac L,Chan H,Basu R,Zhou Z H,Feigon J. 2018. Structure of telomerase with telomeric DNA[J]. Cell,173(5):1179-1190.
Kinoshita T,Nagamatsu G,Saito S,Takubo K,Horimoto K,Suda T. 2014. Telomerase reverse transcriptase has an extratelomeric function in somatic cell reprogramming[J]. The Journal of Biological Chemistry,289(22):15776-15787.
Kosebent E G,Uysal F,Ozturk S. 2018. Telomere length and telomerase activity during folliculogenesis in mammals[J]. The Journal of Reproduction and Development,64(6):477-484.
Kremenskoy M,Kremenska Y,Suzuki M,Imai K,Takahashi S,Hashizume K,Yaqi S,Shiota K. 2006. DNA methylation profiles of donor nuclei cells and tissues of cloned bovine fetuses[J]. The Journal of Reproduction and Development,52(2):259-266.
Lanza R P,Cibelli J B,Blackwell C,Cristofalo V J,Francis M K,Baerlocher G M,Mak J,Schertzer M,Chavez E A,Sawyer N,Lansdorp P M,West M D. 2000. Extension of cell life-span and telomere length in animals cloned from senescent somatic cells[J]. Science,288(5466):665-669.
Lewinska A,Adamczyk-Grochala J,Kwasniewicz E,Wnuk M. 2017. Downregulation of methyltransferase Dnmt2 results in condition-dependent telomere shortening and senescence or apoptosis in mouse fibroblasts[J]. Journal of Cellular Physiology,232(12):3714-3726.
Lin C,Qin Y,Zhang H,Gao M Y,Wang Y F. 2018. EGF upregulates RFPL3 and hTERT via the MEK signaling pathway in non-small cell lung cancer cells[J]. Oncology Reports,40(1):29-38.
Marión R M,Schotta G,Ortega S, Blasco M A. 2011. Suv4-20h abrogation enhances telomere elongation during reprogramming and confers a higher tumorigenic potential to iPS cells[J]. PLoS One,6(10):e25680.
Marión R M,Strati K,Li H,Murga M,Blanco R,Ortega S,Fernandez-Capetillo O,Serrano M,Blasco M A. 2009a. A p53-mediated DNA damage response limits reprogramming to ensure iPS cell genomic integrity[J]. Nature,460(7259):1149-1153.
Marión R M,Strati K,Li H,Tejera A,Schoeftner S,Ortega S,Serrano M,Blasco M A. 2009b. Telomeres acquire em-bryonic stem cell characteristics in induced pluripotent stem cells[J]. Cell Stem Cell,4(2):141-154.
Marx J. 2006. Molecular biology. Protein tail modification opens way for gene activity[J]. Science,311(5762):757.
Mason M,Schuller A,Skordalakes E. 2011. Telomerase structure function[J]. Current Opinion in Structural Biology,21(1):92-100.
Mersaoui S Y,Wellinger R J. 2019. Fine tuning the level of the Cdc13 telomere-capping protein for maximal chromosome stability performance[J]. Current Genetics,65(1);109-118.
Park J I,Venteicher A S,Hong J Y,Choi J,Jun S,Shkreli M,Chang W,Meng Z,Cheung P,Ji H,McLauqhin M,Veenstra T D,Nusse R,McCrea P D,Artandi S E. 2009. Telomerase modulates Wnt signalling by association with target gene chromatin[J]. Nature,460(7251):66-72.
Park J,Lee H,Han N,Kwak S,Lee H T,Kim J H,Kang K,Youn B H,Jeong H J,Kang J S,Kim S Y,Han J W,Youn H D,Cho E J. 2018. Long non-coding RNA ChRO1 facilitates ATRX/DAXX-dependent H3.3 deposition for transcription- associated heterochromatin reorganization[J]. Nucleic Acids Research. doi: 10. 1093/nar/gky923.
Pech M F,Garbuzov A,Hasegawa K,Sukhwani M,Zhang R J,Benayoun B A,Brockman S A,Lin S,Brunet A,Orwig K E,Artandi S E. 2015. High telomerase is a hallmark of undifferentiated spermatogonia and is required for maintenance of male germline stem cells[J]. Genes & Development,29(23):2420-2434.
Peek G W,Tollefsbol T O. 2016. Down-regulation of hTERT and Cyclin D1 transcription via PI3K/Akt and TGF-β pathways in MCF-7 cancer cells with PX-866 and raloxifene[J]. Experimental Cell Research,344(1):95-102.
Qin Q P,Wang S L,Tan M X,Wang Z F,Luo D M,Zou B Q,Liu Y C,Yao P F,Liang H. 2018. Novel tacrine platinum(II) complexes display high anticancer activity via inhibition of telomerase activity,dysfunction of mitochondria,and activation of the p53 signaling pathway[J]. European Journal of Medicinal Chemistry,158:106-122.
Ravindranath N,Dalal R,Solomon B,Djakiew D,Dym M. 1997. Loss of telomerase activity during male germ cell differentiation[J]. Endocrinology,138(9):4026-4029.
Ruan Z,Zhao X,Qin X,Luo C,Liu X,Deng Y,Zhu P,Li Z,Huang B,Shi D,Lu F. 2018. DNA methylation and expression of imprinted genes are associated with the viability of different sexual cloned buffaloes[J]. Reproduction in Domestic Animals,53(1):203-212.
Sado T,Brockdorff N. 2013. Advances in understanding chromosome silencing by the long non-coding RNA XIST[J]. Philosophical Transactions of the Royal Society of London(Series B),368(1609):20110325. doi: 10.1098/rstb.2011.0325.
Sahakyan A,Plath K,Rougeulle C. 2017. Regulation of X-chromosome dosage compensation in human:Mechanisms and model systems[J]. Philosophical Transactions of the Royal Society of London(Series B),372(1733). Pii:20160363. doi:10.1098/rstb.2016.0363.
Schaetzlein S,Lucas-Hahn A,Lemme E,Kues W A,Dorsch M,Manns M P,Niemann H,Rudolph K L. 2004. Telomere length is reset during early mammalian embryogene-sis[J]. Proceedings of the National Academy of Sciences of the United States of America,101(21):8034-8038.
Schaetzlein S,Rudolph K L. 2005. Telomere length regulation during cloning, embryogenesis and ageing[J]. Reproduction,Fertility,and Development,17(1-2):85-96.
Shen C J,Cheng W K,Wu S C,Chen H L,Tsai T C,Yang S H,Chen C M. 2012. Differential differences in methylation status of putative imprinted genes among cloned swine genomes[J]. PLoS One,7(2):e32812.
Shiels P G,Kind A J,Campbell K H,Waddington D,Wilmut I,Colman A,Schnieke A E. 1999. Analysis of telomere lengths in cloned sheep[J]. Nature,399(6734):316-317.
Silveira M M,Salgado Bayão H X,Dos Santos Mendonça A,Borges N A,Vargas L N,Caetano A R,Rumpf R,Franco M M. 2018. DNA methylation profile at a satellite region is associated with aberrant placentation in cloned calves[J]. Placenta,70:25-33. doi:10.1016/j.placenta.2018. 08.007.
Smogorzewska A,van Steensel B,Bianchi A,Oelmann S,Schaefer M R,Schnapp G,de Lange T. 2000. Control of human telomere length by TRF1 and TRF2[J]. Molecular and Cellular Biology,20(5):1659-1668.
Solter D. 2000. Mammalian cloning:Advances and limitations[J]. Nature Reviews. Genetics,1(3):199-207.
Su Y,Chen C,Guo L,Du J,Li X,Liu Y. 2018. Ecological balance of oral microbiota is required to maintain oral mesenchymal stem cell homeostasis[J]. Stem Cells,36(4):551-561.
Takasawa K,Arai Y,Yamazaki-Inoue M,Toyoda M,Akutsu H,Umezawa A,Nishino K. 2018. DNA hypermethylation enhanced telomerase reverse transcriptase expression in human-induced pluripotent stem cells[J]. Human Cell,31(1):78-86.
Tilborghs S,Corthouts J,Verhoeven Y,Arias D,Rolfo C,Trinh X B,van Dam P A. 2017. The role of nuclear factor-kappa B signaling in human cervical cancer[J]. Critical Reviews in Oncology/Hematology,120:141-150.
Tsai M D,Chen P R,Tien L T,Cai Y J,Lee Y J. 2013. Nuc-lear condensation and cell cycle arrest induced by telome-rase siRNA in neuroblastoma cells[J]. Journal of Neu-ro-oncology,111(3):265-272.
Upton H E,Chan H,Feigon J,Collins K. 2017. Shared subunits of tetrahymena telomerase holoenzyme and replication protein a have different functions in different cellular complexes[J]. The Journal of Biological Chemistry,292(1):217-228.
Wang F,Yin Y,Ye X,Liu K,Zhu H,Wang L,Chiourea M,Okuka M,Ji G,Dan J,Zou B,Li M,Zhang Q,Liu N,Chen L,Pan X, Gagos S,Keefe D L,Liu L. 2012. Molecular insights into the heterogeneity of telomere reprogramming in induced pluripotent stem cells[J]. Cell Research,22(4):757-768.
Wu X,Li Y,Xue L,Wang L,Yue Y,Li K,Bou S,Li G P,Yu H. 2011. Multiple histone site epigenetic modifications in nuclear transfer and in vitro fertilized bovine embryos[J]. Zygote,19(1):31-45.
Xu J,Yang X. 2001. Telomerase activity in early bovine embryos derived from parthenogenetic activation and nuc-lear transfer[J]. Biology of Reproduction,64(3):770-774.
Yang W,Wu Z,Yang K,Han Y,Chen Y,Zhao W,Huang F,Jin Y,Jin W. 2019. BMI1 promotes cardiac fibrosis in ischemia-induced heart failure via the PTEN-PI3K/AKT-mTOR signaling pathway[J]. American Journal of Physio-logy,316(1):H61-H69.
Yang X Z,Smith S L,Tian X C,Lewin H A,Renard J P,Wakayama T. 2007. Nuclear reprogramming of cloned embryos and its implications for therapeutic cloning[J]. Nature Genetics,39(3):295-302.
Yano S,Takehara K,Tazawa H,Kishimoto H,Urata Y,Kagawa S,Fujiwara T,Hoffman R M. 2016. Efficacy of a cell-cycle decoying killer adenovirus on 3-D gelfoam®-histoculture and tumor-sphere models of chemo-resistant stomach carcinomatosis visualized by FUCCI imaging[J]. PLoS One,11(9):e0162991.
Yatsenko S A,Hixson P,Roney E K,Scott D A,Schaaf C P,Ng Y T,Palmer R,Fisher R B,Patel A,Cheung S W,Lupski J R. 2012. Human subtelomeric copy number gains suggest a DNA replication mechanism for formation:Beyond breakage-fusion-bridge for telomere stabilization[J]. Human Genetics,131(12):1895-1910.
Zheng L,Suzuki H,Nakajo Y,Nakano A,Kato M. 2018. Re-gulation of c-MYC transcriptional activity by transforming growth factor-beta 1-stimulated clone 22[J]. Cancer Scien-ce,109(2):395-402.
Zvereva M I,Shcherbakova D M,Dontsova O A. 2010. Telomerase:Structure,functions,and activity regulation[J]. Biochemistry,75(13):1563-1583.
(責任编辑 陈 燕)

