长江中下游麦区小麦地方种质千粒重全基因组关联分析
程宇坤,李 健,姚方杰,蒋云峰,江千涛,李 伟,蒲至恩,邓 梅,魏育明,陈国跃*
(1.四川农业大学小麦研究所,成都 611130;2.四川农业大学农学院,成都 611130)
小麦是世界第一大粮食作物,在全世界范围内有三分之一的人口将小麦作为主食。在中国是仅次于水稻的第二大粮食作物,具有非常重要的基础性地位[1]。提高小麦产量是小麦育种的最重要的目标之一。小麦的产量构成因子由单位面积穗数、穗粒数及千粒重组成,改良任何一个产量构成因子均能提高小麦产量。在产量三因子中,更多的遗传研究集中在千粒重。小麦千粒重是典型的多基因控制的数量性状,同时也受环境的影响。迄今为止,国内外已有大量学者利用家系连锁作图(family-based linkage mapping)对小麦千粒重进行了遗传研究,并对控制小麦千粒重的QTL 区段进行了定位和遗传解析。该方法通过双亲杂交,建立遗传作图群体,绘制高密度分子遗传连锁图谱, 遗传群体表型性状,基于连锁分析, 将控制千粒重的QTL(quantitative trait locus)定位在遗传连锁图谱上[2-3]。郑有良等[4](1993)对多小穗品系“10 阿”进行研究发现,粒重低受 5A、1B、2B、6B、2D 和 7D 等染色体上的隐性基因所控制;从其减小效应的程度来看,1B 和2B 染色体上的基因表现为强效;5A、6B、2D 和 7D 染色体上的基因表现为弱效。P.Ramya 等[5]利用RIL 群体对控制小麦千粒重进行QTL 定位分析, 定位到10 个控制千粒重的 QTL, 分布在 1A、1D、2B、2D、4B、5B和6B 染色体上。M.Golabadi 等[6]利用单标记分析方法在干旱胁迫和正常灌溉条件下进行千粒重QTL检测,其中Xcfa2114-6A 能够解释千粒重表型变异率的20%;另外,在3B、7B 染色体上分别检测到3个标记与千粒重紧密连锁。近年来,多个研究团队利用不同的遗传群体均发现,在SSR 标记Xgwm332附近存在控制小麦千粒重的QTL,且均表现为主效QTL[7-12]。Li C.L.等[13]利用宁 7840×Clark 构建的 RIL群体分析在该标记附近发现了一个与小麦千粒重相关的QTL 区段QC-7AL,该区段位于小麦7AL 染色体上的IWA7406-IWA6535 之间,区间长度为6.5 cM,在不同环境中解释千粒重的表型变异为10.9%~14.88%。孙宇慧等[14]进一步对该QTL 进行了精细定位和遗传分析, 结果表明,QC-7AL 主要位于小麦7AL 染色体的 IWA7406-IWA5913 标记区间, 长度为3.1 cM,对应的物理距离约为5.63 Mb,其中包含了473 个SNP 标记和81 个基因。利用家系连锁作图方法对小麦千粒重进行遗传解析往往受群体双亲遗传差异的制约,只能在双亲的背景中分析遗传差异,研究结果有一定的局限性。近年来,随着小麦全基因组测序研究的完成及高通量分子标记的开发,另一种用于解析植物复杂性状基因型的重要方法——自然群体关联作图(association mapping)应运而生。关联作图亦称关联分析,以自然群体为研究对象,以长期重组后保留下来的基因(位点)间连锁不平衡(1inkage disequilibrium,LD)为基础,对自然群体中目标性状的遗传变异与基因多样性进行关联,无须构建遗传作图群体,可直接鉴定出与表型变异密切相关且具有特定功能的基因位点或标记位点[15]。该方法可以同时检测自然群体中的多个等位基因,具有低耗费、高精度、强实用性等优点,目前已广泛应用于水稻、小麦、玉米、油菜等作物复杂性状的遗传解析中[16-18]。近年来,小麦的关联作图主要围绕抗病(条锈病、叶锈病、白粉病等)、产量(株高、穗粒数、可育小穗数、千粒重)、品质(蛋白质含量、湿面筋含量等)相关性状进行。R.R.Mir 等[19]利用关联分析发现了11 个与小麦粒重相关联的SSR、AFLP和 SAMPL 新标记。Zhang D.L.等[20]发现 Xgwm311-A500、Xgwml31-A110 和 Xcfe273-A306 等 23 个增加小麦籽粒数的优异等位变异。武玉国等[21]选用108对多态性高的SSR 标记对黄淮麦区175 份小麦进行关联分析,得到23 个与株高、产量相关性状显著关联的标记。此外,张国华等[22]以128 份黄淮麦区小麦品种(系)为材料,通过关联分析表明获得38 个在2 个或以上环境或均值下与产量相关性状显著关联的位点;并进一步发掘了一批与产量相关性状的优异等位变异。但迄今为止,尚未有关于长江中下游麦区小麦地方种质千粒重全基因组关联分析的报道。
本研究团队在前期研究中利用SSR 分子标记和DArT-seq 芯片技术, 对长江中下游麦区的小麦地方品种进行全基因组分子扫描,发现长江中下游麦区的小麦地方品种在分子水平上具有丰富的遗传多样性[23]。因此,本研究拟通过对188 份长江中下游麦区的小麦地方品种在多环境下的千粒重进行鉴定,结合覆盖小麦全基因组的7 099 个DArT-seq标记开展关联分析,以期发掘控制长江中下游麦区的小麦地方品种千粒重的显著性位点或区段,为小麦产量分子辅助育种提供分子依据。
1 材料和方法
1.1 供试材料
供试的188 份长江中下游麦区小麦地方品种来自 9 个省区,包括:江苏省(61 份)、浙江省(58份)、湖北省(34 份)、安徽省(8 份)、陕西省(7 份)、江西省(5 份)、湖南省(5 份)、上海市(4 份)、河南省(4 份)(表 1)。以上地方种质均由国家种质资源库(北京)提供,四川农业大学小麦研究所保存。
1.2 表型鉴定
供试材料分别于2017年和2018年分别种植于四川农业大学崇州试验基地(简称2017CZ、2018CZ)和绵阳农业科学院小麦试验基地(2017MY)。按照完全随机区组设计,每份材料3 行区、行长2 m、行距0.3 m。单粒点播,株距10 cm,每行20 粒种子,生长过程中按常规田间管理。5 月中旬成熟后,每份材料混收、脱粒、去除杂质后晒干至含水量达到14%以下。每份材料种子随机数粒1 000 粒进行称量,3次重复,取其平均值为其千粒重(g)。
表1 供试小麦地方种质Table 1 Wheat landraces used in this study

(续表1)
1.3 基因型鉴定
2018年,于小麦三叶期,每份材料代表性植株8 株,每株取1 片新鲜嫩叶,采用改良的CTAB 法[24]提取基因组DNA。DNA 提取后质控其纯度和浓度,并将其稀释至80 ng/μL 备用。将制备好的188 份长江中下游麦区小麦地方品种基因组DNA 送到澳大利亚 DArT-seq 公司(Diversity Arrays Technology,http://www.diversityarrays.com/) 进行全基因芯片扫描,获得原始芯片测序数据;进而对原始数据进行质控:①剔除一致性<95%的等位基因标记;②剔除标记数据缺失率在10%以上的数据。
1.4 数据分析
通过利用Excel 对千粒重表型数据进行描述性统计;采用SPSS 16.0 软件对表型数据进行方差及相关性分析[25];基于公式 H2=VG/(VG+VE)计算千粒重的广义遗传力(broad-sense heritability),其中VG 代表遗传方差,VE 代表环境方差[26]。在研究团队前期群体结构、 亲缘关系和连锁不平衡分析基础上,基于3 个环境下小麦千粒重及其最佳线性无偏估计值(best linear unbiased estimates,BLUE),结合DArT 分子标记,采用一般线性模型(Q+K)(general linear model,GLM,Q+K)进行全基因组关联分析[27-28]。获得的显著关联标记位点(-log10(P-value)>3.0)进一步发掘其优异等位变异,解析各等位变异的表型遗传效应[29]。
以LD 距离作为候选基因的预测区间, 在该距离内的关联位点为同一个数量性状位点(QTL)[30-31];QTL 的命名方法按照QTL+性状+sicau+染色体。其中,QTL 以 Q 表示, 性状以英文缩写表示,sicau 代表四川农业大学,染色体以小麦染色体的名称表示,位于同一染色体的相同性状的QTL 在性状后面用1、2 等区分。采用 Excel、R 软件(R version 3.0.3)[32]及ggplot2 作图。
2 结果与分析
2.1 长江中下游麦区地方种质千粒重表型特征分析
188 份长江中下游麦区地方种质千粒重在3 个环境下均表现出丰富的表型变异,其变异幅度分别为22.69~49.95 g、17.71~42.55 g、17.92~50.93 g(表 2)。进一步分析发现,来自上海的丈四红、江苏的镇3-96 和浙江的松花蒲3 份地方种质在不同环境中均表现出较高的千粒重,分别为43.20、41.79 和40.67 g。基于千粒重BLUE 值发现, 共有87 份小麦地方种质千粒重>33.0 g,主要来自于江苏(31 份)、浙江(24 份)和湖北(18 份),其余 14 份来自其他省区(图1)。方差分析显示千粒重在基因型间存在显著或极显著差异, 表明千粒重既受基因型的控制,也易受环境影响。3 个环境下千粒重的广义遗传力(H2)均较高,分别达到 0.90、0.86 和 0.92,揭示长江中下游麦区地方种质千粒重主要受遗传控制。千粒重的峰度和偏度均接近0,说明千粒重基本符合正态分布,揭示千粒重是多基因控制的数量性状(表2)。供试材料的千粒重在3 个环境中相关性均达到极显著水平(P<0.001)。其中,2017 绵阳和 2018年崇州相关程度最高,其相关系数达到0.707 2;2017年绵阳和2017年崇州相关程度最低, 其相关系数为0.676 1(表3)。
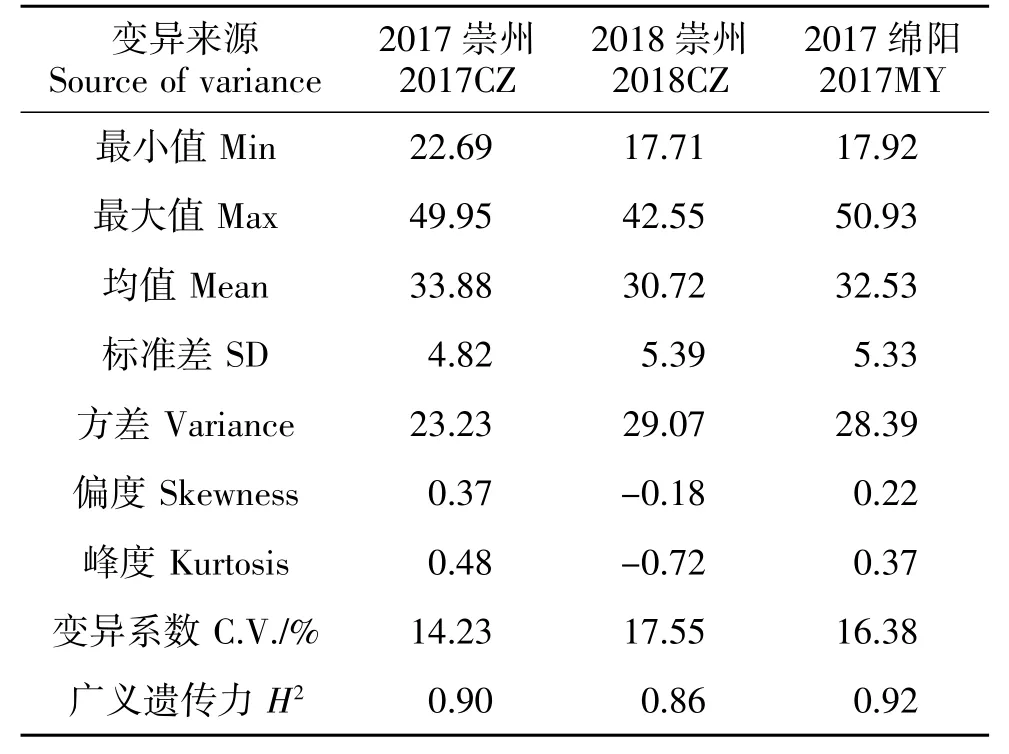
表2 188 份材料在3 个环境下的千粒重表型变异Table 2 Variation of thousand kernel weight in 188 wheat accessions detected in three environments
2.2 小麦千粒重全基因组关联分析
本研究团队在前期研究中, 利用DArT 芯片技术对供试材料进行全基因组分子扫描,共获得9730个DArT 分子标记。进一步对获得的初始标记进行质控,最终获得7 099 个高质量的有效DArT 标记。同时,利用这些有效标记,对188 份长江中下游麦区小麦地方种质进行群体结构分析,将供试材料划分为3 个亚群[23]。本研究在此基础上,结合千粒重表型,基于一般线性模型(Q+K)对来自长江中下游麦区的188 份小麦地方种质进行全基因组关联分析。
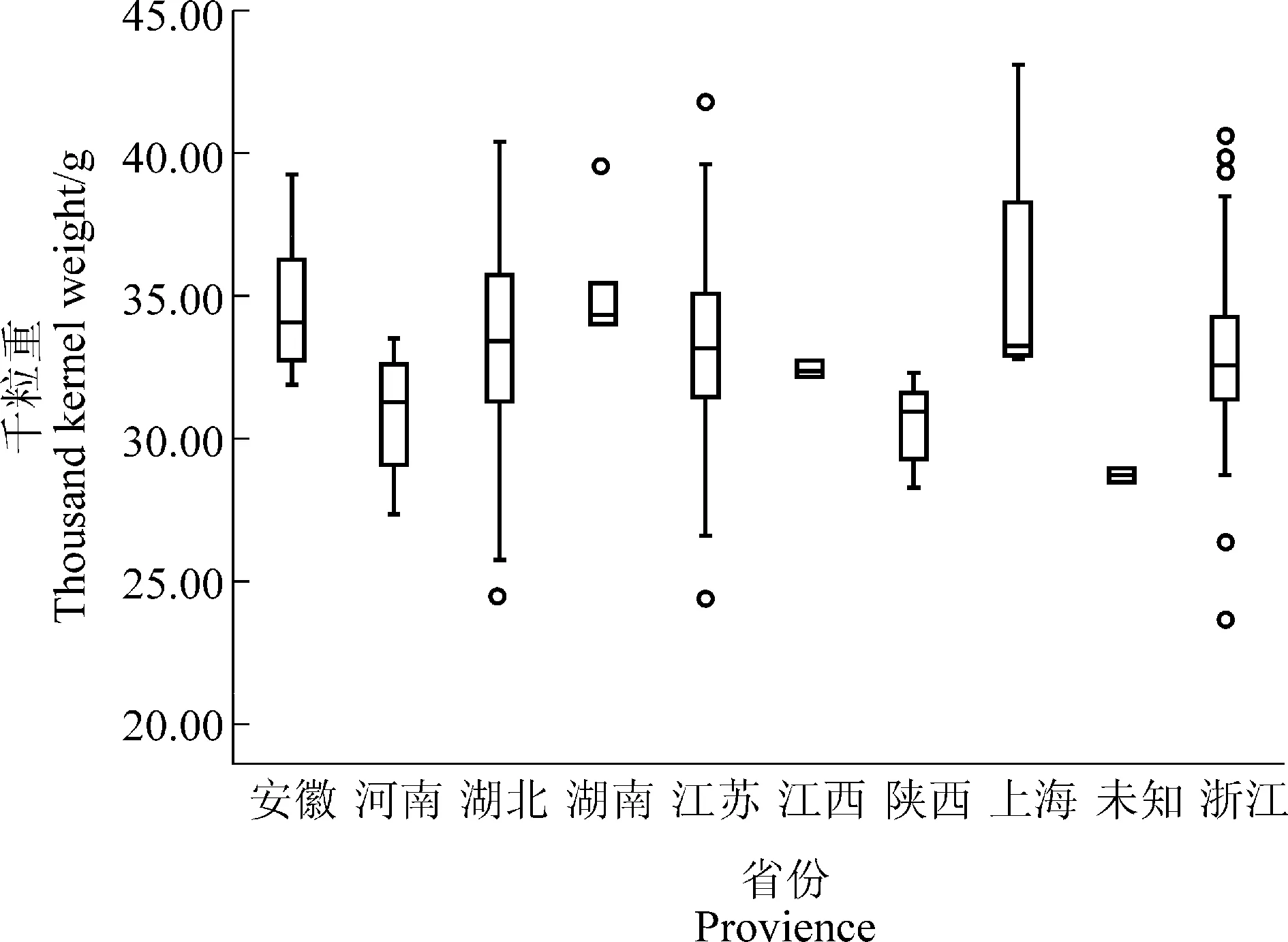
图1 不同省份千粒重BLUE 值比较Figure 1 Comparison of thousand kernel weight BLUE value in different provinces
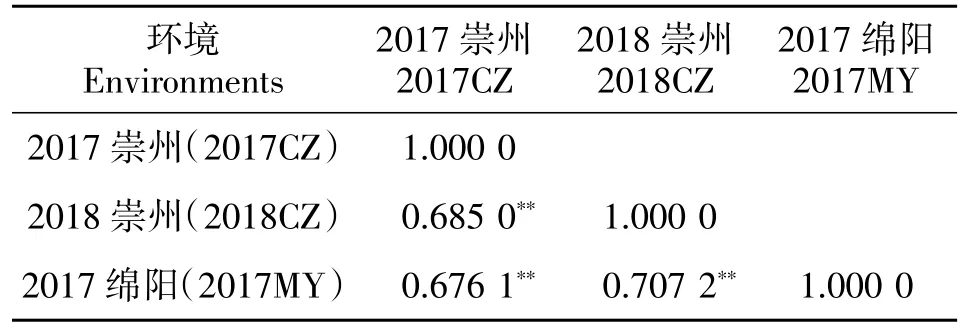
表3 不同环境下千粒重相关性分析Table 3 Thousand kernel weight correlation coefficients in different environments
通过关联分析发现, 在2017年崇州共检测到121 个与千粒重关联的位点, 分布在除3D、4D、6B和6D 以外的17 条染色体上;2017年绵阳检测到与千粒重相关的位点48 个,分布在11 条染色体上(1B、1D、2A、2B、2D、3B、4A、4B、5B、6B 和 6D);2018年崇州检测到与千粒重相关的位点46 个,分布在1A、1B、1D、2A、2B、2D、3B、4B、5B、6B 和 6D 染色体上;与BLUE 值关联检测到与千粒重相关的位点6 个,分布在 1D、3B、4A 和 4B 染色体上。综合分析 3 个环境及BLUE 值关联位点, 共鉴定出8 个DArT 标记在2 个或2 个以上的环境中与小麦千粒重显著相关(表4)。其中,标记 1219741 在 3 个环境下和BLUE 值中被重复检测到,其贡献率(R2)为10.94%。4 个位点:3533475、1252691、7348209、3939196、1095647 和 1233875,则分别在 2017年崇州、绵阳和2018年崇州3 个环境下均被重复检测到,R2范围从9.68%到12.65%。而1377569 则在2017年崇州和2018年崇州以及BLUE 值下被重复检测到,其 R2为 9.19%。
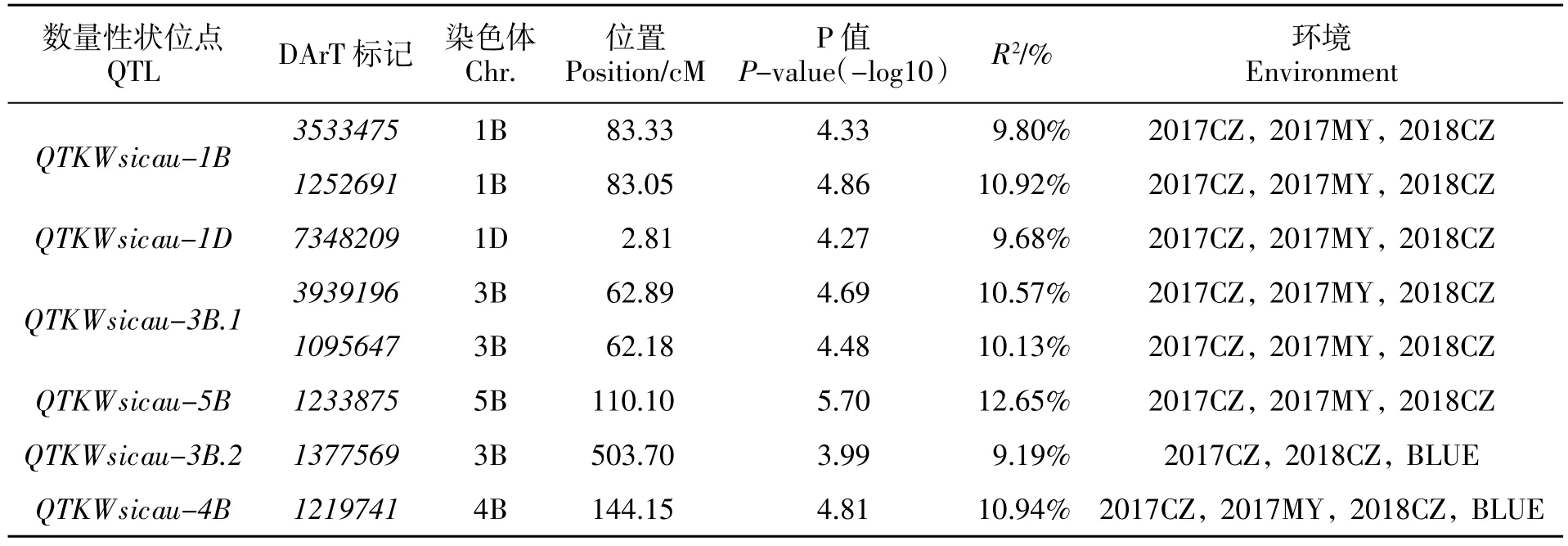
表4 与小麦千粒重显著关联位点Table 4 Significant loci associated with thousand kernel weight in 188 wheat accessions
2.3 控制小麦千粒重显著关联位点遗传效应分析
结合表型变异,对上述获得的8 个与小麦千粒重显著关联位点进行遗传效应分析,结果发现,5 个显著关联位点对小麦千粒重具有正向遗传效应。其中,1219741 可使千粒重增加1.8 g, 其在群体中出现的频率占40%;位点3939196 可使千粒重增加0.6 g, 在群体中出现频率为 29%;而 7348209 和3533475 均可使千粒重增加0.3 和0.1 g, 在群体中所占比例为27%和77%;1377569,千粒重增加0.2 g,所占群体比例最高为91%。另外3 个低频率显著关联位点 1252691、1095647 和 1233875, 则对小麦千粒重具有负向遗传效应,分别使千粒重降低1.6、0.3和2.2 g, 在群体中所占比例分别为24%、35%和31%(图 2)。
3 讨论与结论
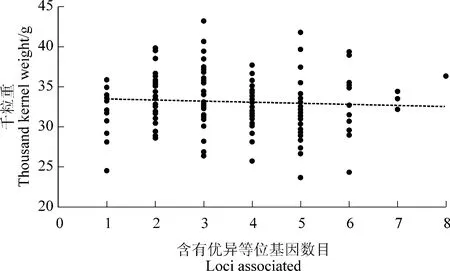
图2 显著关联位点在供试材料中的分布及其与千粒重之间的线性回归Figure 2 Distribution of the eight loci associated with thousand kernel weight contained in 188 accessions and linear regression relationships between loci associated and thousand kernel weight
千粒重是小麦产量的主要构成因素之一,该性状受数量性状基因控制,因此其表型选择往往费力费时。利用分子标记辅助选择将是一个有效的方法。在过去的十多年间,利用双亲群体已定位了较多的控制小麦千粒重QTL, 分布在小麦的1A、1D、2A、2B、2D、3A、3B、3D、4B、5A、5B、5D、6A、6D、7A和7D 染色体上[33-43]。本研究利用DArT 芯片测序技术对188 份长江中下游小麦地方种质千粒重进行了关联分析,共鉴定出8 个与小麦千粒重稳定关联的位点、 共涉及6 个染色体区段, 分布在1B、3B、4B、5B 和 1D 染色体上(表4)。其中,在 1B 染色体上鉴定出一个控制小麦千粒重位点QTKWsicau-1B,其解释的表型变异率为9.80%~10.92%。蒲艳艳等[44]在1B 染色体上也发现一个控制小麦千粒重的区段QTgw.1-1B,通过标记共线性比较分析发现,该区段与本研究获得的与小麦千粒重关联位点QTKWsicau-1B 距离较近, 很可能是同一控制小麦千粒重的位点(区段)。廖祥政等[45]以人工合成小麦Am3 为供体亲本, 普通小麦莱州953 为轮回亲本,通过连续回交和自交,构建一个含85 个株系的F2∶3群体,并利用该群体,检测到3 个千粒重QTL。其中一个QTL(QGw.caas-4B)位于被定位在染色体4BL上标记 Xgwm513~Xwmc657 区间内。张学勇等[17]也在该区间内检测到提高千粒重1.91 g 的QTL。本研究在4B 染色体上也鉴定到另一个与小麦千粒重显著关联的位点QTKWsicau-4B, 其可解释的表型变异率为10.94%。另外,张桂芝和高明刚[46-47]分别在附近区域上发现与穗粒数相关的QGns-4B 以及与千粒重相关的QTgw-4B.2。参照和对比D.Somers 等[48]小麦遗传连锁图谱和DArT 分子标记高密度连锁图谱, 尽管本研究鉴定的QTL 与前人研究的QTL 在相近区域内, 但其定位位置并不重叠。因此推测,QTKWsicau-4B 是一个不同于已鉴定的控制小麦千粒重QTL。有意思的是,在位于1D 染色体上,我们鉴定了另一个控制小麦千粒重的QTL(QTKWsicau-1D)。在这个区段内,蒲艳艳和高明刚等[44,47]人发现该区段与小麦产量相关性状有关,并分别鉴定出控制小麦每平米穂数相关位点QSn.1-1D 和千粒重的QTgw-1D。前人对该区段进行研究显示,该区段与小麦条锈病抗性也有关联,并分别定位了QYr.caas-1DS_Naxos、QYrst.orr-1DS_Stephens、QYr.sun-1D_CPI133 和QYrdr.wgp-1DS_IWA226 等控制条锈病抗性的QTL[49-53]。在本研究中,在3B 染色体上共鉴定出2 个控制小麦千粒重关联位点:QTKWsicau-3B.1 和QTKWsicau-3B.2,在这两个位点附近,也发现其与条锈病抗性基因相关[42-43]。这一现象揭示这些QTL 区段可能不仅仅控制着单一目标性状,它们既控制着产量相关性状,同时还携带着抗病基因或位点。

