基于CRISPR/Cas9技术的水稻抗稻瘟病基因Pita突变体的创制
吴 凡, 王 月, 陈 闽, 刘 佳, 杭悦宇, 孙小芹
〔江苏省中国科学院植物研究所(南京中山植物园), 江苏 南京 210014〕
水稻(OryzasativaLinn.)的稻瘟病是由稻瘟病菌(Magnaportheoryzae)引起的一种真菌性病害[1]。目前,研究人员已经鉴定出约100个抗稻瘟病基因,定位于除3号染色体外的其他11条染色体上[2],并成功克隆出Pib、Pita、Pb1、Pi9、Pi2、Pizt和Pid2等27个抗稻瘟病基因[3-4]。Pita基因是继Pib基因之后第2个被克隆出来的抗稻瘟病基因,其表达产物与稻瘟病菌的无毒基因AVR-Pita的表达产物相互作用,激活抗病防御基因表达,从而抑制水稻稻瘟病发生[5]。Pita基因是一个包含2个外显子和1个内含子的单拷贝显性基因,定位于水稻12号染色体近着丝点的区域[6];Pita基因编码NBS-LRR类蛋白,其功能受到氨基酸位点的控制,当第918位氨基酸由丙氨酸突变为丝氨酸时,该基因功能丧失[6]。此外,不同水稻个体的Pita基因存在大量的单核苷酸多态性位点,导致水稻对稻瘟病菌的抗性存在个体间差异[7-8]。
CRISPR/Cas9是一种基因编辑系统,能够为农作物遗传改良提供高效、快速的遗传操作工具[9],已应用于多种模式植物和农作物的基因敲除[10-14]。杨海河等[15]利用CRISPR/Cas9技术对粳稻品种‘日本晴’(O.sativasubsp.japonica‘Nipponbare’)的pi21基因进行编辑,从T1代转基因株系中筛选获得pi21纯合突变体;韩娇等[16]利用CRISPR/Cas9技术对水稻Pht3亚家族磷转运蛋白基因OsPht进行编辑,成功获得目标基因碱基缺失突变体;黄忠明等[17]利用CRISPR/Cas9技术对粳稻温敏不育基因TMS5进行编辑,获得tms5突变体;白建江等[18]利用CRISPR/Cas9技术对水稻2号染色体上的淀粉分支酶基因SBE3进行编辑,获得高抗性淀粉纯合突变体。Pita基因为稻瘟病抗性基因,对Pita基因进行研究有利于研究者对水稻抗稻瘟病机制的了解,然而,目前尚未见利用CRISPR/Cas9技术编辑水稻抗稻瘟病基因Pita的研究报道。
鉴于此,本研究利用CRISPR/Cas9技术对粳稻品种‘日本晴’Pita基因的第6至第25位碱基进行定点编辑,对突变体进行鉴定,筛选出不含T-DNA区的纯合突变体,并对该基因纯合突变体的稻瘟病抗性及6个稻瘟病病程相关基因的表达水平进行分析,以期明确Pita基因在水稻抵御稻瘟病中的作用,为水稻良种选育及品质改良提供材料。
1 材料和方法
1.1 材料
本研究以稻瘟病感病品种‘日本晴’为转基因受体材料;供试的粳稻品种‘日本晴’和籼稻品种‘9311’(O.sativasubsp.indica‘9311’)均由南京大学生命科学学院田大成教授馈赠。实验使用的真核表达载体pGREB31购自美国Addgene公司;大肠杆菌DH5α购自宝日医生物技术(北京)有限公司,农杆菌LBA4404为本实验室保存的菌株;稻瘟病菌小种CH199(Magnaportheoryzaestrain CH199)由中国科学院上海生命科学研究院植物生理生态研究所何祖华教授提供。实验涉及的所有引物均由南京金斯瑞生物科技有限公司合成。
1.2 实验方法
1.2.1Pita基因靶位点设计 利用CRISPR-P在线工具(http:∥crispr.hzau.edu.cn/cgi-bin/CRISPR2/SCORE)设计Pita基因的靶位点[19-21],选择评分较高且位于Pita基因5′端的第6至第25位碱基序列(5′-GCCGGCGGTCAGTGCATCGC-3′)作为靶位点。根据Cas-OFFinder和BLAST分析结果确认不存在脱靶效应。
1.2.2 pTGE1表达载体的构建 利用CRISPR-P在线工具设计Pita基因的gRNA序列(g11S序列为5′-GGCAGCCGGCGGTCAGTGCATCGC-3′,g11AS序列为5′-AAACGCGATGCACTGACCGCCGGC-3′);将合成的g11S和g11AS等量混合,制备双链接头;与经过BsaⅠ酶切的pGREB31载体连接,获得pTGE1表达载体(图1)。
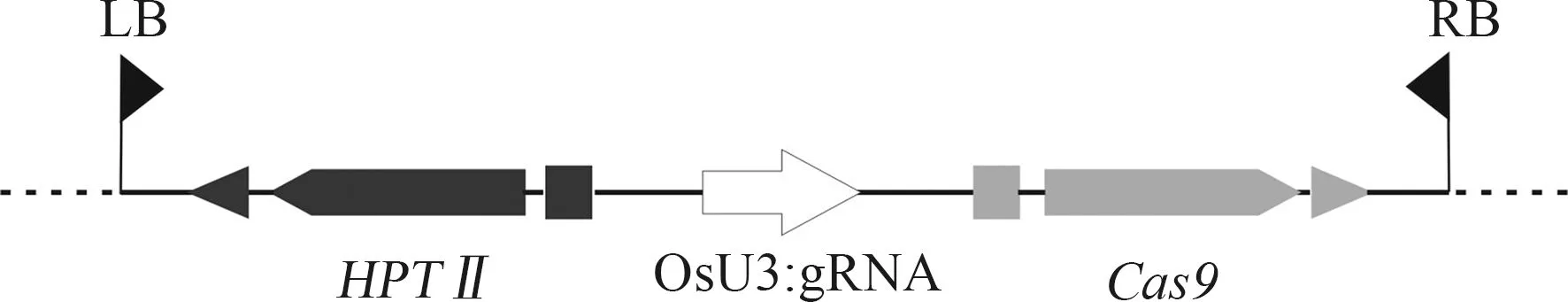
LB: T-DNA的左边界Left boundary of T-DNA;HPTⅡ: 潮霉素磷酸转移酶基因HygromycinBphosphotransferasegene; OsU3:gRNA: OsU3启动子与引导RNA的融合结构Fusion structure of OsU3 promoter and guide RNA;Cas9: CRISPR相关蛋白核酸酶9基因CRISPR-relatedproteinnuclease9gene; RB: T-DNA的右边界Right boundary of T-DNA.
图1Pita基因编辑载体pTGE1的T-DNA区
Fig. 1 T-DNA region ofPitagene editing vector pTGE1
1.2.3 T0代阳性转基因植株的获得与验证 以野生型‘日本晴’的成熟胚为外植体,将构建的pTGE1表达载体转化到农杆菌LBA4404中并进行侵染,采用质量浓度50 mg·L-1潮霉素进行筛选,获得再生组培苗,炼苗后移栽到土壤中,置于温室内进行培养,培养条件为昼温30 ℃、夜温28 ℃、空气相对湿度70%、光照度30 000 lx、光照时间16 h·d-1。
取移栽后存活再生苗的新鲜叶片,采用CTAB法[22]提取总DNA。以T-DNA区域的Cas9基因序列为模板设计1对PCR扩增引物进行阳性鉴定。其中,引物Cas9S的序列为5′-TGGTGGAAGAGGATAA
GAAGC-3′,引物Cas9AS的序列为5′-TCAAACAGT
GTCAGGGTCAGC-3′。扩增条件:95 ℃预变性4 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸40 s,35个循环;72 ℃延伸10 min。
1.2.4 突变体鉴定 设计1对跨越靶位点的引物对T0代阳性转基因植株的Pita基因进行扩增。其中,引物Pita787的序列为5′-TGCCTACTTCTTTCCACAT
CA-3′,引物Pita1900的序列为5′-ATCCGAAGACTG
ATGCTGATGTT-3′。将扩增产物进行测序,用Sequencher 4.5软件对测序结果进行序列比对,统计突变类型和突变体类型。
1.2.5 不含T-DNA区的纯合突变体筛选 利用引物Pita787和Pita1900扩增Pita基因编辑位点上、下游的DNA片段,对T-DNA阴性植株进行Pita基因突变位点鉴定。具体操作如下:播种T0代纯合突变体植株自交获得的T1代种子,根据PCR扩增结果确定T-DNA阴性植株,从T1代T-DNA阴性植株各株系材料中分别随机选择3株植株,对其DNA进行PCR扩增,将扩增产物进行TA克隆,随机选取4个单克隆进行测序。若4个单克隆中Pita基因编辑位点的碱基突变完全一致,则判定为纯合突变体。按照上述方法对T2代植株进行检测,确定Pita基因编辑材料是否稳定遗传。
1.2.6 纯合突变体对稻瘟病的抗性分析 将稻瘟病菌小种CH199进行活化培养,待培养至第12天时洗脱并收集稻瘟病菌的孢子,调整至1 mL溶液中包含2.5×105个孢子[23],用于样株的稻瘟病菌喷雾接种。分别以抗稻瘟病的籼稻品种‘9311’和野生型‘日本晴’为对照进行稻瘟病抗性分析。取健康饱满的‘9311’、野生型‘日本晴’和基因编辑材料V1-38-5的T2代植株种子进行萌发,发芽5 d后移栽到土壤中,在同一盆中分区种植,每个区种植10株;移栽8 d后进行稻瘟病菌喷雾接种,接种稻瘟病菌5 d后对每个区的样株分别进行病级调查及评定,最终计为样株的平均病级[24]。
1.2.7 纯合突变体稻瘟病病程相关基因的相对表达量分析 分别在接种稻瘟病菌小种CH199的0(未接种)、12和24 h时采集基因编辑材料V1-38-5的T2代植株和野生型‘日本晴’植株的嫩叶,用TRIzol试剂(美国Invitrogen公司)提取总RNA,并反转录成cDNA;以EF-257基因为内参基因[15],采用qRT-PCR技术检测水稻稻瘟病菌病程相关基因PR2[25]、PR1b[26]、PR3[27]、PDR(引物F的序列为5′-ATCCACTCACCGCAAGTAACAG-3′,引物R的序列为5′-CTCCAGGAAGACCAACTAAAGCA-3′)、PBZ1[28]和E2F[29]的表达水平。采用2-ΔΔCt法计算各基因的相对表达量。
1.3 数据统计分析
使用SPSS 16.0统计分析软件对病级和各病程相关基因的相对表达量进行差异显著性分析。
2 结果和分析
2.1 水稻Pita基因突变体鉴定
利用CRISPR/Cas9技术对‘日本晴’的Pita基因进行编辑,利用农杆菌介导的水稻转化方法将Pita基因编辑载体pTGE1转化到野生型‘日本晴’的成熟胚中,经过潮霉素筛选及PCR扩增,共获得8株T0代阳性转基因植株。
使用引物Pita787和Pita1900对获得的8株T0代阳性转基因植株进行PCR扩增,测序和鉴定结果(表1)显示:有7株T0代阳性转基因植株的Pita基因发生了突变,突变率为87.5%;经鉴定,在这7株突变体中,有1株为杂合体,6株为纯合体,纯合突变率为85.7%。突变类型包括碱基置换、碱基插入和碱基缺失。
表1 粳稻品种‘日本晴’Pita基因突变体的测序和鉴定结果
Table 1 Sequencing and identification results of mutants ofPitagene inOryzasativasubsp.japonica‘Nipponbare’

编号No.突变位置1)Mutation position1)突变情况Mutation status突变类型Mutation type突变体类型Mutant typeV1-2621st,22ndAT置换成G AT substituting to G碱基置换Base substitution纯合体 HomozygoteV1-2921st,22ndAT置换成G AT substituting to G碱基置换Base substitution纯合体 HomozygoteV1-3520th,21stT插入 T insertion碱基插入Base insertion纯合体 HomozygoteV1-3820th,21stT插入 T insertion碱基插入Base insertion纯合体 HomozygoteV1-3920th,21stT插入 T insertion碱基插入Base insertion纯合体 HomozygoteV1-4720th,21stT插入 T insertion碱基插入Base insertion纯合体 HomozygoteV3-5520th,21stCA缺失 CA deletion碱基缺失Base deletion杂合体 Heterozygote
1)从起始密码子开始的碱基位置Base position from start codon.
2.2 不含T-DNA区的纯合突变体筛选
根据PCR扩增结果共获得42株T1代T-DNA阴性植株(部分检测结果见图2),经鉴定这42株T1代T-DNA阴性植株全部为纯合突变体。同样,从来源于基因编辑材料T1代V1-38-5自交的T2代植株中,随机选取8株进行PCR扩增及测序分析,结果(图3)显示这8株T2代植株全部为纯合突变体,说明Pita基因编辑材料遗传稳定。
2.3 纯合突变体对稻瘟病的抗性分析
以抗稻瘟病籼稻品种‘9311’和野生型‘日本晴’为对照,对Pita基因编辑材料V1-38-5的T2代(即纯合突变体)进行稻瘟病菌小种CH199接种,并对其稻瘟病抗性进行分析,结果(图4)显示:接种稻瘟病菌小种CH199后,‘9311’的叶片没有出现病斑,平均病级为0.0±0.0,说明‘9311’未感病;野生型‘日本晴’的叶片病斑面积较小,平均病级为3.0±0.2,表现为轻度至中度感病;V1-38-5的T2代植株叶片病斑面积较大,平均病级为4.1±0.2,表现为重度感病。V1-38-5的T2代植株的平均病级与野生型‘日本晴’和‘9311’在统计学上存在极显著(P<0.01)差异,说明Pita基因被编辑后 ‘日本晴’对稻瘟病菌的抗性降低。
2.4 纯合突变体稻瘟病病程相关基因的相对表达量分析
qRT-PCR分析结果(图5)表明:与未接种稻瘟病菌(0 h)时相比,基因编辑材料V1-38-5的T2代植株PR2、PR1b、PR3、PDR、PBZ1和E2F6个稻瘟病病程相关基因的相对表达量均在接种稻瘟病菌12和24 h时明显升高;野生型‘日本晴’PR2基因的相对表达量在接种稻瘟病菌24 h时明显升高,其PR3和E2F基因的相对表达量在接种稻瘟病菌12和24 h时也明显升高,而其PR2基因的相对表达量在接种稻瘟病菌12 h时明显降低,其PR1b、PDR和PBZ1基因的相对表达量在接种稻瘟病菌12和24 h时也明显降低。

M: DNA标记 DNA marker; 1: 阴性对照 Negative control; 2-12,14-24: T-DNA阳性植株 T-DNA positive plants; 13: T-DNA阴性植株T-DNA negative plant.
图2 粳稻品种‘日本晴’Pita基因编辑材料V1-38-5的T-DNA阴性植株的部分检测结果
Fig. 2 Partial detection result of T-DNA negative plants ofPitagene editing material V1-38-5 ofOryzasativasubsp.japonica‘Nipponbare’
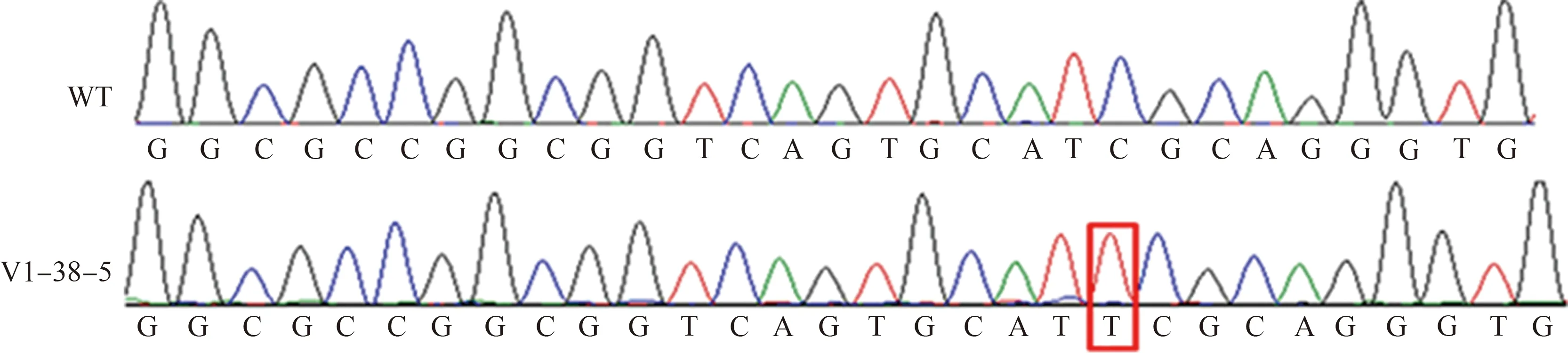

图3 粳稻品种‘日本晴’Pita基因编辑材料V1-38-5的T2代植株纯合突变体鉴定
Fig. 3 Homozygous mutant identification of T2generation plants ofPitagene editing material V1-38-5 ofOryzasativasubsp.japonica‘Nipponbare’

9311: 籼稻品种‘9311’Oryzasativasubsp.indica‘9311’; WT: 野生型 ‘日本晴’ Wild type of ‘Nipponbare’; V1-38-5:Pita基因编辑材料V1-38-5的T2代植株T2generation plants ofPitagene editing material V1-38-5.
图4 接种稻瘟病菌小种CH199后粳稻品种‘日本晴’Pita基因编辑材料V1-38-5的T2代植株抗性分析
Fig. 4 Resistance analysis on T2generation plants ofPitagene editing material V1-38-5 ofOryzasativasubsp.japonica‘Nipponbare’ after inoculatingMagnaportheoryzaestrain CH199


图5 接种稻瘟病菌小种CH199后粳稻品种‘日本晴’Pita基因编辑材料V1-38-5的T2代植株病程相关基因的表达分析
Fig. 5 Analysis on expression of pathogenesis-related genes in T2generation plants ofPitagene editing material V1-38-5 ofOryzasativasubsp.japonica‘Nipponbare’ after inoculatingMagnaportheoryzaestrain CH199
比较发现,与野生型‘日本晴’相比,基因编辑材料V1-38-5的T2代植株PR2和PR1b基因的相对表达量在接种稻瘟病菌12 h时较低,其PR2、PR1b、PR3和E2F基因的相对表达量在接种稻瘟病菌24 h时也较低。
3 讨论和结论
稻瘟病既是水稻易遭受的严重病害之一,也是限制水稻高产和稳产的重要因子。Pita基因是水稻中的单拷贝抗稻瘟病基因,因此,研究Pita基因有助于人们更好地了解稻瘟病的发生机制。CRISPR/Cas9技术可通过编辑目的基因改良农作物的农艺性状,利用CRISPR/Cas9技术开发的遗传材料常存在多种突变类型[30],可为植物育种研究提供丰富的遗传材料。本研究利用CRISPR/Cas9技术对水稻Pita基因进行了编辑,结果表明:T0代阳性转基因植株的Pita基因突变率达87.5%,且T0代的纯合突变率高达85.7%;突变类型包括碱基置换、碱基插入和碱基缺失,并且这些突变可稳定遗传。
相关研究[5-6]表明:Pita基因通过特异性识别AVR-Pita可产生超敏反应,激活抗病防御基因的表达,并且Pita基因第918位氨基酸的突变决定了该基因对AVR-Pita的识别特异性,当该位点的丙氨酸被丝氨酸取代后,Pita基因与AVR-Pita的互作被削弱,从而导致水稻感染稻瘟病。本研究使用的转基因受体材料为‘日本晴’,其Pita基因的第918位为丝氨酸,接种稻瘟病菌IC17后表现为重度感病[7],接种稻瘟病菌小种81278ZB15和GUY11后表现为抗病[31],本研究接种稻瘟病菌小种CH199后则表现为轻度至中度感病,推测这可能是由于使用的稻瘟病菌小种不同所致。本研究中,‘日本晴’Pita基因编辑材料V1-38-5的T2代植株表现为重度感病,这可能是因为Pita基因的N端发生了碱基非3整数倍的替代、插入或缺失,致使该基因的编码框提前终止,从而导致植株丧失抗稻瘟病菌的能力。另外,‘日本晴’Pita基因编辑材料V1-38-5的T2代植株各病程相关基因的相对表达量总体上明显低于野生型‘日本晴’,说明水稻Pita基因的突变可能影响病程相关基因对稻瘟病菌的敏感性,导致植株抗病能力下降,更易感病。
综上所述,利用CRISPR/Cas9技术对水稻Pita基因进行定点编辑,能够获得稳定遗传且更易感病的纯合突变体材料,这些材料为水稻抗性品种选育和品质改良奠定了材料基础,从而在一定程度上加速水稻定向分子育种的进程。

