分蘖相关基因在再生稻腋芽萌发期的表达分析
朱容 刘歆 吴建伟 刘念 梁大成 刘章勇


摘要:【目的】明確分蘖相关基因在再生稻腋芽萌发期的表达动态变化,为阐明水稻再生季腋芽发育的调控机理提供理论依据。【方法】以再生分蘖能力强的材料i21和再生分蘖能力弱的材料i89为研究对象,运用实时荧光定量PCR(qRT-PCR)检测8个分蘖相关基因在水稻头季(分蘖始期和拔节孕穗期)和再生季不同时期(头季去穗后1、24、48和72 h)的表达动态变化,并分析分蘖相关基因间及其与最大再生分蘖的相关性,筛选出与腋芽生长相关性高的基因。【结果】自头季稻稻穗收割后,材料i21和i89的再生分蘖数的变化趋势基本一致,均随着腋芽的生长再生分蘖数逐渐增多,收割后20 d达最多并趋于稳定,但材料i21再生季发苗多且快,再生分蘖发生速度明显高于材料i89;材料i21和i89的最大再生分蘖数为33和10个。LAX1、LAX2和MOC1基因的相对表达量在不同材料不同时期间均存在明显差异,其中自头季去穗后,除MOC1基因在头季去穗后72 h的相对表达量外,LAX1、LAX2和MOC1基因在水稻再生分蘖形成期均表现为材料i21中的相对表达量极显著高于材料i89(P<0.01,下同),MOC1基因在头季去穗后1 h达峰值,LAX1和LAX2基因在头季去穗后24 h达峰值,随后显著降低(P<0.05,下同)并趋于稳定。头季去穗后,除TAD1基因在头季去穗后24 h时外,材料i89中D10、HTD1、OsTB1和TAD1基因的相对表达量均极显著高于材料i21中的相对表达量,D10和HTD1基因在头季去穗后1 h达峰值,OsTB1和TAD1基因在头季去穗后24和48 h达峰值,D27基因在材料i21和i89中的相对表达量均先升高后降低,且头季去穗后24~72 h,D27基因在材料i21中的表达量极显著高于材料i89。最大再生分蘖数分别与LAX2和MOC1基因的表达水平呈极显著和显著正相关,与D10、HTD1、OsTB1和TAD1基因的表达量呈极显著负相关。此外,不同基因的表达水平也存在不同相关性。【结论】LAX1、LAX2和MOC1基因高表达有利于水稻再生季分蘖的形成,D10、HTD1、OsTB1和TAD1基因高表达会抑制水稻再生季分蘖的形成。不同分蘖相关基因间存在相互作用,共同调控水稻再生季分蘖的形成。
关键词: 分蘖基因;再生稻;腋芽;表达分析
中图分类号:S511.035.3 ; 文献标志码: A 文章编号:2095-1191(2021)10-2632-09
Abstract:【Objective】 The expression of tiller-related genes during axillary bud outgrowth in ratoon rice were profiled to provide insight into the molecular mechanisms underlying axillary bud development in the ratoon season. 【Method】 The rice varieties i21 and i89 with high and low ratooning abilities, respectively, were used as the study materials. Real-time fluorescence quantitatire PCR (qRFRCR) was used to characterize the expression dynamics of eight tiller-related genes at the stages of tiller initiation, elongation and booting of the first season and at 1, 24, 48 and 72 h after heading removal in the ratoon season. The correlation between tiller-related genes and maximum ratoon tillers was analyzed and the genes with high correlation with axillary bud growth were selected.【Result】The number of ratoon tillers in i21 and i89 increased gradually with the growth of axillary buds and reached a stable maximum 20 d after the first-season harvest. However, i21 ratoon tillers grew much faster than those of i89 and the maximum number of ratoon tillers of i21 and i89 were 33 and 10, respectively. The relative expression levels of LAX1, LAX2 and MOC1 genes differed significantly in the two varieties. The relative expression levels of LAX1, LAX2 and MOC1 genes in i21 were significantly higher than those in i89 during the ratooning period (P<0.01, the same below). MOC1 reached peak levels at 1 h after beheading, whereas the expressions of LAX1 and LAX2 peaked at 24 h after beheading then decreased significantly (P<0.05, the same below) to stable levels thereafter. During ratooning, the relative expression levels of D10, HTD1, OsTB1 and TAD1 genes in i89 were significantly higher than those in i21, except for TAD1 at 24 h after beheading. D10 and HTD1 genes peaked at 1 h after beheading, while OsTB1 and TAD1 genes reached their peak values later at 24 and 48 h after beheading. The relative expression levels of D27 in i21 and i89 increased initially but decreased at 24-72 h after heading removal, although this decrease in i21 was significantly less than that in i89. The maximum tiller number was positively and significantly correlated with the expression levels of LAX2 and MOC1 and negatively correlated with those of D10, HTD1, OsTB1 and TAD1. In addition, different gene expression levels also had different correlations.【Conclusion】The high expression of LAX1, LAX2 and MOC1 genes was beneficial to the formation of ratoon tillers, while the high expression of D10, HTD1, OsTB1 and TAD1 genes inhibited ratoon till outgrowth. This work indicates that the different tiller-related genes interacted to jointly regulate ratoon tiller formation.
Key words:tiller gene; ratoon rice; axillary bud; expression analysis
Foundation item:Key Scientific and Technological Project of Hubei (2020BBA044)
0 引言
【研究意义】水稻(Oryza sativa L.)是我国乃至全世界重要的粮食作物,其产量一直备受关注(刘贵富等,2018)。目前增加水稻播种面积和单产日益困难,增加复种指数成为提高稻谷产量的关键举措(Ray Foley,2013;Dong et al.,2017)。再生稻作为一种土地高效利用、资源节约型的栽培模式,具有米质优、低污染、低能耗的特点,是提高复种指数、增加稻谷产量的有效途径(朱永川等,2013;徐富贤,2015;Jiang et al.,2016;Tao et al.,2016;刘歆等,2020)。由于分蘖力是水稻的重要农艺性状,直接影响稻谷产量(李学勇等,2003;李经勇等,2009;孙秀红和胡波,2020),因此,腋芽萌发能力是再生稻产量的关键因素(李贵勇等,2015;习敏等,2017;胡香玉等,2019;汪浩等,2019)。对再生稻分蘖力进行分子生物学方面的研究,以期解析再生稻分蘖产生和调控的分子机理,对选育强再生力的水稻品种具有重要的指导意义。【前人研究进展】目前,已研究证实水稻分蘖相关基因MONOCULM1(MOC1)、LAX1、LAX2、D10、HTD1、D27、OsTB1和TAD1的表达影响水稻分蘖形成,其中MOC1基因是第一个发现的控制水稻分蘖的单蘖基因,其等位基因为GNP6(Li et al.,2003;Shao et al.,2019;Zhang et al.,2021),编码植物特有的GRAS家族蛋白(Bolle,2004),在腋生分生组织的发生、分蘖芽的形成过程中发挥促进作用;LAX1基因在植株的侧生分生组织的起始发育发挥重要作用,编码非典型的bHLH(basic-Helix-Loop-Helix)型转录因子,在顶端分生组织与新形成的分生组织之间的边界区域表达,主要调节水稻腋芽原基的形成(Maekawa et al.,2001;Komatsu et al.,2003;Oikawa and Kyozuka,2009);LAX2基因编码一个核蛋白,该蛋白含有植物特有的保守结构域,能与LAX1发生蛋白互作,Gnp4为其等位基因,LAX2/Gnp4基因编码一个功能未知的核蛋白,调控水稻腋生分生组织的形成(Tabuchi et al.,2011;Zhang et al.,2011);D10、HTD1和D27基因参与独脚金内酯(Strigolactones,SLs)的生物合成,SLs是一种控制植物分枝的新激素,其可抑制植物分枝和侧芽生长(Gomez-Roldan et al.,2008;Umehara et al.,2008);D10基因与拟南芥的MAX4、豌豆的RMS1和矮牵牛的DAD1是直系同源基因,编码胡萝卜素裂解双加氧酶OsCCD8,该酶抑制侧芽的向外生长从而使水稻分蘖数减少(Arite et al.,2007);HTD1基因编码胡萝卜素裂解双加氧酶OsCCD7,该酶抑制水稻分枝的生长从而减少水稻分蘖数(Zou et al.,2005,2006);D27基因编码定位于叶绿体上的α-β类胡萝卜素异构酶含铁蛋白,主要在茎顶端和根部表达,在SLs生物合成中发挥关键作用,使水稻分蘖数减少(Lin et al.,2009;Adrian et al.,2012);OsTB1/FC1位于同一基因位点,作用于SLs的下游,编码含TCP结构域的bHLH转录因子,对分蘖发挥负调控作用;OsTB1基因对分蘖数的负调控作用是通过控制腋芽的生长过程来实现,腋芽中OsTB1基因过量表达会阻碍其形成,最终导致水稻分蘖数显著减少,而OsTB1基因功能丧失型突变体fc1分蘖数显著增加(Takeda et al.,2003;Minakuchi et al.,2010);TAD1基因编码细胞分裂后期启动复合物(Anaphase-promoting complex,APC/C)的共激活子,与MOC1基因作用于同一途径,通过细胞周期依赖的方式降低靶标MOC1基因的表达水平,抑制腋生分生组织的起始和形成,从而抑制其分蘖(Lin et al.,2012;Xu et al.,2012)。以上研究表明MOC1、LAX1和LAX2基因促进水稻分蘖的形成,而D10、HTD1、D27、OsTB1和TAD1基因抑制水稻分蘖的发生。【本研究切入点】虽然分蘖相关基因的功能及其对水稻分蘖形成影响已有大量研究报道,但目前鲜见有关分蘖相关基因在水稻再生季的表达特性的研究报道。【拟解决的关键问题】以本课题组筛选出的再生力极端表型水稻品种为材料,利用实时荧光定量PCR(qRT-PCR)检测水稻分蘖相关基因在极端表型材料中再生季腋芽发生不同时期的表达差异,以期为水稻再生季穗数形成的分子機理提供理论依据。
1 材料与方法
1. 1 试验材料
192份核心水稻种质材料,由中国农业科学院作物科学研究所提供,用于筛选再生能力极端表型的材料。植物总RNA提取试剂盒E.Z.N.A.® Plant RNA Kit购自上海北诺科技生物科技有限公司;2×EasyTaq PCR SuperMix(AS111-01)购自北京全式金生物技术有限公司;cDNA反转录试剂盒Fast Quant RT Kit(with gDNA)和荧光定量试剂盒SuperReal PreMix Plus(SYBR Green)购自天根生化科技(北京)有限公司;100 bp DNA Ladder、1 kb DNA Ladder、4S Green Plus染料和6×Loading Buffer均购自生工生物工程(上海)股份有限公司;Tris碱、硼酸和EDTA购自北京索莱宝科技有限公司。5×TBE贮存液是由54 g/L Tris碱、27.5 g/L硼酸、20 mL 0.5 mol/L EDTA(pH 8.0)配制而成,将其稀释10倍成0.5×TBE;50×TAE贮存液用242 g/L Tris碱、37.2 g/L EDTA和57.1 mL醋酸加水定容至1 L配制。主要设备仪器:超速离心机(Eppendorf,德国)、PCR仪(Bio-Rad,美国)、实时荧光定量PCR仪(Bio-Rad CFX96TM Real-Time System,美国)、凝胶成像系统(Bio-Rad,美国)、Nanodrop ND-1000分光光度计(美国)和水平琼脂糖凝胶电泳仪(北京君意东方电泳设备有限公司)。
1. 2 试验方法
1. 2. 1 材料筛选 192份核心水稻种质材料于2018年分别在荆州、海南陵水和广东英德共3个生态点种植,调查头季和再生季最大分蘖数,筛选出再生能力极端表型的材料,编号为i21和i89,其中,i21为再生分蘖能力强的材料,i89为再生分蘖能力弱的材料。
1. 2. 2 田间种植 筛选出的材料于2019年夏季在长江大学农学院试验基地采用盆栽方式种植,塑料盆上直径36 cm、高31 cm,每盆装18±0.3 kg干土,土表距离盆口约4 cm。3月31日播种,4月25日移栽,每盆种4穴,单穴单苗,每个品种3盆,3次重复,正常水肥管理。
1. 2. 3 再生季分蘖动态调查 自头季稻去穗后,每隔5 d调查5株水稻,挂牌记录再生分蘖数,直至达到最大分蘖为止。
1. 2. 4 取样方法 设6次取样时期,分别为头季分蘖始期(CK)、头季拔节孕穗期(R1)、头季去穗后1 h (R2)、头季去穗后24 h(R3)、头季去穗后48 h(R4)和头季去穗后72 h(R5),取来自5个单株节间倒2节腋芽的混合样品(CK为5个单株水稻基部2 cm左右的混合样品,统一在水稻达到4个分蘖时取样),取样后立即经液氮冷冻30 min并于-80 ℃冰箱保存。每个时期设3个生物学重复。
1. 2. 5 总RNA提取及反转录合成cDNA 利用E.Z.N.A.® Plant RNA Kit植物总RNA提取试剂盒提取水稻样品总RNA,用1%琼脂糖凝胶电泳检测总RNA的完整性,取1 µL用Nanodrop ND-1000分光光度计测定其纯度和浓度,结果显示RNA提取效果好,且RNA无降解;OD260/OD280为1.8~2.0,纯度较好,可满足后续的试验要求。
利用Fast Quant RT Kit(with gDNA)试剂盒反转录合成cDNA。以Actin为内参基因,采用普通PCR检测各材料cDNA质量,结果证实cDNA无gDNA污染,质量较好,可用于后续试验。
1. 2. 6 引物设计 通过国家水稻数据中心(http://www.ricedata.cn/)和已报道的分蘖基因的相关文献资料(Komatsu et al.,2003;Li et al.,2003;Takeda et al.,2003;Arite et al.,2007;Hong et al.,2009),查找分蘖相关基因信息,然后通过NCBI数据库(https://www.ncbi.nlm.nih.gov/)获取分蘖相关基因LAX1、LAX2、MOC1、D10、HTD1、D27、OsTB1和TAD1基因序列,最后通过BLAST比对确定其编码蛋白的保守区域,然后利用Primer Premier 5.0设计长度合适、GC含量合理、无发卡结构、无错配及无杂带的引物(表1)。
1. 2. 7 实时荧光定量PCR检测 以Actin作为内参基因,采用实时荧光定量PCR检测8个水稻分蘖相关基因的表达情况。按照SuperReal PreMix Plus说明配制反应体系20.0 µL:10 µL 2×SuperReal PreMix Plus,cDNA模板2.0 µL,10 µmol/L上、下游引物各0.6 µL,ddH20补足至20.0 µL。反应结束后,通过检测熔解曲线确定反应产物特异性,采用比较CT值法(2-△△Ct)计算基因的相对表达量(Gomez-Roldan et al.,2008)。
1. 3 统计分析
使用Excel 2007进行数据整理,用SPSS 21.0对数据进行显著性分析。
2 结果与分析
2. 1 再生分蘖特性比较结果
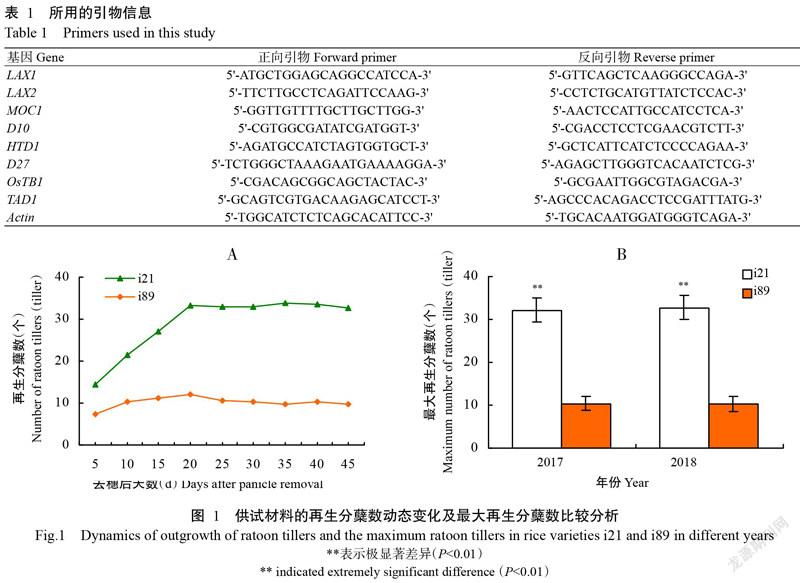
由图1-A可知,自头季稻稻穗收割后,材料i21和i89再生分蘖数的变化趋势基本一致,均随着腋芽的生长再生分蘖数逐渐增多,收割后20 d达最多并趋于稳定,但材料i21再生季发苗多且快,再生分蘖发生速度明显高于材料i89。由图1-B可知,这2个材料的最大再生分蘖数存在极显著差异(P<0.01,下同),其中材料i21的最大再生分蘖数为33个;材料i89的最大再生分蘖数为10个,且不同年限间2个材料的再生能力表现稳定(图1)。
2. 2 分蘖正调控基因在不同材料不同时期间的表达分析结果
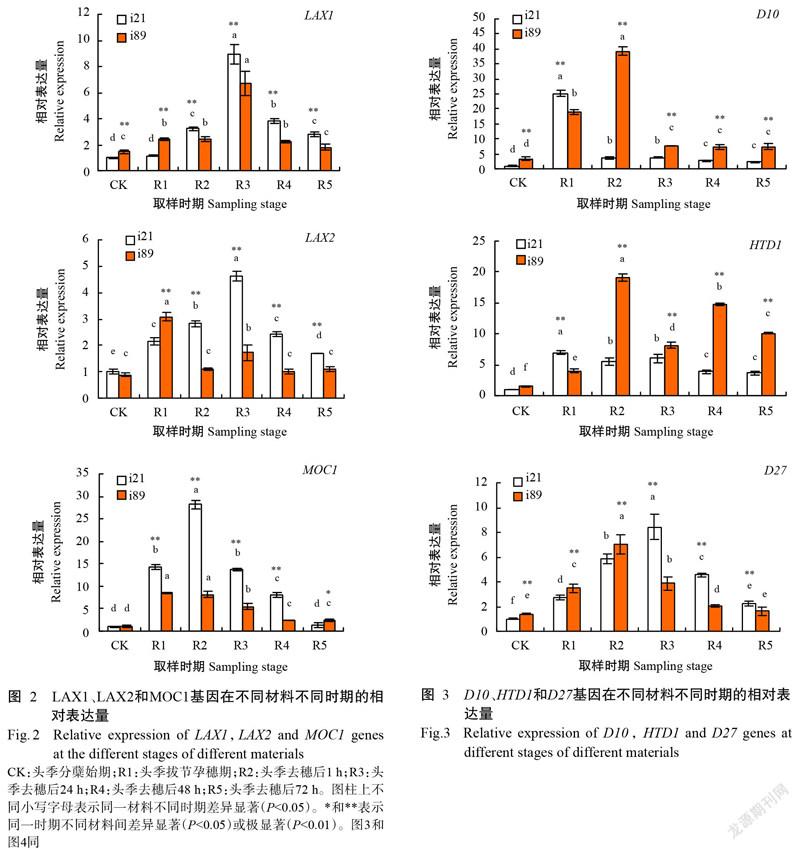
由图2可知,自头季去穗后,LAX1基因在材料i21和i89的相对表达量显著升高(P<0.05,下同),至头季去穗后24 h达峰值,随后显著降低并趋于稳定;LAX2基因在材料i21的相對表达量变化趋势与LAX1基因基本一致,均呈先升高再降低的变化趋势,在头季去穗后24 h达峰值,但LAX2基因在材料i89中的表达量自头季去穗后呈降低─升高─降低的变化趋势,最后逐渐趋于稳定;MOC1基因在材料i21和i89的相对表达量的变化趋势与LAX2基因基本一致。
由图2还可知,自头季去穗后,除MOC1基因在头季去穗后72 h外,LAX1、LAX2和MOC1基因在水稻再生分蘖形成期(R2~R5)均表现为再生分蘖能力强的材料i21中的相对表达量极显著高于分蘖能力弱的材料i89。可见,LAX1、LAX2和MOC1基因高表达可促进水稻再生季分蘖形成,表明这3个基因是参与水稻分蘖芽形成的关键基因,对分蘖芽的形成发挥促进作用,可提高水稻分蘖数。
2. 3 分蘖负调控基因在不同材料不同时期的表达分析结果
2. 3. 1 SLs合成基因的表达差异 由图3可知,自头季去穗后,D10和HTD1基因在材料i21中的相对表达量显著降低并逐渐趋于稳定;D10基因在材料i89的相对表达量显著升高,头季去穗后1 h达峰值,随后显著降低并趋于稳定;材料i89中HTD1基因的相对表达量在去头季穗后1 h显著升高并达峰值,随后先降低再升高并逐渐趋于稳定,且其在材料i89的相对表达量均高于材料i21;D27基因在材料i21和i89中的相对表达量先升高后降低,且头季去穗24 h后其在材料i21中的相对表达量极显著高于材料i89,说明D10和HTD1基因高表达会抑制水稻再生季分蘖形成。
2. 3. 2 OsTB1和TAD1基因的表达差异 由图4可知,自头季去穗后,OsTB1基因在材料i21中的相对表达量出现2次先升高后降低,头季去穗后48 h达最大值,OsTB1基因在材料i89中的相对表达量先升高后降低,头季去穗后24 h达峰值,随后显著降低,且OsTB1基因在材料i21中的相对表达量均显著低于材料i89;TAD1基因在材料i21和i89中的相对表达量均呈先升高后降低趋势,但在材料i21中的相对表达量在头季去穗后24 h达峰值,在材料i89中的相对表达量在头季去穗后48 h达峰值,且其在材料i89中的相对表达量显著高于材料i21,说明OsTB1和TAD1基因高表达可抑制水稻再生季分蘖形成。
2. 4 分蘖相关基因间及其与最大再生分蘖数间的相关分析结果
由表2可知,最大再生分蘖数分别与LAX2和MOC1基因的表达水平呈极显著和显著正相关,与TAD1、OsTB1、D10和HTD1基因的表达水平呈极显著负相关。此外,不同基因的表达水平也存在不同相关性,D27基因与LAX1、LAX2和MOC1基因呈极显著正相关;LAX1基因与LAX2基因、LAX2基因与MOC1基因、D10基因与HTD1基因均呈极显著正相关;LAX2基因与HTD1基因、D27基因与OsTB1基因均呈极显著负相关;OsTB1基因与MOC1和LAX2基因均呈显著负相关;D27基因与TAD1基因也呈显著负相关。可见,分蘖相关基因的表达水平与水稻再生季分蘖形成密切相关,而且不同分蘖相关基因也存在相互作用,共同调控水稻再生季分蘖的形成。同时,进一步证实LAX1、LAX2和MOC1基因的表达水平升高有利于水稻再生季分蘖形成,D10、HTD1、OsTB1和TAD1基因的表达水平升高会抑制水稻再生季分蘖形成。
3 讨论
水稻分蘖是一个受遗传基因和环境双重影响的复杂数量性状(谭震波等,1997;杨川航等,2012;李兴星等,2016)。已有研究表明,LAX1、LAX2和MOC1基因均能促进水稻腋芽的生长萌发,正向调控水稻分蘖的形成(Maekawa et al.,2001;Li et al.,2003;Tabuchi et al.,2011)。本研究发现,除MOC1基因在头季去穗后72 h的相对表达量外,LAX1、LAX2和MOC1基因在水稻再生分蘖形成期(R2~R5)均表现为再生分蘖能力强的材料i21中的相对表达量极显著高于分蘖能力弱的材料i89,说明LAX1、LAX2和MOC1基因对于水稻再生分蘖的形成发挥促进作用,与其在水稻头季的研究结果一致,均能正向调节水稻分蘖的形成。MOC1基因在头季去穗后72 h的相对表达量表现为再生分蘖能力强的材料i21显著低于分蘖能力弱的材料i89,与头季去穗后其他时期(R2~R4)刚好相反,其原因可能是头季去穗后72 h水稻再生腋芽已萌发,处于伸长生长阶段,MOC1基因的相对表达量升高,与其他通路的基因存在关联,抑制了分蘖,具体的机制尚不清楚,有待进一步研究探索。
D10、HTD1、D27、OsTB1和TAD1基因是从一系列水稻突变体材料定位克隆出的分蘖相关基因,均可抑制水稻侧芽的生长,从而负调节水稻分蘖的形成(Takeda et al.,2003;Zou et al.,2005;Arite et al.,2007;Lin et al.,2009;Xu et al.,2012)。本研究中,D10、HTD1、OsTB1和TAD1基因均在再生分蘗能力弱的材料i89中相对表达量较高,说明这些基因可抑制水稻再生分蘖的形成,与其在水稻头季的研究结果一致,均能负向调节水稻分蘖的形成。D27基因在头季拔节孕穗期和头季去穗后1 h的相对表达量呈现出i89高于i21,在头季去穗后24~72 h的相对表达量为i21高于i89,与前人研究结果不一致,导致无法判断其对再生分蘖的形成是否存在影响,其原因可能是D27基因在无效分蘖的控制中起作用。造成再生季中基因表达差异的原因很多,其一,再生季的生长过程本身是一个复杂过程,其生长环境不同,基因表达调控也会存在很大差异;其二,材料本身的遗传背景差异很大,造成最终分蘖结果存在差异;其三,头季去穗后会产生基因表达量无变化或产生与基因功能不一致的结果,这可能是植株本身对外界变化的适应能力体现(Cline and Oh,2006)或是再生季的调控过程本身的不同所导致,推测水稻再生季通过其他途径调控再生苗的生长。
水稻分蘖相关基因形成一个复杂的调控网络,且存在组织特异性、生长时期特异性及特异性表达。Tabuchi等(2011)通过对稀穗突变体lax1与lax2的研究发现,LAX2是通过与LAX1相互作用从而调控水稻分蘖的形成;Xu等(2012)研究表明TAD1基因可通过细胞周期依赖的方式降低MOC1基因的表达水平从而抑制水稻分蘖的发生。本研究相关分析结果显示,LAX1基因与LAX2基因的呈极显著正相关(相关系数为0.780**),MOC1基因与TAD1基因呈显著负相关(相关系数为-0.490*),与前人的研究结果(Tabuchi et al.,2011;Xu et al.,2012)一致。此外,本研究还发现,不同分蘖相关基因间存在复杂的相关关系,说明水稻再生分蘖的形成可能与水稻头季分蘖一样,存在着复杂的分子调控机制,且不同基因之间存在相互作用,共同调控水稻再生季分蘖的形成,但其具体的分子遗传机理还有待进一步研究。
本研究分析了分蘖相关基因在再生稻腋芽萌发期的表达差异,后续的研究工作可从以下两方面开展:一方面可利用本课题已有的遗传多样性丰富的材料群体进行重测序,鉴定其基因型,并结合再生稻分蘖能力的表型进行全基因组关联分析,获得显著性SNP位点;另一方面,构建F2群体,对其进行重测序,鉴定基因型,结合表型进行QTL分析,从而鉴定再生季分蘖发生的关键调控基因。借助分子手段深入研究水稻再生季分蘖的基因调控网络,对于揭示再生稻的遗传机理具有重要意义。
4 结论
LAX1、LAX2和MOC1基因高表达有利于水稻再生季分蘖的形成,D10、HTD1、OsTB1和TAD1基因高表达会抑制水稻再生季分蘖的形成。不同分蘖相关基因间存在相互作用,共同调控水稻再生季分蘖的形成。
参考文献:
胡香玉,钟旭华,彭碧琳,刘彦卓,黄农荣,梁开明,潘俊峰,傅友强. 2019. 不同氮肥运筹下低桩机收再生稻的产量和经济效益[J]. 中国稻米,(4):1-7. doi:10.3969/j.issn.1006-8082. 2019.04.004. [Hu X Y,Zhong X H,Peng B L,Liu Y Z,Huang N R,Liang K M,Pan J F,Fu Y Q. 2019. Grain yield and profit of machine-harvested low stubble ratoon rice under different nitrogen management[J]. China Rice,(4):1-7.]
李贵勇,谭丽明,刘玉文,黄洁,陈双云,夏琼梅,龙瑞平,杨从党. 2015. 再生稻的产量形成及其高产群体分析[J]. 江西农业大学学报,37(4):577-583. [Li G Y,Tan L M,Liu Y W,Huang J,Chen S Y,Xia Q M,Long R P,Yang C D. 2015. Analysis on yield formation and its higher yielding population quality of ratooning rice[J]. Acta Agriculturae Universitatis Jiangxiensis,37(4):577-583.] doi: 10. 13836/j.jjau. 2015089.
李经勇,张洪松,唐永群. 2009. 中国再生稻研究与应用[J]. 南方农业,3(3):88-92. [Li J Y,Zhang H S,Tang Y Q. 2009. Research and application of regenerated rice in China[J]. South China Agriculture,3(3):88-92.] doi:10. 19415/j.cnki.1673-890x.2009.03.030.
李兴星,郑剑,周军杰,杨永清,秦小健,张汉马,梁永书. 2016. 利用籼粳交“热粳35/协B”F2群体对水稻再生力的QTL分析[J]. 分子植物育种,14(9):2383-2391. [Li X X,Zheng J,Zhou J J,Yang Y Q,Qin X J,Zhang H M,Liang Y S. 2016. QTL analysis of ratooning ability by using Indica-japonica “Rejing 35/Xie B” F2 populations in rice[J]. Molecular Plant Breeding,14(9):2383-2391.] doi:10.13271/j.mpb.014.002383.
李学勇,钱前,李家洋. 2003. 水稻分蘖的分子机理研究[J]. 中国科学院院刊,18(4):274-276. [Li X Y,Qian Q,Li J Y. 2003. Progress in elucidating the molecular mechanism of rice tillering[J]. Bulletin of the Chinese Academy of Sciences,18(4):274-276.] doi:10.16418/j.issn.1000-3045. 2003.04.009.
劉贵富,陈明江,李明,吕慧颖,葛毅强,魏珣,杨维才. 2018.水稻育种行业创新进展[J]. 植物遗传资源学报,19(3):416-429.[Liu G F,Chen M J,Li M,Lü H Y,Ge Y Q,Wei X,Yang W C. 2018. Advances and innovation of rice breeding[J]. Journal of Plant Genetic Resources,19(3):416-429.] doi:10.13430/j.cnki.jpgr.2018.03.006.
刘歆,朱容,杨梅,刘章勇. 2020. 再生稻高产种质资源筛选及高产群体分析[J]. 作物杂志,(2):28-33. [Liu X,Zhu R,Yang M,Liu Z Y. 2020. Screening of high-yield germplasms for ratoon rice and analysis of high yield composition[J]. Crops,(2):28-33.] doi:10.16035/j.issn.1001-7283.2020.02.005.
孙秀红,胡波. 2020. 影响水稻分蘖发生的主要栽培措施和环境因素研究进展[J]. 现代农业科学,(7):6-8. [Sun X H,Hu B. 2020. Research progress on main cultivation measures and environmental factors affecting tillering of rice[J]. Modern Agricultural Science,(7):6-8.] doi:10.3969/ j.issn.1007-5739.2020.07.002.
谭震波,沈利爽,袁祚廉,陆朝福,陈英,周开达,朱立煌. 1997. 水稻再生能力和头季稻产量性状的QTL定位及其遗传效应分析[J]. 作物学报,23(3):289-295. [Tan Z B,Shen L S,Yuan Z L,Lu C F,Chen Y,Zhou K D,Zhu L H. 1997. Identification of QTLs for ratooning ability and grain yield traits of rice and analysis of their genetic effects[J]. Acta Agronomica Sinica,23(3):289-295.] doi:10.3321/j.issn:0496-3490.1997.03.005.
汪浩,張强,张文地,李思宇,黄健,朱安,刘立军. 2020. 腋芽萌发能力对再生稻产量影响的研究进展[J]. 中国水稻科学,34(3):205-216. [Wang H,Zhang Q,Zhang W D,Li S Y,Huang J ,Zhu A,Liu L J. 2020. Advances in the effects of the ability of axillary bud germination on grain yield in ratoon rice[J]. Chinese Journal Rice Science,34(3):205-216.] doi:10.16819/j.1001-7216.2020.9097.
习敏,吴文革,汪靖桂,汪宏伟,陈刚,许有尊. 2017. 再生稻种植产量差形成的研究[J]. 华北农学报,32(1):104-110. [Xi M,Wu W G,Wang J G,Wang H W,Chen G,Xu Y Z. 2017. Study on formation of grain yield differences in ratooning rice cultivation[J]. Acta Agriculturae Boreali-Sinica,32(1):104-110.] doi:10.7668/hbnxb.2017.01.017.
徐富贤,熊洪,张林,朱永川,蒋鹏,郭晓艺,刘茂. 2015. 再生稻产量形成特点与关键调控技术研究进展[J]. 中国农业科学,48(9):1702-1717. [Xu F X,Xiong H,Zhang L,Zhu Y C,Jiang P,Guo X Y,Liu M. 2015. Progress in research of yield formation of ratooning rice andIts high-yielding key regulation technologies[J]. Scientia Agricultura Sinica,48(9):1702-1717.] doi:10.3864/j.issn.0578-1752.2015.09.04.
杨川航,王玉平,涂斌,李婷,胡亮,李仕贵. 2012. 利用RIL群体对水稻再生力及相关农艺性状的QTL分析[J]. 作物学报,38(7):1240-1246. [Yang C H,Wang Y P,Tu B,Li T,Hu L,Li S G. 2012. QTL analysis of rice ratooning ability and related agronomic traits by using RIL populations[J]. Acta Agronomica Sinica,38(7):1240-1246.] doi:10.3724/SP.J.1006.2012.01240.
朱永川,熊洪,徐富贤,郭晓艺,张林,刘茂,周兴兵. 2013. 再生稻栽培技术的研究进展[J]. 中国农学通报,29(36):1-8. [Zhu Y C,Xiong H,Xu F X,Guo X Y,Zhang L,Liu M,Zhou X B. 2013. Progress on research of ratoon rice cultivation technology[J].Chinese Agricultural Science Bulletin,29(36):1-8.] doi:10.11924/j.issn.1000-6850. 2013-0363.
Adrian A,Muhammad J,Mattia M,Mark B,Martina V,Peter B,Sandro G,Harro B,Peter B,Salim A B. 2012. The path from β-carotene to carlactone,a strigolactone-like plant hormone[J]. Science,335(6074):1348-1351. doi:10.1126/ science.1218094.
Arite T,lwata H,Ohshima K,Maekawa M,NakajimaM,Kojima M,Sakakibara H,Kyozuka J. 2007.DWARF10,an RMS1/MAX4/DAD1 ortholog,controls lateral bud outgrowth in rice[J]. The Plant Journal,51(6):1019-1029. doi:10.1111/j.1365-313X.2007.03210.x.
Bolle C. 2004. The role of GRAS proteins in plant signal transduction and development[J]. Planta,2(18):683-692. doi:10.1007/s00425-004-1203-z.
Cline M G,Oh C. 2006. A reappraisal of the role of abscisic acid and its interaction with auxin in apical dominance[J]. Annals of Botany,98(4):891-897. doi:10.1093/aob/mcl173.
Dong H L,Chen Q,Wang W Q,Peng S B,Huang J L,Cui K H,Nie L X. 2017. The growth and yield of a wet-seeded rice-ratoon rice system in central China[J]. Field Crops Research,208:55-59. doi:10.1016/j.fcr.2017.04.003.
Gomez-Roldan V,Fermas S,Brewer P B,Puech-Pages V,Dun E A,Pillot J P,Letisse F,Matusova R,Danoun S,Portais J C,Bouwmeester H,Becard G,Beveridge C A,Rameau C,Rochange S F. 2008. Strigolactone inhibition of shoot branching[J]. Nature,455(7210):189-194. doi:10.1038/nature07271.
Hong C Y,Chao Y Y,Yang M Y,Cheng S Y,Cho S C,Kao C H. 2009. NaCl-induced expression of glutathione reductase in roots of rice(Oryza sativa L.) seedlings is mediated through hydrogen peroxide but not abscisic acid[J]. Plant and Soil,320:103-115. doi:10.1007/s11104-008-9874-z.
Jiang Q W,Wang W Q,Chen Q,Peng S B,Huang J L,Cui K H,Nie L X. 2016. Response of first flood irrigation timing after rice dry-direct-seeding:Productivity and greenhouse gas emissions in Central China[J]. Agricultural Water Management,177:241-247. doi:10.1016/j.agwat.2016. 08. 006.
Komatsu K,Maekawa M,Ujiie S,Satake Y,Furutani I,Okamoto H,Shimamoto K,Kyozuka J. 2003. LAX and SPA:Major regulators of shoot branching in rice[J]. Procee-dings of the National Academy of Sciences of the United States of America,2003,100(20):11765-11770. doi:10. 1073/pnas.1932414100.
Li X Y,Qian Q,Fu Z M,Wang Y H,Xiong G S,Zeng D L,Wang X Q,Liu X F,Teng S,Yuan M,Luo D,Han B,Li J Y. 2003. Control of tillering in rice[J]. Nature,422(6932):618-621. doi:10.1038/nature01518.
Lin H,Wang R X,Qian Q,Yan M X,Meng X B,Fu Z M,Yan C Y,Jiang B,Su Z,Li J Y,Wang Y H. 2009. DWARF27,an iron-containing protein required for the biosynthesisof strigolactones,regulates rice tiller bud outgrowth[J]. The Plant Cell,21(5):1512-1525. doi:10.11 05/tpc.109.065987.
Lin Q B,Wang D,Dong H,Gu S H,Cheng Z J,Gong J,Qin R Z,Jiang L,Li G,Wang J L,Wu F Q,Guo X P,Zhang X,Lei C L,WangHY,WanJM. 2012. Rice APC/CTE controls tillering by mediating the degradation of MONOCULM 1[J]. Nature Communications,(3):644-656. doi:10.1038/ncomms1716.
Maekawa M,Komatsu M,Shimamoto K,Kyozuka J. 2001. The LAX1 and FRIZZY PANICLE 2 genes determine the inflorescence architecture of rice by controlling rachis-branch and spikeletdevelopment[J]. Developmental Biology,231(2):364-373. doi:10.1006/dbio.2000. 9988.
Minakuchi K,Kameoka H,Yasuno N,Umehara M,Luo L,Kobayashi K,Hanada A,Ueno K,Asami T,Yamaguchi S,Kyozuka J. 2010. FINE CULM1(FC1) works downstream of strigolactones to inhibit the outgrowth of axillary buds in rice[J]. Plant and Cell Physiology,51(7):1127-1135. doi:10. 1093/pcp/pcq083.
Oikawa T,Kyozuka J. 2009. Two-step regulation of LAX PANICLE1 protein accumulation in axillary meristem formation in rice[J]. The Plant Cell,21(4):1095-1108. doi:10.1105/tpc.108.065425.
Ray D K,Foley J A. 2013. Increasing global crop harvest frequency:Recent trends and future directions[J]. Environmental Research Letters,8:44041-44050. doi:10.1088/1748-9326/8/4 /044041.
Shao G N,Lu Z F,Xiong J S,Wang B,Jing Y H,Meng X B,Liu G F,Ma H Y,Yan L,Yu H. 2019. Tiller bud formation regulators MOC1 and MOC3 cooperatively promote tiller bud outgrowth by activating FON1 expression in rice[J].Molecular Plant,12(8):1090-1102. doi:10.1016/j.molp. 2019.04.008.
Tabuchi H,Zhang Y,Hattori S,Omae M,Shimizu-Sato S,Oikawa T,Qian Q,Nishimura M,Kitano H,Xie H,Fang X H,Yoshida H,Kyozuka J,Chen F,Sato Y. 2011. LAX PANICLE2 of rice encodes a novel nuclear protein and regulates the formation of axillary meristems[J].The Plant Cell,23(9):3276-3287. doi:10.1105/tpc.111.088 765.
Takeda T,Suwa Y,Suzuki M,Kitano H,Ueguchi-Tanaka M,Ashikari M,Matsuoka M,Ueguchi C. 2003. The OsTB1 gene negatively regulates lateral branching in rice[J].The Plant Journal,33(3):513-520. doi:10.1046/j.1365-313X. 2003.01648.x.
Tao Y,Chen Q,Peng S B,Wang W Q,Nie L X. 2016. Lower global warming potential and higher yield of wet direct-seeded rice in Central China[J].Agronomy for Sustaina-ble Development,36(2):24. doi:10.1007/s13593-016-0361-2.
Umehara M,Hanada A,Yoshida S,Akiyama K,Arite T,Takeda-Kamiya N,Magome H,Kamiya Y,Shirasu K,Yoneyama K,Kyozuka J,Yamaguchi S. 2008. Inhibition of shoot branching by new terpenoid plant hormones[J]. Nature,455(7210):195-200. doi:10.1038/nature07272.
Xu C,Wang Y H,Yu Y C,Duan J,Duan J B,Liao Z G,Xiong G S,Meng X B,Liu G F,Qian Q,Li J Y. 2012. Degradation of MONOCULM1 by APC/C(TAD1) regulates rice tillering[J]. Nature Communications,(3)743-750. doi:10.1038/ncomms1743.
Zhang Z Y,Li J J,Yao G X,Zhang H L,Dou H J,Shi H L,Sun X M,Li Z C. 2011. Fine mapping and cloning of the grain number per-panicle gene(Gnp4) on chromosome 4 in rice(Oryza sativa L.)[J]. Agricultural Scien-ces in China,10(12):1825-1833. doi:10.1016/S1671- 2927 (11)60182-X.
Zhang Z Y,Sun X M,Ma X Q,Xu B X,Zhao Y,Ma Z Q,Li G L,Khan N U,Pan Y H,Liang Y T,Zhang H L,Li J J,Li Z C. 2021. GNP6,a novel allele of MOC1,regulates panicle and tiller development in rice[J]. The Crop Journal,9(1):57-67. doi:10.1016/j.cj.2020.04.011.
Zou J H,Chen Z X,Zhang S Y,Zhang W P,Jiang G G,Zhao X F,Zhai W X,Pan X B,Zhu L H. 2005. Characterizations and fine mapping of a mutant gene for high tillering and dwarf in rice(Oryza sativa L.)[J]. Planta,222(4):604-612. doi:10.1007/s00425-005-0007-0.
Zou J H,Zhang S Y,Zhang W P,Li G,Chen Z X,Zhai W X,Zhao X F,Pan X P,Xie Q,Zhu L H. 2006. The rice HIGH-TILLERING DWARF1 encoding an ortholog of Arabidopsis MAX3 is required for negative regulation of the outgrowth of axillary buds[J]. The Plant Journal,48(5):687-698. doi:10.1111/j.1365-313X.2006.02916.x.
(責任编辑 陈 燕)

