温度和药剂胁迫下黄翅绢野螟实时荧光定量PCR内参基因筛选
孟倩倩 孙世伟 王政 高圣风 苟亚峰 刘爱勤


摘 要:為筛选黄翅绢野螟(Diaphania caesalis)逆境胁迫下稳定表达的内参基因,使该虫抗逆基因的定量表达分析标准化。本研究根据黄翅绢野螟成虫转录组注释结果,利用qPCR技术分析了9个分别编码ACT、β-TUB、GAPDH、G6PDH、RPS3a、RPL13a、EF1α、EIF4A、β-ACT的候选内参基因在温度及药剂胁迫下mRNA的表达水平,并通过geNorm、NormFinder、BestKeeper和RefFinder软件(算法)分析各基因的稳定性。结果表明:温度胁迫下,geNorm分析表明ACT和RPS3a为表达最稳定的基因,NormFinder和BestKeeper分析结果表明GAPDH和EIF4a的表达最稳定。药剂胁迫下,geNorm和NormFinder分析结果均表明EIF4a和ACT为最稳定的内参基因,而BestKeeper分析结果表明GAPDH和RPS3a为最稳定内参基因。全样品条件下,geNorm和NormFinder分析结果相似,表明EF1α和EIF4a为最稳定的内参基因,BestKeeper表明GAPDH和RPL13a为最稳定的内参基因。根据geNorm软件成对变异值Vn/Vn+1<0.15的原则,确定3种条件下对靶标基因表达进行标准化时需引入的内参基因的数目均为2个。利用在线分析软件RefFinder综合评价上述3个分析结果,最终确定温度胁迫下最合适的内参基因为EIF4A和GAPDH,药剂胁迫下最适内参基因为ACT和EIF4A,全样品下最适内参基因为EF1α和EIF4A。本研究结果为利用qPCR技术分析黄翅绢野螟温敏及抗药性相关基因表达差异提供了稳定的内参基因,进而为基因的功能分析奠定了基础。
关键词:黄翅绢野螟;温度胁迫;药剂胁迫;内参基因;稳定性
中图分类号:S433.4 文献标识码:A
Abstract: In order to normalize the expression of stress resistant genes, this study aims to select the reference genes stably expressed of Diaphania caesalis under different stress conditions. According to the annotation information of the transcriptome of D. caesalis, the mRNA expression levels of nine candidate genes predicted to code proteins of ACT, β-TUB, GAPDH, G6PDH, RPS3a, RPL13a, eEF1α, EIF4A and β-ACT, were analyzed by qPCR under temperature and pesticide stress. The stability of each gene was evaluated by geNorm, NormFinder, BestKeeper and RefFinder softwares (algorithms), respectively. In temperature stress, the most stable genes identified by geNorm were ACT and RPS3a. However, NormFinder and BestKeeper recommended that GAPDH and EIF4a were the most stable genes. In pesticide stress, EIF4a and ACT were ranked as the most stable genes by geNorm and NormFinder, but GAPDH and RPS3a were recognized as the most suitable reference genes referred from the analysis with software of BestKeeper. In all samples, the results analyzed by the softwares of geNorm and NormFinder were similar, which both found that the ideal internal control were EF1α and EIF4a. But GAPDH and RPL13a were considered as the most stable genes based on the result of BestKeeper. Complying with the principle of Vn/Vn+1<0.15 from geNorm software, the two internal reference genes should be introduced in the three treatments above for normalization of target gene expression, respectively. The online software of RefFinder was used to comprehensively evaluate the results of the three softwares. Finally, the most suitable reference genes in temperature stress were confirmed as EIF4A and GAPDH. ACT and EIF4A were determined as the most stable reference genes in pesticide stress, and EF1α and EIF4A were recommended as the most suitable reference genes in all samples. This study would provide suitable internal reference genes for qPCR analysis of the expression of target genes associated to temperature sensitivity and insecticide resistance. Furthermore, it could also lay the foundation for gene functional analysis.
Keywords: Diaphania caesalis; temperature stress; pesticide stress; reference gene; stability
DOI: 10.3969/j.issn.1000-2561.2021.01.026
实时荧光定量PCR(quantitative real-time PCR, qPCR)因其灵敏度高、重复性好、特异性强和快速准确等技术优点,被广泛应用于基因的特异性扩增、mRNA表达水平分析、限制性片段长度多态性分析和单核苷酸多态性分析等研究领域[1]。然而,qPCR结果的准确性和可靠性易受生物信号、引物扩增效率,模板的产量和质量等因素影响[2-5],需要引入稳定的内参基因对定量结果进行校正和标准化处理,以减小实验误差,保证结果的准确性和可靠性[6]。因此,选择稳定的内参基因是进行qPCR过程中至关重要的一步。一般认为,理想化的内参基因应是维持细胞正常生命代谢的管家基因,它们在生物和非生物条件下都能相对稳定表达[7-9]。传统的管家基因包括18S核糖体RNA基因(18S rRNA)、微管蛋白基因(tu bulin)、甘油醛-3-磷酸脱氢酶基因(glyceraldehyde- 3-phosphate dehydrogenase)和肌动蛋白基因(actin)等,它们常被作为内参基因用于许多物种不同处理条件下基因的表达分析[10-12]。但越来越多的研究表明,并没有绝对稳定的内参基因能在不同物种、不同组织及不同实验条件下均稳定表达,它们只能在特定物种或实验条件下相对稳定的表达[13-16]。因此,在利用qPCR对某物种基因进行转录水平表达分析前,需对候选内参基因在特定条件下的表达稳定性进行评估,以选择最适内参基因。
黄翅绢野螟(Diaphania caesalis)属鳞翅目(Lepidoptera),螟蛾科(Pyralidae),绢野螟属(Diaphania)。主要分布在热带和亚热带地区,在我国主要分布在海南、云南、广西、广东等省(区)。该虫以幼虫钻蛀取食菠萝蜜(Artocarpus heteroyllus Lam.)、榴莲蜜(Artocarpus champeden Spreng.)和面包果(Artocarpus altilis Fosberg.)等富含淀粉的“木本粮食作物”[17-19]。蛀虫果易引起果蝇幼虫进入取食,使果实受害部位变褐腐烂,导致果实发育不良、脱落,严重影响作物的产量和质量,造成的经济损失达30%~50%。该虫在海南全年都有发生,且无明显越冬现象,一年发生约8代,7—9月为发生高峰期[20-22]。目前关于黄翅绢野螟的研究主要包括其发生为害特点、分布情况及天敌种类等[19, 23-24]。尚无黄翅绢野螟qPCR内参基因的筛选研究。随着研究的深入,基因表达水平对于研究黄翅绢野螟的寄主选择、适应性、抗药性和神经调节等相关基因的调控机理方面具有重要作用。因此,在分析黄翅绢野螟基因表达水平之前,首先需评估不同处理条件下内参基因的表达稳定性。本研究拟检测黄翅绢野螟9个候选内参基因在不同温度和药剂胁迫下mRNA的表达水平,并利用geNorm、NormFinder、BestKeeper和RefFinder 4个软件(算法)分析温度胁迫、药剂胁迫和全样品条件下各候选基因的稳定性,筛选出不同处理情况下最稳定的内参基因,以期为黄翅绢野螟温度和药剂胁迫下基因表达水平的研究提供合适的内参基因。
1 材料与方法
1.1 供试昆虫
黄翅绢野螟采自海南省琼中县菠萝蜜种植园(19°3′24″N, 109°50′58″E),室内饲养2代后作为供试昆虫。幼虫食料为未施农药的菠萝蜜叶片,采自中国热带农业科学院香料饮料研究所试验基地(18°44′29″N, 110°12′11″E)。将采集的幼虫置于保鲜盒中饲喂,待羽化为成虫后转入网笼,喂食10%的蜂蜜水。向笼中放置菠萝蜜叶片或鲜果收集卵粒,待孵化后挑出幼虫至干净的保鲜盒内,放入新鲜菠萝蜜叶片进行继代饲养,饲养条件:温度(26±1)℃,相对湿度70%±5%,光周期14 L∶10 D。
1.2 温度处理
设置低温处理4 ℃、高温处理37 ℃、超高温处理42 ℃及对照处理26 ℃。分别将黄翅绢野螟2~3日龄成虫置于上述处理下刺激4 h,室温26 ℃恢复30 min,每个处理3头成虫,设置3个重复,置于液氮?80 ℃下冻存,供提取总RNA。
1.3 药剂处理
根据黄翅绢野螟防治常用药剂,设置高效氯氟氰菊酯(lambda-cyhalothrin)进行药剂处理。采用药膜法处理黄翅绢野螟成虫,具体方法如下:将2.5%高效氯氟氰菊酯水乳剂(安徽省阴山药业有限公司)用丙酮依次稀释40 000、60 000和80 000,吸取5 mL稀释好的药液滴入三角瓶中,迅速顺时针转动三角瓶,确保药剂均匀形成药膜,待丙酮挥发后放入6头2~3日龄成虫处理4 h,每个处理重复3次,对照组为未经药剂处理的黄翅绢野螟成虫,将对照和处理样本置于液氮?80 ℃下冻存,供总RNA提取。
1.4 总RNA提取及cDNA第一链合成
采用TRIzol Reagent试剂(Invitrogen公司,美国)分别进行上述各处理黄翅绢野螟总RNA的提取,并用1%琼脂糖凝胶电泳检测RNA的完整性,利用Biotek H1荧光酶标仪(Biotek公司,美国)测定总RNA的浓度和纯度,记录OD260/OD280波长处的吸光度,比值在1.8~2.2之间的样品用于cDNA合成。每个样品取2 μg RNA,按照FastKing RT Kit反转录试剂盒[天根生化科技(北京)有限公司]步骤合成cDNA第一链,稀释5倍于-20 ℃保存備用。
1.5 候选内参基因的确定及引物设计
基于黄翅绢野螟转录组注释结果,选择以下9个基因作为候选内参基因。经NCBI比对后,分别为编码肌动蛋白基因(Actin, ACT)、β-微管蛋白基因(β-tubulin, β-TUB)、甘油醛-3-磷酸脱氢酶基因(glyceraldehyde 3-phosphate dehydrogenase, GAPDH)、葡萄糖-6-磷酸脱氢酶基因(glucose 6 phosphate dehydrogenase, G6PDH)、核糖体蛋白S3a基因(ribosomal protein S3a, RPS3a)、核糖体蛋白L13a基因(ribosomal protein L13a, RPL13a)、延伸因子1α(elongation factor 1 alpha, EF1α)、真核生物启动因子4A(eukaryotic initiation factor 4A, EIF4A)和β-肌动蛋白基因(β-Actin, β-ACT)。根据基因序列信息,利用NCBI Primer-BLAST设计qPCR引物,基因及引物信息见表1。
1.6 候选内参基因引物特异性及标准曲线绘制
以1.2中对照处理RNA反转录的cDNA为模板,通过qPCR检测各基因的mRNA表达水平,qPCR反应使用TB GreenTM Premix Ex TaqTM II(TaKaRa,日本)酶,扩增结束后在60~95 ℃通过熔解曲线分析,检测各引物的扩增特异性。标准曲线的绘制使用5 U/mL Pfu Taq DNA聚合酶(TaKaRa,日本)对9个候选内参基因进行PCR扩增,产物胶收后分别连接于pMD19-T载体(TaKaRa,日本)于16 ℃过夜,后转化至感受态细胞DH5α(TaKaRa,日本)中,通过在LB液体培养基上扩大培养提取质粒DNA,质粒DNA抽提采用EZNA Plasmid Mini Kit试剂盒(Omega公司,美国),后依次稀释5个梯度,每个梯度稀释10倍,以各浓度梯度质粒DNA为模板进行qPCR。根据qPCR检测结果绘制标准曲线得到线性相关系数(R2)和斜率(slope),并计算引物的扩增效率(E),E=(10[-1/slope]-1)×100%,qPCR和PCR扩增反应条件、连接、转化等步骤具体参照王政等[7]的方法。
1.7 候選内参基因表达稳定性分析
分别以1.4中合成的cDNA为模板,利用qPCR分析9个候选内参基因在不同处理下的mRNA表达量,反应体系为20 ?L,反应条件同1.6。采用geNorm[25]、NormFinder[26]、BestKeeper[27]和RefFinder[28]软件对候选内参基因在温度胁迫、药剂胁迫以及2种处理条件下全样品的表达稳定性进行分析。geNorm软件通过计算平均变异度(mean variability, M)和配对变异值(pairwise variation, V)来确定最佳的内参基因及数目。geNorm软件要求M<1.50的基因为可用内参基因,M值越小,候选基因越稳定;最佳内参基因的数目要求Vn/Vn+1<0.15,表明最佳内参基因数目为n个。NormFinder根据各基因表达稳定值(stability value, SV),SV越小表明基因表达越稳定。BestKeeper软件计算每个基因产生配对的相关系数(r)、标准偏差(standard deviation, SD)和变异系数(coefficient of variation, CV),根据SD和CV越小则基因越稳定的原则,最终对各基因进行排名,SD<1的基因被视为表达水平较为稳定。最后,根据geNorm软件Vn/Vn+1推荐的稳定内参的数量,利用RefFinder在线分析程序(https://www.heartcure.com.au/reffinder/?type=reference)对上述4种方法的分析结果进行综合评价,最终确定各处理条件下适用的内参基因。
2 结果与分析
2.1 候选内参基因引物特异性和扩增效率检测
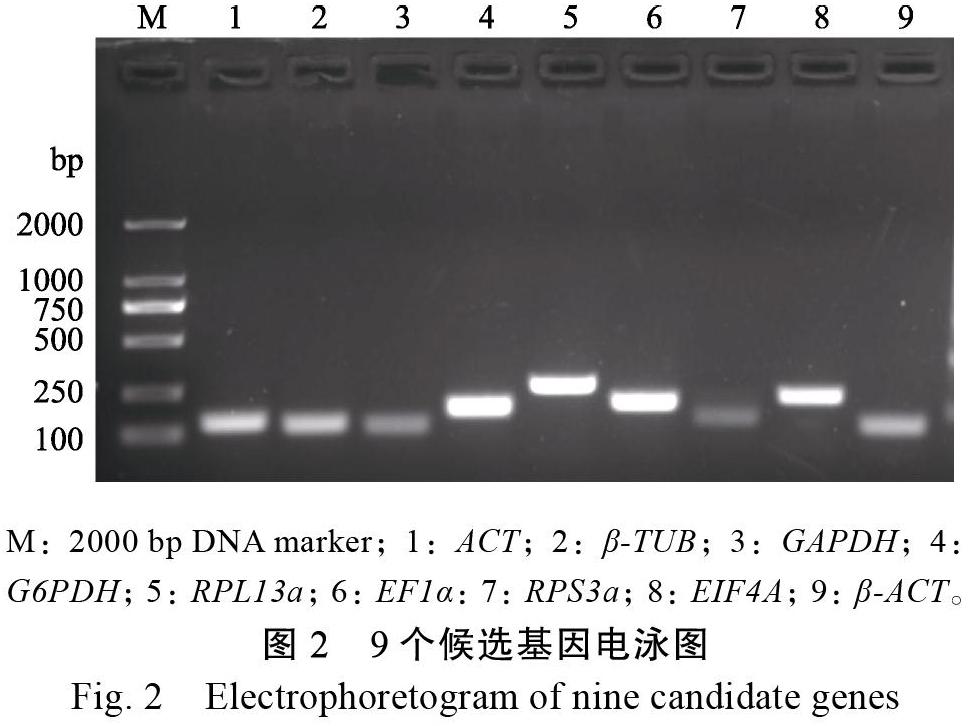
根据qPCR结果,9个候选内参基因的溶解曲线均为单一峰(图1),PCR产物经1.5%琼脂糖凝胶电泳后条带单一且明亮,长度与引物设计预期长度一致(图2),无非特异性扩增,经测序与黄翅绢野螟转录组序列一致。表明各基因序列正确,引物特异性良好,可用于后续试验。以各基因质粒DNA进行梯度稀释为模板进行标准曲线绘制,结果显示9个候选基因的线性相关系数R2均在0.998以上,表明模板DNA浓度与相应的Ct值线性关系明确,绘制的标准曲线可信。各基因的引物扩增效率为95.10%~107.5%(表1),表明各基因引物设计合理,使用该引物进行qPCR扩增是有效的。
2.2 不同样品中候选内参基因mRNA表达水平分析
qPCR检测9个候选内参基因在7个处理(3个温度处理,3个药剂处理,1个对照组)中的mRNA表达水平结果显示,各基因在不同样品中表达量均有差异,温度胁迫条件下平均Ct值范围为18.23(EF1α)~25.45(β-ACT),表达量由高到低依次为EF1α(17.19~19.49)>RPL13a(17.58~20.72)>GAPDH(18.49~19.95)>RPS3a(18.19~21.04)>EIF4A(19.52~22.04)>ACT(20.5~23.37)>β-TUB(20.92~24.48)>G6PDH(21.13~24.78)>β-ACT(21.94~27.47)(图3A);药剂胁迫条件下平均Ct值范围为18.12(EF1α)~ 25.26(β-ACT),表达量由高到低依次为EF1α(16.90~19.87)>RPL13a(17.58~20.28)>RPS3a(18.11~20.51)>GAPDH(18.49~20.58)>EIF4A(18.66~21.84)>ACT(19.75~22.18)>β-TUB(20.55~24.48)>G6PDH(21.52~25.64)>β-ACT(22.24~27.47)(图3B);全样品条件下平均Ct值范围为18.22(EF1α)~25.20(β-ACT),表达量由高到低依次为EF1α(16.90~19.87)>RPL13a(17.58~20.72)>GAPDH(18.49~20.58)>RPS3a(18.11~21.04)>EIF4A(18.66~22.04)>ACT(19.75~23.37)>β-TUB(20.55~24.48)>G6PDH(21.13~25.64)>β-ACT(20.55~27.47)(图3C)。不同处理条件下,各基因在组间的表达量差异较大,其中GAPDH的表达量组间差异最小,β-ACT的表达量组间差异最大。
2.3 温度胁迫下内参基因稳定性分析
温度胁迫下,geNorm软件分析结果表明,9个候选内参基因平均变异度M值均小于1.5,表明候选内参基因在温度胁迫条件下均可作为内参基因使用,其中ACT和RPS3a的M值(0.115)最小,是排名最稳定的内参基因;β-ACT的M值(0.983)最大,为相对最不稳定的内参基因;基因稳定性排序为EF1α(0.303)>EIF4A(0.368)> RPL13a(0.508)>G6PDH(0.576)>GAPDH (0.667)>β-TUB(0.861)(图4A)。NormFinder软件分析结果显示SV值最小的基因为EIF4A(0.193),因此EIF4A被推荐为表达最稳定的内参基因,基因稳定性排序依次为GAPDH(0.354)> EF1α(0.371)>RPL13a(0.618)>G6PDH(0.636)>ACT(0.716)>RPS3a(0.819)>β-TUB(1.211)>β-ACT(1.292)(图4B);BestKeeper软件分析结果表明,9个基因SD值均小于1,因此都可作为内参基因使用,其中GAPDH的SD值(0.198)最小;基因稳定性排序为GAPDH(0.198)>EIF4A(0.398)>EF1α(0.451)>ACT(0.605)>RPL13a(0.639)>RPS3a(0.639)>G6PDH(0.676)>β-TUB(0.754)>β-ACT(0.825)(图4C)。
2.4 药剂胁迫下内参基因稳定性分析
geNorm软件计算结果表明,9个基因的M值均小于1.5,均可作为药剂胁迫下基因表达分析的内参基因。其中EIF4A和ACT的M值(0.087)最小,表达最稳定,基因稳定性排序依次为EF1α(0.117)>RPS3a(0.286)>GAPDH(0.332)> RPL13a(0.350)>G6PDH(0.541)>β-TUB(0.728)>β-ACT(0.913)(图5A);NormFinder软件分析9个基因稳定性排序与geNorm结果一致(图5B);BestKeeper軟件分析结果与geNorm和Norm Finder分析结果差异较大,其中β-ACT和G6PDH的SD值均大于1,因此其稳定性预测结果不可靠,9个基因稳定性排序依次为GAPDH(0.285)>RPS3a(0.387)>EIF4A(0.486)>ACT(0.489)>RPL13a(0.519)>EF1α(0.538)>β-TUB(0.798)>G6PDH(1.266)>β-ACT(1.396)(图5C)。
2.5 全样品条件下内参基因稳定性分析
全样品条件下,geNorm软件分析9个基因M值均小于1.5,表明所有基因都可用作全样品条件下的内参,基因稳定性排序依次为EF1α/EIF4A(0.283)>ACT(0.332)>RPS3a(0.386)>RPL13a(0.477)>GAPDH(0.558)>G6PDH(0.700)>β-TUB(0.824)>β-ACT(0.953)(图6A);NormFinder分析结果表明,最稳定和最不稳定的内参基因分别为EF1α(SV值为0.187)和β-ACT(SV值为1.265),基因稳定性排序依次为EIF4A(0.193)>RPL13a(0.461)>ACT(0.545)>GAPDH(0.547)>RPS3a(0.675)>G6PDH(0.951)>β-TUB(1.039)(图6B);BestKeeper分析结果表明,GAPDH的SD值(0.288)和CV值(1.501)均为最小,为最稳定内参基因,而β-ACT和G6PDH的SD值均大于1,不能将其作为内参基因使用,其余基因稳定性排序依次为EIF4A(0.496)>EF1α(0.520)>RPL13a(0.571)>RPS3a(0.592)>ACT(0.602)>β-TUB(0.692)(图6C)。
2.6 黄翅绢野螟不同条件下最佳内参基因的确定
内参数目可参照geNorm软件成对变异值Vn/Vn+1来确定。即当Vn/Vn+1<0.15时,则该条件下最佳内参基因的数目为n个。geNorm软件结果表明,上述不同处理下成对变异值V2/V3均小于0.15(图7),因此,研究温度胁迫、药剂胁迫和全样品条件下基因表达分析时,需引入2个内参基因进行标准化。RefFinder综合geNorm、NormFinder、BestKeeper 3个软件的基因稳定性排序结果,最终确定各基因在不同处理条件下的几何平均值,几何平均值越小,稳定性越好。因此,温度胁迫下,最终确定的内参基因为EIF4A、GAPDH;药剂胁迫下,最终确定的内参基因为ACT、EIF4A;全样品下,最终确定的内参基因为EF1α、EIF4A(表2)。在进行各条件下基因表达分析时,可引入上述基因作为内参基因。
3 讨论
黄翅绢野螟因其钻蛀为害寄主植物的特性,化学防治效果欠佳且生态弊端显著,利用分子生物学手段揭示黄翅绢野螟的生理及行为学机制,有助于开发防控新技术。qPCR技术是分子生物学领域研究基因功能、揭示生理机制的重要技术手段,通常采用相对定量的方法对基因表达水平进行检测,即引入内参基因作为标准校正目的基因的表达量[7, 29]。目前,尚无某个理想的内参基因在不同物种和不同处理条件下均能稳定表达。因此,对某一物种进行基因表达分析前,筛选出在特定实验条件下稳定表达的内参基因至关重要[30, 13]。黄翅绢野螟是菠萝蜜、面包果和尖蜜拉等富含淀粉的热带果树上重要的钻蛀性害虫,国内外未见其内参基因筛选的报道。因此,本研究根据前期转录组数据,筛选出9个候选内参基因进行表达稳定性分析。在进行基因表达分析前,首先对其引物特异性及扩增效率进行评估。一般认为引物特异性高且扩增效率介于90%~110%之间时,可用于qPCR试验的开展[31],本研究中9个候选基因引物均无非特异性扩增,且各基因的引物扩增效率介于95.10%~107.5%之间,表明各基因引物设计合理,符合qPCR扩增要求,检测结果有效。
本研究分别采用geNorm、NormFinder和BestKeeper软件对候选内参基因稳定性进行分析,结果发现:基因稳定性排序在3种软件之间存在差异。温度胁迫下,NormFinder和BestKeeper分析结果较为相似,而geNorm分析结果与NormFinder、BestKeeper分析结果差异较大。例如,ACT/RPS3a在geNorm中排名为最稳定的内参基因,而在NormFinder中排名分别为第6、第7位,BestKeeper中分别为第4、第6位。药剂胁迫下,geNorm和NormFinder分析结果排序结果均一致,排名前3位的基因均为EIF4A、ACT和EF1α,而这3个基因在BestKeeper中分别排在第3、第4和第6位。全样品条件下,geNorm和NormFinder分析结果表明,EF1α和EIF4A为排名前2位的基因,排在后3位的基因均为G6PDH、β-TUB和β-ACT,其他基因排列顺序有少许差异,而BestKeeper分析的基因排序结果显示,排在前2位的基因为GAPDH和EIF4A,与geNorm和NormFinder差异较大。造成3种软件分析结果不一致的原因是各软件计算原理不同,這种情况在瓜实蝇(Bactrocera cucurbitae)[32]、松墨天牛(Monochamus alternatus)[33]、大灰象甲(Sympiezomias velatus)[34]、扶桑绵粉蚧[35]等昆虫中也存在。为了消除不同软件分析结果的差异,需利用RefFinder在线分析软件综合评估内参基因的稳定性,得到最终排名结果,从中选出最合适的内参基因。
根据RefFinder最终评价结果,发现温度胁迫、药剂胁迫和全样品条件下推荐的稳定内参基因并不完全相同。其中,仅EIF4A在3种条件下都可作为稳定内参使用。但使用单一内参基因进行qPCR数据校准,可能会影响结果的精确性,因此qPCR分析过程中往往使用多个内参基因进行校正[25, 36]。所需内参基因的数目可根据geNorm软件基因配对差异值Vn/Vn+1来确定。依据Vn/Vn+1< 0.15的原则,本研究中温度胁迫、药剂胁迫和全样品条件下进行qPCR时均需引入2个内参基因,因此,上述3种处理下推荐的另一个内参基因分别是GAPDH、ACT和EF1α。EIF4A为真核生物启动因子,参与mRNA与核糖体的结合[37],已被证实可用作许多鳞翅目昆虫温度胁迫或抗药性基因的表达分析,如家蚕(Bombyx mori)中肠抗菌肽基因的表达[38]、粉纹夜蛾(Trichoplusia ni)抗药蛋白基因的表达[39]、虫草蝙蝠蛾(Thitarodes armoricanus)抗寒基因的表达[40]等。本研究中EIF4A的适用范围则更广泛,说明相同的基因在不同物种中可作为内参基因的适用范围并不一致。另外,GAPDH和ACT作为传统内参基因并非在所有条件下均表达稳定,如在番茄木虱(Bactericera cockerelli)[41]、黑腹果蝇(Drosophila melanogaster)[42]、二化螟(Chilo suppressalis)[12]、小菜蛾(Plutella xylostella)[43]等昆虫中二者仅在特定条件下才可作为内参基因使用,本研究结果证实了这一点,GAPDH仅在温度胁迫下稳定,而ACT仅在药剂胁迫下稳定。值得注意的是,王政等[7]发现茶刺盲蝽在温度胁迫下最佳内参基因为EIF4A和RPS3a,药剂胁迫和全样品条件下最佳内参基因为RPS3a和RPL13a,尽管本研究部分实验方法参照此文,但即使在相同处理条件下结果却完全不同,再次表明不同物种的内参基因表达稳定性存在差异。因此,在进行特定条件靶标基因qPCR分析时,必须首先对内参基因稳定性进行筛选,以确保结果的准确性和可靠性。
本研究仅对黄翅绢野螟温度胁迫、药剂胁迫及全样品条件下的内参基因进行筛选,其结果是否适用其他处理条件或其他物种,有待进一步验证。本研究结果为利用qPCR技术分析黄翅绢野螟温敏及抗药性相关基因表达差异提供了稳定的内参基因,进而为基因的功能分析奠定了基础。
参考文献
Wang Z, Meng Q Q, Zhu X, et al. Evaluation and validation of reference genes for quantitative real-time PCR in Helopeltis theivora Waterhouse (Hemiptera: Miridae)[J]. Scientific Reports, 2019, 9(1): 13291.
Jiang H C, Qian Z J, Lu W, et al. Identification and characterization of reference genes for normalizing expression data from red swamp crawfish Procambarus clarkii[J]. International Journal of Molecular Sciences, 2015, 16(9): 21591-21605.
Hu Y N, Fu H T, Qiao H, et al. Validation and evaluation of reference genes for quantitative real-time PCR in Macrobrachium Nipponense[J].International Journal of Molecular Sciences, 2018, 19(8): 2258-2273.
Shu B S, Zhang J J, Cui G F, et al. Evaluation of reference genes for real-time quantitative PCR analysis in larvae of Spodoptera litura exposed to azadirachtin stress conditions[J]. Frontiers in Physiology, 2018, 9: 372.
Zhang Y T, Peng X R, Liu Y, et al. Evaluation of suitable reference genes for qRT-PCR normalization in strawberry (Fragaria × Ananassa) under different experimental conditions[J]. BMC Miology, 2018, 19(1): 8.
Shekh K, Tang S, Niyogi S, et al. Expression stability and selection of optimal reference genes for gene expression normalization in early life stage rainbow trout exposed to cadmium and copper[J]. Aquatic Toxicology, 2017, 190: 217-227.
王 政, 朱 茜, 孟倩倩, 等. 溫度和药剂胁迫下茶刺盲蝽内参基因稳定性分析[J]. 植物保护学报, 2019, 46(3): 530-541.
Pan H P, Yang X W, Bidne K, et al. Selection of reference genes for RT-qPCR analysis in the monarch butterfly, Danaus plexippus (L.), a migrating bio-indicator[J]. PLoS One, 2015, 10(6): e0129482.
Chen H, Yang Z Q, Hu Y, et al. Reference genes selection for quantitative gene expression studies in Pinus massoniana L.[J]. Trees, 2016, 30(3): 685-696.
Suzuki T, Higgins P J, Crawford D R. Control selection for RNA quantitation[J]. Biotechniques, 2000, 29(2): 332-337.
Zhang L, Zhang Q L, Wang X T, et al. Selection of reference genes for qRT-PCR and expression analysis of high-altitude-related genes in grassland caterpillars (Lepidoptera: Erebidae: Gynaephora) along an altitude gradient[J]. Ecology and Evolution, 2017, 7(21): 9054-9065.
Xu J, Lu M X, Cui Y D, et al. Selection and evaluation of reference genes for expression analysis using qRT-PCR in Chilo suppressalis (Lepidoptera: Pyralidae)[J]. Journal of economic entomology, 2017, 110(2): 683-691.
Volkov R A, Panchuk I I, Sch?ffl F. Heat-stress-dependency and developmental modulation of gene expression: the potential of house-keeping genes as internal standards in mRNA expression profiling using real-time RT-PCR[J]. Journal of Experimental Botany, 2003, 54(391): 2343-2349.
Najat D, Ghows A, Andrade M L. Assessment of Aedes albopictus reference genes for quantitative PCR at different stages of development[J]. PLoS One, 2018, 13(3): e0194664.
Li H B, Dai C G, Zhang C R, et al. Screening potential reference genes for quantitative real-time PCR analysis in the oriental armyworm, Mythimna separata[J]. PLoS One, 2018, 13(4): e0195096.
Wang G H, Chen Y F, Zhang X Y, et al. Selection of reference genes for tissue/organ samples on day 3 fifth-instar larvae in silkworm, Bombyx mori[J]. Archives of Insect Biochemistry and Physiology, 2018, 98(2): e21458.
王平远. 中国绢螟属(Diaphania Hübner)记述[J]. 昆虫学报, 1963, 12(3): 358-367.
罗永明, 金启安. 海南岛两种热带果树害虫记述[J]. 热带作物学报, 1997, 18(1): 71-78.
Kallekkattil S, Krishnamoorthy A, Shreevihar S, et al. First report of a hymenopteran parasitoid complex on jackfruit shoot and fruit borer Diaphania caesalis (Lepidoptera: Crambidae) from India[J]. Journal of Biocontrol Science and Technology, 2019, 29(11): 1037-1052.
刘爱勤, 桑利伟, 孙世伟, 等. 海南省菠萝蜜主要病虫害识别与防治[J]. 热带农业科学, 2012, 32(12): 64-69, 74.
王 政, 孟倩倩, 谭乐和, 等. 黄翅绢野螟蛹和成虫雌雄形态的快速鉴定[J]. 环境昆虫学报, 2017, 39(5): 1185- 1190.
孟倩倩, 王 政, 谭乐和, 等. 黄翅绢野螟触角感器的扫描电镜观察[J]. 热带作物学报, 2017, 38(7): 1323-1327.
Soumya K, Krishnamoorthy A, Patil P, et al. Evaluation of jackfruit germplasm against jack shoot and fruit borer, Diaphania caesalis (Wlk.) (Lepidoptera: Pyralidae)[J]. Pest Management in Horticultural Ecosystems, 2015, 21(1): 8-10.
Rajkumar M B, Gundappa B, Tripathi M M, et al. Pests of Jackfruit[M]//Omkar. Pests and Their Management. Springer, Singapore, 2018: 589-593.
Vandesompele J, de Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): 467-470.
Andersen C L, Jensen J L, ?rntoft T F. Normalization of real-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250.
Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-excel-based tool using pair-wise correlations[J]. Biotechnology Letter, 2004, 26(6): 509-515.
Xie F L, Xiao P, Chen D L, et al. MiRDeepFinder: A miRNA analysis tool for deep sequencing of plant small RNAs[J]. Plant Molecular Biology, 2012, 80(1): 75-84.
楊 苓, 胡晓静, 徐志峰, 等. 桃蛀螟实时荧光定量PCR内参基因的筛选[J]. 昆虫学报, 2017, 60(11): 1266- 1277.
刘凡奇, 万贵钧, 曾路影, 等. 近零磁场下灰飞虱转录表达分析稳定性内参基因筛选[J]. 中国农业科学, 2019, 52(19): 3346-3356.
Taylor S, Wakem M, Dijkman G, et al. A practical approach to RT-qPCR-publishing data that conform to the MIQE guidelines[J]. Methods, 2010, 50(4): 1-5.
王凤英, 杨 朗, 黎柳锋, 等. 不同温度胁迫下瓜实蝇RT-qPCR内参基因筛选[J]. 环境昆虫学报, 2018, 40(5): 1097-1105.
冯 波, 郭前爽, 毛必鹏, 等. 松墨天牛化学感受组织荧光定量PCR内参基因的鉴定与筛选[J]. 昆虫学报, 2016, 59(4): 427-437.
李 晓, 李建文, 成 波, 等. 大灰象甲实时定量PCR内参基因的筛选[J]. 昆虫学报, 2018, 61(11): 1284-1294.
陈 芳, 陆永跃. 热胁迫下棉花粉蚧内参基因的筛选[J]. 昆虫学报, 2014, 57(10): 1146-1154.
Concha C, Edman R M, Belikoff E J, et al. Organization and expression of the Australian sheep blowfly (Lucilia cuprina) hsp23, hsp24, hsp70 and hsp83 genes[J]. Insect Biochemistry and Molecular Biology, 2012, 21(2): 169-180.
Svitkin Y V, Pause A, Haghighat A, et al. The requirement for eukaryotic initiation factor 4A (eIF4A) in translation is in direct proportion to the degree of mRNA 5secondary structure [J]. RNA, 2001, 7(3): 382-394.
Wu S, Zhang X, He Y, et al. Expression of antimicrobial peptide genes in Bombyx mori gut modulated by oral bacterial infection and development[J]. Developmental and Comparative Immunology, 2010, 34(11): 1191-1198.
Simmons J, D Souza O, Rheault M, et al. Multidrug resistance protein gene expression in Trichoplusia ni caterpillars[J]. Insect Molecular Biology, 2013, 22(1): 62-71.
Liu G Q, Qiu X H, Cao L, et al. Evaluation of reference genes for reverse transcription quantitative PCR studies of physiological responses in the ghost moth, Thitarodes armoricanus (Lepidoptera, Hepialidae)[J]. PLoS One, 2016, 11(7): e0159060.
Ibanez F, Tamborindeguy C, Selection of reference genes for expression analysis in the potato psyllid, Bactericera cockerelli[J]. Insect Molecular Biology, 2016, 25(3): 227-238.
Ponton F, Chapuis M P, Pernice M, et al. Evaluation of potential reference genes for reverse transcription-qPCR studies of physiological responses in Drosophila melanogaster[J]. Journal of Insect Physiology, 2011, 57(6): 840-850.
Teng X L, Zhang Z, He G L, et al. Validation of reference genes for quantitative expression analysis by real-time RT-PCR in four lepidopteran insects[J]. Journal of Insect Science, 2012, 12(60): 1-17.
責任编辑:谢龙莲

