串联反向CTCF位点的系列删除揭示增强子调控HOXD基因簇表达的平衡
王玲,李金环,黄海燕,吴强
研究报告
串联反向CTCF位点的系列删除揭示增强子调控基因簇表达的平衡
王玲,李金环,黄海燕,吴强
上海交通大学系统生物医学研究院比较生物医学研究中心,系统生物医学教育部重点实验室,上海 200240
三维基因组染色质架构蛋白CTCF (CCCTC-binding factor)能够介导增强子与基因启动子的远距离染色质相互作用,也可以结合调控区域的绝缘子发挥增强子绝缘功能,对发育中的基因表达调控具有重要作用。同源框基因家族(Homeobox gene family,)编码一类控制动物发育的关键转录因子,在发育中主要沿胚胎首尾轴(head-to-tail axis)呈时空线性表达。在哺乳动物中,基因分为、和四个基因簇,在中枢神经系统、骨骼和四肢发育中发挥重要功能。基因簇主要调控四肢发育,受位于其两侧调控域内的增强子调节,沿肢体近远轴(proximal-distal axis)呈时空线性表达。在人类基因组中,基因簇及其两侧的调控区域分布有串联排列的CTCF结合位点(简称CTCF位点),参与9个基因的表达调控。本研究以基因簇为模式基因,探究CTCF对发育基因(developmental genes)转录调控的影响。利用CRISPR DNA片段编辑技术在人HEK293T细胞中获得一系列的串联反向CTCF位点删除的单细胞克隆株。RNA-seq实验揭示CTCF位点删除后基因表达下降。定量高分辨率染色体构象捕获实验显示,与上游增强子簇的远距离染色质相互作用增强,与下游增强子簇的远距离染色质相互作用减弱。综上所述,串联反向的CTCF位点通过其绝缘子功能维持上下游增强子簇对基因簇表达调控的平衡,为探究动物发育过程中基因表达的精准调控机制提供参考。
基因簇;CTCF位点;调控平衡;增强子;绝缘子
染色质架构蛋白CTCF(CCCTC-binding factor)是一类结合DNA的锌指蛋白,在哺乳动物中可与基因组上调控区域内大量的绝缘子结合,对发育基因的精准调控起到关键作用[1~6]。CTCF最初在鸡中被发现是一种转录抑制因子或共抑制因子[7],CTCF结合位点(CTCF-binding site,CBS)序列在脊椎动物中高度保守,超过30多万的CBS广泛分布在整个基因组中[8]。CTCF与cohesin复合物通过环挤出模型(loop extrusion model)共同介导远距离染色质相互作用,参与基因的调控[3,9,10]。CBS序列的方向性直接影响染色质远距离互作,分别被正向和反向CBS元件锚定的增强子和启动子在CTCF/cohesin介导下形成远距离染色质环[2,3,5,11~14]。成对的反向–正向CBS (背靠背)通常分布在拓扑相关结构域(topological- associated domain, TAD)边界,发挥绝缘子的作用,阻止增强子跨区域异位激活基因表达[3]。CTCF除了能与cohesin一起介导染色质环,还能与其他的非编码调控元件及架构蛋白(如YY1)共同搭建三维基因组结构[1,15,16]。CTCF参与调控的染色质三维空间结构是生物体发挥正常生理功能的基础,包括神经系统中原钙粘蛋白(protocadherin,)基因簇启动子的选择[4,13,14,17]、尿苷二磷酸葡萄糖醛酸转移酶1 (UDP-glucuronosyltransferase 1,)的表达[12]、免疫球蛋白(Immunoglobulin,)和T细胞受体(T cell receptor,)的V(D)J重组[18~20]、四肢发育过程中的同源框基因家族(homeobox gene family,)控制[21~23]以及生物学的其他方面[1,24]。
脊椎动物和果蝇基因簇内的CTCF结合位点经过数亿年进化仍然存在,这暗示了在两侧对称动物进化历程中CTCF参与基因表达调控的古老起源[25]。基因簇编码的转录因子调控着细胞分化的多个方面,影响生物体四肢发育、器官形成等[26]。在哺乳动物中,基因分布在四个簇中,分别命名为、、和,根据序列同源性及在簇中的位置,基因分为13组(~) (图1A),躯干和四肢的不同区域表达基因的不同组合[27]。在发育的最初阶段,基因簇不表达,在发育过程中主要沿胚胎首尾轴(head-to-tail axis)呈时空线性表达[28]。
基因簇包括、、、~,整体位于两个拓扑结构域边界,受上游增强子簇(upstream enhancer cluster)和下游增强子簇(downstream enhancer cluster)调控(图1,A和B)。在小鼠胚胎肢芽(limb bud)发育早期,位于基因簇3′端粒侧TAD(telomeric domain, T-DOM)处于激活状态,控制~在四肢的近端转录[29];随后,在胚胎肢芽(limb bud)远端细胞中,位于5′中心粒侧TAD (centromeric domain, C-DOM)处于激活状态,负责~在手指细胞中的表达[30,31]。在小鼠胚胎肢芽中,上游增强子簇中的5个增强子(Island1~5)包含许多保守的非编码调控元件,同时富集有H3K4me1、H3K27ac(活性增强子标记),与~有很强的互作[31]。删除部分增强子只会影响它们与~基因的互作,但对~基因的表达影响不大;当完全删除上游增强子簇,~基因表达才会完全消失。这些研究表明这些Island之间可能存在冗余性和互补性[31]。在小鼠生殖器结节(genital tubercle,GT)中,上游增强子簇中的GT1和GT2与启动子也有远距离DNA相互作用,但GT2仅在生殖器中表现出较强的活性,在肢端细胞中不能显著激活基因转录[32]。在小鼠肢芽近端细胞中,下游增强子簇中的CS38-41 (conserved sequence element 38-41)参与调控~的表达[21]。这些上下游增强子簇通过转录因子或架构蛋白介导的染色质环与基因启动子实现远程互作,从而完成特定的基因调控[33~35]。然而,目前对于基因簇调控机制的研究主要借助于在小鼠中对其调控区域或者基因进行大片段删除或反转[21,22,31,32,36],对于人细胞中基因簇调控机制的研究较少。
本研究以人基因簇为模式基因,探索CTCF对发育基因(developmental genes)转录调控的影响。利用CRISPR DNA片段编辑系统[37~40]在人HEK293T细胞中获得一系列的串联反向CBS删除的单细胞克隆株。进行RNA-seq实验检测CBS删除后基因表达水平的变化,最后通过定量高分辨率染色体构象捕获(quantitative high resolution chromosome conformation capture copy, QHR-4C)[13]进一步观测CBS删除对基因簇三维基因组结构的影响,发现成串反向排列的CTCF位点具有平衡上下游增强子簇对基因簇转录调控的作用,为揭示哺乳动物基因转录的复杂调控提供参考。
1 材料与方法
1.1 实验材料
人胚胎肾细胞系HEK293T从中国科学院细胞库购买;胰酶、青霉素/链霉素双抗购自美国Gibco公司;DMEM培养基购自美国HyClone公司;胎牛血清购自南美ExCell公司;Lipofectamine 3000购自美国Invitrogen公司;pcDNA3.1-Cas9-WT质粒来源于北京大学席建忠教授;Ⅰ限制性内切酶、T4 DNA连接酶、Ⅱ限制性核酸内切酶、RNA文库制备试剂盒、Q5 Hot Start HiFi PCR Master Mix均从美国NEB公司采购;pGL3-U6-sgRNA-Puromycin- BsaⅠ质粒由上海科技大学黄行许教授赠予;质粒小量提取试剂盒、DNA凝胶回收试剂盒均从美国AXYGEN公司采购;MinElute Gel Extraction kit从德国QIAGEN公司采购;Green Taq Mix、dNTPs、高保真Phanta DNA聚合酶购自南京诺唯赞生物科技有限公司;pClone007 Versatile Simple Vector购自北京擎科新业生物技术有限公司;sgRNA、测序引物由上海生物工程(上海)股份有限公司合成;Triton X-100、10%SDS、DNA提取液、NP40等从美国SIGMA公司采购;三氯甲烷、硼酸和乙醇购自上海沪试实验室器材股份有限公司;异丙醇和氯化钠购自上海凌峰化学试剂有限公司;链霉亲和素磁珠从美国Invitrogen公司采购;PCR文库纯化试剂盒从瑞士Roche公司采购;甲醛溶液、RNA酶A、糖原等均从美国Thermo公司采购;TRIzol Reagent从美国Ambion公司采购;AMPure XP Beads从美国BeckmanCoulter公司采购;CTCF抗体从英国Abcam公司采购。

图1 CTCF位点在Hox基因簇及其调控区域内的分布
A:果蝇及哺乳动物基因家族的基因组结构。果蝇基因簇分为触足复合群(ANT-C)和双胸复合群(BX-C),其中ANT-C包含、、、和基因,BX-C包含、和基因。哺乳动物具有4个基因簇:、、和,共包含39个基因,其中基因簇包括、、、~。果蝇及哺乳动物相同颜色的基因为直系同源基因(orthologues),它们起源于同一祖先基因。B:人胚胎肾细胞系HEK293T中的CTCF蛋白、架构蛋白YY1、增强子标记H3K4me1和H3K27ac、启动子标记H3K4me3、转录活性标记PolⅡ和p300在人基因簇及其调控区域内的分布。红色虚线框指示增强子Island2、Island5、GT2、CS38-41和基因簇所在区域。哺乳动物的基因簇位于3′端粒侧TAD (T-DOM)和5′中心粒侧TAD (C-DOM)交界处,并受到上下游增强子簇(upstream enhancer cluster和downstream enhancer cluster)的调控。C:人类基因簇区域CTCF位点的分布。人肾脏和胚胎肾细胞系HEK293T的CTCF ChIP-seq结合峰分布图:基因簇C-DOM和T-DOM交界区域均具有串联排列的CTCF位点。红色虚线框依次指示CBS a、b、c和e所在区域。D:小鼠基因簇区域CTCF位点的分布。小鼠肾脏和第12.5天胚胎肢芽CTCF ChIP-seq数据显示小鼠基因簇中心粒侧对应的位置具有串联反向排列的4个CTCF位点。图C和D中箭头代表CTCF位点,其中红色箭头代表正向CTCF位点,蓝色箭头代表反向CTCF位点。
1.2 细胞培养
HEK293T细胞的培养基为89% DMEM完全培养液、10%胎牛血清和1%青霉素/链霉素双抗的混合液,在37℃、5% CO2的恒温培养箱中培养。
1.3 HEK293T细胞CTCF的ChIP-seq
收集2×107细胞后,首先用1%甲醛交联细胞,再加入终浓度为0.125 mol/L的甘氨酸溶液终止反应,然后用预冷的ChIP缓冲液裂解细胞两次(裂解条件均为4℃,缓慢旋转10 min),离心后再用预冷的ChIP缓冲液重悬细胞,冰上孵育10 min。用非接触式超声仪进行超声,凝胶电泳鉴定超声后DNA片段长度为100~10,000 bp。将细胞悬液离心后的上清转移至新的离心管中并加入50 µL的protein A/G磁珠孵育2 h。用磁力架弃去磁珠,将上清转移至新的离心管中加入4 µg的CTCF抗体,4℃缓慢旋转孵育过夜。第二天加入50 µL的protein A/G磁珠,4℃缓慢旋转孵育3 h。然后用ChIP缓冲液、高盐缓冲液、无盐缓冲液、LiCl缓冲液清洗磁珠(每次4℃缓慢旋转孵育10 min),最后用洗脱缓冲液洗脱DNA。含有DNA的溶液经过RNA酶A (37℃震荡孵育2 h)、蛋白酶K (55℃静置孵育2 h)及酚氯仿试剂纯化后,DNA沉淀用无核酸酶的水溶解,最后用诺唯赞公司的ChIP-seq试剂盒ND607-02构建DNA文库。文库通过苏州金唯智生物科技有限公司的Illumina HiSeq平台进行测序,根据index序列(P7-index序列见表1)对数据进行拆分,通过Bowtie2比对到GRCh37/hg19基因组,用BamCoverage进行归一化处理,标准化到RPKM (reads per kilobase per million mapped reads),共做两个重复,每个样品文库测序约2000万条序列。

续表
sgRNA引物中带下划线部分为构建质粒所需的粘性末端。P7-index中粗体带下划线部分为index。
1.4 HEK293T细胞中组蛋白修饰、转录活性标志及CTCF的ChIP-seq数据分析
人胚胎肾细胞系HEK293T的H3K4me1和H3K27ac ChIP-seq数据来源于ENCODE数据库,数据编号分别为ENCSR000FCG和ENCSR000FCH;YY1、H3K4me3、PolⅡ、p300来源于GEO,数据编号分别为GSM3636215、GSM945288、GSM935534、GSM1239071;人肾脏CTCF ChIP-seq数据的GEO编号为GSM1006886;小鼠肾脏及第12.5天胚胎肢芽CTCF ChIP-seq数据的GEO编号分别为GSE91529、GSM4665698;将数据分别比对到GRCh37/hg19及GRCm38/mm10基因组进行分析。
1.5 构建pGL3-U6-sgRNA质粒
按照表1合成单链sgRNA (共10条,分别合成双链sgRNA a1、sgRNA b1、sgRNA c1、sgRNA e1和sgRNA e2),按照如下体系(总体积为20 µL,)进行退火步骤:2 µL sgRNA-F,2 µL sgRNA-R,2 µL 10 × NEB Buffer 2,14 µL ddH2O;退火条件:95℃预变性5 min,缓慢降温(从第二个循环开始,每个循环20 s、降温0.2℃,共351个循环)。环状pGL3- U6-sgRNA-Puromycin-BsaⅠ质粒经过Ⅰ限制性核酸内切酶处理后,得到线性的pGL3-U6-sgRNA- Puromycin载体。将退火后的双链sgRNA引物与线性化的pGL3-U6-sgRNA-Puromycin载体在室温进行连接,经感受态细胞转化培养后,挑取单菌落进行培养(每种sgRNA至少挑取3个单菌落),用质粒小量提取试剂盒纯化质粒并测序。
1.6 细胞转染与单克隆化
待12孔板内的HEK293T细胞汇合度达到70%~ 90%左右时进行转染,一个离心管中加入Lipofectamine 3000与MEM混合均匀室温静置5 min,另一个管中加入Cas9质粒、两种sgRNA质粒、p3000试剂和MEM培养基混合均匀后室温静置5 min (此步骤中sgRNA组合:sgRNA a1/sgRNA e2或sgRNA b1/sgRNA e2或sgRNA c1/sgRNA e2或sgRNA e1/sgRNA e2),然后将Lipofectamine 3000加入其中,混合均匀后室温孵育约15 min,滴入培养细胞中。
待转染时间达到48 h后,换用含有2 μg/mL嘌呤霉素的培养基对转染细胞进行药物筛选。药物筛选大约4天后,更换为DMEM完全培养基继续培养2天,然后用胰酶消化收集适量细胞至PCR管中,加入10 µL碱裂解液,混匀置于PCR仪中,98℃ 30 min使细胞裂解,再加入10 µL中和液,获得细胞基因组。在sgRNA上下游位置设计PCR引物,鉴定转染后的细胞中是否含有目的片段删除的细胞,若存在,则对12孔板中剩余的转染细胞进行计数,用培养基稀释至浓度约为1个细胞每100 µL培养液,转移至96孔板中培养。待细胞生长一周左右,显微镜下查看单一细胞团并做好标记。sgRNA和PCR引物序列见表1。
1.7 单细胞克隆株鉴定
将标记的单细胞克隆株(共1056个)用胰酶消化,转移适量细胞到含有10 µL碱裂解液的PCR小管中进行裂解反应获取细胞基因组。两个sgRNA对大片段DNA进行删除时,会造成两个切口,需要通过特异性引物鉴定排除片段反转的情况[37,41]。通过PCR反应(PCR引物序列见表1)及DNA琼脂糖凝胶电泳实验挑选出目的片段删除的纯合子单细胞克隆株,将细胞目的片段删除型的扩增产物通过凝胶电泳分离,切胶回收并送测。若测序结果为双峰,则需要进行TA克隆测序鉴定单细胞克隆株的基因型。
按照TA克隆产品推荐用量将PCR产物与pClone007 Versatile Simple Vector混合均匀,室温孵育约10 min;在离心管中将连接产物与50 µL感受态细胞混合均匀,冰上静置30 min后,进行42℃热激45 s,然后迅速转移至冰上,2 min后加入500 µL的LB无抗培养基,然后将离心管放在37℃摇床上培养1 h。离心后去除450 µL上清,重悬细胞后均匀涂布在含有氨苄抗性的LB固体培养基上,37℃恒温箱中培养约16 h,次日挑取8~10个单菌落测序,然后将桑格测序结果与目的片段序列比对,确定单细胞克隆株基因型。最终获得CBS e删除、CBS c-e删除、CBS b-e删除和CBS a-e删除的单细胞克隆株各两株。
1.8 RNA-seq实验
待12孔板中处于对数生长期的HEK293T细胞、单细胞克隆株汇合度达到90%时,先用1 × PBS清洗2遍,然后加入500 µL/孔TRIzol溶液处理15 min,然后转移至无核酸酶的离心管中,用三氯甲烷、异丙醇、乙醇提取纯化总RNA。根据试剂说明书进行如下操作:使用Poly(A) mRNA Magnetic Isolation Module试剂盒提取mRNA,利用磁珠对mRNA片段进行分离与片段化;通过PCR反应先进行cDNA第一链合成,再进行cDNA第二链合成反应;双链cDNA连接Adaptor之后,用AMPure XP Beads和新鲜配制的80%的乙醇对DNA进行纯化,进行文库扩增反应;扩增后产物用AMPure XP Beads和新鲜配制的80%的乙醇进行纯化,最后用无核酸酶的水洗脱DNA文库。用Qubit3 Fluorometer测量终产物浓度后送至苏州金唯智生物科技有限公司的Illumina HiSeq平台进行测序,根据特异性index序列(表1)拆分数据,用STAR、cufflinks等软件分析数据[42]。本研究共有30个RNA-seq样品,每个样品文库测序约1000万条序列。
1.9 定量高分辨率染色体构象捕获实验QHR-4C
用10 cm培养皿扩增培养HEK293T细胞、单细胞克隆株,待细胞汇合度达到80%~90%且生长良好时,用胰酶消化离心收集细胞;用1%甲醛交联细胞,再加入终浓度为0.125 mol/L的甘氨酸溶液终止反应;用预冷的1 × PBS重复清洗细胞,然后配制4C裂解液(1 × inhibitor、pH 7.5 50 mmol/L Tris-HCl、150 mmol/L NaCl、5 mmol/L EDTA、0.5% NP-40和1% Triton X-100)重复裂解细胞;通过Triton X-100、Ⅱ限制性内切酶处理使DNA片段化,用T4 DNA连接酶捕捉相互邻近的DNA片段;纯化连接后的DNA,然后进行超声处理(不同长度的DNA片段经琼脂糖凝胶电泳鉴定呈弥散状分布且主要片段长度小于1000 bp)、PCR线性扩增反应及接头连接反应,最后进行DNA文库扩增。文库送至苏州金唯智生物科技有限公司的Illumina HiSeq平台进行测序,根据index(序列见表1)对测序结果进行拆分,通过Bowtie2、Samtools、r3C-seq等方法处理数据[13]。本研究每个实验三个重复,共有78个4C样品,每个样品文库测序约800万条序列。
1.10 高通量测序数据信息
本研究的相关结果数据已收录在国家基因库生命大数据平台(CNGBdb)[43]的国家基因库序列归档系统(CNSA)[44],项目编号:CNP0001773。
2 结果与分析
2.1 CTCF位点在HOXD基因簇的分布高度保守
基因簇最初因引起黑腹果蝇翅膀数目改变而被发现,在两侧对称动物中高度保守[45]。果蝇基因组中含有一个基因簇,由8个基因组成,分为触足复合群(ANT-C)和双胸复合群(BX-C),其中ANT-C包含、、、和基因,BX-C包含、和基因(图1A)。而哺乳动物基因组具有四个基因簇:、、和,共包括39个基因[27](图1A),这可能是由进化过程中的两次DNA片段复制过程产生[46,47]。本研究以人HEK293T细胞为模型,通过分析ChIP-seq数据发现位于C-DOM和T-DOM边界的基因簇串联排列着多个CTCF位点,提示CTCF可能对的转录调控发挥重要作用(图1B)。
利用ChIP-seq数据进一步分析了与转录调控密切相关的转录因子和各组蛋白修饰在基因簇及其两侧调控区域内富集的情况,包括CTCF蛋白、架构蛋白YY1、增强子标记H3K4me1、H3K27ac,启动子标记H3K4me3,转录活性标记PolⅡ、p300(图1B):基因簇所在的区域有大量的CTCF蛋白、YY1蛋白富集以及H3K27ac、H3K4me3组蛋白修饰,是一个转录活跃区域;Island2、Island5处有CTCF蛋白结合峰,但是没有相应的组蛋白修饰,也没有转录活性标记(PolⅡ和p300),提示Island2、Island5在HEK293T细胞中可能没有增强子活性。GT2处没有CTCF蛋白结合,有YY1的结合峰、H3K4me1和H3K27ac等组蛋白修饰(图1B),说明在HEK293T中增强子GT2可能不完全依赖CTCF激活基因转录;CS38-41处有CTCF、YY1结合以及H3K27ac组蛋白修饰,同时富集PolⅡ和p300,因此在HEK293T细胞中具有较强的增强子活性。
为了分析基因簇处CTCF分布的保守性,本研究比较了人肾脏和人胚胎肾细胞系HEK293T中CTCF的ChIP-seq结合峰分布,发现基因簇C-DOM和T-DOM交界区域均分布着成簇排列的CBS,靠近C-DOM处具有5个串联排列的反向CBS,分别记为CBS a、b、c、d和e(图1C)。此外,分析小鼠肾脏及第12.5天胚胎肢芽CTCF的ChIP-seq数据发现,小鼠基因簇中心粒侧对应的位置串联排列着类似的CBS簇(图1D)。综上所述,C-DOM和T-DOM边界处串联排列的反向CBS簇在哺乳动物中高度保守,CTCF可能通过结合该CBS簇参与指导两侧调控区域内的增强子对基因簇的转录表达调控。
2.2 边界处单个CTCF位点删除影响HOXD基因簇表达
为了研究反向串联排列的CBS a、b、c、d和e对人基因簇调控的作用,首先用CRISPR/ Cas9编辑技术对CBS e进行删除。我们针对CBS e设计一对sgRNA (sgRNA e1和sgRNA e2),Cas9核酸酶在sgRNA介导下特异性识别CBS e两侧靶向序列后进行切割,经过修复后能够获得CBS e删除的编辑细胞(图2A)。利用PCR筛选单细胞克隆株(图2B),对获得的单细胞克隆株进行TA克隆并测序确定其基因型(图2C)。获得的e#113存在两种不同的染色体基因型,Allele 1是853 bp片段删除的基因型,其切口连接处有小片段DNA删除,Allele 2是Cas9蛋白在PAM位点上游第3位切割后精确DNA修复产生的831 bp片段删除的基因型[41,48,49];e#126也存在两种不同的染色体基因型,其中一种是Cas9蛋白精准切割在PAM位点上游第3位后修复,另一种可能是Cas9蛋白切割在PAM位点上游第3位和第4位[48]:基因型鉴定及测序结果均证明e#113和e#126细胞中CBS e已被删除(图2C)。
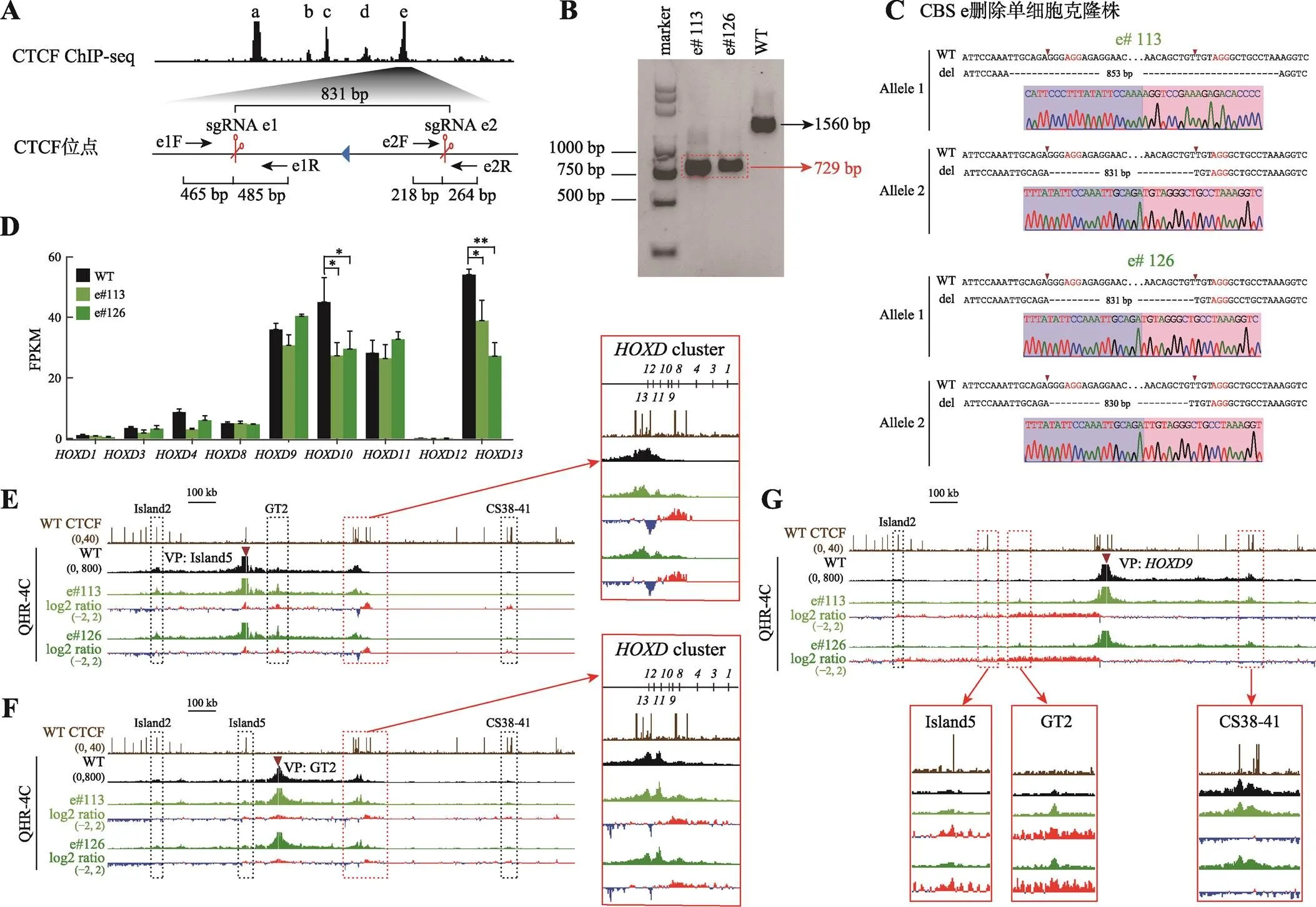
图2 CBS e删除改变增强子与启动子间的远程互作从而影响HOXD基因簇的基因表达
A:利用CRISPR DNA片段编辑技术获得CBS e删除的单细胞克隆株(e#113和e#126)的示意图。针对CBS e设计一对sgRNA(sgRNA e1和sgRNA e2),Cas9核酸酶在sgRNA e1和sgRNA e2的介导下特异性识别CBS e两侧靶向序列后进行切割,形成的两个切口被修复后连接在一起获得CBS e删除的编辑细胞。B:采用特异性引物PCR后进行凝胶电泳实验鉴定单细胞克隆株。引物对e1F/e2R在野生型(WT)细胞中扩增出1560 bp的片段,在e#113和e#126细胞株中扩增出CBS e删除后的729 bp片段(红色虚线框指示目的条带)。C:TA克隆并进行桑格测序确定单细胞克隆株的基因型。引物对e1F/e2R在e#113和e#126细胞株中扩增的产物经TA克隆后的测序结果图。D:RNA-seq数据分析比较WT细胞、e#113和e#126细胞株中基因簇表达水平。*:<0.05;**:<0.01;FPKM:fragments per kilobase of transcript per million mapped reads。E:在染色质构象捕获(QHR-4C)实验中,以增强子Island5为观测点(viewpoint,VP),分析e#113和e#126细胞株中Island5与基因簇启动子及调控元件Island2、GT2和CS38-41的远程互作。将e#113和e#126细胞株的数据分别与WT进行log2处理,红色实线框内为Island5与基因簇启动子之间的染色质相互作用,黑色虚线框依次指示调控元件Island2、GT2和CS38-41。F:以增强子GT2为VP,分析CBS e删除后GT2与基因簇启动子的远程互作。红色实线框内为GT2与基因簇启动子间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、Island5和CS38-41。G:以启动子为VP,分析e#113和e#126细胞株中调控元件Island2、Island5、GT2、CS38-41与启动子之间的染色质相互作用。黑色虚线框指示Island2,红色实线框内分别为Island5、GT2和CS38-41与启动子之间的染色质相互作用放大图。
为了研究单个CBS e删除是否会影响基因转录,对获得的单细胞克隆株进行RNA-seq实验。在HEK293T细胞中,~以及表达量较高;CBS e删除后及的表达水平降低最显著(图2D)。为了探究CBS e删除对基因表达影响的机制,对获得的e#113和e#126单细胞克隆株进行QHR-4C实验,分别以Island5、GT2及为观测点,分析CBS e删除后细胞内基因簇染色质高级结构变化。在WT细胞中,增强子Island5、GT2与基因簇之间存在显著的DNA相互作用(图2,E和F),启动子主要与下游增强子簇中的CS38-41有远距离DNA相互作用(图2G)。删除CBS e后~启动子与Island5之间相互作用有显著增加(图2E)。除了与,其余基因启动子与GT2的相互作用略微增加(图2F)。启动子与Island5、GT2的远程互作有明显增加,与前述结果一致,其与CS38-41的相互作用略微减少(图2G)。综上所述,单个CBS e删除引起基因簇启动子与增强子之间的远距离DNA相互作用改变,导致及表达水平降低。
2.3 边界处多个CTCF位点删除对HOXD基因簇远距离DNA相互作用的影响具有叠加效应
为了探究多个CBS删除对基因转录水平的影响,我们对CBS c-e (图3)和CBS b-e (图4)进行删除。分别设计一对sgRNA (sgRNA c1/sgRNA e2和sgRNA b1/sgRNA e2)对HEK293T细胞进行编辑(图3A和图4A),再用特异性引物PCR进行凝胶电泳鉴定(图3B和图4B)。将目的条带切胶纯化、TA克隆并进行桑格测序鉴定细胞的基因型,最终获得了CBS c-e删除的c-e#49和c-e#54单细胞克隆株(图3C)以及CBS b-e删除的b-e#59和b-e#80单细胞克隆株(图4C)。
通过RNA-seq实验发现,在CBS c-e和CBS b-e删除的细胞(这些细胞的已删除)中,、及的转录水平下降,其中下降最显著;的转录水平分别降低了约40%和56%(图3D和图4D)。QHR-4C实验显示,无论在CBS c-e还是CBS b-e删除的细胞中,~与Island5之间的远程互作均增加(图3E和图4E),与GT2的互作也增加,且幅度比CBS e单独删除型细胞大(图3F和图4F)。在CBS b-e删除的细胞中,启动子与增强子Island5及GT2的远程互作相对CBS c-e删除型细胞进一步增强(图3G和图4G),并且在Island5介导的远程互作中新增了下游增强子簇(CS38-41)(图3E和图4E);启动子与CS38-41之间的相互作用没有进一步减弱(图3G和图4G)。综上所述,边界处CBS删除的数量对上游增强子簇与基因簇远程互作的影响有叠加效应,但不会递进式地影响整个基因(除了)的转录表达水平。
2.4 边界处CTCF位点的全部删除破坏了增强子调控HOXD基因簇表达的平衡
CTCF常富集于TAD区域边界,对维持相关拓扑结构域的稳定具有重要作用。为了探究整个CBS a-e对基因簇表达调控的影响,设计一对sgRNA(sgRNA a1和sgRNA e2)将CBS a-e删除(图5A),进行PCR鉴定(图5B)、TA克隆并进行桑格测序(图5C)获得a-e#93和a-e#108单细胞克隆株。对CBS a-e删除的细胞进行RNA-seq实验发现,、与的基因表达显著降低,其中最显著,降低了约70% (图5D)。QHR-4C实验发现,Island5与下游增强子簇(CS38-41)的互作进一步增加,原本与CBS a-e整体的互作进一步转移至下游的一个反向CBS附近,从而使其与基因簇内启动子的互作从~扩展至~(图5E);GT2与~启动子的互作增强,同时与T-DOM的染色质远距离互作增强(图5F)。以Island5、GT2为观测点发现一个有意思的现象:有CTCF结合的Island5与基因簇的染色质相互作用区域具有明显的边界(图2E、3E、4E和5E),而没有CTCF结合的增强子GT2与基因簇区域的染色质相互作用呈现广泛弥散式分布(图2F、3F、4F和5F),没有明显的边界,这提示具有CTCF结合的调控元件以及没有CTCF结合的调控元件与基因启动子之间的染色质相互作用模式是不同的。以启动子为VP发现,在HEK293T野生型细胞中,启动子主要与下游增强子簇有DNA相互作用;而在CBS a-e删除的细胞中,启动子与上游增强子簇(Island2、Island5、GT2、GCR和Prox)的远程互作普遍显著增强,与下游增强子簇(CS38-41)的远程互作普遍减弱,CBS a-e删除改变了的转录调控模式,由最初主要受下游增强子簇调控转变为上下游两个增强子簇调控(图5G),原有的调控平衡被破坏,这可能是导致~转录水平急剧降低的原因。
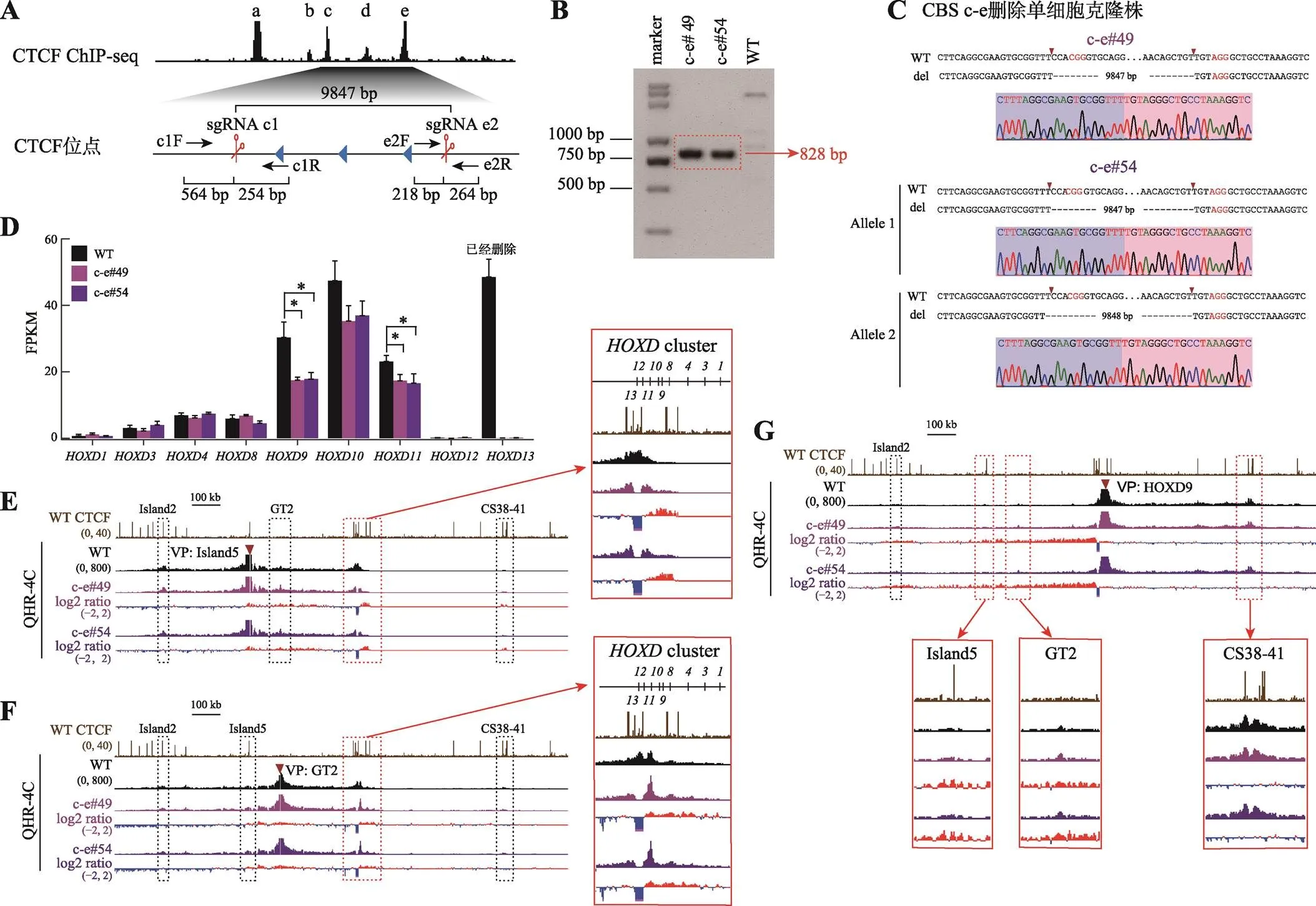
图3 CBS c-e删除引起上游增强子与近端HOXD基因相互作用的增强
A:设计一对sgRNA(sgRNA c1和sgRNA e2)对HEK293T野生型细胞进行编辑获得CBS c-e删除的单细胞克隆株(c-e#49和c-e#54)的示意图。B:用引物c1F和e2R PCR鉴定c-e#49和c-e#54单细胞克隆株的凝胶电泳图。C:TA克隆鉴定c-e#49和c-e#54单细胞克隆株基因型的测序结果图。D:RNA-seq数据分析比较WT细胞、c-e#49和c-e#54细胞中基因簇表达水平。E:QHR-4C实验中,以Island5为VP,分析c-e#49和c-e#54细胞株中Island5与基因簇启动子的远程互作。红色实线框内为Island5与基因簇启动子之间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、GT2和CS38-41。F:以GT2为VP,分析CBS c- e删除后GT2与基因簇启动子的远程互作。红色实线框内为GT2与基因簇启动子间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、Island5和CS38-41。G:以启动子为VP,c-e#49和c-e#54细胞株中Island2、Island5、GT2、CS38-41与启动子之间的染色质相互作用。黑色虚线框指示Island2,红色实线框内分别为Island5、GT2和CS38-41与启动子之间的染色质相互作用放大图。
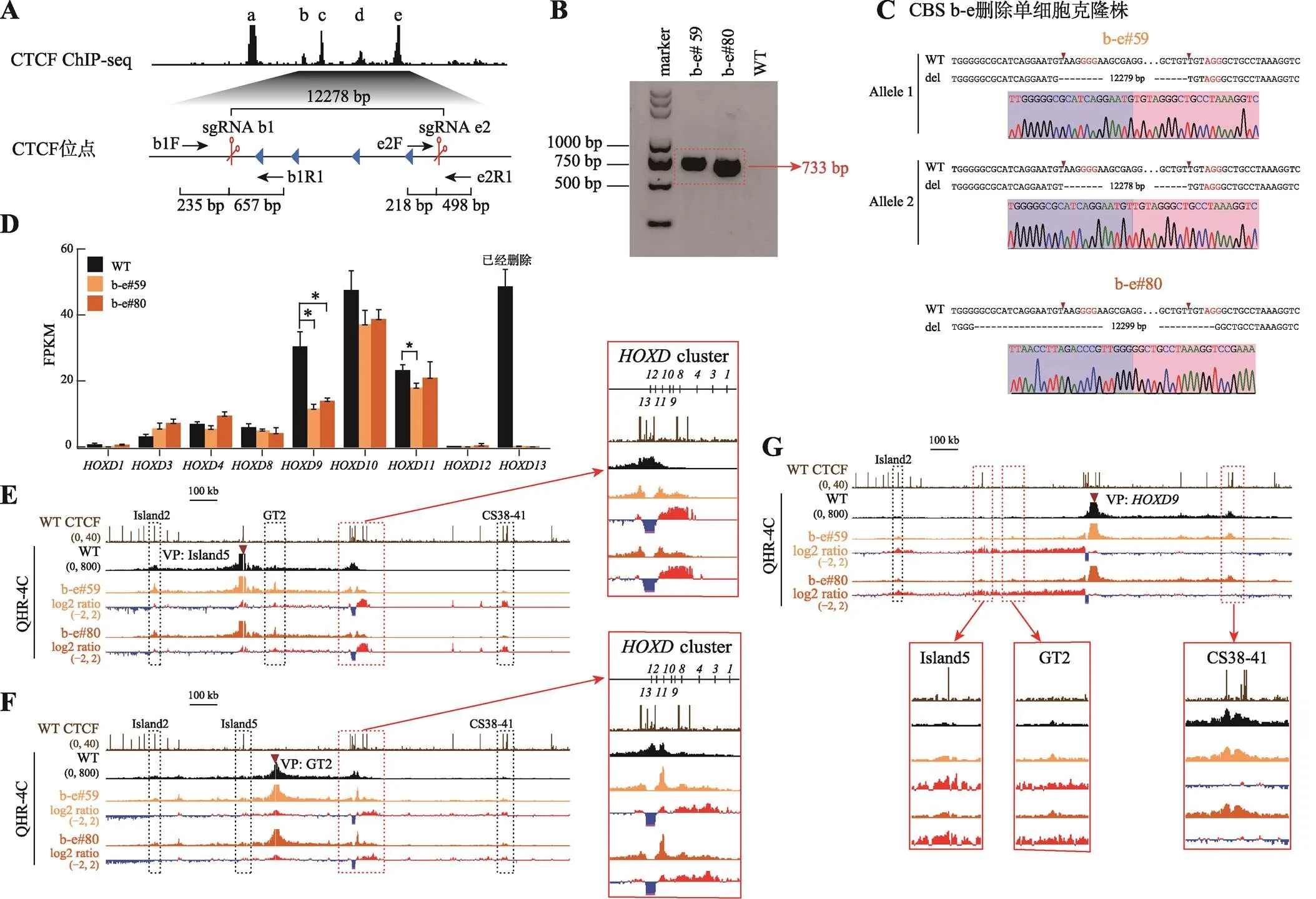
图4 串联排列CTCF位点对增强子远程互作和HOXD基因表达具有叠加效应
A:利用CRISPR/Cas9编辑系统获得CBS b-e删除的单细胞克隆株(b-e#59和b-e#80)的示意图。B:用引物b1F和e2R1进行PCR鉴定b-e#59和b-e#80单细胞克隆株的凝胶电泳图。C:TA克隆鉴定b-e#59和b-e#80单细胞克隆株基因型的测序结果图。D:RNA-seq数据分析比较WT细胞、b-e#59和b-e#80细胞株细胞中基因簇表达水平。E:QHR-4C实验中,以Island5为VP,分析CBS b-e删除对Island5与基因簇启动子远程互作的影响。红色实线框内为Island5与基因簇启动子之间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、GT2和CS38-41。F:以GT2为VP,分析b-e#59和b-e#80细胞株中GT2与基因簇启动子的远程互作。红色实线框内为GT2与基因簇启动子间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、Island5和CS38-41。G:以启动子为VP,分析b-e#59和b-e#80细胞株中Island5、GT2、CS38-41与启动子间的染色质相互作用。黑色虚线框指示Island2,红色实线框内分别为Island5、GT2和CS38-41与启动子之间的染色质相互作用放大图。
以上结果说明,C-DOM和T-DOM边界处的反向串联排列的CBS主要调控~及的基因表达。在HEK293T细胞中,Island2、Island5没有增强子活性,GT2不完全依赖于CTCF调控的表达,上游增强子簇与基因簇启动子的远程互作增加,削弱了下游增强子簇中的CS38-41对基因簇的激活强度。边界处单个CBS删除就会影响基因簇原有的三维基因组结构,改变和的基因表达。多个CBS删除对基因簇三维基因组结构以及转录的影响具有叠加效应,但是没有大幅度改变其他基因的表达。当边界处整个CBS a-e全部删除后,基因簇与CS38-41的染色质相互作用进一步减弱,转录水平急剧降低,并且上下游增强子簇之间出现了远距离相互作用。综上所述,基因簇靠近中心粒侧区域的反向串联排列CTCF位点具有绝缘子功能,通过阻断上游增强子簇与的染色质远程互作,维持基因簇表达调控的平衡,使基因能够精准有序地表达。

图5 删除全部反向CTCT位点破坏增强子调控HOXD基因表达的平衡
A:获得CBS a-e删除的单细胞克隆株(a-e#93和a-e#108)的示意图。B:用引物a1F和e2R PCR鉴定a-e#93和a-e#108细胞株的凝胶电泳图。C:TA克隆鉴定a-e#93和a-e#108单细胞克隆株基因型的测序结果图。D:RNA-seq数据分析比较WT细胞、a-e#93和a-e#108细胞株细胞中基因簇转录水平。E:QHR-4C实验中,以Island5为VP,分析CBS a-e删除对Island5与基因簇启动子及两侧调控区域染色质相互作用的影响。红色实线框内为Island5与基因簇启动子之间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、GT2和CS38-41。F:以GT2为VP,分析a-e#93和a-e#108细胞株中GT2与基因簇启动子的染色质相互作用。红色实线框内为GT2与基因簇启动子间的染色质相互作用放大图,黑色虚线框依次指示调控元件Island2、Island5和CS38-41。G:以启动子为VP,分析CBS a-e删除后的表达调控模式。黑色虚线框指示Island2,红色实线框内为Island5、GT2、CS38-41与9基因簇启动子间的染色质相互作用放大图。
3 讨论
在哺乳动物中,增强子的数量远远超过基因的数量,复杂基因的表达往往受多个增强子组合调控,防止在其适当范围之外的异位表达[50]。增强子活性的经典模式是相对独立的、自主的并且可叠加的,但是顺式调控区域内存在功能有冗余性(redundancy)的增强子[51,52]。增强子激活需要结合多个转录因子[53],转录因子结合增强子后,对基因表达起抑制作用或激活作用取决于结合的转录因子[54]。例如,转录因子RFX5与增强子的结合对原钙粘蛋白基因簇的表达起抑制作用[55]。增强子活性的强弱取决于招募的共激活蛋白对染色质的修饰和增强子的重塑,包括组蛋白乙酰基转移酶(例如p300/CBP)、组蛋白甲基转移酶(例如MLL3/4和CARM1)、染色质重塑因子(例如Brg1和CHD7),以及在启动子处促进转录的相关因子(例如中介体复合物)[56]。本研究中,CBS a-e删除后,基因簇与上游增强子簇中的Island5、GT2的远程互作显著增加,而的表达下降,这表明Island5、GT2与启动子之间有互作,但是它们没有增强子活性,这可能是因为在这两个增强子上结合了抑制性的共激活蛋白或转录因子[35,57]。小鼠胚胎发育过程中基因的表达就需要结合不同的转录因子[58]。

图6 HOXD基因簇的表达调控模式图
A:野生型(WT)细胞中基因簇的表达调控示意图。在野生型细胞中,CTCF位点具有绝缘子作用,阻碍了上游增强子簇中的Island2、Island5、GT2与基因簇启动子的远程相互作用,基因簇主要受下游增强子簇中的CS38-41调控。B:CTCF位点删除的细胞中基因簇的表达调控示意图。在CBS删除的细胞中,CTCF位点绝缘子作用消失,上游增强子簇与基因簇之间产生远距离DNA相互作用,导致基因簇双边调控的平衡被破坏、基因表达降低。
CTCF与cohesin的结合是一个动态过程[59],成串的CBS可以阻碍渗透的cohesin滑动,形成类似于“葫芦”结构的染色质环[1,17],由此介导的远距离增强子与启动子的相互作用可有效持久地调控基因的表达。我们先前利用小鼠和多种细胞模型对原钙粘蛋白和珠蛋白模式基因进行系统的研究[13],发现单个CTCF位点可确保适当的增强子绝缘和启动子激活,而串联排列CTCF位点的绝缘作用可维持基因组三维空间的动态平衡及准确的启动子选择。在本研究中,我们进一步利用HEK293T细胞模型对基因簇中的CTCF位点进行了系统的组合性删除实验,发现串联排列CTCF位点通过其绝缘功能维持上下游增强子簇调控基因表达的平衡。另外我们观察到一个有趣的现象,包含CTCF位点的增强子Island5与基因的染色质相互作用具有明显的边界,而不包含CTCF位点的增强子GT2与基因的染色质相互作用区域则分布较广泛,这和原钙粘蛋白基因簇中的增强子HS5-1 (包含CTCF位点)、HS7 (不包含CTCF位点)与基因染色质互作[13]的特点一致。
在本研究中,我们将基因簇C-DOM与T-DOM边界处CTCF位点进行逐步删除,随着剩余的CTCF位点数量减少,基因簇启动子与上游增强子簇DNA远程互作逐渐增强,与下游增强子簇的DNA远程互作逐渐减弱。当边界串联反向的CTCF位点全部删除后,基因启动子由最初与下游增强子簇有相互作用转变为与上下游增强子簇均有相互作用,并且上下游增强子簇之间出现了DNA远程互作(图6)。
综上所述,基因簇中串联反向CTCF位点扮演着绝缘子的角色,阻碍上游增强子簇跨区域调控近端基因的表达,对基因簇在肢体发育中时空线性表达的精细调控具有重要意义,为深入研究基因簇复杂调控网络提供了参考。
[1] Wu Q, Liu PF, Wang LY. Many facades of CTCF unified by its coding for three-dimensional genome architecture., 2020, 47(8): 407–424.
[2] Guo Y, Monahan K, Wu HY, Gertz J, Varley KE, Li W, Myers RM, Maniatis T, Wu Q. CTCF/cohesin-mediated DNA looping is required for protocadherin alpha promoter choice., 2012, 109(51): 21081– 21086.
[3] Guo Y, Xu Q, Canzio D, Shou J, Li JH, Gorkin DU, Jung I, Wu HY, Zhai YN, Tang YX, Lu YC, Wu YH, Jia ZL, Li W, Zhang MQ, Ren B, Krainer AR, Maniatis T, Wu Q. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function., 2015, 162(4): 900– 910.
[4] Zhai YN, Xu Q, Guo Y, Wu Q. Characterization of a cluster of CTCF-binding sites in a protocadherin regulatory region., 2016, 38(4): 323–336.
翟亚男, 许泉, 郭亚, 吴强. 原钙粘蛋白基因簇调控区域中成簇的CTCF结合位点分析. 遗传, 2016, 38(4): 323–336.
[5] Yin M, Wang J, Wang M, Li X, Zhang M, Wu Q, Wang Y. Molecular mechanism of directional CTCF recognition of a diverse range of genomic sites., 2017, 27(11): 1365–1377.
[6] Guo Y, Wu Q. Inversion of CTCF binding sites by DNA fragment editing alters genome topology and enhancer/ promoter functions., 2015, 37(10): 1073–1074.
郭亚, 吴强. 采用DNA片段编辑技术反转CTCF结合位点改变基因组拓扑结构和增强子与启动子功能. 遗传, 2015, 37(10): 1073–1074.
[7] Filippova GN, Fagerlie S, Klenova EM, Myers C, Dehner Y, Goodwin G, Neiman PE, Collins SJ, Lobanenkov VV. An exceptionally conserved transcriptional repressor, CTCF, employs different combinations of zinc fingers to bind diverged promoter sequences of avian and mammalian c-myc oncogenes., 1996, 16(6): 2802–2813.
[8] Chen HB, Tian Y, Shu WJ, Bo XC, Wang SQ. Comprehensive identification and annotation of cell type-specific and ubiquitous CTCF-binding sites in the human genome., 2012, 7(7): e41374.
[9] Nasmyth K. Disseminating the genome: Joining, resolving, and separating sister chromatids during mitosis and meiosis., 2001, 35: 673–745.
[10] Kim Y, Shi ZB, Zhang HS, Finkelstein IJ, Yu HT. Human cohesin compacts DNA by loop extrusion., 2019, 366(6471): 1345–1349.
[11] Lu YJ, Shou J, Jia ZL, Wu YH, Li JH, Guo Y, Wu Q. Genetic evidence for asymmetric blocking of higher-order chromatin structure by CTCF/cohesin., 2019, 10(12): 914–920.
[12] Zheng XF, Huang HY, Wu Q. Chromatin architectural protein CTCF regulates gene expression of thecluster., 2019, 41(6): 509–523.
郑晓飞, 黄海燕, 吴强. 染色质架构蛋白CTCF调控基因簇的表达. 遗传, 2019, 41(6): 509–523.
[13] Jia ZL, Li JW, Ge X, Wu YH, Guo Y, Wu Q. Tandem CTCF sites function as insulators to balance spatial chromatin contacts and topological enhancer-promoter selection., 2020, 21(1): 75.
[14] Wu YH, Jia ZL, Ge X, Wu Q. Three-dimensional genome architectural CCCTC-binding factor makes choice in duplicated enhancers atlocus., 2020, 63(6): 835–844.
[15] Dekker J, Mirny L. The 3D genome as moderator of chromosomal communication., 2016, 164(6): 1110– 1121.
[16] Nichols MH, Corces VG. A tethered-inchworm model of SMC DNA translocation., 2018, 25(10): 906–910.
[17] Wu Q, Jia ZL. Wiring the brain by clustered protocadherin neural codes., 2021, 37(1): 117–131.
[18] Lin SG, Ba ZQ, Alt FW, Zhang Y. RAG chromatin scanning during V(D)J recombination and chromatin loop extrusion are related processes., 2018, 139: 93–135.
[19] Chen L, Carico Z, Shih HY, Krangel MS. A discrete chromatin loop in the mouse Tcra-Tcrd locus shapes the TCRdelta and TCRalpha repertoires., 2015, 16(10): 1085–1093.
[20] Majumder K, Koues OI, Chan EAW, Kyle KE, Horowitz JE, Yang-Iott K, Bassing CH, Taniuchi I, Krangel MS, Oltz EM. Lineage-specific compaction ofrequires a chromatin barrier to protect the function of a long-range tethering element., 2015, 212(1): 107–120.
[21] Rodríguez-Carballo E, Lopez-Delisle L, Zhan Y, Fabre PJ, Beccari L, El-Idrissi I, Huynh THN, Ozadam H, Dekker J, Duboule D. Thecluster is a dynamic and resilient TAD boundary controlling the segregation of antagonistic regulatory landscapes., 2017, 31(22): 2264– 2281.
[22] Rodríguez-Carballo E, Lopez-Delisle L, Yakushiji- Kaminatsui N, Ullate-Agote A, Duboule D. Impact of genome architecture on the functional activation and repression ofregulatory landscapes., 2019, 17(1): 55.
[23] Rodríguez-Carballo E, Lopez-Delisle L, Willemin A, Beccari L, Gitto S, Mascrez B, Duboule D. Chromatin topology and the timing of enhancer function at thelocus., 2020, 117(49): 31231– 31241.
[24] Jia ZL, Wu Q. Clustered protocadherins emerge as novel susceptibility loci for mental disorders., 2020, 14: 587819.
[25] Heger P, Marin B, Bartkuhn M, Schierenberg E, Wiehe T. The chromatin insulator CTCF and the emergence of metazoan diversity., 2012, 109(43): 17507–17512.
[26] Lewis EB. A gene complex controlling segmentation in Drosophila., 1978, 276(5688): 565–570.
[27] Mallo M. Reassessing the role ofgenes during vertebrate development and evolution., 2018, 34(3): 209–217.
[28] Kmita M, Duboule D. Organizing axes in time and space; 25 years of colinear tinkering., 2003, 301(5631): 331–333.
[29] Andrey G, Montavon T, Mascrez B, Gonzalez F, Noordermeer D, Leleu M, Trono D, Spitz F, Duboule D. A switch between topological domains underliesgenes collinearity in mouse limbs., 2013, 340(6137): 1234167.
[30] Beccari L, Yakushiji-Kaminatsui N, Woltering JM, Necsulea A, Lonfat N, Rodríguez-Carballo E, Mascrez B, Yamamoto S, Kuroiwa A, Duboule D. A role forproteins in the regulatory switch between TADs at thelocus., 2016, 30(10): 1172–1186.
[31] Montavon T, Soshnikova N, Mascrez B, Joye E, Thevenet L, Splinter E, de Laat W, Spitz F, Duboule D. A regulatory archipelago controlsgenes transcription in digits., 2011, 147(5): 1132–1145.
[32] Lonfat N, Montavon T, Darbellay F, Gitto S, Duboule D. Convergent evolution of complex regulatory landscapes and pleiotropy atloci., 2014, 346(6212): 1004–1006.
[33] Long HK, Prescott SL, Wysocka J. Ever-changing landscapes: Transcriptional enhancers in development and evolution., 2016, 167(5): 1170–1187.
[34] Schoenfelder S, Fraser P. Long-range enhancer-promoter contacts in gene expression control., 2019, 20(8): 437–455.
[35] Kim S, Shendure J. Mechanisms of interplay between transcription factors and the 3D genome., 2019, 76(2): 306–319.
[36] Noordermeer D, Leleu M, Splinter E, Rougemont J, De Laat W, Duboule D. The dynamic architecture ofgene clusters., 2011, 334(6053): 222–225.
[37] Li JH, Shou J, Guo Y, Tang YX, Wu YH, Jia ZL, Zhai YN, Chen ZF, Xu Q, Wu Q. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9., 2015, 7(4): 284–298.
[38] Chang N, Sun C, Gao L, Zhu D, Xu X, Zhu X, Xiong JW, Xi JJ. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos., 2013, 23(4): 465–472.
[39] Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9., 2014, 346(6213): 1258096.
[40] Liu PF, Wu Q. Probing 3D genome by CRISPR/Cas9., 2020, 42(1): 18–31.
刘沛峰, 吴强. CRISPR/Cas9基因编辑在三维基因组研究中的应用. 遗传, 2020,42(1): 18–31.
[41] Li JH, Shou J, Wu Q. DNA fragment editing of genomes by CRISPR/Cas9., 2015, 37(10): 992–1002.
李金环, 寿佳, 吴强. CRISPR/Cas9系统在基因组DNA片段编辑中的应用. 遗传, 2015,37(10): 992–1002.
[42] Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L. Differential gene and transcript expression analysis of RNA-seq experiments with tophat and cufflinks., 2012, 7(3): 562–578.
[43] Guo XQ, Chen FZ, Gao F, Li L, Liu K, You LJ, Hua C, Yang F, Liu WL, Peng CH, Wang LN, Yang XX, Zhou FY, Tong JW, Cai J, Li ZY, Wan B, Zhang L, Yang T, Zhang MW, Yang LL, Yang YW, Zeng WJ, Wang B, Wei XF, Xu X. CNSA: A data repository for archiving omics data., 2020; 2020: baaa055.
[44] Chen FZ, You LJ, Yang F, Wang LN, Guo XQ, Gao F, Hua C, Tan C, Fang L, Shan RQ, Zeng WJ, Wang B, Wang R, Xu X, Wei XF. CNGBdb: China National Genebank Database., 2020, 42(08): 799–809.
陈凤珍, 游丽金, 杨帆, 王丽娜, 郭学芹, 高飞, 华聪, 谈聪, 方林, 单日强, 曾文君, 王博, 王韧, 徐讯, 魏晓锋. CNGBdb: 国家基因库生命大数据平台. 遗传, 2020, 42(8): 799–809.
[45] Pearson JC, Lemons D, McGinnis W. Modulatinggene functions during animal body patterning., 2005, 6(12): 893–904.
[46] Lonfat N, Duboule D. Structure, function and evolution of topologically associating domains (TADs) atloci., 2015, 589(20): 2869–2876.
[47] Holland PW, Garcia-Fernàndez J, Williams NA, Sidow A. Gene duplications and the origins of vertebrate development., 1994, 125–133.
[48] Shou J, Li J, Liu Y, Wu Q. Precise and predictable CRISPR chromosomal rearrangements reveal principles of Cas9-mediated nucleotide insertion., 2018, 71(4): 498–509 e4.
[49] Fu YF, Foden JA, Khayter C, Maeder ML, Reyon D, Joung JK, Sander JD. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells., 2013, 31(9): 822–826.
[50] Perry MW, Boettiger AN, Levine M. Multiple enhancers ensure precision of gap gene-expression patterns in the Drosophila embryo., 2011, 108(33): 13570–13575.
[51] Frankel N, Davis GK, Vargas D, Wang S, Payre F, Stern DL. Phenotypic robustness conferred by apparently redundant transcriptional enhancers., 2010, 466(7305): 490–493.
[52] Barolo S. Shadow enhancers: Frequently asked questions about distributed cis-regulatory information and enhancer redundancy., 2012, 34(2): 135–141.
[53] Buecker C, Wysocka J. Enhancers as information integration hubs in development: Lessons from genomics., 2012, 28(6): 276–284.
[54] Jolma A, Yin YM, Nitta KR, Dave K, Popov A, Taipale M, Enge M, Kivioja T, Morgunova E, Taipale J. DNA- dependent formation of transcription factor pairs alters their binding specificity., 2015, 527(7578): 384–388.
[55] Wang N, Jia ZL, Wu Q. RFX5 regulates gene expression of thecluster., 2020, 42(8): 760–774.
王娜, 甲芝莲, 吴强. RFX5调控原钙粘蛋白基因簇的表达. 遗传, 2020, 42(8): 760–774.
[56] Malik S, Roeder RG. The metazoan mediator co-activator complex as an integrative hub for transcriptional regulation., 2010, 11(11): 761–772.
[57] Bolt CC, Duboule D. The regulatory landscapes of developmental genes., 2020, 147(3): dev171736.
[58] Neijts R, Deschamps J. At the base of colineargene expression: Cis-features and trans-factors orchestrating the initial phase ofcluster activation., 2017, 428(2): 293–299.
[59] Xu DF, Ma RS, Zhang JH, Liu ZJ, Wu B, Peng JH, Zhai YN, Gong QG, Shi YY, Wu JH, Wu Q, Zhang ZY, Ruan K. Dynamic nature of CTCF tandem 11 zinc fingers in multivalent recognition of DNA as revealed by NMR spectroscopy., 2018, 9(14): 4020–4028.
Serial deletions of tandem reverse CTCF sites reveal balancedregulatory landscape of enhancers
Ling Wang, Jinhuan Li, Haiyan Huang, Qiang Wu
The genome architectural protein CTCF (CCCTC-binding factor) not only mediates long-distance chromatin interactions between distal enhancers and target promoters, but also functions as an important insulator-binding factor to block improper enhancer activation of non-target promoters, and is thus of great significance to transcriptional regulation of developmental genes.The(Homeobox) gene family plays an important role in the development of the brain, bones, and limbs. The spatiotemporal colinear expression of thecluster along the proximal-distal axis of limbs is regulated by two clusters of enhancers known as super-enhancers located in the flanking regulatory regions. We focused on thecluster to explore the architectural role of CTCF in transcriptional regulation of developmental genes. Thecluster contains 9 paralogous genes intermixed with a series of CBS (CTCF-binding site) elements. Using the CRISPR DNA-fragment editing system, we generated a series of single-cell HEK293T clones with deletion of increasing numbers of reverse CBS elements. RNA-seq experiments revealed decreased levels ofgene expression. In addition, chromosome conformation capture experiments revealed increased long-distance chromatin interactions betweenand the upstream enhancer cluster and corresponding decreased interactions betweenand the downstream enhancer cluster. Thus, tandem reverse CTCF sites function as insulators to maintainregulatory balance between the upstream and downstream enhancer clusters. This study has interesting implications on the precise gene expression control of thefamily during animal development.
gene cluster; CTCF site; balanced regulatory mechanism; enhancer; insulator
2021-04-11;
2021-05-12
国家自然科学基金项目(编号:31800636,31630039,91940303)和上海市科学技术委员会项目(编号:19JC1412500)资助[Supported by the National Natural Science Foundation of China (Nos. 31800636, 31630039, 91940303), and Science and Technology Commission of Shanghai Municipality (No. 19JC1412500)]
王玲,硕士研究生,专业方向:生物学。E-mail: wanglingmail0613@163.com
黄海燕,博士,副研究员,研究方向:药物分子遗传学。E-mail: hy_huang@sjtu.edu.cn
吴强,博士,教授,研究方向:基因表达调控及神经发育。E-mail: qwu123@gmail.com
10.16288/j.yczz.21-132
2021/6/30 14:14:38
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210630.1109.002.html
(责任编委: 李大力)

