乙型肝炎病毒相关肝细胞癌的关键基因筛选及临床意义*
曹旭,李小科,2,糟小宾,3,杨先照,2,张嘉鑫,李志国,杜宏波,2,叶永安,2**
(1.北京中医药大学东直门医院北京100700;2.北京中医药大学肝病研究所北京100700;3.北京中医药大学东直门医院中医内科学教育部/北京市重点实验室北京100700;4.北京市丰台中西医结合医院北京100072)
肝细胞癌(Hepatocellular Carcinoma,HCC,简称肝癌)是目前全球范围内公认的难治性恶性肿瘤之一。近一个世纪以来,发病率显著升高[1]。据2018年全球肿瘤统计数据显示,肝癌占癌症死亡原因的8.2%,排名第4位[2]。2019年国家癌症中心数据显示,2015年肝癌新发病人数37万,是我国第4位常见恶性肿瘤和第2位肿瘤致死病因,严重威胁着人类的生命和健康[3,4]。我国约80%肝癌由乙型肝炎病毒(Hepatitis B Virus,HBV)感染引起,致使HBV相关肝癌成为我国重大公共卫生问题之一,但其发病机制尚未完全阐明[4]。在传统的HBV相关肝癌治疗上,慢性HBV感染与肝脏恶性肿瘤被视作存在因果关系但又相对独立的两个方面,因此其治疗也被划分为针对HBV的长期积极抗病毒治疗,以及介入、靶向和手术等针对肝癌的治疗。现有的西医药治疗方案大致属于“组合”方案而非“综合”方案。中医药治疗由于其辨证论治的属性与对疾病的总体观,可发挥综合作用,在控制HBV慢性感染背景下的疾病进展,包括阻断癌前病变、弥补单纯西医治疗的局限性、延长生存期及改善生活质量等方面具有独特优势。但由于中药作用网络的高度复杂性,对于药物靶点的探索以及有效药物的筛选是当前的重要挑战。本研究利用基因芯片数据挖掘和生物信息学技术,通过对GEO数据库中包含HBV感染的肝癌组织和癌旁组织数据进行分析,分别找到两个数据集的差异表达基因(Differentially Expressed Genes,DEGs)并取重叠DEGs进行后续分析。通过多种数据分析软件及模型,对重叠DEGs进行筛选并得到HBV相关肝癌中的关键基因与潜在药物,初步揭示了关键基因之间表达相关性及调控机制。综上所述,本研究为临床HBV相关肝癌的中、西医治疗和生存预后判断提供了新的靶点与思路。
1 材料与方法
1.1 材料
美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)基 因 表 达 数 据 库(Gene Expression Omnibus Database,GEO)[5](https://www.ncbi.nlm.nih.gov/gds/)中HBV相关HCC数据集,包括GSE121248[6]与GSE49713数据集[7]。GSE121248数据集(Platforms:GPL570)有107个样本(37个非肝癌组织,70个肝癌组织),GSE49713数据集(Platforms:GPL11269)有10个样本(5个非肝癌组织,5个肝癌组织)。
1.2 方法
1.2.1 筛选基因表达谱芯片的差异表达基因(Differentially Expressed Genes,DEGs)
以在线分析工具GEO2R对GSE121248和GSE49713数据集进行分析,两组条件均设置为P<0.01,|logFC|≥1.5,剔除数据集中没有注释的探针及同时对应多个基因的探针。若多个探针对应同一个基因则取绝对值最大值,对获得的DEGs取交集,进行韦恩图绘制[sangerbox(http://sangerbox.com/)]。
1.2.2 DEGs功能注释(Gene Ontology,GO)和通路富集 分 析(Kyoto Encyclopedia of Gene and Genome,KEGG)
以DAVID 6.8数据库[8-9](https://david.ncifcrf.gov/summary.jsp)对DEGs进行GO及KEGG分析,P<0.05为有统计学意义。GO分析包含生物学过程(Biological Process,BP)、分 子 功 能(Molecular Function,MF)和细胞组分(Cellular Component,CC)。气泡图绘制于R包ggplot2(https://cran.r-project.org/web/packages/ggplot2/)。
1.2.3 构建蛋白互作网络、筛选hub基因
通过String 11.0数据库[10](String-db.org)对DEGs进行蛋白互作(Protein-Protein Interaction,PPI)网络构建,设置medium confidence>0.4的蛋白互作关系导入Cytoscape 3.8.0软件[11],以MCODE插件筛选hub基因,设置条件degree cutoff:2,cluster finding:haircut,node score cutoff:0.2,k-core:2。
1.2.4 获取并验证关键基因
通过Kaplan-Meier Plotter数据库[12](kmplot.com/analysis/)对筛选得到的hub基因进行再筛选,在111位HBV感染的肝癌患者数据里进行验证,得到总体生存(Overall Survival,OS)有意义的关键基因,设置条件为liver cancer;auto select best cutoff;survival:OS;follow up threshold:all;risk factors:alcohol consumption:none;hepatitis virus:yes,P<0.05为有统计学意义。以肿瘤基因组图谱(The Cancer Genome Atlas,TCGA)的分析网站UALCAN数据库[13](http://ualcan.path.uab.edu/)对关键基因在421位HCC患者数据里进行验证,设 置 条 件 为TCGA analysis;gene symbol;liver hepatocellular carcinoma;explore;expression。
1.2.5 关键基因的分析
通过cBioPortal数据库[14](www.cbioportal.org)对关键基因进行基因突变、表达相关性及生存预后分析。关键基因表达相关性通过String 11.0数据库进行可视化,设置条件为liver;liver hepatocellular carcinoma(TCGA,Firehose legacy,442 samples);oncoprint;mutual exclusivity;survival。
1.2.6 筛选关键基因的潜在药物
通过比较毒物遗传学数据库(comparative toxicogenomics database,CTD)[15](http://ctdbase.org/)筛选对关键基因具有潜在作用的化合物,设置条件为search;chemical-gene Interaction query;decrease;gene equals protein,选择能够降低关键基因蛋白活性或表达量的潜在化合物。利用Cytoscape 3.8.0软件绘制靶基因与潜在化合物的关系图,以Network Analyzer插件分析网络相关属性,得到关键基因和化合物。
2 结果
2.1 分析流程和GSE121248与GSE49713数据集中DEGs分析
本研究中HBV相关肝癌关键基因及化合物的相关分析流程,如图1(A)所示。首先,通过GEO2R工具对GEO数据库GSE121248与GSE49713数据集中DEGs进行分析,其中GSE121248数据集中得到240个DEGs,GSE49713数据集中得到1285个DEGs,同时两个数据集取交集,共得到93个重叠DEGs,并绘制韦恩图,如图1(B)所示。

图1 分析流程和GSE121248与GSE49713数据集中DEGs分析
2.2 重叠DEGs的GO与KEGG通路富集分析
通过DAVID 6.8数据库对93个重叠DEGs进行GO和KEGG通路富集,分析结果如图2(A-D)所示,具体见附表1。其中,GO富集BP结果显示重叠DEGs主要富集在氧化还原、蛋白质水解和有丝分裂核分裂等29个过程,MF重叠结果与血红素结合、铁离子结合和染色质结合等11个功能相关,CC重叠结果显示主要集中在细胞外小体、细胞外区域和细胞外隙等8个部分;KEGG通路富集表明重叠基因主要集中在代谢途径,丙氨酸、天冬氨酸和谷氨酸代谢和色氨酸代谢3条通路。以上结果表明筛选得到的相关基因与HBV肝癌的发生发展密切相关。

表1 重叠DEGs的GO功能注释和KEGG通路富集分析

图2 重叠DEGs的GO与KEGG通路富集分析
2.3 重叠DEGs的PPI网络构建和hub基因筛选
通过String 11.0数据库对93个重叠DEGs进行PPI网络可视化构建,观察到密切交互区域,如图3(A)所示。其中密切交互区域中存在13个hub基因网状连接,如图3(B)所示,分别为:ANLN、CDK1、CDKN3、EZH2、FAM83D、KIF20A、PTTG1、TOP2A、UBE2T、ZWINT、NEK2、NUSAP1和PRC1。

图3 重叠DEGs的PPI网络构建和hub基因筛选
2.4 HBV相关肝癌关键基因的确定和验证
基于Hub基因筛选结果,进一步分析这些基因与HBV相关肝癌患者总体生存的相关性。结果表明,表达值对HBV相关肝癌患者预后预测有意义的基因分别为ANLN、CDK1、CDKN3、EZH2、FAM83D、KIF20A、PTTG1、TOP2A、UBE2T和ZWINT,如图4(A-J)所示。对预后有意义的关键基因之间的相关性进行分析,结果表明10个关键基因之间存在一定表达相关性,如图4(K)所示,表达相关性存在统计学意义(P<0.05)的组别如附表2所示。进一步通过UALCAN数据库对这10个关键基因进行验证,发现相比于非肝癌组织,这些关键基因在肝癌组织中均存在高表达(P<0.001),如图5(A-J)所示,并与肿瘤分期有一定的相关性,对患者预后不利,如图6(A-K)所示。以上结果提示10个关键基因在HBV相关肝癌中发挥重要作用。

图5 HBV相关肝癌关键基因的确定和验证
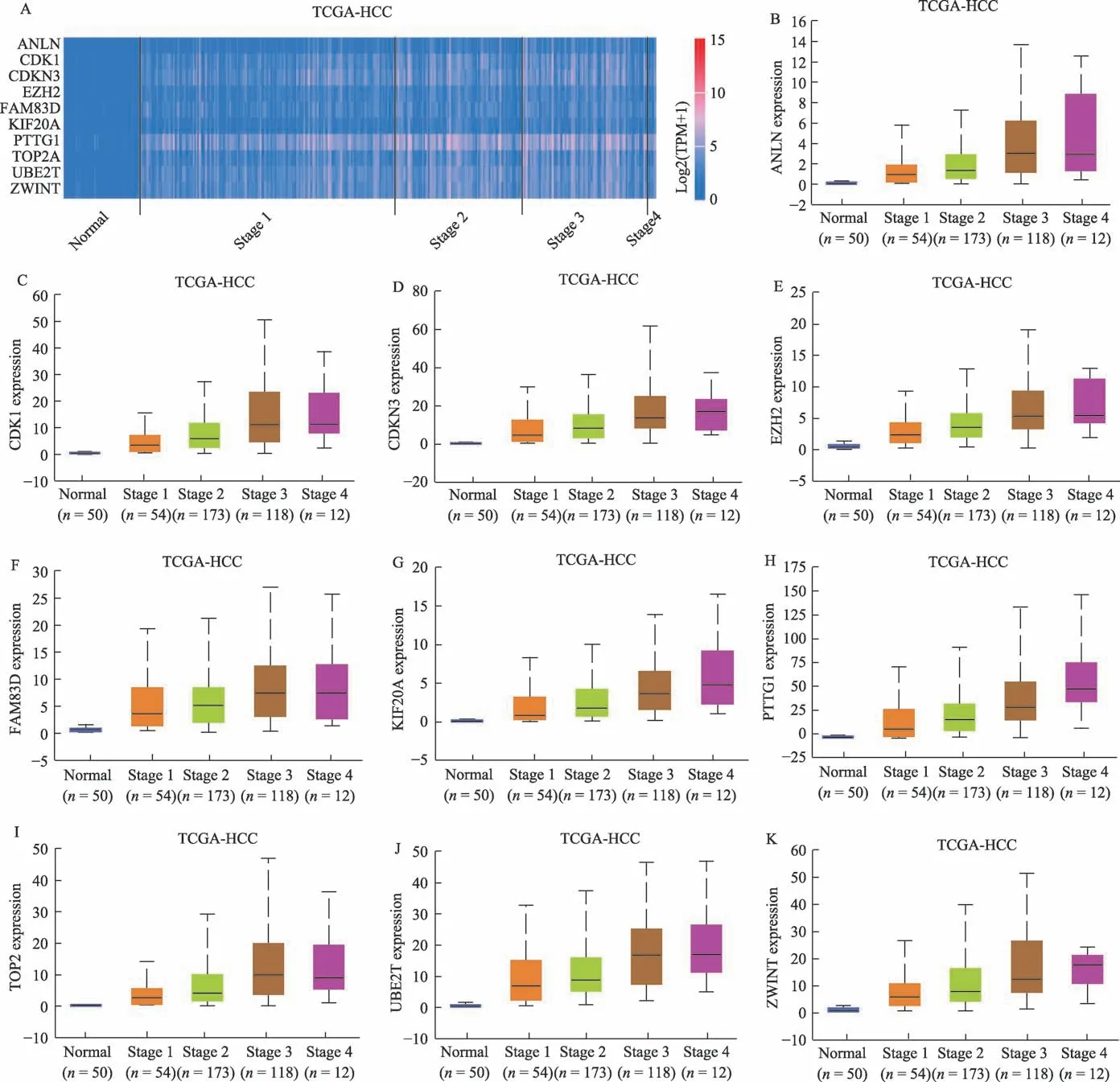
图6 HBV相关肝癌关键基因的确定和验证
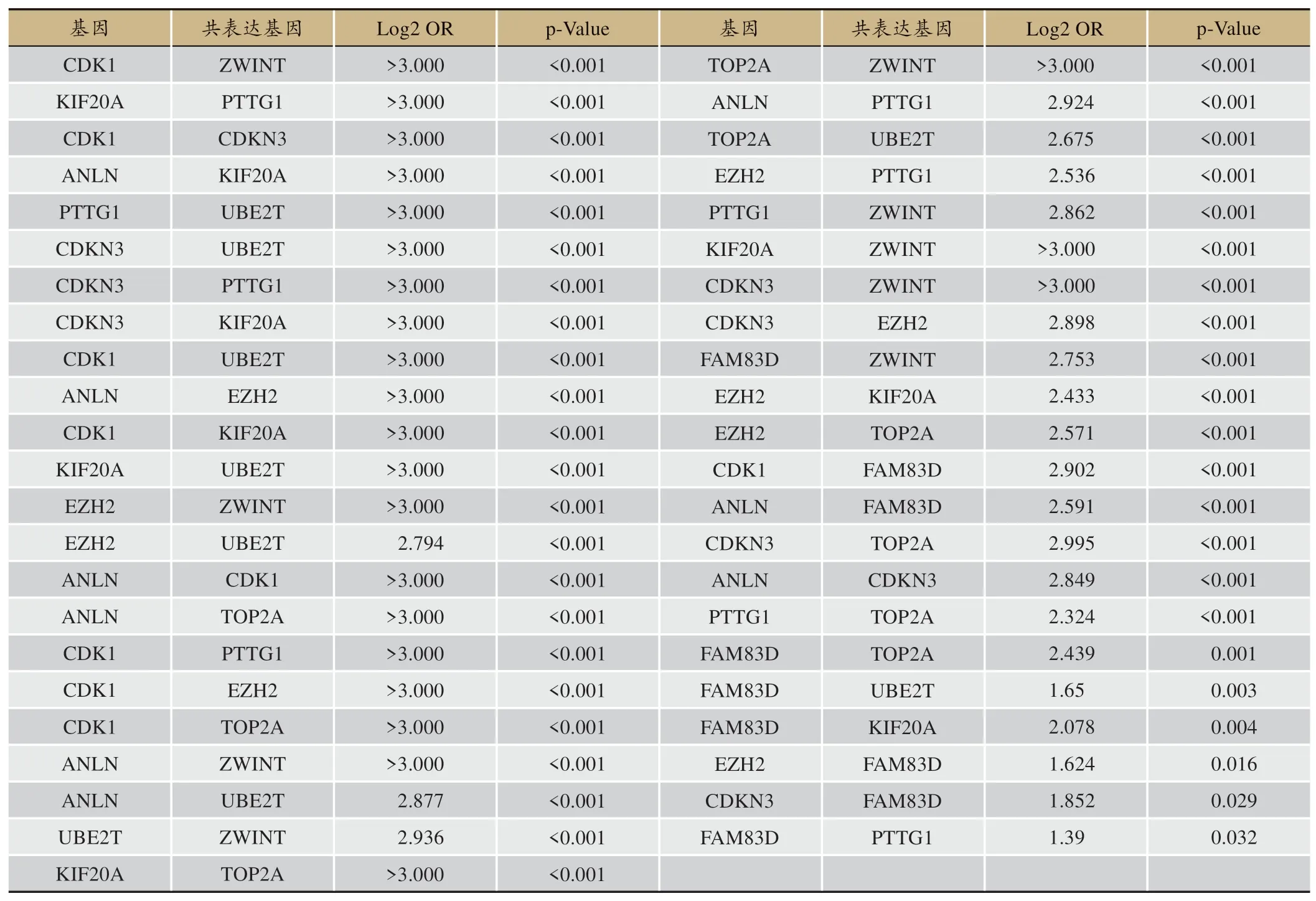
表2 关键基因之间的表达相关性

图4 关键基因的预后和表达相关性分析
2.5 关键基因在肝癌中的突变及预后分析
通过cBioPortal数据库对关键基因进行基因突变分析,结果表明,在肝癌组织中这10个关键基因均有一定的突变发生率,如图7(A)所示。将10个基因均无突变样本归为一组(unaltered group),将基因突变样本归为一组(altered group)并分析对肝癌患者预后的影响。结果表明,基因突变组肝癌患者的总体生存率和无病生存(disease free survival,DFS)率相较于无突变组更低,且存在统计学意义(P<0.001),如图7(BC)所示。以上结果提示,筛选得到的关键基因表达及预后相关性与基因突变有关,是一项重要的调控机制。

图7 关键基因在肝癌中的突变及预后分析
2.6 HBV相关肝癌潜在治疗药物筛选
根据上述结果,关键基因高表达的患者预后较差,所以减少关键基因表达或活性的化合物可能存在治疗作用。通过CTD数据库得到减少关键基因表达量或活性的潜在作用化合物,见附表3。以Cytoscape 3.8.0软件绘制关键基因和潜在化合物的互作图,Analyse Network得到互作图的网络信息,根据网络连接密度来设置关键基因图标的大小,图标的大小与Degree值成正比。如图8所示,排名前三位的基因是CDK1、TOP2A、EZH2;得到作用于2个以上关键基因的化合物是三氧化二砷、白藜芦醇、红豆碱、玫瑰树碱、多柔比星、氯化镉、甲基苯丙胺、帕博西尼、对苯二酚、吡唑嵌蒽酮、雷公藤甲素、伏立诺他、乙酰半胱氨酸、重铬酸钾、二乙基己基邻苯二甲酸和亚砷酸钠。以上结果表明通过结合数据库分析,能够筛选得到多种关键基因、靶点及相应中医药,具体的作用调控、作用机制需要进一步研究。

图8 HBV相关肝癌潜在治疗药物筛选

表3 关键基因和相关化合物
3 讨论
全球每年因HBV感染相关疾病死亡人数将近88.7万,其中HBV相关肝癌约占38%。在我国,HBV导致的HCC比例高达84%[16]。目前HBV相关肝癌发生发展的分子机制研究方兴未艾,但仍缺乏针对性治疗靶点、治疗药物及预后评估的有效标记物。本研究通过临床样本数据挖掘与生物信息学分析相结合的方式,筛选得到在HBV相关肝癌与癌旁中存在表达差异的93个基因,在HBV相关肝癌发生发展中发挥重要作用的10个关键基因,并完成了针对关键基因的潜在治疗化合物的预测,为HBV相关肝癌的治疗及预后评估提供了新的靶点与思路。
在发病机制方面,GO富集分析BP结果提示筛选得到的93个DEGs参与了氧化还原、蛋白质水解和有丝分裂核分裂等多个过程,均与肝癌的发生、发展和转移相关。近年研究发现肿瘤微环境多处于缺氧状态,Warburg效应提示糖酵解是肿瘤细胞的主要能量来源[17]。缺氧诱导因子在缺氧条件下转录活性增强,导致参与细胞功能的相关基因表达增强,从而促进体内细胞代谢、增殖和迁移[18]。CC结果显示筛选得到的93个DEGs集中在细胞外小体、细胞外区域和细胞外隙等,提示HBV相关肝癌微环境中细胞间交互可能存在重要意义。KEGG通路富集结果表明筛选得到的93个DEGs主要集中在代谢途径,这与GO富集分析MF结果具有一致性。这些差异基因在HBV相关肝癌中发挥的作用需要进一步验证。
在治疗靶点及预后标志物方面,通过PPI网络互作分析得到了13个hub基因。利用TCGA-HCC数据对这些hub基因表达进行肝癌生存分析发现,ANLN、CDK1、CDKN3、EZH2、FAM83D、KIF20A、PTTG1、TOP2A、UBE2T和ZWINT这10个基因对HBV相关肝癌患者的总体生存均有影响,在肝癌组织中基因高表达组患者的预后更差(P<0.05)。相比于非肝癌组织,这10个基因在肝癌组织中的表达均显著上调(P<0.001)且与肝癌分期有一定相关性,这与既往文献报道结果相一致[19-28]。通过进一步对关键基因的表达和突变情况分析发现,这10个基因在肝癌组织中存在一定的表达相关性,且均有突变发生。针对突变样本与非突变样本的预后分析结果表明,关键基因突变不利于肝癌患者的预后,而突变往往带来基因表达与蛋白活性的改变,提示靶向这些关键基因表达及蛋白活性药物研究的重要性。
在潜在药物筛选方面,以关键基因与潜在化合物的网络互作图得到连接度最高的前3个基因,包括CDK1,TOP2A,EZH2。筛选得到作用于2个以上关键基因的目标化合物,如三氧化二砷、白藜芦醇、红豆碱、雷公藤甲素、玫瑰树碱等。基于肿瘤药物敏感性基因组学数据库(Genomics of Drug Sensitibity in Cancer,GDSC)[29]的分析结果提示EZH2基因突变会显著增加Afuresertib、Venetoclax药物治疗肿瘤的敏感性。研究表明,EZH2表达与HBV病毒的转录和复制密切相关,并且在HBV相关肝癌中发挥较为重要的作用[30]。因此,可以降低EZH2表达量的穿心莲内酯可能成为针对HBV相关肝癌的潜在治疗药物。除此之外,针对CDK1基因为靶点的黄芩提取物、水飞蓟宾、槲皮素、姜黄素、芹菜素、木犀草素、金雀异黄素及葫芦素I等药物,以及针对TOP2A基因为靶点的隐丹参酮、熊果酸、金丝桃素、乳香酸、薄荷醇、飞燕草色素、齐墩果酸、花青素B2及杨梅黄酮均为HBV相关肝癌潜在治疗药物。这些药物与HBV及HBV相关肝癌的相互作用及机制需要进一步研究。

续表

续表

续表

续表

续表

续表

续表
目前我国仍存在很高比例的HBV感染肝癌患者,这也是我国肝癌防治策略不同于西方国家的重要考量。基于这一重要而普遍的始动因素,我们在对肝癌进行新药筛选的过程中,应将肝癌相关基因调控及分子通路的分析融入到HBV感染的背景中加以分析。在中医治疗中强调“整体观念”“辨证论治”,常采用辨病与辨证相结合的策略,对HBV相关肝癌的治疗也确非“抗病毒”与“抗肿瘤”的简单叠加,从药物的选择上亦是如此。基于本研究结果,发现其内涵可能与HBV相关肝癌的特殊调控机制相关,是值得进一步探索、发掘的方向。从服务新药开发的角度来看,上述研究对优化处方,明确临床有效处方的起效机制、相关靶点,聚焦关键药物,以及建立可供实验进一步证实的单体和复方均具有一定指导意义;另一方面对HBV相关肝癌及肝癌前病变过程中,药物治疗的疗效评价和结局预测提供参考。因此,本研究利用生物信息学方法筛选出HBV相关肝癌的关键基因及潜在的中药药物,为后续HBV相关肝癌发病机制及治疗靶点研究奠定了重要的基础。

