基于隐马尔可夫模型的全外显子测序拷贝数变异检测算法研究
刘 妮 刘 晗 赵阿曼 徐凡丁 刘文宇 段君博*
1(西安交通大学生命科学与技术学院,教育部生物医学与信息工程重点实验室,西安 710049)
2(苏州大学附属儿童医院检验科,江苏 苏州 215003)
引言
拷贝数变异(copy number variation,CNV)属于基因组结构变异,一般指长度大于1 kb的基因组大片段的拷贝数增加或减少[1]。有研究表明,CNV至少占到了人体基因组的12%[2],说明 CNV 不仅是基因组多态性的重要来源之一,而且相比单核苷酸多态性等基因变异类型,对人类健康的影响更为显著。大量研究发现,CNV与癌症之间存在相关性[3-5]。因此,CNV的准确识别,对于癌症等疾病的预防和治疗具有重要的现实意义。
高通量测序技术能一次并行对几十万到几百万条DNA分子进行序列测定,大大提高了测序的效率。目前CNV检测算法大多都是基于高通量测序技术[6],该技术又可以被划分为全外显子组测序技术与全基因组测序技术。全外显子组测序技术相较于全基因组测序技术,价格更低、耗时更短,且对基因疾病的研究更为有效[7-8],所以在临床诊断和学术研究中使用得更为广泛。
隐马尔可夫模型(hidden Markov model,HMM)是比较经典的机器学习模型,现已应用于语音识别、行为识别以及生物信息等领域[9]。HMM在拷贝数变异检测中也得到广泛应用,与其相关的算法众多但各有优劣,这使得在进行拷贝数变异检测时算法的选择成为一个重要问题。本研究选取5个具有代表性的基于HMM的CNV检测算法,对其性能进行评估,并与实际情况进行结合,最终得出了不同应用场景的算法选取指南。
1 方法
首先,构建仿真数据集与真实数据集;然后,选择一些具有代表性的基于HMM的CNV检测算法,从真阳性率(TPR)、假发现率(FDR)和计算性能等方面,对所选取的 CNV 检测算法进行性能评估;最后,将被选择的CNV 检测算法的性能评估结果与实际应用场景结合,获得可供应用的临床使用指南。
1.1 数据集
本研究的数据集分为仿真的和真实的全外显子数据集,以此来评估CNV检测算法的性能。
对于仿真数据集的构建,首先选择hg19版本的人体10号染色体作为仿真数据的参考基因序列,然后使用SimulateCNVs软件进行拷贝数变异仿真[10]。在外显子区域模拟了不同的覆盖值(X2、X20、X40、X70和X100),每种覆盖值都包含10个参考样本和30个待测样本,每个待测样本随机产生拷贝数变异,并且每个样本中重复和缺失拷贝数的变异数量相同。对于覆盖度为X100的样本,额外生成两组拷贝数变异密度不同的数据集。
对于真实数据集,从NCBI数据库中的SRP007198项目,下载来自5个个体的WES数据SRR292250、SRR303332、SRR303335、SRR303338和SRR303340。在Krumm等的研究中,这5个样本共产生了32个拷贝数变异[11],将其作为金标准来评估CNV检测工具的准确性。
1.2 算法选择
为了帮助研究人员根据需求采用合适的基于HMM的CNV检测方法,笔者选择了具有代表性的算法。在此过程中,有两个选择算法的标准。首先是基于读深度方法来检测CNV,其次是基于HMM方法。此外,还需考虑算法文章的引用量以及源码是否公开。基于这些条件,确定了CNV检测候选算法[12-15],但部分算法由于无法下载或版本过旧,故予以删除,如M-HMM、EXCAVATOR、CoNVex和CONDEX等。最后,选择了5种算法进行检测,分别为XHMM[16]、ADTex[17]、CANOES[18]、ExomeCopy[19]和Exome Depth[20],基本信息如表1所示。

表1 研究选取算法的相关信息Tab.1 Selected representative CNV calling methods
就算法实现而言,XHMM主要采用主成分分析法对整个样本组进行分析,根据它们的读深信号变化来判断样本是否存在CNV;ADTex采用覆盖深度对比法,一个样本作为正常样本,另一个则作为待测样本,将两个样本的读深信号化为一个个窗,对窗与窗的覆盖深度进行对比,从而找出待测样本的CNV;CANOES与XHMM相似,也是采用了主成分分析法;ExomeCopy采用基于GC、覆盖度和窗负二项回归模型,ExomeDepth则基于GC矫正的β-二项分布模型,这两者的原理相似,都是经过GC矫正,再将读深信号转化为Grange信号进行分析。
1.3 评价指标
为了更全面地评价CNV检测工具,选择真阳性率(TPR)、假发现率(FDR)、计算性能等作为评价指标。真阳性率TPR为正确检测出的CNV的数量除以CNV总数量,是正确识别真阳性结果比例的统计量,FDR为误检测到的CNV的数量除以检测出的CNV总数量,是正确识别假阳性结果比例的统计量。
此外,为了更全面地评估这些算法,计算性能也是一个重要的指标,主要包括时间复杂度和空间复杂度。以算法运行时间来表征时间复杂度,以中央处理器和内存的占用情况(即计算机资源使用量)来表征空间复杂度。时间复杂度和空间复杂度越低,则说明算法优化得越好。
2 结果
根据评价指标,对5种CNV检测工具的性能进行评价。
2.1 仿真参数
全外显子组测序数据的覆盖深度和CNV的密度可能会对CNV检测结果有影响,故将二者作为仿真系数,评测其对检测性能的影响。
2.1.1覆盖深度
覆盖深度为测序得到的碱基总量与基因组大小的比值,如X20表示覆盖深度为20。为了评估覆盖深度对这些算法的CNV检测性能的影响,在外显子区域模拟了不同的覆盖值,包含X2、X20、X40、X70和X100共5种情况,每种情况下生成的CNV的缺失和重复数均相同。使用5种选定的算法,进行CNV检测。检测结果的TPR和FDR如图1所示,其中(a)和(b)分别显示覆盖深度对5种检测算法TPR和FDR的影响。根据图1(a)可以得到以下结论:一是随着数据覆盖深度的增加,这5种检测算法的TPR先迅速增加,后保持稳定;二是在低覆盖深度下,ADTex和XHMM无法获得CNV检测结果;三是ADTex和XHMM的CNV检测结果的TPR明显逊于ExomeCopy、ExomeDepth和CAONES。根据图1(b)可以得到以下结论:一是检测算法的FDR随着覆盖深度的增加而减少;二是XHMM的FDR最高,其他4种算法的FDR相对接近。从上述结果可以看出,在实际应用中,100X的覆盖深度对拷贝数变异检测来说已经足够。

图1 覆盖深度对5种检测算法性能的影响;(a)对TPR的影响;(b)对FDR的影响Fig.1 The changes of tools′ performances with respect to the coverage. (a) The changes of these tools′ TPR; (b) The changes of these tools′ FDR
2.1.2CNV密度
为了评估外显子密度对这些算法的CNV检测性能的影响,基于X100覆盖深度模拟了一系列CNV,每1000、500和330个外显子组平均会产生一个CNV,其中重复和缺失CNV的数量相同。 然后,使用选定的算法从这些数据中检测CNV, 计算出的TPR结果如图2所示。可以看出,随着拷贝数变异密度的增加,XHMM、CANOES、ExomeDepth和ExomeCopy的TPR降低。ExomeDepth在高密度下有明显的下降,而其他3种算法则是略有下降。 相反,ADTex的TPR随外显子组密度的增加而增加,表明其在高外显子组密度下具有检测CNV的优势。

图2 CNV密度对TPR的影响Fig.2 The changes of TPR with respect to the CNV density
2.2 计算性能
为了全面地评估这几种CNV检测算法,在对检测算法进行了统计学评估之后,将计算性能也作为算法的评价标准之一。以运行时间和计算机资源使用量来表征计算性能,5种算法的结果如图3所示。
从图3(a)可以看出,在相同条件下XHMM所花费时间明显高于其他4种算法。ADTex和ExomeDepth所花费的时间大致相同,CANOES所花费时间高于前两者,ExomeCopy所花费时间是最少的。
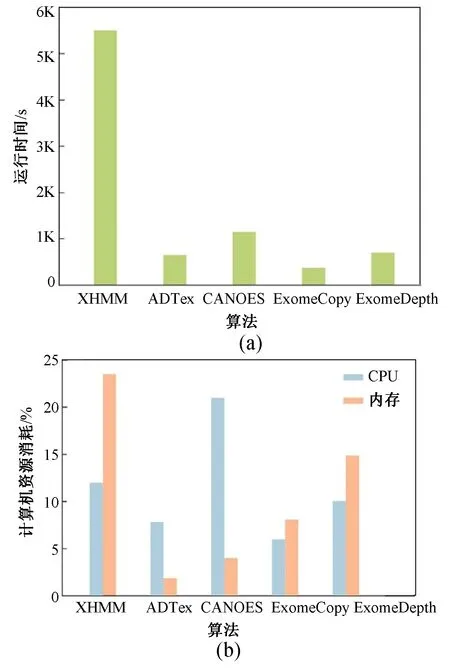
图3 不同算法的计算性能对比。(a)5种工具的运行时间;(b)5种工具计算资源消耗的对比Fig.3 Computational performance comparison of the five tools. (a) Running times of the five tools;(b) Computer resources consumption of the five tools
从图3(b)可以看出,在相同条件下XHMM占用了最高的电脑资源,CPU和内存使用率最高,说明其算法优化尚待提高。ADTex性能最佳、CPU和内存使用率最低,CAONES具有较高的CPU使用率和非常低的内存使用率,ExomeCopy的结果仅次于ADTex,ExomeDepth的CPU和内存使用率都很高,研究人员可以根据自己的计算机配置选择合适的算法。
2.3 数据比较
对真实数据进行拷贝数变异检测,以Krumm等的研究结果[11]作为金标准,分别用5种工具进行检测,并且比较测得拷贝数变异重复和缺失的能力,结果如图4所示。从图4(a)中可以看出,ADTex、ExomeCopy和ExomeDepth的准确率较高,其中ADTex为68.75%,与模拟数据的结果相似。ExomeCopy和ExomeDepth的精度分别为75%和81.25%,不如模拟数据的结果。其中,ExomeDepth的真实数据结果与高密度外显子组的模拟数据相似。CAONES的精度为56.25%,明显低于模拟数据;XHMM的准确率最差为25%,结果也与模拟的数据相似。从图4(b)可以看到,不同算法对缺失型和重复型的拷贝数变异检测的能力不同。CAONES和ExomeCopy对于缺失型和重复型拷贝数变异的检测水平基本相同。 ExomeDepth可以检测更多重复型拷贝数变异,同时对于缺失型拷贝数变异也有着很高的检测水平;ADTex具有最佳的缺失型拷贝数变异检测水平,几乎可以检测到所有缺失型拷贝数变异,但对于重复型拷贝数变异的检测水平较差;XHMM的性能最差,几乎没有检测到缺失型拷贝数变异,而且重复型拷贝数变异检测水平也很低。

图4 真实数据的检测结果。(a)拷贝数变异检测数量;(b)重复和缺失拷贝数变异检测数量Fig.4 Detection results of real data. (a) Number of detected CNV. (b) Numbers of duplication and deletion CNV
3 讨论
本研究对于5种算法在各种情况下的检测性能进行了系统比较,作为发布时间最早的算法,XHMM在TPR、FDR以及真实数据的表现上不是很理想。ADTex在TPR上的表现较差,并且每次只能进行一对参考样本和测试样本的对比,这对参考样本的质量提出了要求,但如果研究人员的样本数量受限,则可以选择ADTex算法。
CANOES开发年代较早,也没有进行后续更新,但在拷贝数密度较高时的检测效果比较好。ExomeCopy和ExomeDepth至今一直都有开发者进行更新与维护,因此能很好地适应测序技术的发展。ExomeCopy在检测拷贝数变异时较为保守,在模拟数据中往往将拷贝数变异的长度大为压缩;ExomeDepth在各种情况下表现都很突出,因此在没有特殊需求时是研究人员的首选。
综上所述,笔者根据不同的应用场景选择合适的CNV检测工具,形成了推荐指南,如表2所示。

表2 不同场景下CNV检测算法的推荐指南Tab.2 The recommended tool for different requirements
4 结论
在本研究中,首先选择了5种基于HMM的CNV检测工具:ExomeDepth、ExomeCopy、XHMM、ADTex和CANOES;然后,对所选的5种CNV检测工具的性能进行了综合评价和比较;最后,通过对实验结果的分析,根据实际的应用需求,分别推荐了合适的检测工具。对多种CNV检测进行比较,有助于CNV检测算法的临床应用,在一定程度上保证了CNV检测结果的准确率与可靠性。但在一些方面仍有待改进,比如本研究只选取了5种基于HMM的CNV检测算法进行比较,真实数据样本量较少,等等。在未来的工作中,可以在这些方面进一步完善,进行更多样化的比较。

