高寒湿地土壤微生物区系组成对氮添加的响应
徐润宏, 谭梅, 刘泽华, 朱锦福
高寒湿地土壤微生物区系组成对氮添加的响应
徐润宏, 谭梅, 刘泽华, 朱锦福*
青海师范大学生命科学学院, 西宁 810008
土壤微生物是土壤养分循环的关键驱动者, 对土壤环境变化感应明显, 氮是陆地生态系统的限制元素之一, 其改变可能会给生态系统物种多样性造成一定影响。为了解高寒湿地土壤微生物组成对氮添加的响应, 以青海湖流域高寒湿地为研究对象, 通过(0 g·m-2、0.5 g·m-2、1 g·m-2、1.5 g·m-2、2 g·m-2、2.5 g·m-2、5 g·m-2、10 g·m-2)8个水平的模拟氮添加试验, 采用高通量测序技术, 分析土壤微生物区系组成对氮添加的响应。结果表明: 氮添加使细菌、真菌和古菌的Chao1指数均下降, 细菌和古菌的Shannon指数显著增加(<0.05), 真菌Shannon指数显著下降(<0.05); 氮沉降改变微生物群落结构, 变形菌门()、放线菌门()、广古菌门()和泉古菌门()的相对丰度均减少, 而酸杆菌门()和子囊菌门()的相对丰度增加; 真菌群落对于氮添加的响应变化较低, 对环境的适应性可能要优于细菌和古菌群落。由此可见, 氮添加对不同土壤微生物的影响存在一定差异, 氮沉降是不断积累的过程, 长时间的氮沉降势必会引起土壤微生物群落的改变, 影响生态系统的稳定。
高寒湿地; 氮添加; 土壤微生物; 群落结构
0 前言
目前人类活动对生态系统已产生重要影响, 其中氮沉降的急剧上升引起了人们的普遍关注[1]。近几十年, 由于经济迅速发展和人口增长, 工农业规模的不断扩大及矿物燃料和农业化肥的大量使用, 我国已成为全球氮沉降最严重的地区之一[2-4]。目前我国的氮沉降区域已逐渐从东南沿海发达地区向西北乃至全国迅速扩展[5], 给我国的生态环境造成了非常严重的影响[6]。氮沉降不仅会改变土壤性质[7], 引起土壤酸化、pH下降, 还会改变植物及微生物的多样性[8]和群落组成[9]。
土壤微生物是陆地生态系统最重要也是最活跃的组分之一[10-11], 在生态系统的物质转化、能量流动中起关键作用[12], 是生态系统不断进化演替的主要驱动力之一[13]。土壤微生物对环境变化较敏感[14],可对环境变化做出较强的响应, 是影响土壤健康评价的关键因素之一[15]。目前针对氮沉降对生态系统的研究已有大量报道, 主要集中于林地和草原生态系统[16]。大多研究表明, 氮沉降可在一定范围内促进微生物生长[17-18], 如, 杨建琴在东北松嫩草原羊草草甸的试验发现, 施氮处理后, 土壤微生物量碳、氮与对照相比均提高了20%以上[19]。但也有一部分研究表明氮沉降使微生物生长受到抑制[20-21], 被认为是氮添加使土壤有效氮含量增加, 促进了植物对氮的需求, 进而使微生物获取养分减少[20]。Deforest等在密歇根阔叶林的研究发现, 施氮处理使土壤微生物量减少了68%[22]。Demoling等在针叶林中研究发现, 施氮处理下土壤微生物量平均降低了40%, 并且真菌生物量降低更明显[23](41)氮沉降对土壤微生物的影响机制复杂, 国际上关于氮沉降的研究主要集中于欧洲和北美温带林地[24], 结果存在一定的区域和局限性, 不同生态系统土壤微生物对氮添加的响应机制还应继续研究探讨。
湿地生态系统的单位碳存储远远高于其他生态系统[25], 在全球碳循环中扮演着重要角色[26]。青海湖位于青藏高原北部, 是维系青藏高原东北部生态安全的重要水体[27], 同时也是全球变化的敏感区和生态系统典型脆弱区[28], 具有特殊的生物地理区位和生态重要性。在维护我国西北部及青藏高原生态平衡方面起着不可忽视的作用[29]。基于此, 本文以青海湖流域小泊湖高寒湿地氮添加土壤为研究对象, 通过人工氮添加以模拟大气氮沉降, 采取高通量测序的方法, 研究土壤微生物对氮沉降的响应规律。本文拟解决以下问题: (1)不同土壤微生物多样性在氮添加条件下如何变化; (2)不同土壤微生物优势菌门在氮添加条件下的变化趋势; (3)不同土壤微生物对氮添加的响应敏感度有何区别。
1 材料与方法
1.1 研究区概况
小泊湖湿地(100˚46'E, 37˚15'N), 位于青海湖东岸青海湖湖东种羊场西北约6 km处, 是青海湖水位下降后遗留下来的沼泽草甸湿地, 具有典型的高原大陆性气候, 干燥少雨, 雨热同季, 降水多集中在5—9月份。土壤类型为草甸土, 优势植物主要有华扁穗草 () 、禾叶嵩草 ()和高原嵩草() 等[30]。
1.2 实验设计及样品采集
根据我国青藏高原大气氮沉降现状[31], 本实验共设置N0: 0 g·m-2, N0.5: 0.5 g·m-2, N1: 1 g·m-2, N1.5: 1.5 g·m-2, N2: 2 g·m-2, N2.5: 2.5 g·m-2, N5: 5 g·m-2, N10: 10 g·m-28个不同浓度氮添加处理。每个处理设置三个重复, 一共24个1×1 m样方, 样方间隔1 m最为缓冲带。本实验施加氮源为NH4NO3, 按不同施氮梯度, 称取相应的NH4NO3, 溶于500 mL水中, 用喷壶喷洒于相应样方中, 对照只喷洒等量的水, 从2019年6月份起至8月份止, 每月中旬取样后施药, 按上述方法共施氮3次。
于2019年8月中旬植物生长旺盛时采样, 在每块样方中按照五点取样法进行采样, 采用螺旋取土钻, 按照0—15 cm, 15—30 cm两层进行取样, 尽可能去除植物根系及凋落物, 放入无菌自封袋中, 约400 g左右。将土样迅速带回实验室, 并将新鲜土样放入冰箱中4 ℃冷藏保存, 用于土壤微生物多样性分析。
1.3 土壤微生物测定
1.3.1 NDA提取试剂盒和扩增引物
使用MN NucleoSpin 96 Soil试剂盒/PowerSoil DNA Isolation kit强力土壤DNA提取试剂盒提取土壤微生物NDA。
细菌16S rRNA (V3+V4)区域引物: 正向引物5'- ACTCCTACGGGAGGCAGCA-3'; 反向引物5'- GGACTACHVGGGTWTCTAAT-3'。真菌ITS1 区域引物: 正向引物5'-CTTGGTCATTTAGAGG AAGTAA-3'; 反向引物5'-GCTGCGTTCTTCATCG ATGC-3'。古菌16S rRNA (V3+V4)区域引物: 正向引物5'-GYGCASCAGKCGMGAAW-3'; 反向引物5'-GGACTACVSGGGTATCTAAT-3'。
1.3.2 PCR扩增
目标区域PCR(10 μL体系): 基因组DNA, 50 ng±20%; *Vn F(10 μM), 0.3 μL; *Vn R(10 μM), 0.3 μl; KOD FX Neo Buffer, 5 μl; dNTP (2 mM each), 2 μL ; KOD FX Neo, 0.2 μL; ddH2O , 补至10 μL。反应条件: 95 ℃, 5 min; 95 ℃, 30 s, 50 ℃, 30 s, 72 ℃, 40 s, 20个循环; 72 ℃, 7 min; 4 ℃, ∞。
Solexa PCR: 目的区域PCR 纯化产物, 5 μL; *MPPI-a(2 μM), 2.5 μL; *MPPI-b(2 μM), 2.5 μL; 2×Q5 HF MM, 10 μL; Total, 20 μL。反应条件: 98 ℃, 30 s; 98 ℃, 10 s, 65 ℃, 30 s, 72 ℃, 30 s, 10个循环; 72 ℃, 5 min。1.8%的琼脂糖凝胶。电压120 V, 40 min。(*根据上机安排进行选择index)
将得到的PCR产物根据电泳定量结果, 按质量比1: 1进行混样, 混样后采用OMEGA DNA纯化柱进行过柱纯化。再用1.8%的琼脂糖凝胶检验。基于 Illumina HiSeq 测序平台, 对纯化的样品进行测序分析。
1.4 数据分析
使用FLASH(version 1.2.11)对原始数据进行拼接, 再进行质量过滤, 并用UCHIME(version 8.1)软件去除嵌合体, 得到高质量的Tags序列。使用Usearch软件对Tags在97%的相似度水平下进行聚类、获得OTU, 并基于Silva(细菌)和UNITE(真菌)分类学数据库对OTU进行分类学注释。将OTU的代表序列与微生物参考数据库进行比对可得到每个OTU对应的物种分类信息, 进而在各水平统计各样品群落组成, 利用QIIME软件生成不同分类水平上的物种丰度表, 再利用R语言工具绘制成样品各分类学水平下的群落结构图(只显示丰度水平前10的物种, 其他物种合并为 Others 在图中显示, Unclassified代表未得到分类学注释的物种)。Mothur version v.1.30软件分析样品的α多样性(Alpha diversity), 再基于R语言绘制样品 β 多样性(Beta diversity)分析图。接着由SPSS 20.0对数据进行统计分析, 采用单因素方差分析(One Way ANOVA)和LSD(最小显著差异法)比较各处理水平之间的差异。
1.5 测序结果概述
细菌测序共获得3,122,368对Reads,双端Reads拼接、过滤后共产生3,008,891条Cleantags,每个样品至少产生38,869条Cleantags,平均产生62,685条Cleantags。真菌测序共获得3,594,092对Reads,双端Reads拼接、过滤后共产生3,357,584条Cleantags,每个样品至少产生22,082条Cleantags,平均产生71,438条Cleantags。古菌测序共获得3,409,956对Reads,双端Reads拼接、过滤后共产生2,951,129条Cleantags,每个样品至少产生25,462条Cleantags,平均产生61,482条Cleantags。
2 结果与分析
2.2 氮添加对土壤微生物多样性及群落结构的影响
2.2.1 土壤微生物丰富度变化
不同氮添加处理下, 三种微生物OTU数目均为: 细菌>古菌>真菌, 且差异显著(P<0.05)。0—15 cm土层中, 各处理下细菌OTU平均数在1108—1288之间, 古菌在615—932之间, 真菌在274—459之间; 15—30 cm土层中, 各处理下细菌OTU平均数在1157—1384之间, 古菌在693—915之间, 真菌在201—395之间。在0—15 cm土层中, 除N2处理下细菌OTU数有所增加, 氮处理对土壤微生物OTU数的影响大多为降低, 细菌和古菌OTU数均没有显著变化(>0.05), 但在N1、N1.5、N5和N10处理下真菌OTU数显著减少(<0.05)。15—30 cm土层中, 土壤微生物的OTU数目均下降, 对细菌和古菌OTU数影响不显著(>0.05), 在N5处理下, 真菌OTU数显著减少(<0.05)。
2.2.2 土壤微生物Alpha多样性(Alpha diversity)特征
由表1可知, 0—15 cm土层中, 细菌、真菌和古菌的Chao1指数均有所下降, 真菌达到极显著水平(<0.01), 主要表现在N5处理下, 细菌和古菌的丰富度变化差异不显著(>0.05); 细菌的Simpson指数显著下降(<0.05), 而真菌和古菌的Simpson指数在氮添加条件下变化不显著(>0.05); 三种菌的Shannon指数变化为, 细菌多样性增加并达到极显著水平(<0.01)主要表现在N2、N2.5、N10中高浓度处理下, 而真菌和古菌则呈现出先减少后增加的趋势, 差异均不显著(>0.05)。在15—30 cm土层中, 细菌、真菌和古菌的Chao1指数、Simpson指数和Shannon指数在氮添加条件下的变化均没有达到显著性水平(>0.05)。其中三种菌的Chao1指数均表现为下降趋势, 细菌和古菌的Simpson指数表现为下降趋势, 而真菌则表现为增加趋势, Shannon指数在细菌和古菌中表现为增加趋势, 而在真菌中却表现为相反的趋势。
2.2.3 土壤微生物物种群落结构差异分析
根据β多样性的主成分分析(principalcomponentanalysis, PCA)(图2), 氮添加对土壤三种微生物群落分布情况均产生影响, 细菌、真菌和古菌群落在氮处理下的分布情况规律表现不同。随着氮添加浓度的改变, 真菌群落在0—15 cm土层和15—30 cm土层中分布都较为集中, 群落结构变化不明显。古菌群落在氮添加处理下的分布较为分散, 结构差异最明显, 表明在不同氮添加处理下真菌群落组成对氮添加的响应敏感度最低, 真菌群落对小泊湖湿地环境的改变稳定性最高, 细菌次之, 古菌群落最不稳定。总体上氮添加对土壤微生物群落的影响上层土壤要大于下层土壤。
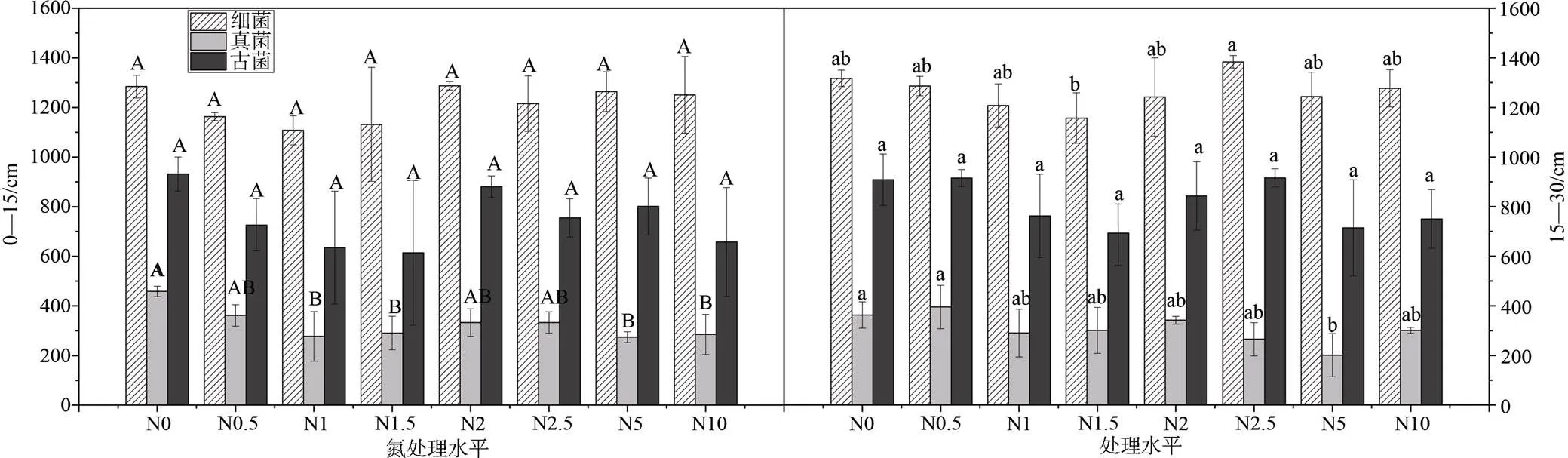
注: 图中不同大写字母表示在0—15 cm土层中的差异显著性(P<0.05), 小写字母表示在15—30 cm土层的差异显著性(P<0.05)。
Figure 1 changes of OTU number of soil microorganisms at different nitrogen treatment levels
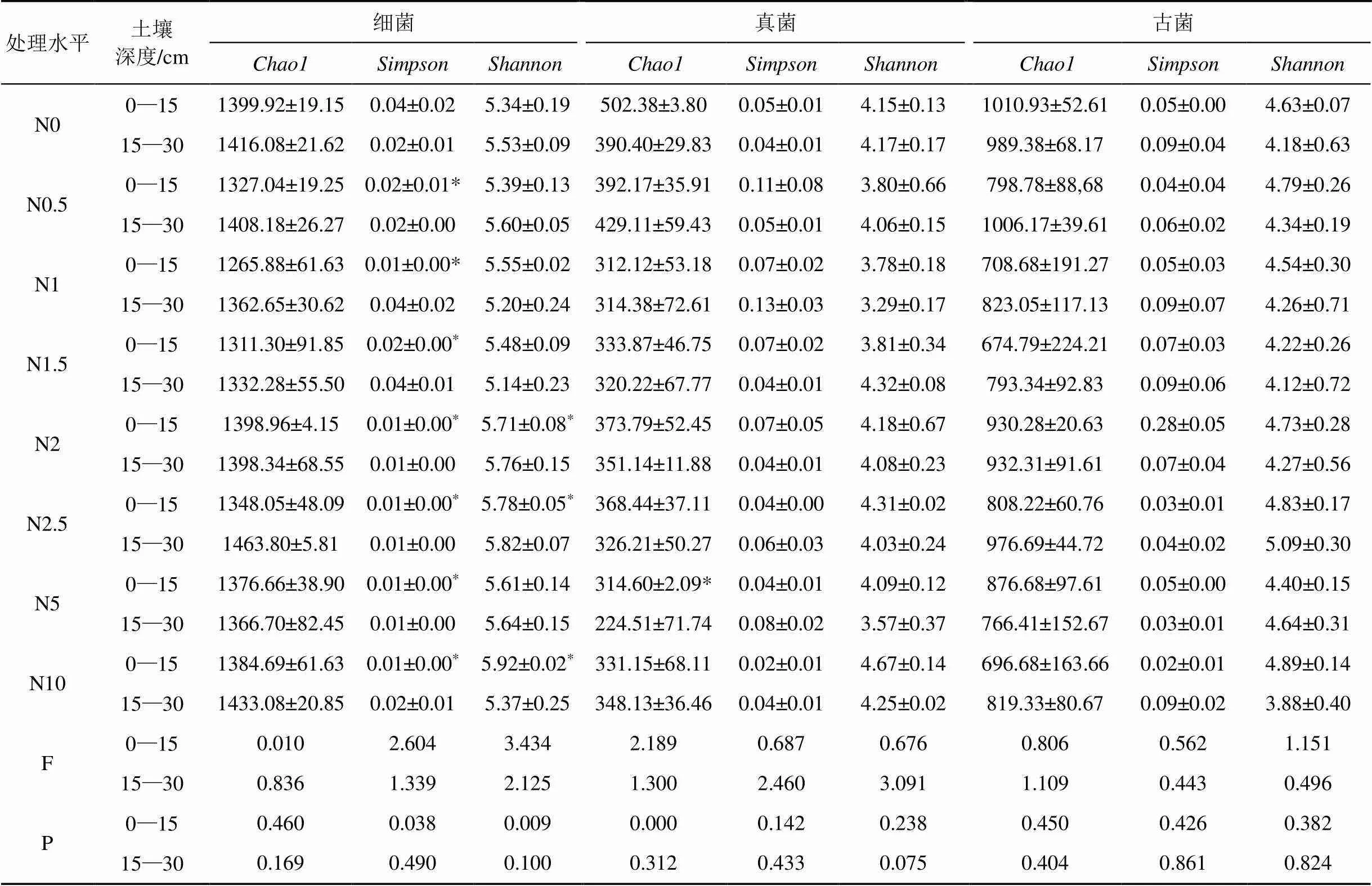
表1 细菌、真菌和古菌的多样性指数
注: *表示在0.05水平下差异显著, **表示在0.01水平下差异极显著。
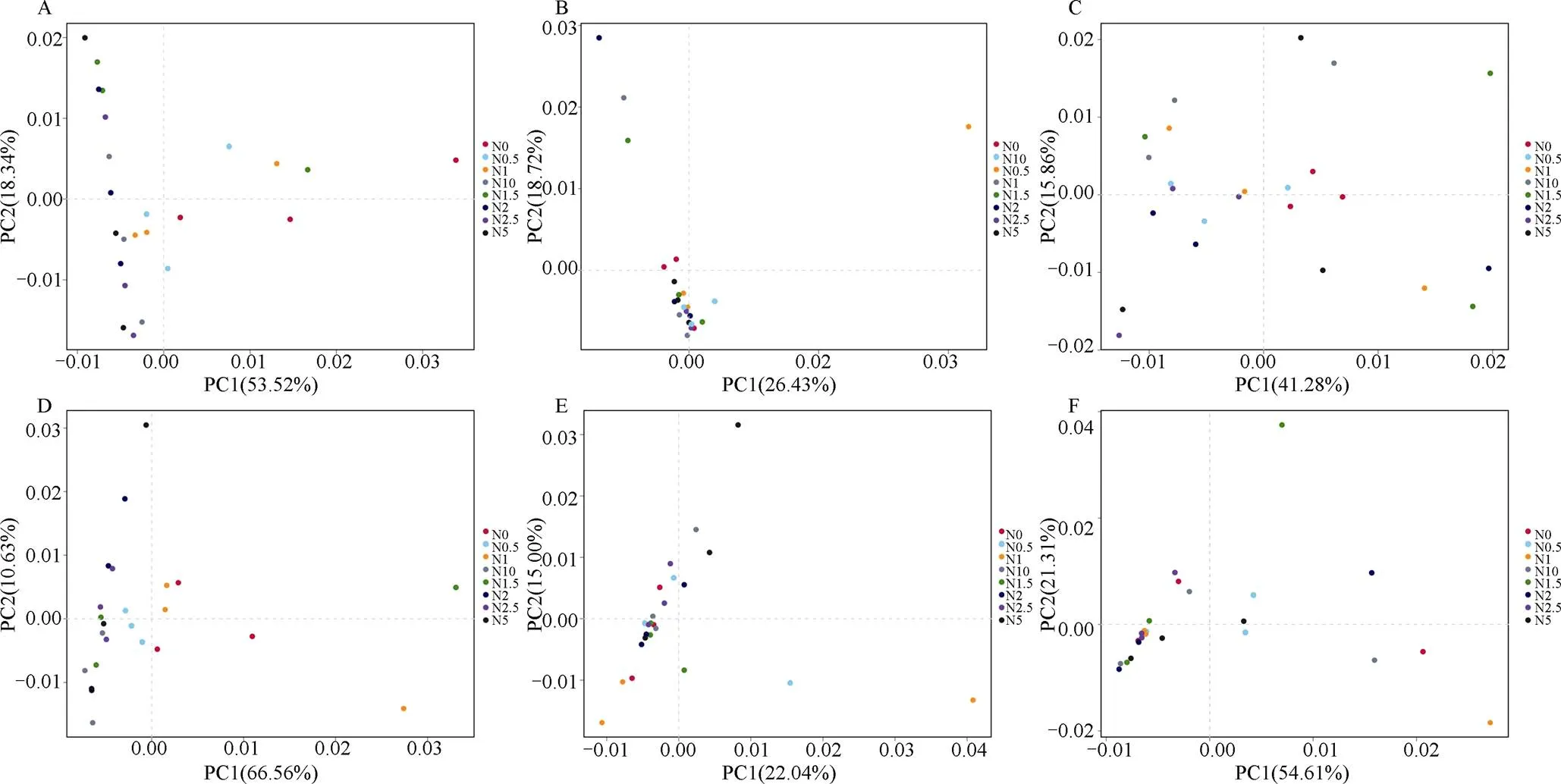
注: A表示0—15 cm 土层细菌群落, B表示0—15 cm 土层真菌群落, C表示0—15 cm 土层古菌群落, D表示15—30 cm土层细菌群落, E表示15—30 cm土层真菌群落, F表示15—30 cm土层古菌群落。下同。
Figure 2 Principal component analysis analysis of soil bacteria fungi and archaea under different nitrogen addition treatments
2.3 不同土壤微生物类群的物种组成变化
在0—15 cm土层中, 细菌优势菌门有变形菌门(Proteobacteria)32.51%—46.26%, 酸杆菌门(Acidobacteria)10.41%—17.55%, 第三为放线菌门(Actinobacteria)11.10%—13.81%。氮添加处理下变形菌门()和放线菌门(Actinobacteria)的相对丰度均有所减少, 而酸杆菌门(Acidobacteria)的相对丰度有所增加(图3A)。真菌优势菌门有占据绝对优势的子囊菌门(Ascomycota)33.33%—64.21%, 其次是被孢霉门(Mortierellomycota)8.68%—13.30%。子囊菌门(Ascomycota)的相对丰度随着氮添加浓度的增加呈现先增加后减少再增加的趋势, 在N10处理下达到最高, N2.5处理下最低; 被孢霉门(Mortierellomycota)的相对丰度除N5处理下减少, 其余处理均增加, 在N1处理下达到最高丰度(图3B)。古菌门类中相对丰度最高的是疣微菌门(Verrucomicrobia)约占整个古菌群落的19.59%—30.23%, 其次是泉古菌门(Crenarchaeota)相对丰度约为4.53%—21.19%, 氮添加条件下, 疣微菌门(Verrucomicrobia)和泉古菌门(Crenarchaeota)的相对丰度均小于N0处理, 且分别在N1.5处理和N2.5处理下达到最低(图3C)。
15—30 cm土层中, 细菌门类相对丰度最高的还是变形菌门(Proteobacteria)约占整个细菌群落的30.58%—48.80%, 其次是绿弯菌门(Chloroflexi)占比约为14.12%—19.40%, 酸杆菌门(Acidobacteria)占比约为9.34%—20.61%, 变形菌门(Proteobacteria)的相对丰度与N0(40.83%)相比, N1和N1.5处理下有所增加, 其余处理下相对丰度均下降, N10处理下最低30.58%; 绿弯菌门(Chloroflexi)的相对丰度与N0(18.16%)相比除N0.5处理下相对丰度增加之外其余处理均下降, 在N10处理下达到最低14.12%; 酸杆菌门(Acidobacteria)的相对丰度在氮添加处理下有所上升(图3D)。真菌门类水平下前10种类别的物种相对丰度如图3E示, 其中子囊菌门(Ascomycota)相对丰度占比约为45.09%—72.99%占据绝对优势, 其次是担子菌门(Basidiomycota)相对丰度占比约为2.54%—8.36%, 子囊菌门(Ascomycota)的相对丰度与N0处理(45.09%)相比, 氮添加处理下均增加了, 并在N10处理达到最高丰度; 担子菌门(Basidiomycota)相对丰度与N0处理(2.95%)相比, 除N0.5处理下减少, 其余处理下均增加。古菌门类物种的相对丰度在氮添加处理下变化较大(图3F), 其中占据主要优势的菌门有广古菌门(Euryarchaeota)相对丰度约占4.63%—38.29%, 疣微菌门(Verrucomicrobia)相对丰度约为14.61%—25.26%, 泉古菌门(Crenarchaeota)相对丰度约为9.23%—26.77%。其中广古菌门(Euryarchaeota)和泉古菌门(Crenarchaeota)的相对丰度在氮添加处理下多表现为减少。
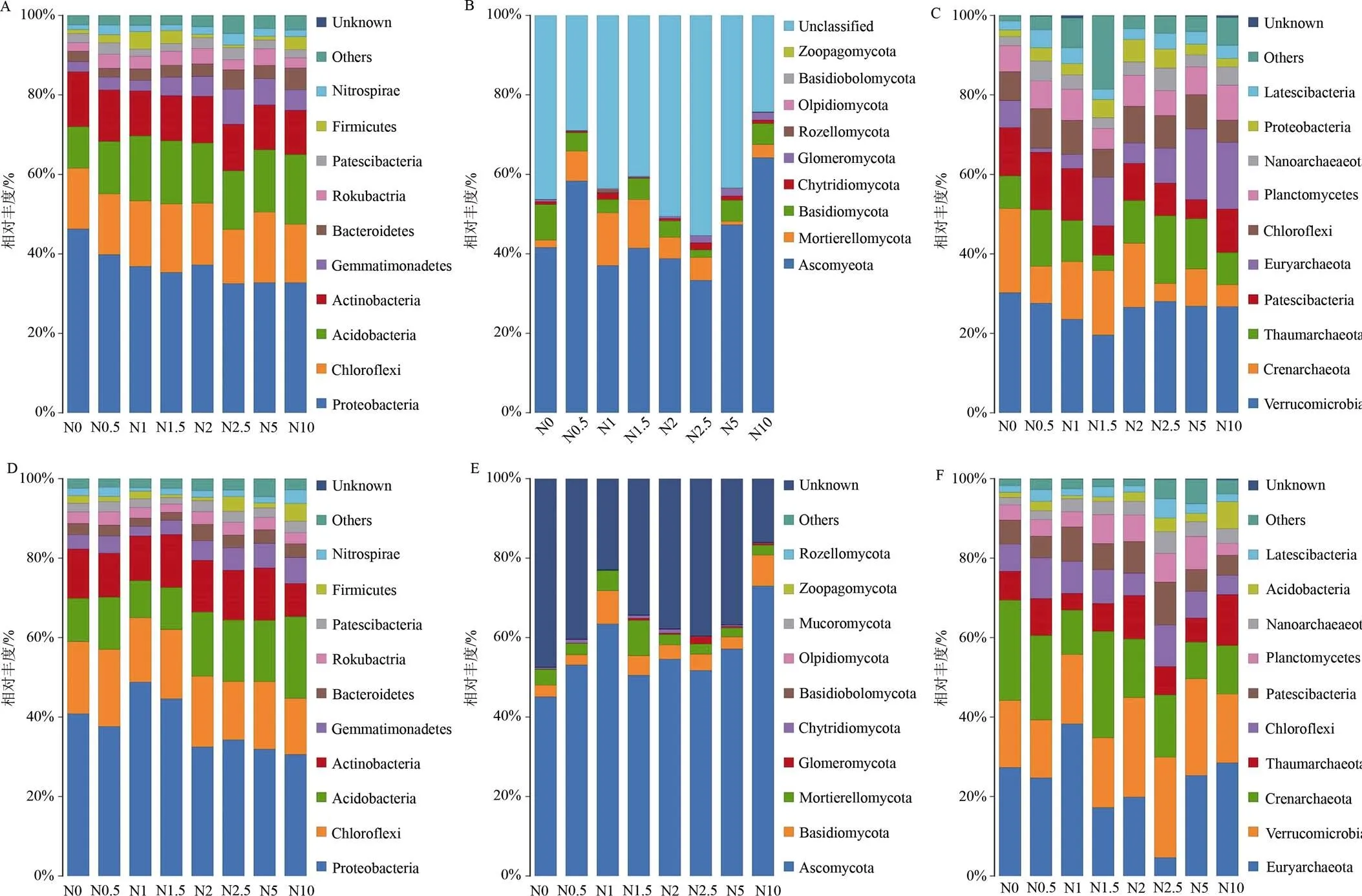
图3 不同氮添加处理下土壤细菌、真菌和古菌的群落组成(门水平)
Figure 3 Community composition of soil bacteria fungi and archaea under different nitrogen addition treatments (phylum level)
综上, 在不同的土层中, 土壤菌群的优势菌门有所变化, 在氮添加条件下细菌和古菌优势菌门的相对丰度有所下降, 而真菌优势菌门的相对丰度呈现出增加的趋势。但是细菌群落中酸杆菌门(Acidobacteria)的相对丰度却与整体优势菌门的变化相反。
3 讨论
3.1 不同土壤微生物类群α和β多样性的特征及差异分析
已有研究表明氮添加引起土壤微生物量的改变[32], 影响微生物多样性和群落结构[33], 多数研究表明施氮降低了细菌多样性[34-35], 本试验得出相反结果, 氮添加降低了细菌OTU数, 但却使细菌Shannon指数上升, 且在N2、N2.5和N10处理下达到显著水平, 细菌的Simpson指数显著下降(表1)。高寒地区环境恶劣, 微生物生长受环境胁迫, 而氮添加改变了土壤元素组成[36], 影响土壤pH[37], 植物生长发育[38]等, 使微生物活性受到抑制, 增加了细菌营养底物, 降低了细菌群落之间的竞争[39], 从而优势物种降低, 多样性上升。土壤真菌菌丝强大, 主要存在于植物根际, 具有强大的纤维素降解能力[40], 是降解纤维素的主要菌类, 生态系统物质循环的主要动力之一[41-42]。多数研究表明, 施加有机肥会降低土壤真菌多样性[43-44], 本试验也得到了类似的结果。真菌Chao1指数和Shannon指数在氮处理下均有显著的响应。真菌多样性随氮添加浓度没有明显的线性关系, 在多数处理下真菌Shannon指数呈下降趋势, 仅在高浓度N10处理下, 两土层的Shannon指数上升。研究显示, 土壤真菌的变化对氮添加的浓度有弹性响应, 这可能受试验地的环境条件、植物群落以及氮添加的时间等多种因素影响[45]。目前, 氮添加对高寒湿地土壤中古菌多样性的变化研究极少。本试验发现, 基于Shannon指数分析, 古菌多样性在N0.5、N2和N2.5处理下两土层中均有增加, 但影响不显著。古菌的变化可能与多种环境耦合作用, 如土壤酶[46]、土壤碳氮含量[47]、植被及土壤类型[48]等。土壤酶是土壤微生物的一个重要指标, 土壤酶活性可以反映土壤有机物的分解强弱。已有研究显示, 氮沉降使土壤酶活性降低, 其中纤维素降解酶和木质素溶解酶最为显著[49]。酶活性降低使植物凋落物分解减慢促进了土壤碳含量的上升。氮沉降对土壤微生物的响应是一个极其复杂的机制, 需要通过多种指标的测定与分析来更为全面的获取土壤微生物对氮沉降的响应机制。
3.2 不同土壤微生物类群的物种组成变化分析
氮沉降促进了土壤微生物结构的改变。氮添加对土壤细菌门类优势菌群产生影响, 如变形菌门(Proteobacteria)和放线菌门(Actinobacteria)均减少, 而酸杆菌门(Acidobacteria)相对丰度增加, 其中在高氮N10处理下酸杆菌门(Acidobacteria)相对丰度增加了 7.41%左右。分析认为, 酸杆菌门与土壤总氮含量呈正相关[50], 氮添加可能使土壤总氮含量增加, 因此, 酸杆菌们的相对丰度上升。说明在高寒地区氮素仍是土壤细菌生长的主要限制因素, 氮添加可能在一定程度上突破了这一限制。变形菌门(Proteobacteria)和放线菌门(Actinobacteria)的相对丰度随土壤pH值的升高而降低[51], 研究表明, 氮沉降降低了土壤pH值[37], 而变形菌门(Proteobacteria)和放线菌门(Actinobacteria)的相对丰度也降低, 这可能与试验环境, 海拔高度等因素相关, 具体机制, 还需进一步探究。氮添加对真菌群落结构也有一定影响, 但影响程度要小于细菌和古菌。真菌门类优势菌群的相对丰度随着氮添加浓度的上升呈现出多种起伏变化, 表明真菌群落可能对氮添加的响应有动态调节作用[52], 已有研究显示, 真菌对环境的适应性可能要优于其他菌类[53], 这在本实验中也得到了支持。子囊菌门(Ascomycota)具有强大的菌丝来吸收营养底物, 在土壤中广泛分布[54], 本试验发现, 子囊菌门(Ascomycota)占据真菌门类的主导地位, 且在氮添加条件下, 子囊菌门(Ascomycota)相对丰度均高于N0空白处理, 真菌群落对于环境的适应性可能与其菌丝生态学特征有关。所以, 真菌群落的稳定性优于细菌和古菌。同时土壤古菌群落结构也发生了明显改变, 疣微菌门(Verrucomicrobia)、广古菌门(Euryarchaeota)和泉古菌门(Crenarchaeota)的相对丰度在氮添加条件下均下降, 使优势菌门丰度呈下降趋势。由于大部分微生物不能独立存在, 需要依靠彼此的细胞外代谢物生存[55]。因此, 微生物可在生态系统中构建复杂的共生网络[56], 而古菌群落在微生物共生网络中占据重要节点[57]。古菌多生于极端环境下, 居于独特的进化地位[58]。由于高寒湿地条件恶劣, 大量古菌类群未被挖掘, 目前古菌的研究也只能借助生物测序, 基于目前研究人们对古菌的了解还不够深入, 应继续注重探索新研究方法关注古菌的生态功能。
试验仅可初步判断高寒湿地微生物区系对氮添加的响应, 湿地具有动态调节能力, 湿地微生物对当前氮添加水平的响应还存在一定的局限性, 今后还应更加注重长期连续的氮添加实验对微生物的累积效应, 以获取更为准确的高寒湿地土壤微生物对氮添加的响应机制。
4 结论
(1)氮添加改变了土壤微生物的多样性。细菌和古菌的多样性指数显著增加, 而真菌多样性下降, 影响不显著。
(2)在氮添加的条件下, 土壤微生物的群落结构有明显变化。细菌和古菌门类水平优势菌群的相对丰度显著下降, 而真菌优势菌门的相对丰度呈现出增加的趋势。
(3)真菌群落对于氮添加的敏感度要小于细菌和古菌群落。
[1] CANFIELD D E, GLAZER A N, FALKOWSKI P G. The Evolution and Future of Earth's Nitrogen Cycle[J]. Science, 2010, 330(6001): 192–196.
[2] 朱潇, 王杰飞, 沈健林, 等. 亚热带农田和林地大气氮湿沉降与混合沉降比较[J]. 环境科学, 2018, 39(6): 2557– 2565.
[3] LIU X, ZHANG Y, HAN W, et al. Enhanced Nitrogen Deposition Over China[J]. Nature, 2013, 494(7438): 459– 462.
[4] GALLOWAY J N, TOWNSEND A R, ERISMAN J W, et al. Transformation of the Nitrogen Cycle: Recent Trends, Questions, and Potential Solutions[J]. Science, 2008, 320(5878): 889–892.
[5] 隋心, 张荣涛, 杨立宾, 等. 应用PLFA技术分析氮沉降对三江平原小叶章湿地土壤微生物群落结构的影响[J]. 中南林业科技大学学报, 2016, 36(12): 91–97.
[6] DENTENER F, DREVET J, LAMARQUE J F, et al. Nitrogen and Sulfur Deposition on Regional and Global Scales: a Multimodel Evaluation[J]. Global Biogeochemical Cycles, 2006, 20(4), doi: 10.1029/2005GB002672.
[7] TIAN D, NIU S, PAN Q, et al. Nonlinear Responses of Ecosystem Carbon Fluxes and Water‐use Efficiency to Nitrogen Addition in Inner Mongolia Grassland[J]. Functional Ecology, 2016, 30(3): 490–499.
[8] BELL T H, KLIRONOMOS J N, HENRY H A L. Seasonal Responses of Extracellular Enzyme Activity and Microbial Biomass to Warming and Nitrogen Addition[J]. Soil Science Society, 2010, 74(3): 820–828.
[9] CHEN W, XU R, CHEN J, et al. Consistent Responses of Surface- and Subsurface Soil Fungal Diversity to N Enrichment are Mediated Differently by Acidification and Plant Community in a Semi-arid Grassland[J]. Soil Biology and Biochemistry, 2018, 127: 110–119.
[10] WANG S, FU B, GAO G, et al. Responses of Soil Moisture in Different Land Cover Types to Rainfall Events in a Re-vegetation Catchment Area of the Loess Plateau, China[J]. Catena, 2013, 101(3): 122–128.
[11] LI J, ZHOU X, YAN J, et al. Effects of Regenerating Vegetation on Soil Enzyme Activity and Microbial Structure in Reclaimed Soils on a Surface Coal Mine Site[J]. Applied Soil Ecology, 2015, 87: 56–62.
[12] 于树, 汪景宽, 李双异. 应用PLFA方法分析长期不同施肥处理对玉米地土壤微生物群落结构的影响[J]. 生态学报, 2008, 28(9): 4221–4227.
[13] LOVETT G M, REINERS W A, OLSON R K. Cloud Droplet Deposition in Subalpine Balsam Fir Forests: Hydrological and Chemical Inputs.[J]. Science, 1982, 218(4579): 1303–1304.
[14] 雷学明, 沈芳芳, 雷学臣, 等. 模拟氮沉降和灌草去除对杉木人工林地土壤微生物群落结构的影响[J]. 生物多样性, 2018, 26(9): 962–971.
[15] VET R, ARTZ RS, CAROU S, et al. A Global Assessment of Precipitation Chemistry and Deposition of Sulfur, Nitrogen, Sea Salt, Base Cations, Organic Acids, Acidity and Ph, and Phosphorus[J]. Atmospheric Environment, 2014, 93: 3–100.
[16] 杨杉杉. 模拟氮沉降对黄河三角洲滨海湿地土壤微生物群落结构及多样性的影响[D]. 长春: 吉林建筑大学, 2018.
[17] LIU X, DUAN L, Mo J, et al. Nitrogen Deposition and Its Ecological Impact in China: an Overview[J]. Environmental Pollution, 2011, 159(10): 2251–2264.
[18] GEISSELER D, SCOW KM. Long-term Effects of Mineral Fertilizers on Soil Microorganisms–a Review[J]. Soil Biology and Biochemistry, 2014, 75: 54–63.
[19] 杨建琴. 松嫩草地土壤微生物量碳、氮对模拟增温及氮沉降的响应[D]. 长春: 东北师范大学, 2011.
[20] 涂利华, 胡庭兴, 张健, 等. 模拟氮沉降对华西雨屏区慈竹林土壤活性有机碳库和根生物量的影响[J]. 生态学报, 2010, 30(9): 2286–2294.
[21] ZECHMEISTER-BOLTENSTERN S, MICHEL K, PFEFFER M. Soil Microbial Community Structure in European Forests in Relation to Forest Type and Atmospheric Nitrogen Deposition[J]. Plant and Soil, 2011, 343(1): 37–50.
[22] DEFORESTt J L, ZAK D R, PREGITZER K S, et al. Atmospheric Nitrate Deposition and the Microbial Degradation of Cellobiose and Vanillin in a Northern Hardwood Forest[J]. Soil Biology and Biochemistry, 2004, 36(6): 965–971.
[23] DEMOLING F, NILSSON L O, BAATH E. Bacterial and Fungal Response to Nitrogen Fertilization in Three Coniferous Forest Soils[J]. Soil Biology and Biochemistry, 2007, 40(2): 370–379.
[24] 周世兴, 黄从德, 向元彬, 等. 模拟氮沉降对华西雨屏区天然常绿阔叶林凋落物木质素和纤维素降解的影响[J]. 应用生态学报, 2016, 27(5): 1368–1374.
[25] WILPISZESKI R L, AUFRECHT J A, RETTERER S T, et al. Soil Aggregate Microbial Communities: Towards Understanding Microbiome Interactions at Biologically Relevant Scales[J]. Applied and Environmental Microbiology, 2019, 85(14).
[26] MA K, ZHANG Y, TANG S, et al. Spatial Distribution of Soil Organic Carbon in the Zoige Alpine Wetland, Northeastern Qinghai-Tibet Plateau[J]. Catena, 2016, 144: 102–108.
[27] 徐晓梅, 王军农. 青海湖流域水文要素初步分析[J]. 科学技术与工程, 2012, 12(29): 7669–7673.
[28] 孙鸿烈, 郑度, 姚檀栋, 等. 青藏高原国家生态安全屏障保护与建设[J]. 地理学报, 2012, 67(1): 3–12.
[29] 刘景华. 青海湖生态价值与生态环境保护研究[J]. 水利经济, 2007(4): 1–2, 6, 81.
[30] 程雷星, 陈克龙, 汪诗平, 等. 青海湖流域小泊湖湿地植物多样性[J]. 湿地科学, 2013, 11(4): 460–465.
[31] 王伟, 刘学军. 青藏高原氮沉降研究现状及草地生态系统响应研究进展[J]. 中国农业大学学报, 2018, 23(5): 151–158.
[32] TIAN D, NIU S. A Global Analysis of Soil Acidification Caused By Nitrogen Addition[J]. Environmental Research Letters, 2015, 10(2): 24019.
[33] EVANS S E, WALLENSTEIN M D, FIERER N. Climate Change Alters Ecological Strategies of Soil Bacteria[J]. Ecology Letters, 2014, 17(2): 155–164.
[34] Sun R, Zhang X, Guo X, et al. Bacterial Diversity in Soils Subjected to Long-term Chemical Fertilization Can Be More Stably Maintained with the Addition of Livestock Manure Than Wheat Straw[J]. Soil Biology and Biochemistry, 2015, 88(4): 9–18.
[35] ZHOU J, GUAN D, ZHOU B, et al. Influence of 34-years of Fertilization on Bacterial Communities in an Intensively Cultivated Black Soil in Northeast China[J]. Soil Biology and Biochemistry, 2015, 90: 42–51.
[36] 刘佳婷, 廖迎春, 谢越, 等. 氮沉降对土壤养分影响研究综述[J]. 中国水土保持, 2019(9): 63–65.
[37] 田圣贤. 氮化合物添加对草地土壤盐基离子和酸缓冲性能的影响[D]. 沈阳; 沈阳大学, 2018.
[38] WANG G, LIU F. Carbon Allocation of Chinese Pine Seedlings Along a Nitrogen Addition Gradient[J]. Forest Ecology and Management, 2014, 334(15): 114–121.
[39] 郭萍萍. 模拟氮沉降对土壤微生物多样性的影响[D]. 福州: 福建农林大学, 2015.
[40] 文亚雄, 谭石勇, 邱尧, 等. 1株秸秆降解高温菌的筛选、鉴定及堆肥应用[J]. 江苏农业科学, 2018, 46(22): 296– 300.
[41] PING W, JUAN C, QINGQIANG Y, et al. Effects of Thermo-chemical Pretreatment Plus Microbial Fermentation and Enzymatic Hydrolysis on Saccharification and Lignocellulose Degradation of Corn Straw[J]. Bioresource Technology, 2015, 194: 165–171.
[42] 田朝光, 马延和. 真菌降解木质纤维素的功能基因组学研究进展[J]. 生物工程学报, 2010, 26(10): 1333–1339.
[43] 蔡艳, 郝明德, 臧逸飞, 等. 不同轮作制下长期施肥旱地土壤微生物多样性特征[J]. 核农学报, 2015, 29(2): 344– 350.
[44] HEDLUND K. Soil Microbial Community Structure in Relation to Vegetation Management on Former Agricultural Land[J]. Soil Biology and Biochemistry, 2002, 34(9): 1299–1307.
[45] ZHAO A, ZHAO A, LIU L, et al. Influences of Canopy Nitrogen and Water Addition on Am Fungal Biodiversity and Community Composition in a Mixed Deciduous Forest of China[J]. Frontiers in Plant Science, 2018, 9, doi: 10.3389/fpls.2018.01842.
[46] WRIGHT C L, SCHATTEMAN A, CROMBIE A T, et al. Inhibition of Ammonia Monooxygenase From Ammonia-oxidizing Archaea By Linear and Aromatic Alkynes[J]. Applied and Environmental Microbiology, 2020, 86(9), doi: 10.1128/AEM.02388-19.
[47] SERGI C, MIREIA F, FREDERIC G, et al. Metabolic Versatility of Freshwater Sedimentary Archaea Feeding on Different Organic Carbon Sources[J]. Plos One, 2020, 15(4), doi: 10.1371/journal.pone.0231238.
[48] MARIE S, CINDY D, VALERIA T, et al. Influence of Plant Genotype and Soil on the Wheat Rhizosphere Microbiome: Evidences for a Core Microbiome Across Eight African and European Soils[J]. Fems Microbiology Ecology, 2020, doi: 10.1093/femsec/fiaa067.
[49] 李焕茹, 朱莹, 田纪辉, 等. 碳氮添加对草地土壤有机碳氮磷含量及相关酶活性的影响[J]. 应用生态学报, 2018, 29(8): 2470–2476.
[50] 魏志文, 李韵雅, 江威, 等. 无锡地区常见树木根际土壤酸杆菌多样性[J]. 生态学杂志, 2018, 37(9): 2649–2656.
[51] 乔沙沙, 周永娜, 刘晋仙, 等. 关帝山针叶林土壤细菌群落结构特征[J]. 林业科学, 2017, 53(2): 89–99.
[52] Xiaoliang L, Tingyao Z, Fei P, et al. Inner Mongolian Steppe Arbuscular Mycorrhizal Fungal Communities Respond More Strongly to Water Availability Than to Nitrogen Fertilization[J]. Environmental Microbiology, 2015, 17(8): 3051–3068.
[53] 王怀玉, 杨万勤. 季节性冻融对亚高山冷杉林土壤微生物数量的影响[J]. 林业科学, 2012, 48(5): 88–94.
[54] FIERER N. Embracing the Unknown: Disentangling the Complexities of the Soil Microbiome[J]. Nature Reviews Microbiology, 2017, 15(10): 579–590.
[55] SAMAY P, CHRISTIAN K. Bacterial Unculturability and the Formation of Intercellular Metabolic Networks[J]. Trends in Microbiology, 2017, 25(5): 349–361.
[56] KAROLIE F, GIPSI L, JEAN-SEBASTIEN L, et al. Cross- biome Comparison of Microbial Association Networks[J]. Frontiers in Microbiology, 2015, 6: 1200.
[57] WEI G, LI M, SHI W, et al. Similar Drivers but Different Effects Lead to Distinct Ecological Patterns of Soil Bacterial and Archaeal Communities[J]. Soil Biology and Biochemistry, 2020, 144, doi: 10.3389/fmicb.2015.01200.
[58] 东秀珠, 李猛, 向华, 等. 探秘生命的第三种形式——我国古菌研究之回顾与展望[J]. 中国科学: 生命科学, 2019, 49(11): 1520–1542.
Response of microbial flora to nitrogen addition in alpine wetlands
XU Runhong, TAN Mei, LIU Zehua, ZHU Jinfu*
College of Life Science, QingHai Normal University, Xining 810008, China
Soil microorganisms are the key drivers of soil nutrient cycle and have obvious induction to soil environmental change. Nitrogen is one of the limiting elements of terrestrial ecosystem, and its change may have a certain impact on the species diversity of the ecosystem. In order to understand the response of soil microbial composition to nitrogen addition in alpine wetland of Qinghai Lake Basin, eight levels of simulated nitrogen addition tests were conducted (0 g·m-2, 0.5 g·m-2, 1 g·m-2, 1.5 g·m-2, 2 g·m-2, 2.5 g·m-2, 5 g·m-2, 10 g·m-2). The results showed that nitrogen addition reduced the Chao1 index of bacterial fungi and archaea, and the Shannon index of bacteria and archaea increased significantly(<0.05). The Shannon index of fungi decreased significantly (<0.05). The nitrogen deposition changed the microbial community structure, and the relative abundance of Proteobacteria, Actinobacteria, Euryarchaeota and Crenarchaeota decreased, while that of Acidobacteria and Ascomycota increased. Fungal communities have a low response to nitrogen addition and may be more adaptive to the environment than bacterial and archaea communities. These results indicate that the effects of nitrogen addition on different soil microorganisms are different. Nitrogen deposition is a process of continuous accumulation, and long-term nitrogen deposition is bound to cause changes in soil microbial community and affect the stability of the ecosystem.
alpine wetland; nitrogen added; soil microorganisms; community structure
10.14108/j.cnki.1008-8873.2022.01.014
徐润宏, 谭梅, 刘泽华,等. 高寒湿地土壤微生物区系组成对氮添加的响应[J]. 生态科学, 2022, 41(1): 120–128.
XU Runhong, TAN Mei, LIU Zehua, et al. Response of microbial flora to nitrogen addition in alpine wetlands[J]. Ecological Science, 2022, 41(1): 120–128.
S157.2
A
1008-8873(2022)01-120-09
2020-06-01
2020-06-18
青海省科技厅项目(2019-ZJ-7068)
徐润宏(1995—), 女, 河北省石家庄人, 硕士研究生, 从事湿地土壤微生物研究, E-mail: 1192387826@qq.com
朱锦福, 男, 副教授, 主要从事生态学研究, E-mail: zjf@qhnu.edu.cn

