希金斯炭疽菌中NPFxD基序蛋白找寻及其生物信息学分析
陈光建 覃悦 韩长志




摘要:希金斯炭疽菌是一种半活体营养型植物炭疽病原真菌,能够侵染多种十字花科植物引起的炭疽病,特别是引起蔬菜炭疽病的重要病原。胞吞在真菌侵染植物过程中发挥着重要作用,该作用一般同具有NPFxD基序的蛋白受体介导有关,然而,目前有关该炭疽菌中胞吞作用相关蛋白尚未明确。基于前人已报道的构巢曲霉中NPFxD蛋白序列,通过关键词搜索以及Blastp比对等方法对希金斯炭疽菌蛋白质数据库进行NPFxD蛋白找寻,明确该菌中存在2个NPFxD蛋白序列,根据蛋白同源性,分别命名为ChMYO1、ChGBP2。同时,对这2个蛋白序列开展了保守结构域及跨膜区结构、蛋白质理化性、信号肽、亚细胞定位以及二级结构等生物信息学分析。结果表明,ChMYO1、ChGBP2蛋白亚细胞定位情况不同,前者定位在质膜,后者定位在核内;两者在亲疏水性、最高氨基酸残基和其位置等均有着一定差异;两者均无明显的信号肽序列;两者均含有无规则卷曲、α螺旋、β卷曲,而缺乏TM螺旋。此外,对多个不同真菌中NPFxD基序同源蛋白序列开展遗传关系分析,明确希金斯炭疽菌中的NPFxD基序蛋白与炭疽菌属中其他病菌中的蛋白具有较高的同源性,本研究为进一步研究炭疽菌属NPFxD基序蛋白的功能提供了理论参考。
关键词:胞吞作用;希金斯炭疽菌;NPFxD基序蛋白;生物信息学
中图分类号: S436.3;S432.4+4文献标志码: A
文章编号:1002-1302(2022)05-0035-06
收稿日期:2021-06-08
基金项目:云南省“兴滇英才支持计划”青年人才专项(编号:YNWR-QNBJ-2020-188);国家自然科学基金(编号:31960211)。
作者简介:陈光建(1987—),男,云南大理人,硕士研究生,从事资源利用与植物保护研究,E-mail:357053147@qq.com;共同第一作者:覃 悦(1992—),男,云南昆明人,硕士研究生,从事资源植物保护与利用研究,E-mail:453276403@qq.com。
通信作者:韩长志,博士,教授,从事经济林木病害生物防治与真菌分子生物学研究。E-mail:hanchangzhi2010@163.com。
希金斯炭疽菌(Colletotrichum higginsanum Sacc)是半活体营养型植物炭疽病原真菌,能够侵染多种十字花科蔬菜引起炭疽病,是生产上危害蔬菜产业健康发展的重要病原菌[1]。前人研究主要集中在生物防治菌株筛选、外源酚酸类物质对羟基苯甲酸(PHBA)控制进化转化以及一些不同营养元素(氮、硅、钾等)控制病害等方面[2]。近年来,随着该菌全基因序列的公布[3],国内外学者展开了丝裂原活化蛋白激酶(MAPK)信号路径相关蛋白的研究,同時,笔者所在研究团队前期也对该菌中的PITP[4]、RGS[5]、14-3-3[6]、septin[7]、AC[8]以及CAZymes等蛋白进行找寻及其生物信息学分析等研究工作[9]。此外,国内诸多学者对该菌中的转化子基因功能开展了研究[10-11]。对于希金斯炭疽菌在侵染十字花科植物过程中,上述效应分子或分泌蛋白如何发挥作用尚不清楚。前人研究发现,胞吞(endocytosis)在生物细胞内部和周围环境之间进行信号通讯过程中发挥着重要作用[12]。不同生物可以释放出一组不同的外膜囊泡,这些囊泡作为运输载体,将效应分子和毒力因子传递给宿主细胞。囊泡的形态情况是通过各种蛋白质模块(复合物)的形成而实现[13],通过酿酒酵母中NPFxD基序蛋白突变试验,明确Stex22p是实现物质摄取的重要蛋白[14]。因此,推测NPFxD基序蛋白在希金斯炭疽菌侵染植物过程中具有重要的作用,特别是对于不同效应分子或分泌蛋白输出到植物中具有重要的功能。
本研究基于前人已报道的构巢曲霉(Aspergillus nidulans)中NPFxD基序蛋白序列[15],利用关键词搜索及同源序列比对等方法,在希金斯炭疽菌蛋白质数据库中开展序列找寻,同时,利用SMART、TMHMM以及Protscale、SignalP、TargetP等生物信息学分析软件,明确该菌中NPFxD基序蛋白的保守结构域、跨膜结构域、理化性质、信号肽以及亚细胞定位情况,此外,对不同真菌中与希金斯炭疽菌中NPFxD基序蛋白同源蛋白序列开展遗传关系分析,以期为进一步开展该菌中胞吞作用研究提供重要的理论基础,也为进一步实现核桃炭疽病病菌相关胞吞蛋白研究提供重要的理论指导。
1 材料与方法
1.1 试验材料
构巢曲霉中NPFxD基序蛋白myo5、gbp2的序列。
1.2 试验方法
1.2.1 希金斯炭疽菌中NPFxD基序蛋白找寻
在炭疽菌属蛋白质数据库中对“NPFxD”和“NPFxD-mediated endocytosis”等关键词进行搜索,并以构巢曲霉中NPFxD基序为参考,选择默认的参数用Blastp进行比对分析,然后,在NCBI上明确该菌中NPFxD的蛋白登录号等基本信息。
1.2.2 保守结构域及跨膜结构域预测
利用SMART网站[16]对NPFxD基序蛋白中所具有的保守结构域进行预测,利用TMHMM Server v. 2.0[17]和HMMTOP version 2.0[18]等分析跨膜结构域。
1.2.3 理化性质分析
根据Protscale[19]对NPFxD基序蛋白开展理化性质分析。
1.2.4 蛋白质信号序列分析
利用SignalP 5.0[20]开展蛋白信号肽预测;利用TargetP 1.1[21]明确蛋白的转运肽。
1.2.5 蛋白质二级结构预测
利用PHD[22]预测程序明确蛋白的二级结构组成情况。
1.2.6 亚细胞位置分析
利用ProtComp v9.0[23]预测蛋白的亚细胞定位情况。
1.2.7 系统进化树构建
基于希金斯炭疽菌(C. higginsanum)中NPFxD基序蛋白序列,利用Blastp软件进行同源序列比对,获得不同真菌中同源蛋白序列,然后,利用ClustalX[24]软件开展多重比对分析研究,再利用MEGA X[25]采用邻近法构建发育树。
2 结果与分析
2.1 希金斯炭疽菌中NPFxD基序蛋白找寻
首先,利用EMBOSS fuzzpro软件开展希金斯炭疽菌中NPFxD基序蛋白序列的找寻[26],得到34条含有NPFxD的蛋白序列。然后,基于构巢曲霉中NPFxD基序蛋白序列[27],利用Blastp比对已经获取的NPFxD基序蛋白开展比对分析,筛选得出共 1 177 条同源序列,同时,利用关键词搜索,并未得到相关蛋白序列。对上述所获得序列,进一步整合分析,并去除重复数据和对比筛选,结果表明,与构巢曲霉(A. nidulans)同源的C. higginsanum中含有NPFxD基序蛋白质序列共2条(表1),其蛋白ID分别为CH063_04448、CH063_04465。根據同源序列相互关系的分析结果,将上述序列分别命名为ChMYO1、ChGBP2。
2.2 保守结构域及跨膜结构域预测
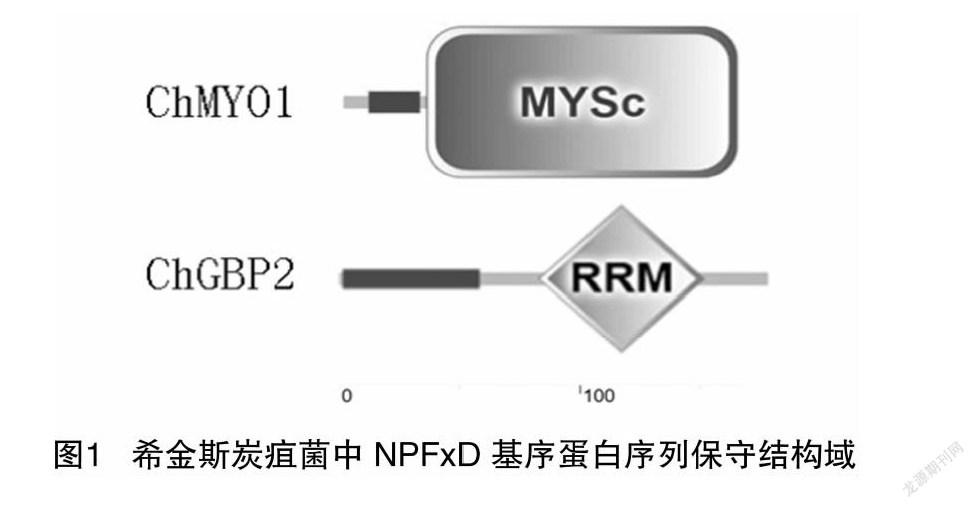
对上述2个NPFxD基序蛋白开展保守结构域分析,结果表明,ChMYO1含有MYSc保守结构域,而ChGBP2含有RRM保守结构域(图1)。进一步分析,MYSc结构域作为ATP酶分子马达,肌肉收缩由肌球蛋白和肌动蛋白之间的周期性相互作用组成,推测该结构域对于胞吞作用的实现发挥着重要功能;RRM结构域则是在许多已知的或应该结合单链RNA的真核蛋白质都包含一个或多个拷贝的大约90个氨基酸的推定RNA结合结构域。同时,开展TMHMM跨膜结构域分析,明确上述2个NPFxD基序蛋白均不含有跨膜结构域,而根据HMMTOP分析,明确ChMYO1具有1个跨膜结构域,起始位置为71、终止位置为90。由于不同的跨膜结构域预测软件所的结果不尽相同,有待于进一步通过试验验证得以明确。
2.3 亚细胞定位分析
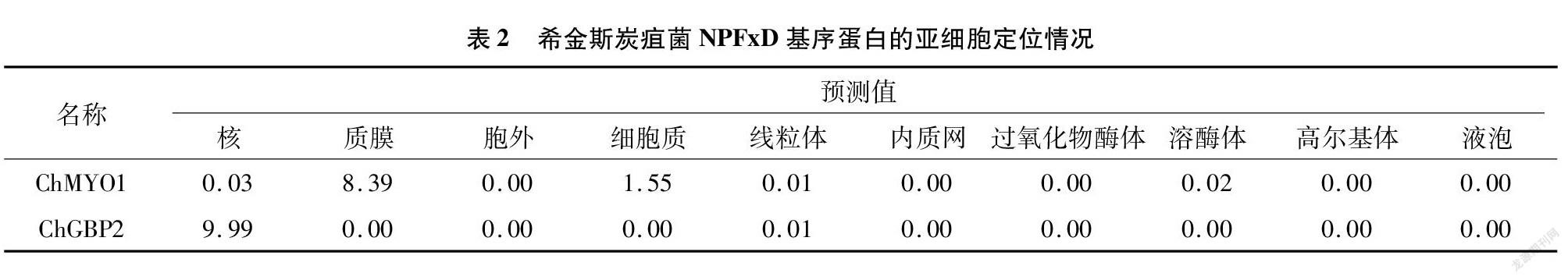
对C. higginsanum中NPFxD基序蛋白进行亚细胞定位分析,结果表明,ChMYO1亚细胞定位在质膜,而ChGBP2蛋白定位在核内(表2)。一般而言,生物中发挥胞吞作用的蛋白经过质膜内陷并形成膜包被的囊泡, 进而实现生理功能,本研究中ChMYO1亚细胞定位在质膜可以较好地解释其功能,而ChGBP2蛋白定位在核内,尚不清楚胞吞作用蛋白定位在核内的原因,推测其还具有其他生物学功能,从而更好地发挥其内吞作用的实现。
2.4 理化性质及疏水性预测
ChMYO1与ChGBP2蛋白在酸碱性氨基酸组成和不带电荷的极性R基氨基酸、非极性R基氨基酸组成方面均存在着一定差异,其中,前者中碱性氨基酸残基K(赖氨酸)、不带电荷的极性R基氨基酸残基G(甘氨酸)的比例较高,均为11.30%,而后者中不带电荷的极性R基氨基酸残基G所占比例最高,为27.30%(表3)。
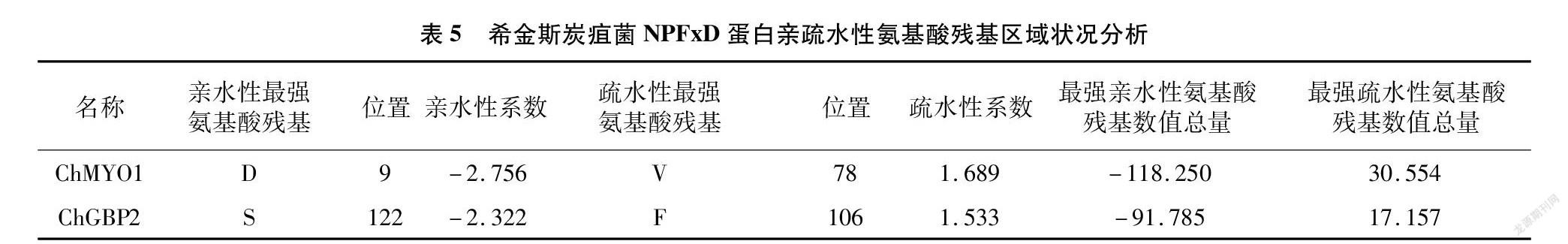
同时,ChMYO1与ChGBP2蛋白在相对分子质量、理论等电点、正电荷氨基酸残基数、负电荷氨基酸残基数、原子质量以及分子式不稳定性系数、总的平均亲水性脂肪族氨基酸指数等方面也具有一定的差异,尽管如此,两者仍具有一定的相似性,其中,两者在不稳定性系数方面数值均小于40,为稳定蛋白;两者的总平均亲水性(GRAVY)数值均小于0,属于亲水性蛋白(表4)。此外,C. higginsianum中ChMYO1与ChGBP2蛋白在亲疏水性、最高氨基酸残基和其位置等均有着一定差异,前者亲水性最强氨基酸残基、疏水性最强氨基酸残基分别为D(天冬氨酸)、V(缬氨酸),后者亲水性最强氨基酸残基、疏水性最强氨基酸残基分别为S(半胱氨酸)、F(苯丙氨酸)(表5)。
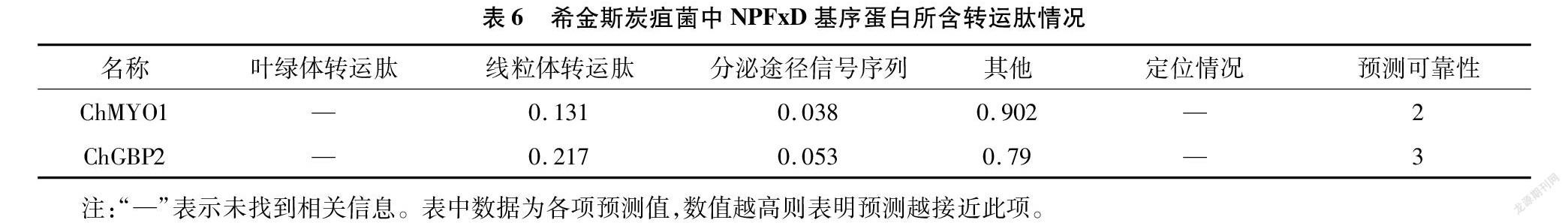
2.5 转运肽及信号序列特征
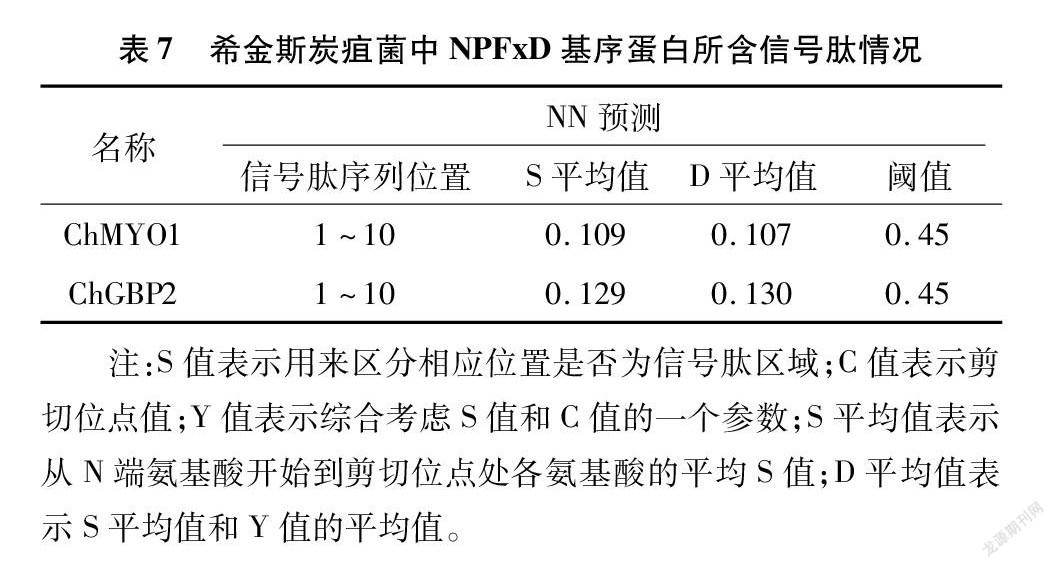
经过TargetP分析,C. higginsianum中ChMYO1、ChGBP2均未获得有效的转运肽定位情况(表6),通过SignalP分析发现,ChMYO1、ChGBP2均没有明显的信号序列(表7)。转运肽(transit peptide)作为一种12~60个氨基酸残基的前导序列,其作用在于引导在细胞溶质中合成的蛋白质输入线粒体和叶绿体。本研究中并未获得ChMYO1、ChGBP2有效的转运肽定位情况,表明上述蛋白并不具有输入到线粒体和叶绿体等功能,这与胞吞作用发挥的功能相一致。同时,作为发挥胞吞作用的蛋白而言,并不具有分泌蛋白特性,因此,与此次所预测的结果——不含有明显的信号肽的预测结果相符合。
2.6 二级结构预测
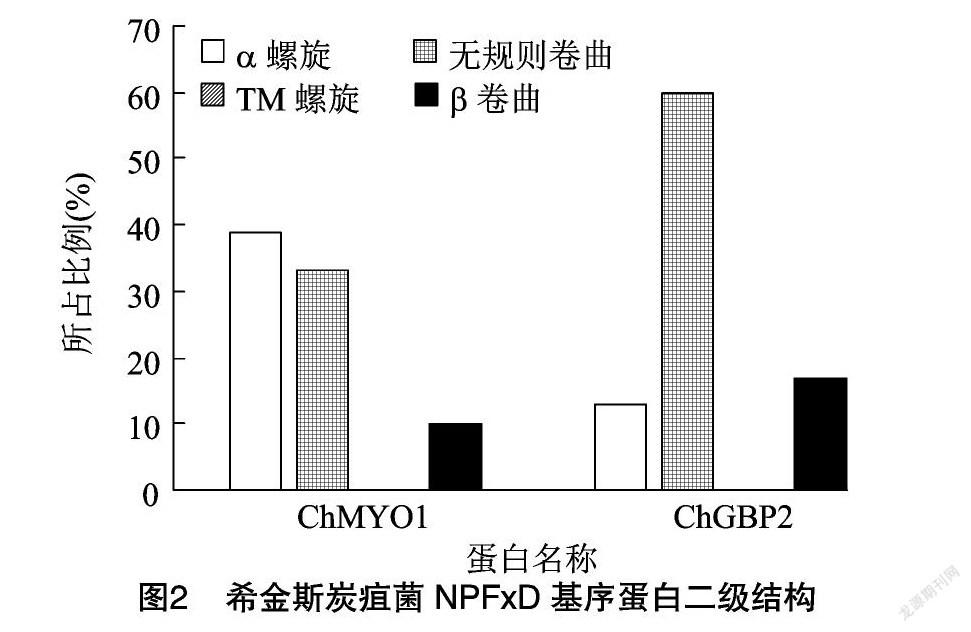
对C. higginsianum中ChMYO1、ChGBP2的二级结构组成情况进行分析,结果表明,上述NPFxD基序蛋白均含有明显的无规则卷曲、α螺旋、β卷曲,且均没有TM螺旋,进一步对上述蛋白二级结构组成展开分析,明确ChMYO1中含有α螺旋的比例高达39%,而ChGBP2中则含有比较多的无规则卷曲,占比为60%(图2)。蛋白质的二级结构指多肽主链骨架原子沿一定的轴盘旋或折叠而形成的特定的构象,即肽链主链骨架原子的空间位置排布,不涉及氨基酸残基侧链。对蛋白质二级结构进行分析,有助于明确其发挥重要作用的构象结构,本研究发现ChMYO1与ChGBP2并不具有相同或相似的蛋白二级结构组成,推测其具有不同的功能。
2.7 遗传关系分析
以C. higginsianum的ChMYO1、ChGBP2蛋白序列作为“种子序列”,在NCBI数据库中开展同源序列搜索,并下载,进一步利用Clust X及MEGA X软件对上述序列进行遗传关系分析,结果显示,在与ChMYO1、ChGBP2同源的18条蛋白序列中,分别以ChMYO1、ChGBP2为中心,明显地分为两大类(图3),说明上述NPFxD基序蛋白序列存在明显的异源关系,这与上述所开展的保守结构域、亚细胞定位以及理化性质分析结果相一致。同时,从遗传关系中可以看出,该菌中的NPFxD基序蛋白与来自于C. tofieldiae、C. sublineola等病菌中的蛋白具有较近的亲缘关系(图3)。
3 讨论与结论
3.1 讨论
植物病原丝状真菌中的胞吞作用,对于其菌丝尖部的快速沿伸具有重要作用。前人研究表明,稻瘟菌中的内吞作用,一般是通过运输Pmk1 MAPK途径的上游膜受体而作用于黏附细胞的产生和侵染等[28],作为同为半活体营养型的希金斯炭疽菌,推测其也采用该胞吞作用机制从而调控着菌丝生长。随着希金斯炭疽菌全基因组序列的公布[3],国内外学者对其致病相关基因开展了诸多解析工作,主要涉及G蛋白信号途径中相关蛋白,如PITP[4]、PLC[29]、RGS[5]、septin[7]、GPCR[30]等蛋白找寻及其基本理化性质、二级结构以及功能特征等。然而,对于上述蛋白如何实现其功能作用的发挥,尚未见学术报道。本研究利用NPFxD基序搜索,共获得了34个蛋白序列,并进一步基于构巢曲霉中的胞吞作用蛋白进行同源比对,最终获得ChMYO1、ChGBP2 2个NPFxD基序蛋白,这2个蛋白在保守结构域、亚细胞定位以及理化性质、遗传关系方面均具有明显的差异性,其在功能上除具有胞吞作用外,是否也具有其他功能方面的差异性,均有待于后续生物学试验验证。同时,其他32个具有NPFxD基序蛋白是否具有胞吞作用,其功能如何,尚不清楚,有待于进一步研究明确。特别是在希金斯炭疽菌侵染植物过程中,具有NPFxD基序蛋白如何协同发挥其功能,均有待于今后进一步明确。
3.2 结论
本研究通过关键词搜索以及Blastp比对找寻,明确了C. higginsianum中具有2个与构巢曲霉中胞吞作用蛋白同源的NPFxD基序蛋白,其蛋白ID分别为CH063_04448、CH063_04465。同时,根据HMMTOP、TMHMM程序对所获取的NPFxD基序蛋白开展跨膜结构域预测解析,结果表明,ChMYO1含有MYSc保守结构域,而ChGBP2则含有RRM保守结构域。对上述蛋白的理化性质、二级结构以及亲疏水性等特征进行分析,明确了上述2个NPFxD基序蛋白在理化性质、亚细胞定位、二级结构组成以及亲疏水性方面均存在着一定的差异性。ChMYO1与ChGBP2蛋白在亲疏水性、最高氨基酸残基和其位置等均有着一定差异,前者亲水性最强氨基酸残基、疏水性最强氨基酸残基分别为D(天冬氨酸)、V(缬氨酸),后者亲水性最强氨基酸残基、疏水性最强氨基酸残基分别为S(半胱氨酸)、F(苯丙氨酸)。ChMYO1亚细胞定位在质膜,而ChGBP2蛋白定位在核内。上述NPFxD基序蛋白均含有明显的无规则卷曲、α螺旋、β卷曲,且均没有TM螺旋,进一步对上述蛋白二级结构组成展开分析,明确ChMYO1中含有α螺旋的比例高达39%,而ChGBP2中则含有比较多的无规则卷曲,比例为60%。此外,对于上述蛋白同源序列开展遗传关系分析明确希金斯炭疽菌中上述NPFxD基序蛋白与C. tofieldiae、C. sublineola等炭疽菌属病菌中的同源蛋白之间的亲缘关系比较相近。上述研究为今后进一步解析该菌以及其他炭疽菌属中的NPFxD基序蛋白功能打下了理论基础。
参考文献:
[1]卢博彬,杨 暹. 菜心炭疽病研究进展[J]. 长江蔬菜,2009(24):1-5.
[2]康云艳,周小萌,杨 暹,等. pHBA对菜心炭疽病的诱导抗性及植株生理特性的影响[J]. 植物病理学报,2014,44(4):393-404.
[3]OConnell R J,Thon M R,Hacquard S,et al. Lifestyle transitions in plant pathogenic Colletotrichum fungi deciphered by genome and transcriptome analyses[J]. Nature Genetics,2012,44(9):1060-1065.
[4]韩长志. 希金斯炭疽菌PITP生物信息学分析[J]. 湖北农业科学,2015,54(3):713-716,721.
[5]韩长志. 希金斯炭疽菌RGS蛋白生物信息学分析[J]. 生物技术,2014,24(1):36-41.
[6]韩长志. 希金斯炭疽菌14-3-3蛋白质生物信息學分析[J]. 湖北农业科学,2014,53(15):3669-3672.
[7]韩长志. 希金斯炭疽菌中5个典型Septin的生物信息学分析[J]. 河南农业科学,2014,43(8):91-96.
[8]韩长志. 希金斯炭疽菌腺苷酸环化酶相关蛋白生物信息学分析[J]. 生物技术,2014,24(6):56-60.
[9]韩长志,祝友朋. 希金斯炭疽菌中碳水化合物酶类蛋白的理化性质分析[J]. 华中农业大学学报,2018,37(3):46-51.
[10]原 田. 希金斯刺盘孢Ch-DES1和Ch-STE12基因的功能分析[D]. 武汉:华中农业大学,2016:10-50.
[11]顾琼楠. 希金斯刺盘孢侵染拟南芥的分子机制研究[D]. 武汉:华中农业大学,2018:39-91.
[12]Marsh M,McMahon H T.The structural era of endocytosis[J]. Science,1999,285(5425):215-220.
[13]McMahon H T,Boucrot E.Molecular mechanism and physiological functions of clathrin-mediated endocytosis[J]. Nature Reviews Molecular Cell Biology,2011,12(8):517-533.
[14]Tan P K,Howard J P,Payne G S.The sequence NPFXD defines a new class of endocytosis signal in Saccharomyces cerevisiae[J]. Journal of Cell Biology,1996,135(6):1789-1800.
[15]Cerqueira G C,Arnaud M B,Inglis D O,et al. The Aspergillus Genome Database:multispecies curation and incorporation of RNA-Seq data to improve structural gene annotations[J]. Nucleic Acids Research,2013,42(D1):D705-D710.
[16]Letinic I,Bork P. 20 years of the SMART protein domain annotation resource[J]. Nucleic Acids Research,2017,46(D1):D493-D496.
[17]Krogh A,Larsson B,von Heijne G,et al. Predicting transmembrane protein topology with a hidden Markov model:application to complete genomes[J]. Journal of Molecular Biology,2001,305(3):567-580.
[18]Simon I.The HMMTOP transmembrane topology prediction server[J]. Bioinformatics,2001,17(9):849-850.
[19]Gasteiger E,Hoogland C,Gattiker A,et al. Protein identification and analysis tools on the ExPASy server[M]//The proteomics protocols handbook.Totowa,NJ:Humana Press,2005:571-607.
[20]Almagro Armenteros J J,Tsirigos K D,Snderby C K,et al. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology,2019,37(4):420-423.
[21]Emanuelsson O,Brunak S,von Heijne G,et al. Locating proteins in the cell using TargetP,SignalP and related tools[J]. Nature Protocols,2007,2(4):953-971.
[22]Kelley L A,Sternberg M J E.Protein structure prediction on the web:a case study using the Phyre server[J]. Nature Protocols,2009,4(3):363-371.
[23]Klee E W,Ellis L B M. Evaluating eukaryotic secreted protein prediction[J]. BMC Bioinformatics,2005,6:256.
[24]Larkin M A,Blackshields G,Brown N P,et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics,2007,23(21):2947-2948.
[25]Kumar S,Stecher G,Li M,et al. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution,2018,35(6):1547-1549.
[26]Rice P,Longden I,Bleasby A.EMBOSS:the European molecular biology open software suite[J]. Trends in Genetics,2000,16(6):276-277.
[27]Schultzhaus Z,Yan H J,Shaw B D. Aspergillus nidulans flippase DnfA is cargo of the endocytic collar and plays complementary roles in growth and phosphatidylserine asymmetry with another flippase,DnfB[J]. Molecular Microbiology,2015,97(1):18-32.
[28]李 瀟. 胞吞调控蛋白MoEnd3和MoCrn在稻瘟病菌致病过程中的功能分析[D]. 南京:南京农业大学,2017:17-20.
[29]韩长志. 希金斯炭疽菌磷脂酰肌醇特异性磷脂酶C生物信息学分析[J]. 江苏农业科学,2016,44(10):177-180.
[30]韩长志. 希金斯炭疽菌GPCR蛋白生物信息学分析[J]. 华中师范大学学报(自然科学版),2015,49(2):246-251.

