2020-2021年河南省猪流行性腹泻病毒流行株遗传进化及S蛋白变异分析
郭子仪,许夕雅,邓梦梦,丁晨梦,高冬生,王永生,陈 陆,阴文奇
(1.河南农业大学 动物医学院,河南 郑州 450046;2.四川省畜牧科学研究院,四川 成都 610066)
猪流行性腹泻(porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)引起的一种急性、高度传染性肠道疾病,以急性水样腹泻、呕吐、脱水为特征,7日龄以下仔猪病死率可达100%[1]。PEDV于1971在英国被首次报道,随后传播到欧洲和亚洲的多个国家[2]。随着基于CV777毒株疫苗的应用,PED在亚洲国家成为地方流行性疾病。2007-2010年,在泰国、越南和中国等地重新出现的PED疫情是由PEDV变异株引起,传统CV777毒株疫苗不能提供有效保护[3]。2013年4月,美国暴发变异株引起的PED疫情迅速蔓延到36个州及墨西哥和加拿大等邻国[4]。PED给世界养猪业造成严重的经济损失[5]。
PEDV是α-冠状病毒属(α-CoV)的成员,具有肠道嗜性[6]。α-冠状病毒属成员感染包括人在内的多种哺乳动物,并导致呼吸道、胃肠道、肝脏和神经系统疾病[7]。冠状病毒S基因编码的S蛋白负责细胞膜融合,S基因的突变、插入和缺失可能会影响冠状病毒的致病性和组织嗜性[8],对于病毒的生物学功能至关重要。SU等[9]对2015-2018年中国22个省市进行PEDV流行病学调查,发现与2011-2014年的PEDV毒株相比,决定PEDV毒力的S1蛋白发生氨基酸突变,突变位点主要位于S1的N-末端结构域(N-terminal domain,NTD)。CHEN等[10]对2011-2017年中国10个省市的腹泻仔猪粪便样品中鉴定出的15株PEDV代表株进行了遗传进化分析,结果显示15株PEDV毒株与当前流行毒株均为GⅡ基因型。这些研究为监测我国PEDV遗传进化情况提供了数据。本试验拟对收集的2020-2021年河南省新生仔猪腹泻临床病料进行RT-PCR鉴定和S基因测序,并对S基因进行同源性比对和遗传进化分析,以期揭示河南省PEDV的最新流行现状及遗传演化情况,为预测PED的流行趋势和防控提供依据。
1 材料与方法
1.1 病料采集及处理采集2020-2021年河南省18个地区499份腹泻仔猪肠道内容物,样品用PBS缓冲液(pH 7.2)按照1∶4的比例混匀,然后10 000×g离心5 min,取上清,-80℃冻存。
1.2 主要试剂M-MLV反转录酶、Recombinant RNAse Inhibitor、2×Taq PCR Mix、DNA Marker均购自宝日医生物技术(北京)有限公司(TaKaRa中国);Oligo dT(18)、Random primer购自生工生物工程(上海)股份有限公司;病毒基因组DNA/RNA提取试剂盒购自天根生化科技(北京)有限公司。
1.3 引物设计与合成根据GenBank 公布的PEDV 变异株序列设计1对针对N基因的引物用于样品检测,设计3对针对S基因的引物用于全基因测序。引物由生工生物工程(上海)股份有限公司合成,引物详细信息见表1。

表1 用于扩增PEDV N基因和S基因的引物
1.4 样品检测取样品上清,利用病毒基因组DNA/RNA提取试剂盒提取样品RNA,然后建立如下反转录体系:5×M-MLV Buffer 4 μL、dNTP(2.5 mmol/L)4 μL、Oligo dT(18)(20 μmol/L)0.5 μL、Random primer(20 μmol/L)0.5 μL、M-MLV反转录酶(200 U/μL)0.5 μL、Recombinant RNase Inhibitor(40 U/μL)0.5 μL、RNA 200 ng,DEPC(焦碳酸二乙酯)水补至20 μL,混匀后置于PCR仪,采用如下反转录程序:42℃处理1 h,70℃处理15 min。反转录后获得的cDNA进行PCR检测,采用50 μL反应体系:25 μL 2×Phanta Max Buffer,dNTP Mix 0.5 μL,上、下游引物(25 μmol/L)各1 μL,Phanta Max Super-Fidelity DNA Polymerasec DNA 1 μL,cDNA 4 μL,ddH2O补至50 μL。反应条件为:95℃预变性5 min;95℃变性30 s,58℃退火30 s,72℃延伸30 s,30个循环;72℃延伸5 min后4℃ 10 min。反应结束后,取PCR扩增产物进行1%琼脂糖凝胶电泳检测。
1.5 PEDVS基因的扩增及测序PCR检测阳性的样品进行S基因扩增及测序。PCR扩增体系和条件同1.4。然后将扩增产物进行胶回收并克隆于pMD18-T载体,挑取阳性质粒送生工生物工程(上海)股份有限公司测序,共测序21株PEDVS基因。
1.6 结果分析将所测序列用CExpress软件拼接,用DNA Star软件中的MegAlign进行同源性比较,用MEGA-X进行遗传进化分析。
2 结果
2.1 样品检测及S基因的扩增测序对处理过的样品进行RT-PCR扩增,电泳鉴定。499份腹泻病料中162份PEDV阳性,在740 bp处出现特异性条带(图1),与预期大小相符,样品阳性率为35.2%。S1、S2和S3片段分别在约1 600 bp位置出现特异性条带(图2),与预期大小相符。对21株特征性 PEDV流行株S1、S2和S3基因片段进行扩增和测序,通过CExpress软件拼接,得到完整的S基因序列,编号命名,并上传至GenBank,登录号为MZ570133~MZ570149和MZ740469~MZ740472。

M.DL1000 DNA Marker;1.PEDV 阳性对照;2.样品;3.阴性对照
2.2 PEDVS基因同源性比对运用DNAstar和MEGA-X软件对本研究毒株、经典毒株以及2011-2019年国内分离毒株进行同源性分析,结果表明21株PEDV的S基因相互之间的核苷酸同源性为96.9%~100%,氨基酸同源性为95.2%~100%;与63株参考毒株之间的核苷酸同源性为92.4%~99.1%,氨基酸同源性为91%~99.4%;与2011-2015年流行毒株核苷酸同源性为96.1%~99.5%,氨基酸同源性为95.9%~99.6%;与2016-2019年流行毒株核苷酸同源性为93.7%~99.7%,氨基酸同源性为93.7%~99.8%。可以看出,PEDV呈现快速进化特点,流行毒株具有多样性。

M.DL2000 DNA Marker;1,2,3.S1~S3基因
2.3 基于S基因的PEDV遗传进化分析通过MEGA-X软件对PEDVS基因序列遗传演化分析,对比对的核苷酸序列采用邻接法(Neighbor-Joining method),通过1 000次重复计算引导值构建系统发育树(图3)。PEDV毒株主要分为GⅠ和GⅡ 2个大群,GⅡ群又分为GⅡa和GⅡb亚群。GⅡ群包括经典毒株、CV777疫苗株。GⅡa群主要包括2011-2015年国内流行毒株;GⅡb群主要包括本试验鉴定的21株毒株以及2015年后国内流行毒株。本试验鉴定的21株毒株位于GⅡb群不同分支,与2019年流行毒株更为接近。可以看出CH-HNXC-03、CH-HNSQ-01、CH-HNXX-02、CH-HNLH-01、CH-HNKF-02、CH-HNXC-01CH-HNXC-04、CH-HNSMX-01、CH-HNXC-02、CH-HNLH-02、CH/HNLH-03、CH-HNJZ-01位于同一个分支,与swun-MY-CH-SCCQ亲缘关系最近;CH-HNJY-01、CH-HNXY-01、CH-HNXX-01、CH-HNNY-01、CH-HNAY-01、CH-KF-03位于同一分支,与swun-MY-CH-SCMY亲缘关系最近;此外CH-HNNY-02、CH-HNPDS-01形成一个独立小分支。结果表明,PEDV呈现快速变异趋势,变异毒株为主流毒株,但也表现出多样性特征。

●表示本试验鉴定2020年毒株;■表示本试验鉴定2021年毒株
2.4 21株PEDV流行株S蛋白氨基酸特征分析将本试验鉴定的21株PEDV S蛋白与2011-2019国内流行的60株毒株氨基酸比对发现,S蛋白氨基酸序列不断发生突变,并逐渐呈现7个规律性变化位点(N139D、P229L、I287M、F345L、F539L、F670I和L998M)(表2)。其中139aa和229aa位于NTD;287aa位于NTD和C-末端结构域(C-terminal domain,CTD)重叠区域 ;345aa和539aa位于CTD区域;而539aa又位于重要抗原表位COE区域;998aa位于七肽重复序列HR1区域(图4)。这些氨基酸变化特征对流行毒株病原特性影响还有待进一步研究。
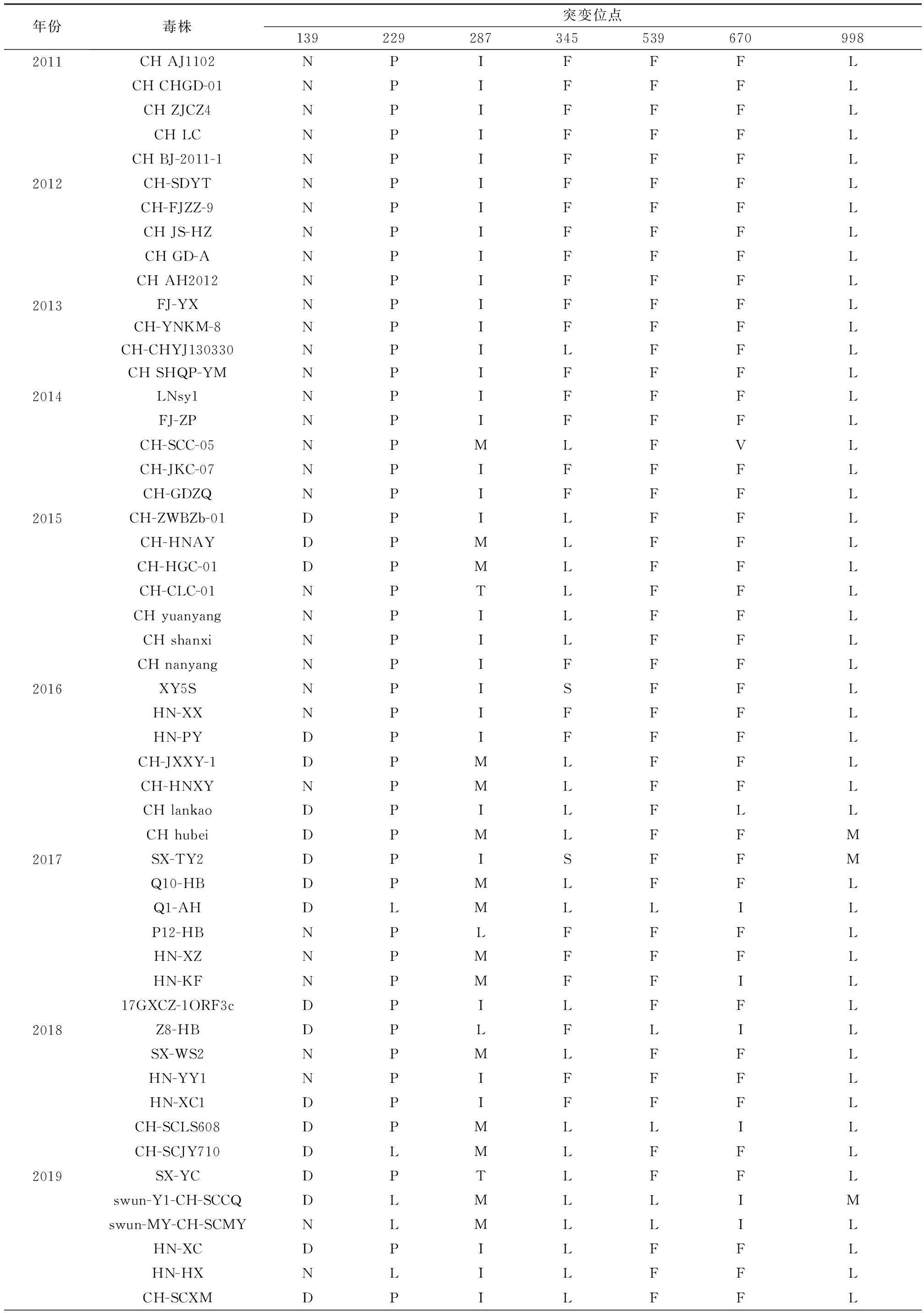
表2 PEDV S蛋白氨基酸突变位点分析

续表2

以CH AJ1102(JX188454)为参考等比例绘制,绿色方框表示氨基酸突变位点
3 讨论
近年来,PEDV的快速变异特性使猪场流行情况变得更加复杂,给现有疫苗的应用带来重大挑战。PED现在仍是中国养猪业最大的威胁之一。本试验对2020-2021年河南省18个地区的499份腹泻样品进行检测分析,得出PEDV总阳性率为32.5%,与黎作华等[11]2017年1月至2018年5月河南省11个地区规模猪场阳性率63.5%相比较低,这可能与在非洲猪瘟模式下,猪场管理加强,人们的生物安全意识提高有着直接关系。
PEDV虽然只有1种血清型,但很多学者根据其全基因组、S基因、ORF3基因或M基因将其分为不同分支来探究其遗传演化规律。DIEP等[12]根据S基因完整序列将其划分为GⅠ和GⅡ 2个大分支,其中GⅠ群又被分为GⅠ-1、GⅠ-2和GⅠ-3等3个亚支,GⅡ群又被分为GⅡ-1和GⅡ-2 2个亚支;ZENG等[13]根据S基因完整序列将其分为 INDEL、GⅠ和GⅡ 3个分支;PARK等[14]根据部分S基因序列将其划分为G1、G2和G3 3个分支。本试验根据遗传演化分析结果将毒株分为2个主要大群,分别为以CV777经典毒株为代表的GⅠ群,以AJ1102等变异毒株为代表的 GⅡ群,GⅡ群又分为GⅡa和GⅡb 2个亚群。GⅠ群包括经典毒株、疫苗毒株。GⅡ群包括本试验鉴定的21株流行毒株以及2011-2019年国流行毒株。并且本试验鉴定的21株毒株又形成2个独立的小分支,与崔建涛等[15]研究结果相似。这些结果表明河南省PEDV的流行情况愈加复杂。
PEDV S蛋白是一种Ⅰ型膜糖蛋白,主要由4个区域构成,从N端至C端依次为:信号肽(signalpeptide,SP)区域(1aa~20aa)、细胞外区域、跨膜区(transmembrane domain,TM)(1 335aa~1 358aa)以及胞质尾巴(cytoplasmic tail,Cyto)(1 358aa~1 385aa)[16]。S蛋白能被宿主蛋白酶切割成S1亚基和S2亚基。S1亚基介导病毒粒子与细胞表面受体结合,S2亚基介导病毒粒子与宿主细胞融合[17]。S1亚基又由4个离散结构域构成,其中包含1个具有与唾液酸结合活性的NTD(20aa~324aa)和1个能与蛋白质受体相互作用的CTD(253aa~638aa)[18]。冠状病毒S1亚基包含有受体结合区域(receptor binding domain,RBD),主要用于识别宿主细胞受体。DENG等[19]通过ELISA和流式细胞术证明PEDV RBD位于S1-CTD区域。最新研究表明,S1蛋白是细胞凋亡的关键诱导剂[20]。截至目前,研究发现S蛋白上至少有4个中和结构域,1个长的核心抗原表位COE(499aa~638aa)和3个线性表位(SS2,748YSNIGVCK755;SS6,764LQDGQVKI771;2C10,1368GPRLQPY1374)[21]。KIM等[20]发现小猪口服NTD231-501蛋白能诱导产生特异性免疫应答,认为S蛋白NTD区域可以作为黏膜免疫疫苗的候选区。本试验发现的流行毒株的7个规律性氨基酸突变位点其中有6个位于S1区,符合S1区变异频率高的特点,其中139aa和229aa位于S1-NTD区域;287aa位于S1-NTD和S1-CTD重叠区域 ;345aa和539aa位于S1-CTD区域;539aa又位于重要抗原表位COE区。S1区氨基酸的突变很可能为病毒逃避获得性免疫反应提供了进化优势[22]。相对于S1,S2亚基在氨基酸序列上更加保守,包括疏水融合肽(FP)、七肽重复区(HR1,978aa~1 117aa和HR2,1 274aa~1 313aa)和C-末端跨膜区(TM)[16]。对于所有的Ⅰ类膜融合蛋白,膜融合都是从疏水融合肽(FP,891aa~908aa)暴露并插入到靶细胞膜开始的,之后融合蛋白被切割成反平行的HR1和HR2区域,形成稳定的六股螺旋结构[23]。在这一转变过程中,融合肽和跨膜结构域变得非常接近,使得病毒和细胞膜能够融合。本试验中998aa位突变位点于HR1区,很可能会影响病毒的膜融合过程。S基因的高变性导致其S蛋白的多样性,S蛋白氨基酸的突变可以使得病毒规避现有疫苗的保护作用,最终导致近几年PEDV疫苗免疫保护作用不强,保护率不高。本试验发现的7个流行毒株氨基酸突变位点有6个位于S基因重要结构域,很大可能会影响毒株的生物学特性,因此根据现行PEDV流行病学特征研制新型疫苗迫在眉睫。
本试验通过对2020-2021河南地区21株特征性PEDV流行毒株S基因进行克隆、测序、遗传进化分析和氨基酸序列比对分析,发现了流行毒株中的7个规律性变化位点,明确了河南省PEDV流行毒株的遗传变异情况,为研发基于PEDV变异株的新型疫苗提供参考。

