31种石斛属植物及金石斛遗传多样性SRAP分析及DNA指纹图谱研究
张迎辉,凡莉莉,杜溶讫,谢德金,杨旺利,陈礼光,郑郁善*
31种石斛属植物及金石斛遗传多样性SRAP分析及DNA指纹图谱研究
张迎辉1,凡莉莉2,杜溶讫2,谢德金2,杨旺利3,陈礼光2,郑郁善2*
1. 福建农业职业技术学院,福建福州 350007;2. 福建农林大学林学院,福建福州 350002;3. 福安市林业局,福建福安 355000
石斛属(Sw.)为兰科(Orchidaceae)兰亚科(Subfam. Orchidoideae)植物,在我国有着广泛的分布与应用,多用于名贵中药材和观赏植物,具有较高的经济价值。由于苛刻的生长条件加上过度的开发利用,导致野生石斛资源破坏严重,大部分种类已近枯竭,这也导致市场上出现的石斛种质混杂、真伪鉴定困难等问题亟待解决。石斛属植物遗传多样性的研究和数字指纹图谱的建立,可以为石斛的品种鉴定与分类、良种选择与保护提供理论依据,对保护药用植物生物多样性也具有十分重要的意义。以31种石斛属植物及金石斛()为研究对象,应用SRAP-PCR分子标记技术对其开展遗传多样性分析并构建DNA指纹图谱。结果表明:利用筛选出来的15对引物进行扩增,共扩增出264个位点,其中多态性位点为251个,平均每对引物扩增出16.73个多态性位点,多态比率达95.08%,其中Me2-Em5、Me4-Em3、Me4-Em5、Me7-Em3、Me7-Ee7等5对引物的多态百分率均为100%,这些引物组合对石斛种类的鉴别效率较高;经过计算31种石斛属植物与金石斛间的观测等位基因数(a)为1.784,有效等位基因数(e)为1.430,Nei’s基因多样性指数()为0.202,Shannon’s信息指数()为0.386,石斛种间存在较高的遗传变异度与丰富的遗传多样性水平;其遗传相似系数的变化范围为0.591~0.851,亲缘关系较近,当遗传相似性系数为0.660时,金石斛单独聚为一类,当遗传相似性系数为0.724时,可将31个石斛属植物进一步分为10组;构建DNA指纹图谱可单独鉴别出31种石斛属植物以及金石斛,为石斛遗传背景分析和快速鉴别石斛种类提供科学依据。
石斛属;SRAP标记;遗传多样性;DNA指纹图谱
石斛属()为兰科(Orchidaceae)植物最大的属之一,约含1000个种,其中我国分布有74个种,2个变种[1]。虽然我国石斛属资源在世界上的占有率较低,但因其具有较高的观赏价值[2-3]和药用价值[4-5],在资源开发与利用方面一直走在前沿。石斛作为传统珍贵中药材,在我国已有2000多年的使用历史。石斛是典型的多基源药材,《中国药典》中收载的石斛来源为兰科植物金钗石斛()、霍山石斛()、鼓槌石斛()或流苏石斛()的栽培种及其同属植物近似种和铁皮石斛()的茎[6]。但由于资源缺乏,目前我国商品流通的药用石斛植物来源有近40种,因此石斛属植物种质混杂、真伪鉴定困难等问题亟待解决。DNA分子标记技术具有操作简单、多态性和重复性好等优点,已被广泛应用于石斛亲缘关系鉴别、基因库构建、指纹图谱库等领域[7-10]。崔学强等[11]采用iPBS分子标记技术分析并构建48份石斛兰种质资源DNA指纹图谱,且利用2对引物可单独进行鉴别。宋爽等[12]利用ISSR和AFLP两种分子标记方法分析了石斛种质资源的遗传多样性,均能有效地将研究材料进行鉴别。蔡莉等[13]对15份金钗石斛栽培种样品运用SRAP分子标记法检测其遗传多样性,认为SRAP分子标记法能有效地反映金钗石斛栽培种的遗传多样性。樊洪泓等[14]利用SRAP和RAPD两种分子标记技术对9份石斛种质进行遗传多样性研究,发现SRAP标记在石斛的遗传多样性与亲缘关系的研究中表现出了极大的优越性。本研究以31种石斛属植物及金石斛为试验材料,应用SRAP-PCR分子标记技术对其开展遗传多样性分析并构建DNA指纹图谱,将为石斛的品种鉴定与分类提供理论和技术依据。
1 材料与方法
1.1 材料
供试的32种石斛样品采自福建省福安旺盛经济林研究所石斛种质资源圃(表1)。每种石斛取样7~10株,每株3~5片嫩叶,混合后装入自封袋中,用变色硅胶(硅胶与叶片体积比例至少10∶1)迅速干燥放入冰盒中,将干燥的样品快速带回实验室,置于–80℃超低温冰箱中保存备用。

表1 供试材料
1.2 方法
1.2.1 石斛总DNA的提取 石斛总DNA采用郑云柯等[15]的改良CTAB法提取,具体如下:取0.1 g嫩叶放入液氮中研磨粉碎并干燥至略发白色;将粉碎的样品粉末放入2 mL的离心管中,立即添加预热的CTAB缓冲液750 µL,16 µL的25 mg/mL R Nase A,65℃水浴50 min,期间轻微颠倒混匀离心管4~6次;取出离心管放至冷却后加入等体积氯仿-异戊醇(24∶1)混合液,轻微颠倒混匀,并静放3 min分层;将混合物于12 000 r/min,4℃中离心12 min;取上清液加800 µL无水乙醇(预冷)轻微充分颠倒混匀,直到有大量白色絮状物析出,放入–20℃冰箱中保存20 min;将离心管于12 000 r/min,4℃中离心5 min,倒出液体后,加入75%乙醇(预冷)清洗2次(12 000 r/min,4℃中离心3 min),室温晾干;加入100 µL已灭菌的ddH2O溶解,将含有DNA的离心管放入–20℃冰箱中保存备用。
1.2.2 SRAP-PCR反应体系 PCR反应体系采用前期研究结果[16]:Mg2+2.5 mmol/L,dNTPs 0.25 mmol/L,DNA聚合酶1.25 U,引物0.4 µmol/L,模板DNA 30 ng,10×PCR buffer 2.5 µL,其余的用灭菌的ddH2O补齐,形成共25 µL的扩增体系。PCR反应程序:94℃预变性5 min,94℃变性1 min,36℃复性1 min,72℃延伸1 min,5次循环,94℃变性1 min,根据不同引物的不同退火温度复性1 min,72℃延伸1 min,30次循环,最终72℃延伸7 min,保存于4℃恒温中。SRAP引物序列见表2。
表2 SRAP引物序列

Tab.2 Primers used in SRAP molecular markers
1.3 数据处理
依据常青等[17]的数据分析方法对电泳图谱进行人工读带,同一位点有条带出现的赋值为“1”,无条带出现的赋值为“0”,将数据输入Excel表格中制成0-1二元矩阵。使用NTSYS Version 2.1软件,结合POPGENE Version 1.32软件计算遗传距离与遗传相似性系数,运用非加权组平均法(UPGMA)进行聚类分析,建立聚类图。
2 结果与分析
2.1 引物扩增多态性及遗传多样性分析
用筛选出来的15对引物对31种石斛属植物及金石斛进行扩增(图1),共扩增出264个位点,其中多态性位点为251个,平均每对引物扩增出16.73个多态性位点,多态率达到95.08%(表3)。31种石斛与金石斛的观测等位基因数(a)、有效等位基因数(e)、Nei’s基因多样性指数()、Shannon’s信息指数()分别为1.784、1.430、0.202、0.386,证明石斛种间存在较高的遗传变异度与丰富的遗传多样性水平。在15对引物组合中,Me2-Em5,Me4-Em3,Me4-Em5,Me7-Em3,Me7-Ee7的多态百分率均为100%,说明这些引物组合对石斛种类的鉴别效率较高,并且SRAP分子标记方法能很好地应用于石斛种类的遗传多样性研究中。
表3 SRAP引物序列及其扩增结果
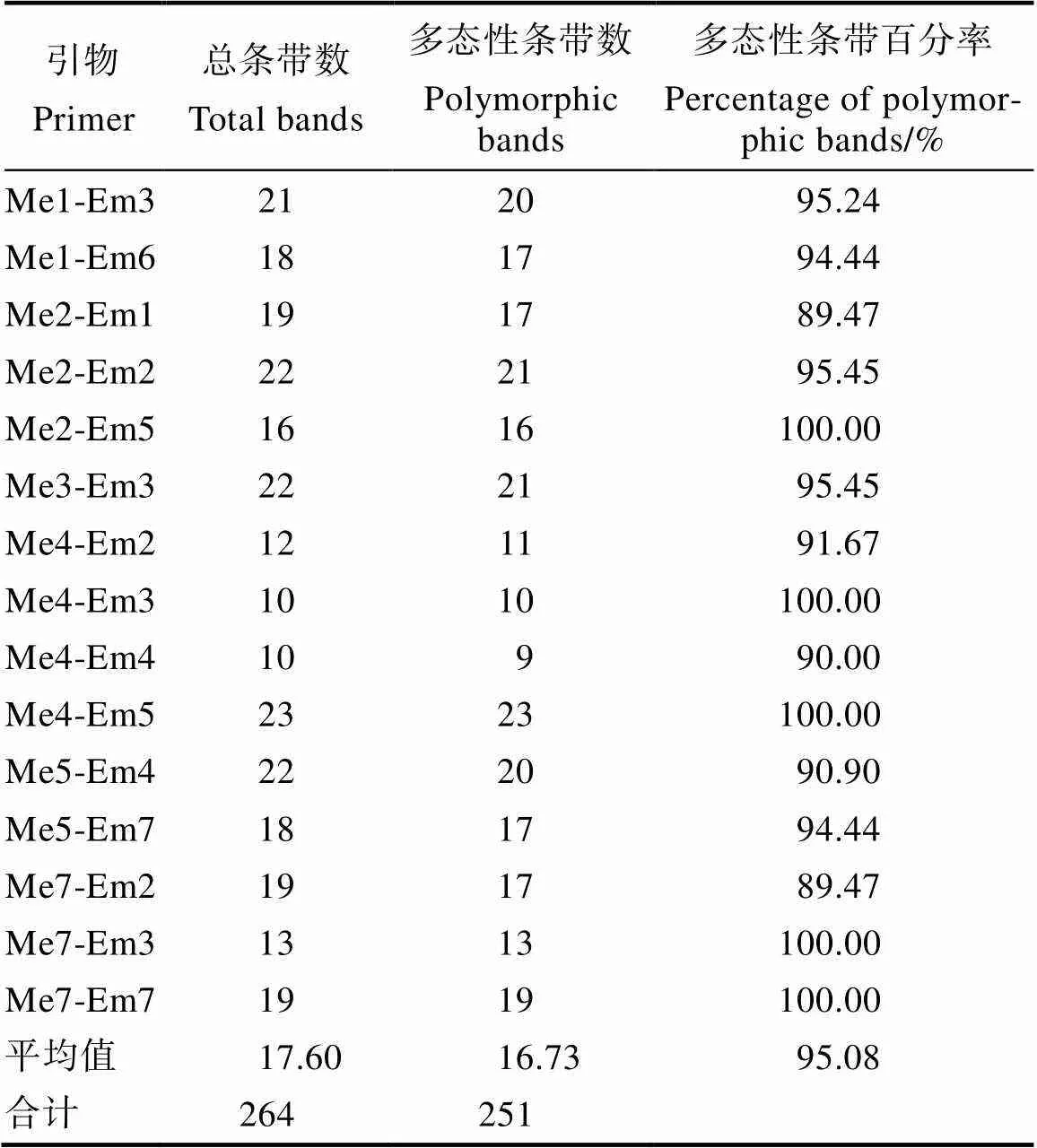
Tab.3 Sequences and amplification results of SRAP primers
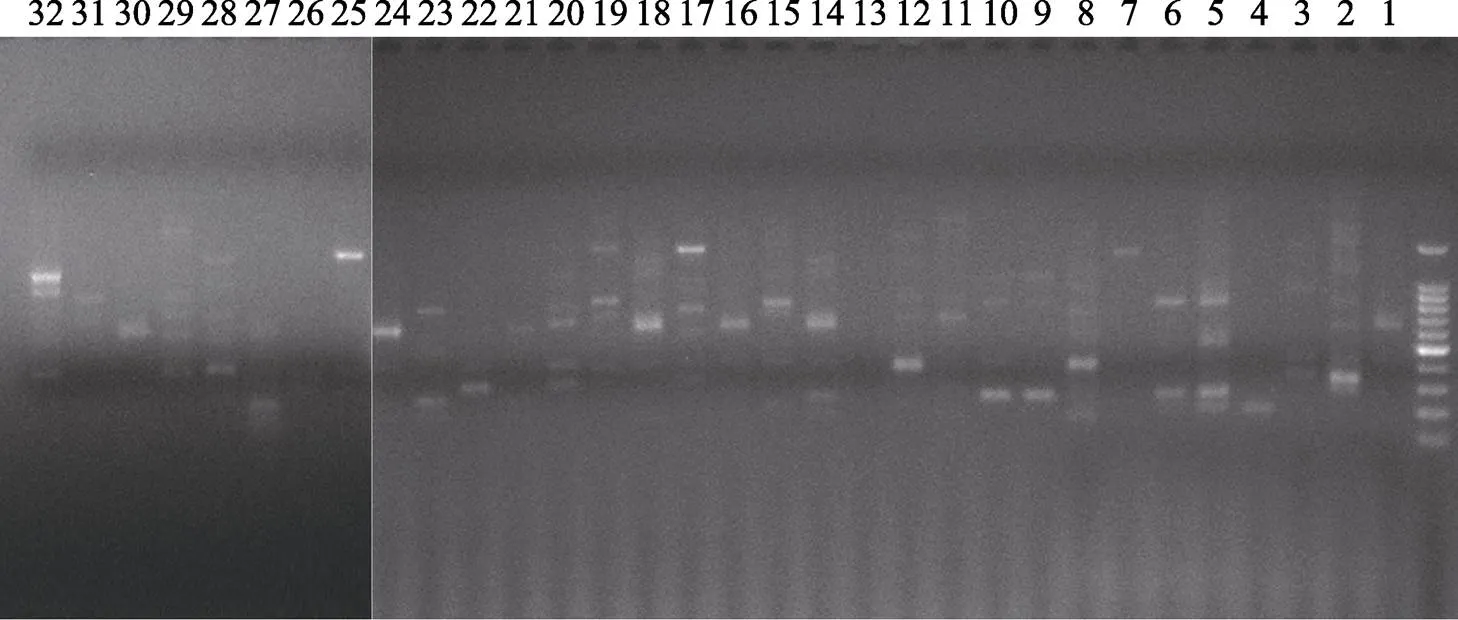
图1 E7M7凝胶成像图
2.2 聚类分析
为了确定石斛种间的遗传关系,将统计好的石斛SRAP-PCR反应数据录入NTSYS Version 2.1软件中进行聚类分析(UPMGA),建立遗传关系聚类图(图2)。31种石斛属植物及金石斛的遗传距离范围为0.138~0.522,均值为0.317,遗传相似性的变化范围为0.591~0.851,均值是0.733,说明31种石斛属植物及金石斛的品种间存在一定的遗传变异。从图2可以看出,当遗传相似性系数为0.660时,31个石斛属植物聚为一类,记为Ⅰ类;金石斛单独聚为一类记为Ⅱ类,表明其遗传基础与石斛属植物存在着较大的差异。当遗传相似性系数为0.724时,Ⅰ类中的31个石斛属植物又可再分为10组,分别记为A、B、C、D、E、F、G、H、I、J,其中A组为海南石斛、少花石斛;B组为长苏石斛、束花石斛、小双花石斛、美花石斛、竹枝石斛、疏花石斛、曲轴石斛、尖刀唇石斛、喇叭唇石斛、金钗石斛、铁皮石斛、霍山石斛、细叶石斛、短棒石斛、杓唇石斛;C组为鼓槌石斛、聚石斛、小黄花石斛;D组为罗河石斛和黄花石斛;E组为剑叶石斛;F组为反瓣石斛;G组为矮石斛、长距石斛;H、I、J组分别为叉唇石斛、钩状石斛和景洪石斛。
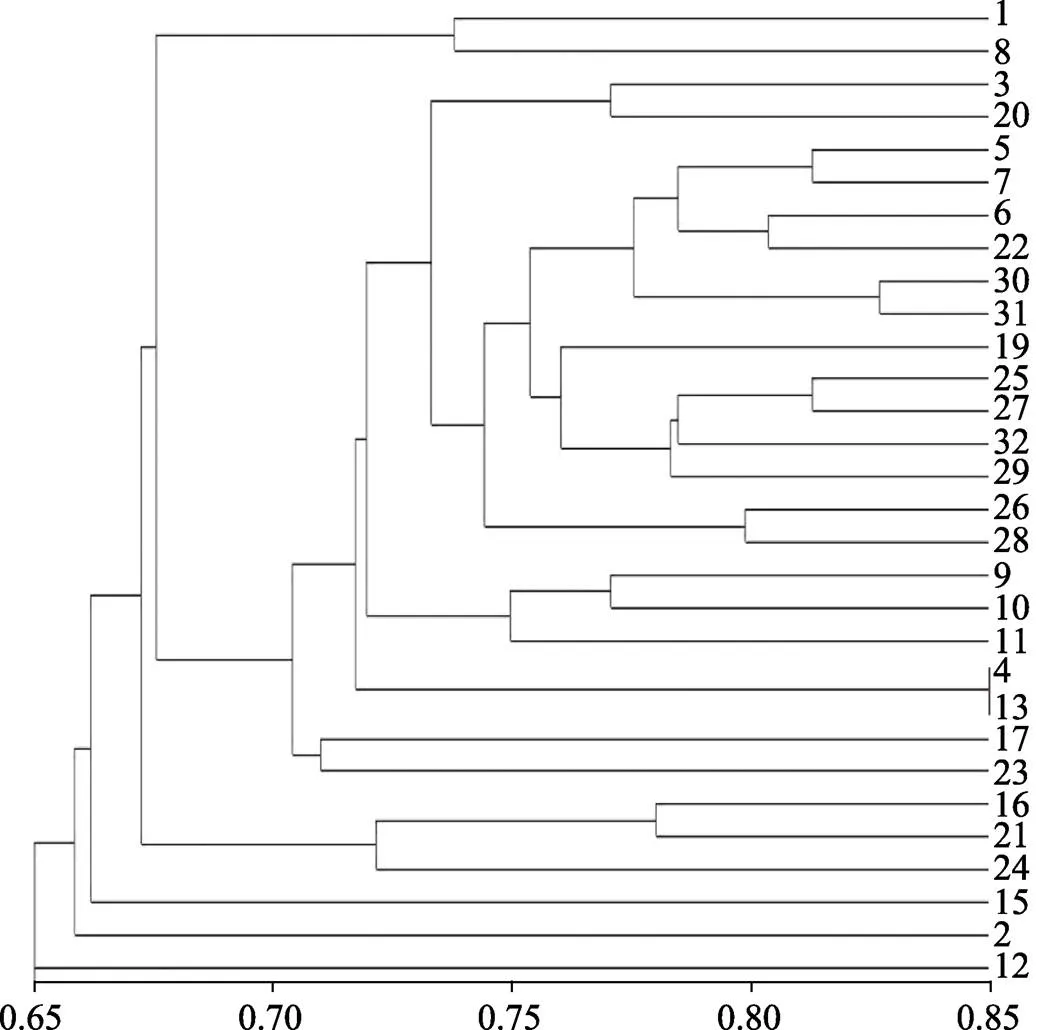
图2 31种石斛属植物及金石斛基于SRAP的遗传相似性聚类分析
2.3 DNA指纹图谱构建
据引物品种鉴别率公式对15个引物组合进行鉴别结果统计,其中引物组合Me1-Em3、Me3-Em3、Me1-Em6均能将31种石斛及金石斛完全鉴别出来,而其他引物组合鉴别率则在68.75%~93.75%之间,未达到完全鉴别。利用引物Me1-Em3构建石斛种类的DNA数字指纹图谱(图3),其中黑色表示该位点有条带,即该位点0-1矩阵中为“1”,无色表示该位点无条带,即该位点在0-1矩阵中为“0”。构建的指纹图谱可以为石斛遗传背景分析和种类鉴定提供依据。
3 讨论
分子标记技术已广泛应用于石斛类植物的研究中。刘玲等[18]采用TRAP分子标记技术,筛选出7对引物可将6个铁皮石斛野生居群以及杂交后代进行鉴别。付涛等[19]开发铁皮石斛EST-SSR分子标记方法,设计的43对引物能够扩增出多态性条带。肖文芳等[20]采用P-M13-SSR分子标记技术将29份石斛兰划分为6个类群,并利用3对引物获得29份石斛兰种质独有、可辨的分子身份证。SRAP标记方法是由美国加州大学蔬菜作物系的LI等[21]创立的一种新型的分子标记方法,该方法的基础虽然也是依靠PCR技术,但其克服了成本高昂、技术繁杂、重复性差等缺点,兼具了简单、可靠、中等信息量和易于对相应片段测序等优点[22],作为理想的分子标记技术在植物遗传多样性分析[23-26]、种质鉴定[27-29]等方面得到广泛应用。

图3 31种石斛属植物及金石斛DNA指纹图谱
杨贞等[30]利用SRAP分子标记法从13份铁皮石斛种质中扩增出的多态性条带为96.75%,遗传相似系数为0.44~0.79,并根据亲缘关系划分为3个类群。林榕燕等[31]从48份石斛兰种质扩增出的多态性条带高达97.48%,多态性信息含量为0.718~0.903,石遗传距离为0.15~0.97,并将48个石斛兰品种分为7大聚类群。朱胜男等[2]采用RAPD分子标记技术对31种石斛属植物进行了遗传多样性分析,根据聚类图结果可以有效地将32个石斛属植物区分开。崔学强等[11]利用iPBS标记对麝香石斛()等18份石斛兰原生种以及30份石斛兰商品种构建的DNA指纹图谱可区分出这48份石斛兰种质资源。本研究采用SRAP分子标记技术对31份石斛属植物原生种以及金石斛开展研究,利用筛选出来的15对引物进行扩增,共扩增出264个位点,其中多态性位点为251个,多态率达到95.08%,其中5对引物的多态百分率达到100%,表明这些石斛属品种的遗传丰富度较高,遗传范围较为广泛。遗传距离与遗传相似性系数是衡量个体间相似程度的重要指标,31种石斛属植物及金石斛的遗传距离范围为0.138~0.522,表明品种间存在丰富的遗传多样性,遗传相似系数的变化范围为0.591~0.851,说明31种石斛及金石斛的亲缘关系较近,其中石斛属植物间罗河石斛与黄花石斛的亲缘关系最近,景洪石斛与金石斛的亲缘关系最远。在聚类分析中金石斛单独聚为一类,剩余的31种石斛属聚为一大类,表明SRAP可有效鉴别出石斛属植物。当遗传相似性系数为0.724时,可将31个石斛属植物为10组,这与植物志中石斛属的分类较为符合。
构建DNA指纹图谱已成为植物品种鉴定的有效方法之一,利用分子标记技术构建石斛兰DNA指纹图谱有少量报道,其中关键是筛选出可鉴别供试材料的引物[2, 18, 20]。本研究筛选的15对引物组合均能鉴别31个石斛属植物及金石斛,应用引物Me1-Em3构建出的DNA指纹图谱,能为石斛的分子鉴别提供参考。
[1] 中国科学院中国植物委员会. 中国植物志(第十九卷)[M]. 北京: 科学出版社, 1999: 69.
Chinese Botanical Committee, Chinese Academy of Sciences. Flora of China (Volume 19)[M]. Beijing: Science Press, 1999: 69. (in Chinese)
[2] 朱胜男, 周振华, 冯尚国, 汪 尚, 索娜娜, 王慧中. 31种石斛属植物的RAPD遗传多样性分析[J]. 杭州师范大学学报(自然科学版), 2011, 10(4): 333-339.
ZHU S N, ZHOU Z H, FENG S G, WANG S, SUO N N, WANG H Z. Analysis on the genetic diversity among 31 species ofbased on RAPD marker[J]. Journal of Hangzhou Normal University (Natural Science Edition), 2011, 10(4): 333-339. (in Chinese)
[3] 鲜小林, 陈 睿, 万 斌, 秦 帆. 西南地区野生春石斛资源搜集、保存与观赏利用价值评价[J]. 西南农业学报, 2013, 26(3): 1184-1189.
XIAN X L, CHEN R, WAN B, QIN F. Wild spring dendrobium resources’ collection, preservation and evaluation of ornamental and use values in Southwest China[J]. Southwest China Journal of Agricultural Sciences, 2013, 26(3): 1184-1189. (in Chinese)
[4] 李 涛, 何 璇. 石斛属27种药用植物的性状鉴定特征比较[J]. 华西药学杂志, 2016, 31(1): 54-57.
LI T, HE X. Comparative study on the macroscopical identification characters of twenty-seven herbal medicines of[J]West China Journal of Pharmaceutical Sciences, 2016, 31(1): 54-57. (in Chinese)
[5] 陈慧玲, 刘宗坤, 杨彦伶, 张新叶. 基于层次分析法的药用石斛种质资源评价[J]. 西南林业大学学报, 2017, 37(1): 82-87.
CHEN H L, LIU Z K, YANG Y L, ZHANG X Y. Evaluating germplasm of medicinalspp. via analytic hierarchy process[J]. Journal of Southwest Forestry University, 2017, 37(1): 82-87. (in Chinese)
[6] 中华人民共和国国家药典委员会. 中华人民共和国药典: 2015年版. 第1部[M].北京: 中国医药科技出版社, 2015: 94, 295.
State Pharmacopoeia Committee of the People’s Republic of China. Pharmacopoeia of the People’s Republic of China: 2015 edition part 1[M]. Beijing: China Medical Science and Technology Press, 2015: 94, 295. (in Chinese)
[7] NGUYEN N H, VU H T, LE N D, NGUYEN T D, DUONG H X, TRAN H D. Molecular identification and evaluation of the genetic diversity ofspecies collected in southern Vietnam[J]. Biology, 2020, 9(4): 76.
[8] 韩晓霞, 章静钢, 张鹏博, 宗 宇, 陈文荣, 郭卫东. 基于SSR的铁皮石斛实生群体遗传多样性研究[J]. 江苏农业科学, 2016, 44(6): 90-93.
HAN X X, ZHANG J G, ZHANG P B, ZONG Y, CHEN W R, GUO W D. Genetic diversity ofseedling population based on SSR[J]. Jiangsu Agricultural Sciences, 2016, 44(6): 90-93. (in Chinese)
[9] LI Q G, MO J M, WU W R, YANG J, LI J J, LAI T X, QU Z L, QIU Z W, GUAN S X, LIAO J J. Genetic diversity, population structure and identification ofcultivars with high polysaccharide contents using SCOT, SCAR and nested PCR markers[J]. Genetic Resources and Crop Evolution, 2019, 66(1): 71-88.
[10] JIANG R B, XU X D, CHEN N P, DING Z S, JIN B. Analysis of the genetic diversity ofby ISSR, SRAP, and scoT[J]. Journal of Biobased Materials and Bioenergy, 2017, 11(6): 590-597.
[11] 崔学强, 唐 璇, 黄昌艳, 邓杰玲, 李秀玲, 卢家仕, 张自斌. 基于iPBS标记的石斛兰种质资源遗传多样性分析及DNA指纹图谱构建[J]. 热带作物学报, 2021, 42(2): 317-324.
CUI X Q, TANG X, HUANG C Y, DENG J L, LI X L, LU J S, ZHANG Z B. Genetic diversity analysis and fingerprinting construction ofgermplasm resources by ipbs marker[J]. Chinese Journal of Tropical Crops, 2021, 42(2): 317-324. (in Chinese)
[12] 宋 爽, 周洋帆, 刘正杰, 赵明富, 杨 娟, 徐绍忠, 文国松. 利用ISSR和AFLP标记分析石斛种质资源的遗传多样性[J]. 云南农业大学学报(自然科学), 2016, 31(4): 688-695.
SONG S, ZHOU Y F LIU Z J, ZHAO M F, YANG J, XU S Z, WEN G S. Genetic diversity amongwith known origins based on the ISSR and AFLP markers[J] Journal of Yunnan Agricultural University (Natural Science), 2016, 31(4): 688-695. (in Chinese)
[13] 蔡 莉, 赵 致, 刘红昌, 李金玲, 杨继勇, 邓贤芬. 基于SRAP分子标记的金钗石斛遗传多样性研究[J]. 时珍国医国药, 2019, 30(8): 1975-1978.
CAI L, ZHAO Z, LIU H C, LI J L, YANG J Y, DENG X F. Genetic diversity ofbased on SRAP molecular markers[J]. Lishizhen Medicine and Materia Medica Research, 2019, 30(8): 1975-1978. (in Chinese)
[14] 樊洪泓, 李廷春, 邱 婧, 林 毅, 蔡永萍. 石斛属几种植物遗传关系的SRAP和RAPD比较分析[J]. 中草药, 2010, 41(4): 627-632.
FAN H H, LI T C, QIU J, LIN Y, CAI Y P. Comparison of SRAP and RAPD markers for genetic analysis of plants inSw[J]. Chinese Traditional and Herbal Drugs, 2010, 41(4): 627-632. (in Chinese)
[15] 郑云柯, 胡翔宇, 宋希强, 王 健. 石斛属植物基因组DNA提取方法的对比[J]. 热带生物学报, 2015, 6(2): 168-172.
ZHENG Y K, HU X Y, SONG X Q, WANG J. Optimized extraction method for genomic DNA fromspecies (Orchidaceae)[J]. Journal of Tropical Biology, 2015, 6(2): 168-172. (in Chinese)
[16] 张迎辉, 杜溶讫, 凡莉莉, 杨旺利, 荣俊冬, 郑郁善, 陈礼光. 石斛属部分植物SRAP-PCR反应体系的建立与优化[J]. 热带作物学报, 2021, 42(1): 25-32.
ZHANG Y H, DU R Q, FAN L L, YANG W L, RONG J D, ZHENG Y S, CHEN L G. Establishment and optimization of SRAP-PCR reaction system in some species of[J]Chinese Journal of Tropical Crops, 2021, 42(1): 25-32. (in Chinese)
[17] 常 青, 周开亚. 分子进化研究中系统发生树的重建[J]. 生物多样性, 1998(1): 55-62.
CHANG Q, ZHOU K Y. Reconstruction of phylogenetic tree in molecular evolution[J]. Chinese Biodiversity, 1998(1): 55-62. (in Chinese)
[18] 刘 玲, 吴 睿, 牛志韬, 薛庆云, 刘 薇, 丁小余. 基于TRAP分子标记的铁皮石斛野生居群的分析与鉴别[J]. 药学学报, 2016, 51(12): 1926−1933.
LIU L, WU R, NIU Z T, XUE Q Y, LIU W, DING X Y. Molecular identification of wild populations ofbased on target region amplification polymorphism[J]. Acta Pharmaceutica Sinica, 2016, 51(12): 1926-1933. (in Chinese)
[19] 付 涛, 胡仲义, 何月秋, 李 文, 林 立. 铁皮石斛EST-SSR分子标记的开发[J]. 核农学报, 2017, 31(4): 663-670.
FU T, HU Z Y, HE Y Q, LI W, LIN L. Development of EST-SSR molecular markers in[J]. Journal of Nuclear Agricultural Sciences, 2017, 31(4): 663-670. (in Chinese)
[20] 肖文芳, 李 佐, 陈和明, 吕复兵. 基于TP-M13-SSR标记的石斛兰种质分子身份证构建[J]. 热带作物学报, 2021, 42(10): 2751-2757.
XIAO W F, LI Z, CHEN H M, LV F B. Molecular identity card establishment ofgermplasms by TP-M13-SSR[J]. Chinese Journal of Tropical Crops, 2021, 42(10): 2751-2757. (in Chinese)
[21] LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in[J]. Theoretical and Applied Genetics, 2001, 103(2/3): 455-461.
[22] 曹墨菊. 植物生物技术概论[M]. 北京: 中国农业大学出版社, 2014.
CAO M J. Introduction to plant biotechnology[M]. Beijing: China Agricultural University Press, 2014. (in Chinese)
[23] QIAO Q, YE M J, WU C, WANG J W, LIU Q Z, TAO J H, ZHANG L, FENG Z. Analysis of leaf morphology variation and genetic diversity via SRAP markers for near-threatened plant[J]. Global Ecology and Conservation, 2022, 33: e01980
[24] KATALIN S, DORU P, SABIN B A, MONICA H. Assessment of genetic diversity and population structure of the endangeredsubsp. transsilvanicus through DNA-Based molecular markers[J]. Plants, 2021, 10(12): 2732.
[25] 王挺进, 袁 璐, 刘 柯, 刘玲娟, 刘胜龙, 陈利萍, 文香英. 浙江省广西越桔组织培养及遗传多样性分析[J]. 核农学报, 2022, 36(3): 613-620.
WANG T J, YUAN L, LIU K, LIU L J, LIU S L, CHEN L P, WEN X Y. Tissue culture and genetic analysis ofin Zhejiang province[J]. Journal of Nuclear Agricultural Sciences, 2022, 36(3): 613-620. (in Chinese)
[26] 林宗铿, 张天翔, 杨俊杰. 基于SRAP的辣木种质资源遗传多样性和亲缘关系分析[J]. 热带作物学报, 2021, 42(4): 945-950.
LIN Z K, ZHANG T X, YANG J J. Assessment of genetic diversity and relationship among 18species based on SRAP marker[J]. Chinese Journal of Tropical Crops, 2021, 42(4): 945-950. (in Chinese)
[27] 林榕燕, 罗远华, 樊荣辉, 叶秀仙, 方能炎, 钟淮钦, 黄敏玲. 文心兰杂交后代的EST-SSR和SRAP分子标记鉴定[J]. 福建农业学报, 2021, 36(12): 1439-1446.
LIN R Y, LUO Y H, FAN R H, YE X X, FANG N Y, ZHONG H Q, HUANG M L. Identification of hybrid progenies fromwith EST-SSR and SRAP molecular markers[J]. Fujian Journal of Agricultural Sciences, 2021, 36(12): 1439-1446. (in Chinese)
[28] 林榕燕, 陈艺荃, 钟淮钦, 林 兵, 叶秀仙. SRAP标记在秋石斛杂交后代鉴定中的应用[J]. 江苏农业科学, 2021, 49(22): 55-59.
LIN R Y, CHEN Y Q, ZHONG H Q, LIN B, YE X X. Application of SRAP marker in identification of hybrid progeny of[J]Jiangsu Agricultural Sciences, 2021, 49(22): 55-59. (in Chinese)
[29] SHAMUSTAKIMOVA A. O, MAVLYUTOV Y. M, KLIMENKO I. A. Application of SRAP markers for DNA identification of Russian alfalfa cultivars[J]. Russian Journal of Genetics, 2021, 57(5): 540-547.
[30] 杨 贞, 蔡友铭, 张永春, 杨柳燕, 王艺程. 基于SRAP分子标记的铁皮石斛遗传多样性分析[J]. 上海农业学报, 2019, 35(5): 23-27.
YANG Z, CAI Y M, ZHANG Y C, YANG L Y, WANG Y C. Genetic diversity analysis ofbased on SRAP molecular markers[J]. Acta Agriculturae Shanghai, 2019, 35(5): 23-27. (in Chinese)
[31] 林榕燕, 叶秀仙, 钟淮钦, 林 兵, 黄敏玲. 基于SRAP分子标记的石斛兰种质资源遗传多样性分析[J]. 福建农业学报, 2018, 33(5):469-473.
LIN R Y , YE X X, ZHONG H Q, LIN B, HUANG M L. Genetic diversity ofgermplasms accessed by SRAP markers[J]. Fujian Journal of Agricultural Sciences, 2018, 33(5): 469-473. (in Chinese)
SRAP Analysis and DNA Fingerprinting of Genetic Diversity of 31 Species ofand
ZHANG Yinghui1, FAN Lili2, DU Rongqi2, XIE Dejin2, YANG Wangli3, CHEN Liguang2, ZHENG Yushan2*
1.Fujian Vocational College of Agriculture, Fuzhou, Fujian 350007, China; 2. College of Forestry, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 3. Forestry Bureau of Fu’an City, Fu’an, Fujian 355000, China
Sw. belongs to the Orchidaceae (Subfam. Orchidoideae), which has a wide distribution and application in China. Most are precious Chinese medicinal materials and ornamental plants with high economic values. Due to harsh growth conditions and excessive development and utilization, wildresources have been severely damaged, and most species have been nearly depleted, which also lead to problems such as mixed germplasm ofin the market and difficulties in authenticity identification. The research on the genetic diversity ofplants and the establishment of digital fingerprints can provide a theoretical basis for the identification and classification ofspecies, the selection, and protection of fine varieties, and it is also of great significance to protect the biodiversity ofresources. Taking 31 species ofandas the research objects, the genetic diversity was analyzed by the SRAP-PCR molecular marker technology and DNA fingerprints were constructed. 15 pairs of primers were used for amplification, and a total of 264 loci were amplified, including 251 polymorphic loci, and an average of 16.73 polymorphic loci with a 95.08% ratio were amplified for each pair of primers. The polymorphism percentage of 5 pairs of primers including Me2-Em5, Me4-Em3, Me4-Em5, Me7-Em3, Me7-Ee7 was 100%, and the primer combinations had higher identification efficiency forspecies. After calculation, the number of observed alleles (a) between 31 species ofandwas 1.784, the number of effective alleles (e) was 1.430, the Nei’s gene diversity index () was 0.202, and Shannon’s information index () was 0.386, which showed there was a high degree of genetic variability and rich genetic diversity amongspecies. The variation range of the genetic similarity coefficient was 0.591–0.851, and the genetic relationship was relatively close. When the genetic similarity coefficient was 0.66,could be clustered into one group. When the genetic similarity coefficient was 0.724, 31species could be divided into 10 groups. The DNA fingerprints constructed by three pairs of primers, including Me1-Em3, Me3-Em3, and Me1-Em6, could identify 31 species ofandindependently, providing a scientific basis for the analysis of the genetic background and the rapid identification ofspecies.
; SRAP markers; genetic diversity; DNA fingerprint
S567.239
A
10.3969/j.issn.1000-2561.2022.10.008
2022-02-16;
2022-03-22
福建省科技创新平台项目(No. 2008Y2001);福建农林大学科技创新专项基金项目(No. CXZX2016043)。
张迎辉(1979—),女,博士,副教授,研究方向:园林植物栽培与应用。*通信作者(Corresponding author):郑郁善(ZHENG Yushan),E-mail:zys1960@163.com。

