甘蓝型油菜SUMO蛋白家族成员鉴定及Bna.SUMO1.C08基因的功能研究
陈 慧 肖 清 汪华栋 文 静 马朝芝 涂金星 沈金雄 傅廷栋 易 斌
甘蓝型油菜SUMO蛋白家族成员鉴定及基因的功能研究
陈 慧 肖 清 汪华栋 文 静 马朝芝 涂金星 沈金雄 傅廷栋 易 斌*
华中农业大学 / 作物遗传改良全国重点实验室 / 国家油菜工程技术研究中心, 湖北武汉 430070
蛋白翻译后修饰对蛋白的功能非常重要。SUMO化修饰就是一种非常重要的蛋白翻译后修饰, 它对植物生长发育的关键过程有很大的影响。甘蓝型油菜作为重要的油料和经济作物在SUMO化修饰方面却鲜有报道。为弥补这一空白, 本研究对甘蓝型油菜中的SUMO化修饰进行了探究。首先通过生物信息学方法在甘蓝型油菜中鉴定到31个SUMO蛋白成员, 分为3类: “典型”群组、“非典型”群组和SUMO-V。然后对甘蓝型油菜中基因的同源基因进行表达模式分析, 发现该基因在根、叶和角果中表达比较高。亚细胞定位结果发现, Bna.SUMO1.C08蛋白定位于细胞核和内质网中。最后在甘蓝型油菜中过表达基因发现其能够增强植株对PEG胁迫的抵抗能力。本研究为后续甘蓝型油菜中SUMO化修饰的研究奠定了一定的基础。
甘蓝型油菜; SUMO化修饰;
类泛素小蛋白修饰物(small ubiquitin-like modifier, SUMO)对真核细胞行使正常功能至关重要。SUMO蛋白是一个长度为100~115个氨基酸的小分子多肽, 包含一个β-grasp fold结构域, 在氨基酸序列上与泛素相似性非常低, 仅为18%, 但在三维结构上却与泛素比较相似, 不同的地方主要在于SUMO蛋白在羧基末端存在几十个氨基酸的延伸[1]。SUMO蛋白可以分为“典型”组和“非典型”组。在典型类群中的SUMO蛋白比较保守, 然而在非典型类群中的SUMO蛋白不仅与典型类群中的SUMO蛋白在氨基酸序列上相似性低而且它们自己之间氨基酸序列相似性也比较低[2]。除上述的SUMO蛋白外, 还存在2种较长的SUMO相关蛋白, 一种是SUMO-V蛋白, 另一种是DUSL蛋白。SUMO-V蛋白与常规SUMO蛋白相比在其β-grasp fold结构域的N段延伸了126~212个氨基酸残基, DUSL蛋白则包含2个β-grasp fold结构域[2]。SUMO蛋白对底物进行SUMO化修饰, 可以改变底物蛋白酶的活性, 促进底物蛋白形成复合物以及改变底物蛋白的亚细胞定位等[3]。大量研究表明SUMO化修饰在控制植物生长、发育、开花、环境胁迫和抵抗病原体侵染等重要生物学过程中起着非常重要的作用。例如: 拟南芥中双突变体和以及单突变体、和都表现出胚胎致死的表型[4]。基因可以通过调控SA和IAA介导的过程来影响细胞的生长和发育[5-6]。Son等[7]在拟南芥的研究发现介导的开花抑制受到SUMO化修饰的调控, 当把FLC的SUMO修饰位点(K154)突变后, 表现出与野生型相似的开花时间。Li对[8]苹果的研究发现SUMO化修饰会影响植株对干旱的耐受。在高温条件下, SUMO化修饰能够稳定DREB2A蛋白增强拟南芥植株的耐热性[9]。SUMO化修饰对铝胁迫也有一定的调控[10]。SUMO化修饰也能通过确保MAPK对底物蛋白的可选择性来调控植物的免疫能力[11]。尽管SUMO化修饰在植物中有着多方面的研究, 但在甘蓝型油菜中对SUMO化修饰却鲜有报道。本研究对甘蓝型油菜SUMO蛋白的鉴定以及对的功能研究, 将为后续甘蓝型油菜中SUMO化修饰的研究提供理论基础。
1 材料与方法
1.1 甘蓝型油菜SUMO蛋白家族成员鉴定
从甘蓝型油菜基因组网站(http://cbi.hzau.edu. cn/bnapus/)下载甘蓝型油菜“ZS11”的基因组序列文件和注释文件。利用Tbtools[12]将拟南芥中鉴定到的SUMO成员的蛋白序列比对到ZS11基因组上筛选出一些候选基因。然后提取这些基因的蛋白序列提交到NCBI (https://www.ncbi.nlm.nih.gov/)数据库中, 利用Blastp做进一步筛选, 在结果中剔除注释不是SUMO蛋白家族的成员。最后将剩余成员的蛋白序列提交到NCBI保守结构域数据库CDD (https:// www.ncbi.nlm.nih.gov/Structure/cdd)中[13], 检查SUMO蛋白保守结构域的完整性, 删除没有完整结构域的成员, 剩余成员即为甘蓝型油菜SUMO蛋白家族成员。
1.2 序列分析
利用在线网站ExPASy (https://www.expasy. org/)[14]和WoLFPSORT (http://www.genscript.com/ tools/wolf-psort)[15]分别对甘蓝型油菜SUMO蛋白成员的分子量和等电点进行计算以及对其亚细胞定位进行预测。利用软件MEGA7.0对甘蓝型油菜SUMO蛋白成员的蛋白序列进行多序列比对, 并使用邻接法构建进化树; Bootstrap设为1000, 其他均为默认参数[16]。利用在线网站Evolview (https://www. evolgenius.info/evolview/)对进化树美化[17]。利用在线工具MEME (https://meme-suite.org/meme/tools/ meme)鉴定甘蓝型油菜SUMO蛋白家族成员的保守基序[18]。将甘蓝型油菜SUMO蛋白家族成员的保守基序和基因组注释文件提交到Tbtools中进行可视化。
1.3 共线性分析
利用TBtools内置的MsScanX软件对拟南芥、甘蓝、白菜以及甘蓝型油菜进行物种内及物种间共线性分析, 并可视化。
1.4 基因表达模式分析和亚细胞定位
根据基因的CDS序列, 在SnapGene上设计荧光定量PCR引物。应用PerfectStart Green qPCR SuperMix试剂(Cat. No. AQ601, 购置于全式金生物技术有限公司), Bio-Rad CFX96荧光定量PCR仪(Bio-Rad, 美国), 以植物各组织(根、茎、叶、花和角果)的cDNA样品为模板进行基因相对表达量分析。
将基因的开放阅读框构建至PMDC83-GFP的载体上, 然后转化农杆菌GV3101。通过农杆菌介导的烟草瞬时转化, 分别与接有红色荧光标签的细胞核Marker和内质网Marker共转入本氏烟草中, 将烟草培养2 d, 用激光共聚焦显微镜观察结果。
基因定量引物:F: 5¢-GTCAGGATGGGAATGAGGTG-3¢;R: 5¢-GAACAAGAAGGCAATGGAGC-3¢。
1.5 植物材料的培养与各种处理
RR材料为甘蓝型油菜波里马细胞质雄性不育恢复系材料, 由华中农业大学油菜工程中心提供。随后将拟南芥基因在甘蓝型油菜中的同源基因的编码区连接于超表达载体PMDC83上, 通过遗传转化甘蓝型油菜RR, 后续对转基因植株中基因的表达量进行检测, 表达量中等的植株用于后续试验。
挑选饱满RR材料的种子和以RR为受体材料的转基因种子, 将这些种子浸水发芽, 等种子都露白后开始处理, 将RR的种子和转基因的种子都分别播种于试验组和对照组的培养盒中进行水培, 对照组中加入水培营养液, 试验组中加入用水培营养液配置的20% PEG溶液, 1周后测定表型。各组别都设置3次重复, 生长条件为: 温度22℃, 16 h光照/8 h黑暗。
1.6 活性氧检测与SOD和POD酶活性检测
分别采用二氨基联苯胺(diaminobenzidiine, DAB)和氯化硝基四氮唑蓝(nitrotetrazolium blue chloride, NBT)组织染色对过表达植株和野生型植株叶片中H2O2和O2܋进行检测。
利用超氧化物歧化酶(SOD)活性检测试剂盒(Cat. No.AKAO001C)和过氧化物酶(POD)活性检测试剂盒(Cat. No.AKAO005C)检测过表达植株和野生型植株中SOD和POD酶活性(试剂盒购置于北京盒子生工科技有限公司)。
2 结果与分析
2.1 甘蓝型油菜SUMO蛋白家族成员鉴定
以拟南芥中SUMO蛋白的蛋白序列为参考, 在甘蓝型油菜中鉴定到了31个SUMO蛋白(具体成员信息详见附表1)。把这31个成员与拟南芥、水稻和玉米中的SUMO蛋白构建进化树, 甘蓝型油菜SUMO蛋白分为3类(图1-A)。Group 1是与拟南芥聚成一类的“典型”群组, 甘蓝型油菜中有14个成员属于这一个群组。Group 2是与Group 1相对的“非典型”群组, “非典型”群组里面包含了15个甘蓝型油菜SUMO蛋白成员。Group 3是SUMO-V, 在甘蓝型油菜中只鉴定到2个成员。
进一步对31个SUMO蛋白进行基因结构分析和保守基序分析(图1-B)。在甘蓝型油菜SUMO蛋白的氨基酸序列中共鉴定到5个保守基序(Motif 1~Motif 5), 其中Motif 1~Motif 3在大多数SUMO蛋白成员中是保守的, 表明这3个Motif可能构成SUMO蛋白的核心结构域。而Motif 4主要存在于“典型”群组中, Motif 5主要存在于“非典型”群组中, 表明典型群组成员和非典型群组成员在功能上有一定的分化。基因结构分析发现典型群组中SUMO蛋白成员基因结构较为相似, 其内含子和外显子数目主要分别为2个和3个, 非典型群组中SUMO蛋白成员的基因结构存在较大的差异, 其内含子和外显子数目主要分别介于0~10个之间和1~11个之间, SUMO-V的基因结构比较复杂, 内含子和外显子个数都超过5个。

(图1)
A: 系统进化树分析。红色标注的Group 1为“典型”群组; 蓝色标注的Group 2为“非典型”群组; 紫色标注的Group 3为SUMO相关蛋白。B: Motif、Domain和基因结构分析。第1列为Motif; 第2列为Domain; 第3列为基因结构。C: 蛋白序列比对。红色长方体标注为β-grasp domain范围; 圆点标注为各作用位点。
A: phylogenetic tree analysis. Group 1 in red is a “canonica” group. Group 2 in blue is a “non-canonica” group. Group 3 in purple is a sumo-related protein. B: the analyses of motif, domain, and gene structure. The first column is Motif; the second column is Domain; the third section is genetic structure. C: protein sequence alignment. The red cuboid is labeled as β-grasp domain range; dots are action sites.
将甘蓝型油菜SUMO蛋白成员与酵母ScSmt3蛋白、人类HsSUMO2蛋白和拟南芥AtSUMO1蛋白进行蛋白序列比对发现, β-grasp domain在甘蓝型油菜SUMO蛋白成员中是比较保守的, 并且在此结构域中包含许多E1激活酶作用位点、E2结合酶作用位点和SUMO化修饰位点, 同时也发现有些SUMO蛋白不存在对底物行使功能的双甘氨酸残基, 说明这些SUMO蛋白可能不具有对底物进行SUMO化修饰的功能(图1-C)。
2.2 甘蓝型油菜SUMO蛋白基因的染色体分布及共线性分析
为了比较直观的观察SUMO蛋白基因在染色体上的分布情况, 本研究将基因在染色体的位置进行可视化(图2-A)。SUMO蛋白基因分布于甘蓝型油菜19条染色体上, 且分布较为均匀, 每条染色体上分布的SUMO蛋白基因个数介于1~4个之间, scaffoldC04染色体上存在个数最多。同时本研究也发现同一类别的SUMO蛋白基因存在不在某一染色体区间聚集的现象。多个基因定位在一条染色体上时, 其定位的位置是不均匀的, 多数情况下这些基因都是定位于染色体的两端。
进一步对拟南芥、白菜、甘蓝和甘蓝型油菜进行了物种内共线性分析和物种间共线性分析(图2-B)。在拟南芥、白菜、甘蓝和甘蓝型油菜中分别鉴定到1、12、8和47对旁系同源基因对, 在拟南芥和甘蓝型油菜、甘蓝和甘蓝型油菜以及白菜和甘蓝型油菜之间分别鉴定到18、78和40对直系同源基因对。同时对甘蓝、白菜和甘蓝油菜SUMO蛋白基因保留率分析发现, 在进化过程中“典型”群组的SUMO蛋白都被100%保留下来, 然而“非典型”群组中的SUMO蛋白存在部分丢失。
2.3 Bna.SUMO1.C08表达模式分析及亚细胞定位
拟南芥中基因对植物的生长发育起着非常重要的作用, 为了在甘蓝型油菜中进一步研究基因, 本研究将拟南芥的基因蛋白序列比对到甘蓝型油菜ZS11基因组上鉴定到了同源性最高的基因。并对基因的表达模式进行了分析。试验结果表明基因在根、叶和角果中都大量表达, 其中在叶中该基因的表达量最高, 然而在茎和花中该基因的表达量比较低(图3-A)。蛋白的亚细胞定位能够显示出蛋白的作用位置, 这对解析基因功能有一定的帮助。因此, 进一步对基因进行亚细胞定位分析发现,基因接的绿色荧光标签散发的绿色荧光能和细胞核Marker与内质网Marker连接的红色荧光标签散发的红色荧光重合, 这表明基因定位于细胞核和内质网中(图3-B)。
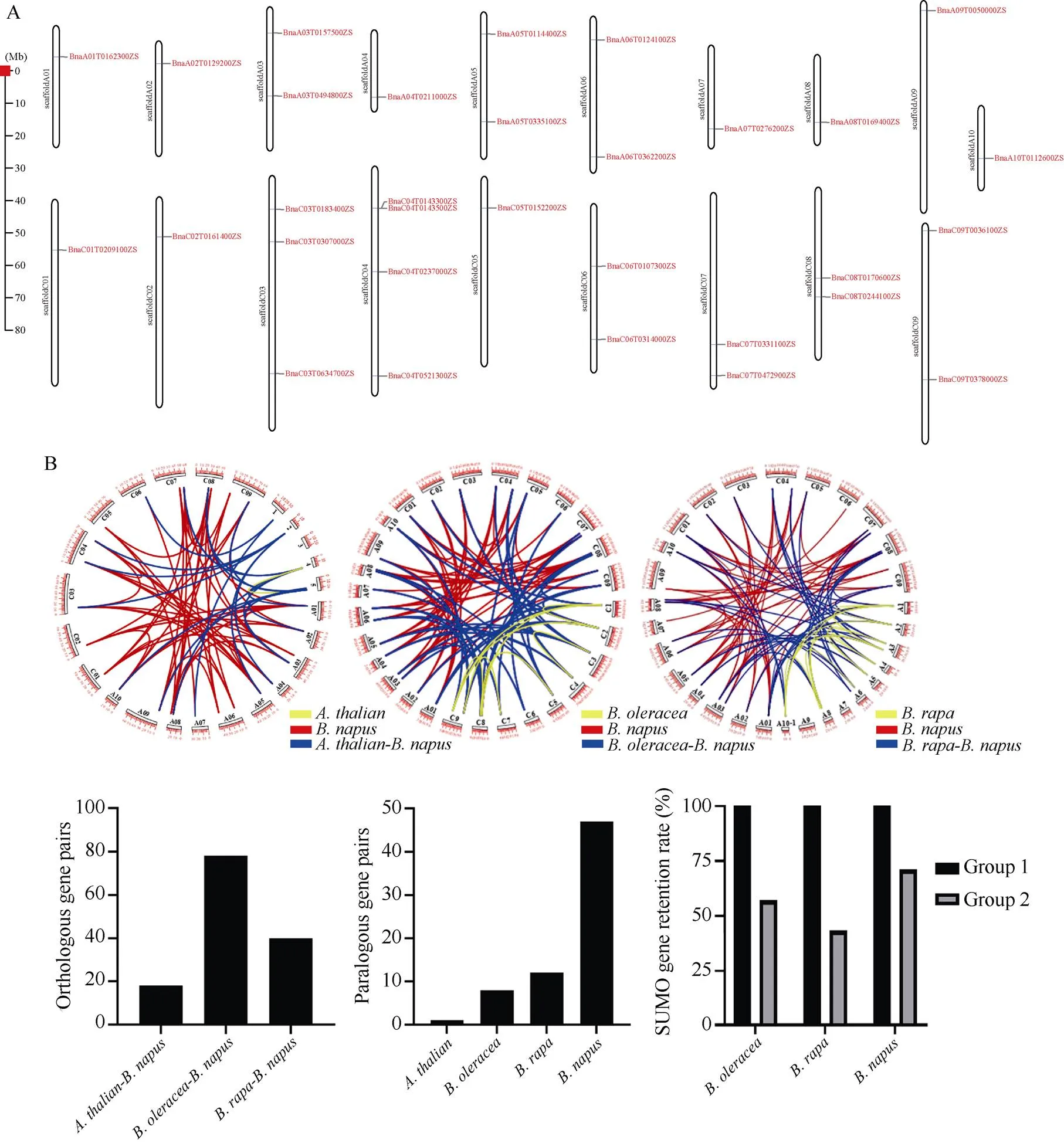
图2 甘蓝型油菜SUMO蛋白基因染色体定位和共线性分析
A: SUMO蛋白基因染色体位置可视化。B: 拟南芥、白菜、甘蓝和甘蓝型油菜物种物种内和物种间共线性分析。
A: the chromosome location visualization of SUMO protein genes. B: the intra-species and interspecies collinearity analysis of,,, and.

图3 Bna.SUMO1.C08表达模式分析和亚细胞定位
A: 表达模式分析。主要在根、叶和角果中表达。B: 亚细胞定位。定位于细胞核和内质网中。标尺: 1 μm。
A: the relative expression pattern.is mainly expressed in roots, leaves, and scones. B: the subcellular localization.locates in the nucleus and endoplasmic reticulum. Bar: 1 μm.
2.4 转基因甘蓝型油菜的分子鉴定
为进一步分析基因在甘蓝型油菜中的基因功能, 本研究将基因的编码区连接到过表达载体PMDC83上(图4-A), 通过遗传转化甘蓝型油菜RR。提取野生型植株和过表达植株RNA进行qRT-PCR, 过表达植株中基因表达量明显高于野生型(图4-B)。本研究挑选表达量接近的3个株系#30、#35和#36进行后续的试验。
2.5 过表达Bna.SUMO1.C08植株在PEG处理下的表型
根据前人研究表明SUMO化修饰能够调控植物的抗旱性。同时对基因ATG上游2000 bp的启动子用plantCARE进行顺势作用元件预测发现存在响应干旱胁迫的元件, 所以本研究对过表达植株和野生型植株用PEG进行模拟干旱处理。通过前期的预试验, 本研究将PEG处理的浓度定为PEG︰水的质量体积比为20%。根系的强弱对植物的抗旱性有着很大的影响, 本研究着重对根系长度进行考察, 对野生型植株和过表达植株在正常条件和20% PEG处理下观察其根系长度并进行根长统计, 结果发现在正常条件这2种材料的根系长度无明显差异, 在20% PEG处理下这2种材料的根系长度都比正常条件下生长的短, 并且这两种材料的根系长度之间存在明显的差异, 过表达基因的植株根系长度显著长于野生型植株(图5-A, B)。

图4 过表达Bna.SUMO1.C08的转基因植株在mRNA水平上的鉴定
A: 过表达基因的载体结构图。B: 过表达植株的表达量检测。
A: the vector structure diagram ofoverexpressed genes. B: the relative expression levels ofin overexpressed plants.
干旱胁迫很多时候会诱导植物体内活性氧大爆发, 清除多余的活性氧在一定程度上能够提升植物的抗旱性。为探究野生型植株和过表达植株中活性氧的含量, 本研究利用二氨基联苯胺(diaminobenzidiine,DAB)和氯化硝基四氮唑蓝(nitrotetrazolium blue chloride, NBT)对2种材料的叶片进行染色, 分别对2种材料中的H2O2和O2܋的含量进行检测。染色结果发现, 无论是在正常条件还是在20% PEG条件下过表达植株的2种染色程度都比野生型植株浅, 在20% PEG处理下的结果更加明显。这表明过表达植株中H2O2和O2܋的含量比野生型植株中低(图5-C, D)。为探究过表达植株叶片内H2O2和O2܋的含量为什么比较低, 本研究对过氧化物歧化酶(SOD)和过氧化物酶(POD)的酶活进行检测。研究结果表明在正常条件下POD和SOD酶的活性没有明显的差异, 但在20% PEG处理下POD和SOD酶的活性在过表达植株中相较于野生型植株有呈显著的提升(图5-E, F)。

图5 PEG处理下过表达Bna.SUMO1.C08植株和野生型植株表型
A, B: 根长表型及统计。C, D: DAB染色和NBT染色。E, F: POD酶和SOD酶活性检测。*< 0.05; **< 0.01; ***< 0.001。
A, B: the phenotype and statistics of root length. C, D: DAB staining and NBT staining. E, F: the enzyme activity detection of POD and SOD. *:< 0.05; **:< 0.01; ***:< 0.001.
3 讨论
有研究表明白菜和甘蓝都属于芸薹属物种, 它们与拟南芥共同的祖先在大约2000万年前开始分化, 随后在大约在1600万年前, 它们的全基因组经历了一个三倍化的过程[19]。按照这个理论, 一个拟南芥基因在白菜和甘蓝中应该存在3个同源基因。而甘蓝型油菜是白菜和甘蓝杂交后进行染色体加倍的产物[20], 同理, 一个拟南芥基因在甘蓝型油菜中应该存在6个同源基因。实际上利用拟南芥的SUMO蛋白通过蛋白同源比对在白菜、甘蓝和甘蓝型油菜中分别鉴定到11个、12个和32个成员(图2), 都比理论上的少, 说明在整个进化过程中SUMO蛋白家族成员存在一定的丢失。本研究鉴定到的31 SUMO蛋白成员可分为3组: “典型”群组、“非典型”群组和SUMO相关蛋白(图1), 这与拟南芥、玉米和大豆等植物中对SUMO蛋白的分类相同[2,21-22]。对“典型”群组成员和“非典型”群组成员的基因保留率分析发现“典型”群组成员在进化过程中被100%保留并且还在进一步扩大, 然而有些“非典型”群组成员却在进化过程中被丢失(图2)。推测在进化过程中SUMO蛋白成员经历了强烈的选择, 而“典型”群组成员能够100%保留, 说明“典型”群组成员在植物的整个生长发育起着非常重要的功能。
干旱胁迫对植物的损害很大一部分是由于干旱诱导产生的ROS打破了植物体内原有ROS平衡, 过量的ROS会对植物细胞产生毒害作用[23]。有研究表明清除干旱胁迫诱导产生的多余ROS, 将在一定程度上增强植物的耐旱性[24]。植物主要通过酶促防御系统来清除干旱诱导产生的ROS[25-26]。为探究过表达基因能够增加甘蓝型油菜抗旱性的原因, 本研究对植株内的ROS和抗氧化酶系统中的2种酶(SOD和POD)的活性进行了检测, 发现在过表达基因的甘蓝型油菜中2种酶的活性都显著上升, 并且ROS的含量比较低(图5)。这说明过表达基因的甘蓝型油菜也可能通过清除植株体内ROS的含量来提升抗旱性。
4 结论
甘蓝型油菜中存在31个SUMO蛋白成员, 分为3组: “典型”群组、“非典型”组和SUMO相关蛋白, 其中典型组成员在进化过程中100%被保留, 可能在植物生长发育过程具有重要大功能。之后对典型组成员的功能进行研究, 表达模式分析发现其主要在根、叶和角果中表达。亚细胞定位结果显示其定位于细胞核和内质网中, 该基因在甘蓝型油菜中过表达发现能够清除活性氧来增强植株的抗旱性。
[1] Vierstra R D. The expanding universe of ubiquitin and ubiquitin-like uodifiers., 2012, 160: 2–14.
[2] Augustine R C, York S L, Rytz T C, Vierstra R D. Defining the SUMO system in maize: SUMOylation is up-regulated during endosperm development and rapidly induced by stress., 2016, 171: 2191–2210.
[3] Augustine R C, Vierstra R D. SUMOylation: re-wiring the plant nucleus during stress and development., 2018, 45: 143–154.
[4] Saracco S A, Miller M J, Kurepa J, Vierstra R D. Genetic analysis of SUMOylation in: conjugation of SUMO1 and SUMO2 to nuclear proteins is essential., 2007, 145: 119–134.
[5] Miura K, Lee J, Miura T, Hasegawa P M. SIZ1 controls cell growth and plant development inthrough salicylic acid., 2010, 51: 103–113.
[6] Miura K, Lee J, Gong Q, Ma S, Jin J B, Yoo C Y, Miura T, Sato A, Bohnert H J, Hasegawa P M. SIZ1 regulation of phosphate starvation-induced root architecture remodeling involves the control of auxin accumulation., 2011, 155: 1000–1012.
[7] Son G H, Park B S, Song J T, Seo H S. FLC-mediated flowering repression is positively regulated by sumoylation., 2014, 65: 339–351.
[8] Li X, Zhou S, Liu Z, Lu L, Dang H, Li H, Chu B, Chen P, Ma Z, Zhao S, Li Z, Nocker S, Ma F, Guan Q. Fine-tuning of SUMOylation modulates drought tolerance of apple., 2022, 20: 903–919.
[9] Wang F, Liu Y, Shi Y, Han D, Wu Y, Ye W, Yang H, Li G, Cui F, Wan S, Lai J, Yang C. SUMOylation stabilizes the transcription factor DREB2A to improve plant thermotolerance., 2020, 183: 41–50.
[10] Fang Q, Zhang J, Zhang Y, Fan N, van den Burg H A, Huang C. Regulation of aluminum resistance ininvolves the SUMOylation of the zinc finger transcription factor STOP1., 2020, 32: 3921–3938.
[11] Verma V, Srivastava A K, Gough C, Campanaro A, Srivastava M, Morrell R, Joyce J, Bailey M, Zhang C, Krysan P J, Sadanandom A. SUMO enables substrate selectivity by mitogen-activated protein kinases to regulate immunity in plants., 2021, 118: e2021351118.
[12] Chen C, Chen H, Zhang Y, Thomas H R, Frank M H, He Y, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020, 13: 1194–1202.
[13] Marchler-Bauer A, Bryant S H. CD-search: protein domain annotations on the fly., 2004, 32: 327–331.
[14] Gasteiger E. ExPASy: the proteomics server for in-depth protein knowledge and analysis., 2003, 31: 3784–3788.
[15] Horton P, Park K J, Obayashi T, Fujita N, Harada H, Adams- Collier C J, Nakai K. WoLF PSORT: protein localization predictor., 2007, 35: W585–W587.
[16] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets., 2016, 33: 1870–1874.
[17] Zhang H, Gao S, Lercher M J, Hu S, Chen W. EvolView, an online tool for visualizing, annotating and managing phylogenetic trees., 2012, 40: W569–W572.
[18] Bailey T L, Boden M, Buske F A, Frith M, Grant C E, Clementi L, Ren J, Li W W, Noble W S. MEME Suite: tools for motif discovery and searching., 2009, 37: W202–W208.
[19] Yu J, Tehrim S, Zhang F, Tong C, Huang J, Cheng X, Dong C, Zhou Y, Qin R, Hua W, Liu S. Genome-wide comparative analysis of NBS-encoding genes betweenspecies and., 2014, 15: 3.
[20] Cheng F, Sun R, Hou X, Zheng H, Zhang F, Zhang Y, Liu B, Liang J, Zhuang M, Liu Y, Liu D, Wang X, Li P, Liu Y, Lin K, Bucher J, Zhang N, Wang Y, Wang H, Deng J, Liao Y, Wei K, Zhang X, Fu L, Hu Y, Liu J, Cai C, Zhang S, Zhang S, Li F, Zhang H, Zhang J, Guo N, Liu Z, Liu J, Sun C, Ma Y, Zhang H, Cui Y, Freeling M R, Borm T, Bonnema G, Wu J, Wang X. Subgenome parallel selection is associated with morphotype diversification and convergent crop domestication inand., 2016, 48: 1218–1224.
[21] Kurepa J, Walker J M, Smalle J, Gosink M M, Davis S J, Durham T L, Sung D, Vierstra R D. The small ubiquitin-like modifier (SUMO) protein modification system in., 2003, 278: 6862–6872.
[22] Li Y, Wang G, Xu Z, Li J, Sun M, Guo J, Ji W. Organization and regulation of soybean SUMOylation system under abiotic stress conditions., 2017, 8: 1458.
[23] Foyer C H, Noctor G. Redox regulation in photosynthetic organisms: signaling, acclimation, and practical implications., 2009, 11: 861–905.
[24] Sun X, Wang P, Jia X, Huo L, Che R, Ma F. Improvement of drought tolerance by overexpressingis mediated by modified antioxidant system and activated autophagy in transgenic apple., 2018, 16: 545–557.
[25] Nadarajah K K. ROS homeostasis in abiotic stress tolerance in plants., 2020, 21: 5208.
[26] You J, Chan Z. ROS regulation during abiotic stress responses in crop plants., 2015, 6: 1092.
附表1 甘蓝型油菜SUMO蛋白家族成员基本信息表
Table S1 Basic information of SUMO protein family members in

基因名Gene name序列号Sequence number基因长度Gene length (bp)蛋白长度Protein length (aa)分子量Molecular weight (Da)等电点Isoelectric point Bna.SUMO1.C08BnaC08T0170600ZS30410111,083.394.91 Bna.SUMO1.A08BnaA08T0169400ZS30410111,124.434.91 Bna.SUMO1.C01BnaC01T0209100ZS31610511,589.984.93 Bna.SUMO1.A03BnaA03T0494800ZS30410111,123.504.91 Bna.SUMO1.A01BnaA01T0162300ZS30310011,120.404.93 Bna.SUMO1.C07BnaC07T0472900ZS2959810,889.234.91 Bna.SUMO1.A05BnaA05T0335100ZS2598610,309.8610.61 Bna.SUMO1.C04BnaC04T0237000ZS256859964.399.18 Bna.SUMO1.C06BnaC06T0107300ZS211708117.079.25 Bna.SUMO1.C03BnaC03T0634700ZS211708158.229.73 Bna.SUMO2.C09BnaC09T0378000ZS31310411,668.174.93 Bna.SUMO2.A10BnaA10T0112600ZS31310411,716.224.93 Bna.SUMO2.A02BnaA02T0129200ZS2959811,017.384.93 Bna.SUMO2.C02BnaC02T0161400ZS48116018,387.545.26 Bna.SUMO3.C03BnaC03T0307000ZS32810912,774.638.69 Bna.SUMO4.C07BnaC07T0331100ZS121340446,871.139.15 Bna.SUMO4.A09BnaA09T0050000ZS244819306.375.26 Bna.SUMO5.A04BnaA04T0211000ZS30410111,358.089.55 Bna.SUMO5.C03BnaC03T0183400ZS36412113,485.519.57 Bna.SUMO5.C04bBnaC04T0521300ZS30410111,358.089.55 Bna.SUMO5.A05BnaA05T0114400ZS30710211,257.989.33 Bna.SUMO5.A03BnaA03T0157500ZS166556198.189.52 Bna.SUMO5.A06BnaA06T0124100ZS32510812,451.378.66 Bna.SUMO5.C04cBnaC04T0143500ZS2959810,885.659.61 Bna.SUMO5.C05BnaC05T0152200ZS31310411,998.788.69 Bna.SUMO5.C08BnaC08T0244100ZS2929711,140.705.04 Bna.SUMO5.C04aBnaC04T0143300ZS30710211,279.039.73 Bna.SUMO6.A06BnaA06T0362200ZS119239745,931.676.45 Bna.SUMO6.C09BnaC09T0036100ZS2719010,505.945.12 Bna.SUM-V.C06BnaC06T0314000ZS104234738,854.965.23 Bna.SUM-V.A07BnaA07T0276200ZS72424127,254.875.32
Identification of SUMO protein family members and functional study ofgene in
CHEN Hui, XIAO Qin, WANG Hua-Dong, WEN Jing, MA Chao-Zhi, TU Jin-Xing, SHEN Jin-Xiong, FU Ting-Dong, and YI Bin*
National Key Laboratory of Crop Genetic Improvement / National Engineering Research Center for Rapeseed / Huazhong Agricultural University, Wuhan 430070, Hubei, China
Post-translational modifications are very important for protein function. SUMOylation is a very important post-translational modification, which has great influence on the key processes of plant growth and development. As an important oil and economic crops, thereis few studies on SUMO modification in. To remedy this gap, SUMOylation inwas investigated in this study. Firstly, 31 SUMO proteins were identified inby bioinformatics methods, which were divided into three groups (“canonical” SUMO, “non-canonical” SUMO, and SUMO-V). Secondly, the relative expression pattern indicated that,, a homologous gene of, was highly expressed in roots, leaves, and silique. Subcellular localization revealed that Bna.SUMO1.C08 was localized in the nucleus and endoplasmic reticulum. Finally,was overexpressed inandcould enhance plant resistance to PEG stress. This study laid a foundation for the subsequent research on SUMO modification in.
; SUMOylation;
10.3724/SP.J.1006.2023.24064
本研究由国家重点研发计划项目(2016YFD01008)资助。
This study was supported by the National Key Research and Development Program of China (2016YFD01008).
易斌, E-mail: yibin@mail.hzau.edu.cn
E-mail: 15661644378@163.com
2022-03-22;
2022-07-21;
2022-08-12.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220812.0910.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

