基于机器学习的分子动力轨迹预测
焦 嘉
(湖南信息职业技术学院 湖南 长沙 410203)
0 引言
分子动力模拟是一种计算分子和原子在相空间模拟运动的轨迹的计算机模拟方法。该方法主要利用计算机来计算模拟分子和原子在某段时间内运动结构变化[1],目前分子动力模拟发展已经比较成熟,能研究具有不同内部自由度的多原子分子、单原子分子,现在也已经成功运用在研究蛋白质、DNA等生物大分子的研究中[2-3]。当在各种条件下发生构象变化时,蛋白质构象在影响其功能中起着重要作用。分子动力学(molecular dynamics,MD)轨迹模拟可以提供蛋白质运动的结构数据,这在蛋白质功能中起着非常重要的作用。
现在比较流行的研究分子动力结构预测的方法是主成分分析(principal component analysis,PCA),在预测蛋白质结构中的用途,从而将前两个(最高)主成分(PC)的线性组合用于生成新结构。与自动编码器相比,对于更刚性的蛋白质,它的性能很好,但对于更柔性的蛋白质,效果不佳[4-6]。本文提出的方法主要就是解决对于柔性蛋白质的预测问题。
1 分子动力模拟结构的分类
本次实验采用可视化分子动力学(visual molecular dynamics,VMD)软件[7],该软件能可视化呈现蛋白质的结构以及对不同轨迹的模拟分析。VMD显示两个轨迹图像的总体视图(见图1),为了显示构象差异,还绘制了两个独立轨迹的RMSD图。
在图1可视化了3D空间中的所有原子点后,根据绘制数据集在空间中的分布大致了解目标数据集的分布情况,确定数据集的分布,并根据数据的特征选择合适的SVM分类器模型。
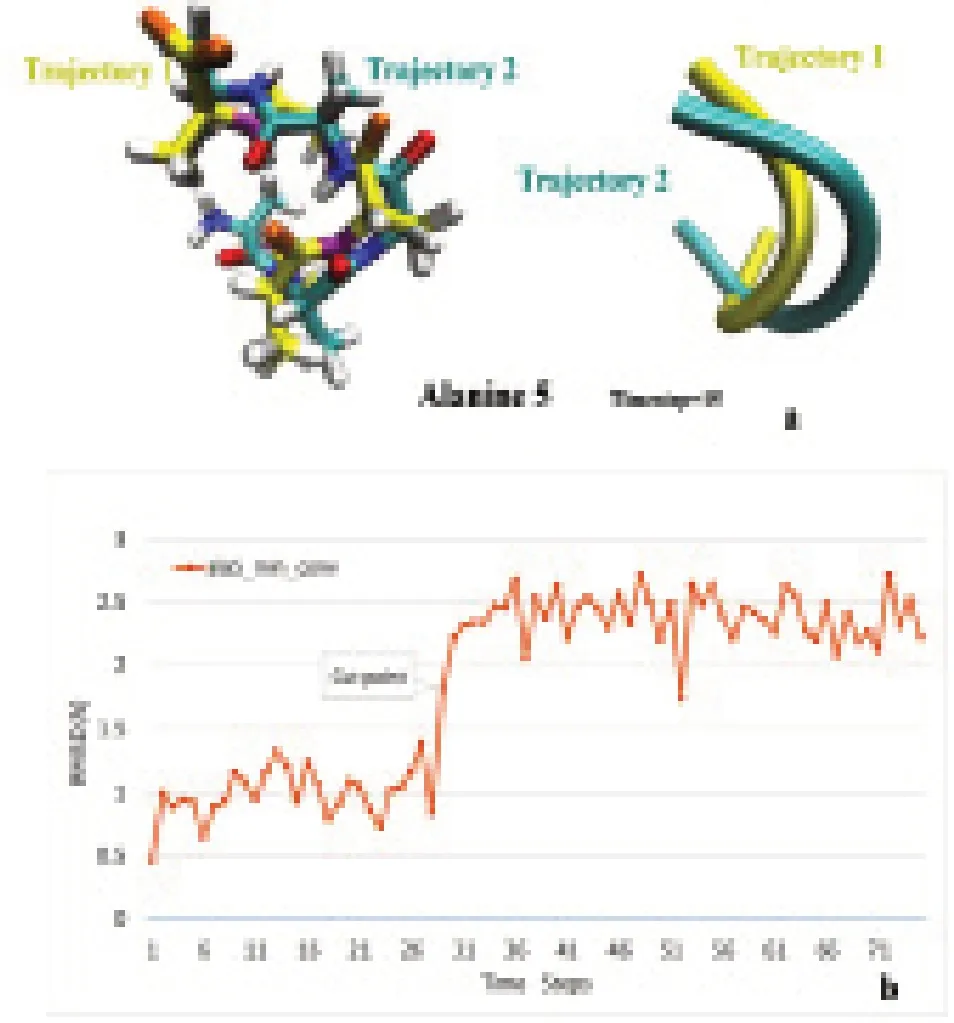
图1 丙氨酸5的不同结构可视化示意图和RMSD示意图
2 分子动力模拟结构的异常检测
2.1 一类支持向量机
在异常检测的任务中,比较常用的就是一类支持向量机(one class support vector machine)[8-9],这个算法的原理就是把所有的训练数据训练出一个最小的超球面,这个超球面就尽量把所有训练数据都“包”起来,当识别新的点的时候,如果落在超球面上,那么就是属于这个类,反之则不是属于这个类别。选择One Class SVM(sklearn中的函数)的默认值RBF内核,以及gamma和nu超参数的默认值。另外,在这个实验中,使用了Grid Search[10]来找到该模型的最佳参数对,并通过使用最佳组合超参数执行一类支持向量机的算法。图2是一类支持向量机的原理图,图中黑点表示数据集,可以看到a、b两个点离超球面的距离非常远,所以这个算法把这两个点视为异常点。

图2 一类支持向量机原理示意图
2.2 孤立森林
我们使用了另一种异常检测方法来区分构象采样中的差异——使用网格搜索通过准确性分数来搜索孤立森林(isolation forest)[11]的最佳组合。为了通过操纵isolation forest找出对性能的不同影响,还对每个超参数使用所有默认值对其进行训练,并获得在RMSD图中显示负点的结果。值得注意的是,将自动设置为污染时,参数行为不能“过时”。因此,将行为从“旧”更改为“新”。最重要的超参数是基本估计量、样本数量和从该数据集中提取的特征,以及训练模型中的估计量。就最大样本数而言,为估计量和特征数设置了默认值,并从50、100、300到500中选择了最大样本数的值来训练该模型。
2.3 离群点检测法
离群点检测法(local outlier factor, LOF)是一种基于距离的异常点检测的算法,这个算法的主要思想是通过目标点T和其每个领域的点的密度来判断被检测点是否属于异常,点T的密度越低就越可能被检测成是异常点。这里的密度,指的是通过点之间的距离来计算的数值,样本中点之间距离越远,密度越低,距离越近,密度越高。同时因为LOF对密度的计算是通过点的第k邻域来计算,而不是需要通过全局扫描式的计算,避免了时间复杂度很高的情况,这也是为什么叫“局部”异常因子算法的原因。下面是局部可达密度(local reachability density)的公式:
在公式(1)中,比值越接近1,说明p和周围的点密度差不多,所以p可能是和周围的点是同类。另外,如果比值小于1,则说明p是密集点,p大于1的话,说明p就是异常点。在异常检测中,直接利用集成好的离群点检测法算法在训练集上,来对比前两种算法的效果,最后得出对于该实验最优的异常检测算法。
3 分子动力模拟结构预测
3.1 改编的自编码模型
这次实验中,为了预测仅给出二维点的高维结构,我们提出了一种符合当前数据情况的编码器模型,该编码器模型通过输入高维的坐标变量({x1,x2,x3…,xn}),转化成二维的隐变量Z(恰好和输入数据一样的维度),然后再利用产生的二维扩展为高维的结构坐标作为解码器(1,2,3。可以计算x和之间的误差,通过控制两编码器和解码器输出的结果之间的误差来训练数据模型:
通过优化公式(2),最初训练了一种具有较小误差(损失)的改进型自动编码器。完善的自动编码器的损失函数的设计如图3所示。其中x是输入向量,x︿是输出向量,尽量地使其与x非常相似,而z是隐向量,z'是新的隐向量对,这些参数通过最小化神经网络中的值来训练。

图3 改编的自编码器结构示意图
改编的自动编码器的思想来自另一个改编的自动编码器,它是变分自动编码器(VAE)[12-13],这是基于自动编码器开发的神经网络的另一种结构。它们之间的主要区别是损失函数,VAE使用Kullback-Leibler散度训练了模型,从而最大限度地减少了编码器分布和解码器分布之间的损失。总的来说,这是一种有效的神经网络结构,该结构以自我监督的方式进行训练,可通过输入大量结构坐标来近似重建几何结构。另外,解码器部分参考给定新的PC向量组(主要成分)的情况下产生新的预测结构的预测模型。通过查阅文献,没有论文研究过这种类型的自动编码器,因此这是一种在传统自动编码器的基础上进行修改的全新自动编码器。
对于预测,我们就可以只利用经过训练的自动编码器中的解码器部分,可以通过训练相似的分布数据集(称为生成模型)来泛化该模型,因此解码器的输出是针对不同数据的预测结果,但是这些数据都具有类似的分布和格式。对于任何潜在分布的采样(PC对),预期此解码器模型可以准确地重建原始输入结构。在实际的实验中,使用Keras在Python 3.8中构建改编的自动编码器,Keras是具有Tensorflow的开源深度学习库。本研究测试了具有3个编码层和3个解码层的网络。在计算机科学方面,模型的损失反映了神经网络的性能。 因此,比较了不同网络产生的损耗结果,以评估结构的性能。图4是整个模型的总体思路。
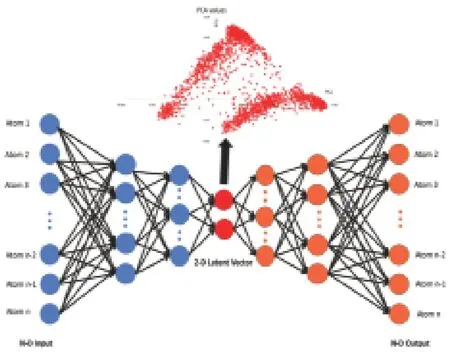
图4 深度学习网络训练结构示意图
3.2 改编的自编码模型结果
本文选择了训练数据之外的12个点作为预测数据,以测试预测的准确性,为了测试改进的自动编码器的性能和稳定性,在预测点周围删除了类似的结构(x的+/-0.002和y的+/-0.003之内的点,y与数据集中的x相比方差略大)。预测12点的每个点时的训练数据集。例如,排除了正方形区域内的点。当预测(0,0.02)的结构时,训练数据中的{(-0.002,0.02),(0.002,0.02),(0,0.017),(0,0.023)}。在划分2 000条轨迹时,仍然采用相同的比率(训练数据为80%,测试数据为20%),并对它们进行了相同的MinMax归一化处理。实验结果如图5所示。
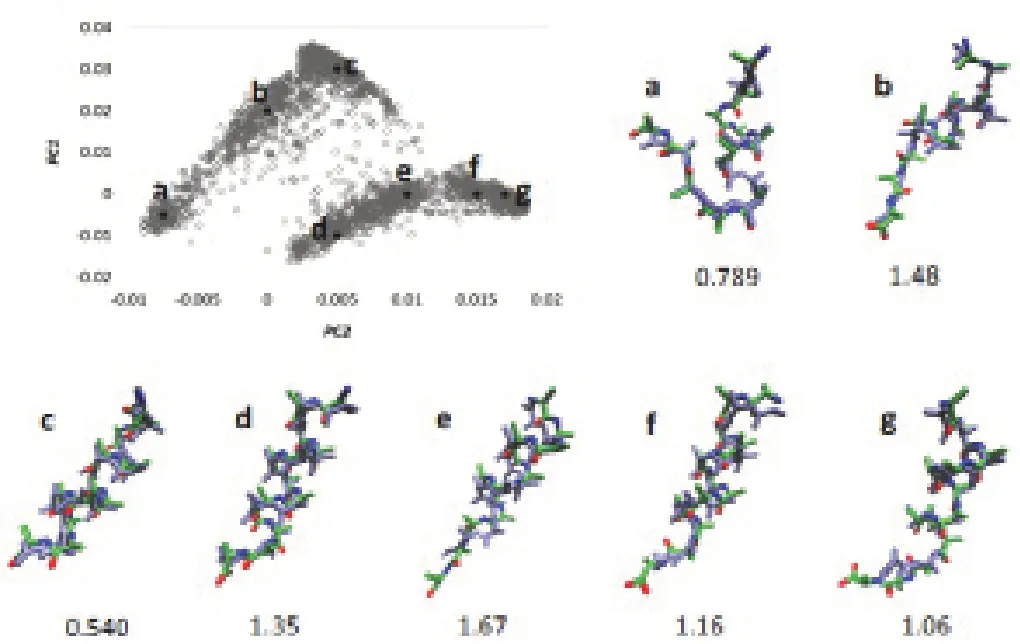
图5 预测结构和真实结构对比图
图5左上方:代表了2D RMSD矩阵(灰色圆圈)的PC1与PC2图,点a~g(黑点)用于测试算法。对于每个预测,将(PC1,PC2)值在(+/-0.002,+/-0.003)以内的点从训练集中排除。对于每个点(a~g),都显示了具有预测结构的原始结构的叠加图,下面的数值代表了其RMSD值。
4 结语
本文通过计算机领域的机器学习和深度学习的使用,在分子动力模拟领域的一系列探究实验,充分证明了机器学习和深度学习在该领域的可行性和可探究性。由于之前很少有人在这个领域用机器学习的方法来对分子运动轨迹进行研究或者预测,之前的一些研究主要是利用类似于PCA的降维方法来观察分子动力模拟轨迹的分布,但是并不能很准确地预测出分子结构,这些问题其实一直困扰着这个领域,所以现在用机器学习和深度学习的方法来进行这些探究实验的意义非常重大,为该领域开辟了一个全新的研究视角。本文提出了运用机器学习和深度学习的方法来对分子动力模拟领域的原子坐标进行探究,成功验证了机器学习算法在蛋白质轨迹分类的实验上的有效性,用三种异常检测算法对不同状态的蛋白质大分子结构进行检测,通过对比不同RMSD值的变化,来判断具体时刻的分子结构变化的程度,从而得到“异常”的蛋白质分子结构,最后提出了一种全新的基于自编码的神经网络模型,通过修改损失函数的组成来构建适合低维输入和高维输出情况的模型。

