自然发酵酸面团发酵过程中微生物菌群多样性分析及细菌功能预测
杨文馨,安飞宇,曹恺欣,乌日娜,3*
(1.沈阳农业大学 食品学院,辽宁 沈阳 110866;2.辽宁省食品发酵技术工程研究中心,辽宁 沈阳 110866;3.沈阳市微生物发酵技术创新重点实验室,辽宁 沈阳 110866)
自然发酵酸面团,是我国特色传统面食发酵剂,有着悠久的历史和丰富的功能特性,在活性干酵母出现的几千年前就已经被广泛地应用于面制品中[1],通过酸面团所制成的面制品也备受人们的喜爱[2]。酸面团中富含多种微生物,是一个复杂的微生态环境,其中主要包含酵母菌、乳酸菌以及霉菌[3-4],通过各种微生物的协同作用使酸面团发酵制品具有更好的风味及质构[5-7],也更有利于人们的肠道健康。酸面团中的微生物具有多种功能特性[8-11],对酸面团制品的感官品质以及营养价值有重要的影响[12-14]。
近年来,随着测序技术的不断进步,高通量测序技术应运而生,其通过对碱基测序和数据收集,可同时完成大量脱氧核糖核酸(deoxyribonucleic acid,DNA)模板的测序,具有高通量、高精度、高速、低费用等特点,被广泛应用于全面解析样品中的微生物群落结构[15-16]。目前,关于通过高通量技术对酸面团发酵时期的细菌和真菌菌群多样性的研究较少。于静等[17]通过传统分离培养的方法对新疆地区囊用酸面团的微生物多样性进行分析,发现有7种乳酸菌和9种酵母菌,优势真菌和细菌分别为酿酒酵母(Saccharomyces cerevisiae)和戊糖乳杆菌(Lactobacillus pentosus);李晓敏等[18]利用高通量测序技术对中国5个省份酸面团的菌群结构和风味物质进行分析,发现酿酒酵母与风味物质之间呈正相关性。
本研究利用高通量测序研究不同发酵阶段自然发酵酸面团中的微生物菌群多样性,并运用PICRUSt对细菌群落的基因功能进行预测,为后期更加全面了解自然发酵酸面中细菌菌群多样性提供理论依据,并为优良发酵功能菌种的筛选提供了数据支持。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
酸面团:从辽宁省沈阳、鞍山、本溪、丹东、桓仁和内蒙古通辽共6个地区分别采集不同发酵时期(12 h、2 d、4 d、6 d)共计24份自然发酵酸面团成品样品。同时,分别取50 g同一发酵时间的自然发酵酸面团样品进行混合,编号为D1、D2、D3、D4。D1样品为发酵前期(12 h),D2、D3样品为发酵中期(2 d、4 d),D4样品为发酵后期(6 d)。
1.1.2 试剂
DNA提取试剂盒:美国OMEGA公司;AxyPrepDNA凝胶回收试剂盒:美国AXYGEN公司;Fastpfu DNA Polymerase:北京TransGen公司;QIA quick聚合酶链式反应(polymerase chain reaction,PCR)引物及扩增体系:北京鼎国昌盛有限公司;Illumina MiSeq platform:美国Illumina公司。其他试剂均为国产分析纯。
1.2 仪器与设备
PCR仪:美国Applied Biosytems公司;DYY-12电泳仪:北京六一生物仪器有限公司;UVP GDS-8000凝胶成像仪:上海精密科学有限公司;Allegra V-15R高速台式冷冻离心机:德国Eppendorf公司;MISEQ测序仪:美国Illumina公司。
1.3 试验方法
1.3.1 DNA提取、PCR扩增及高通量测序
使用DNA提取试剂盒从酸面团样品中提取微生物菌群基因组DNA,以其为模板,采用引物515F(5'-GTGCCGCMGCCGCGG-3')和907R(5'-CCGTCAATTCMTTTRAGTTT-3')对细菌16S rRNA V3-V4区基因序列进行PCR扩增;采用引物SSU0817F(5'-TTAGCATGGAATAATRRAATAGGA-3')和1196R(5'-TCTGGACCTGGTGAGTTTCC-3')对真菌18S rRNA基因进行PCR扩增。PCR扩增体系(30 μL):Phusion Master Mix(2×)15 μL,上下游引物(2 μmol/L)各3 μL,基因组DNA(1 ng/μL)10 μL,双蒸水(ddH2O)2 μL。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55/53 ℃退火30 s,72 ℃延伸45 s,共循环29次;72 ℃再延伸10 min后10 ℃保存。每个样品进行3次重复试验。相同样品的PCR扩增产物用2%琼脂糖凝胶电泳进行检测。在Illumina MiSeq平台上对合格的PCR扩增产物进行双端测序(2×300),按上海美吉生物医药科技有限公司的标准操作规程进行操作。
1.3.2 微生物菌群多样性分析及细菌菌群基因功能性预测
采用Usearch对测序结果进行可操作分类单元(operational taxonomic units,OTU)聚类分析,对相似性≥97%的OTU进行生物信息统计,并使用UCHIME鉴定并去除嵌合序列[19],从而获得有序序列。用核糖体数据库项目(ribosomal database project,RDP)分类器以0.8的置信度阈值[20]对OTU代表序列进行分类学分析,在各个分类水平统计样本群落的组成。利用Alpha多样性评估样品的微生物菌群多样性。采用Unifrac进行Beta多样性分析,比较样品间的差异。根据16S rRNA序列结果,运用PICRUSt预测细菌菌群基因功能[21]。
2 结果与分析
2.1 高通量测序结果
自然发酵酸面团样品微生物菌群的高通量测序结果见表1。
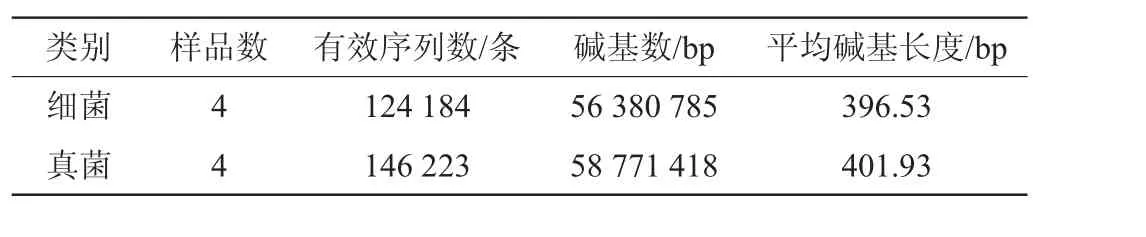
表1 自然发酵酸面团样品微生物菌群高通量测序结果Table 1 High-throughput sequencing results of microflora in spontaneously-fermented sourdough samples
由表1可知,通过高通量测序共得到124 184条细菌有效序列,碱基数为56 380 785 bp,平均碱基长度为396.53 bp;共得到146 223条真菌有效序列,碱基数为58 771 418 bp,平均碱基长度为401.93 bp。真菌和细菌平均碱基长度与设计引物扩增长度接近,质控合格,故可进行后续分析[22]。
2.2 微生物菌群多样性分析
2.2.1α-多样性分析结果
自然发酵酸面团样品微生物菌群的Alpha多样性分析结果见表2。
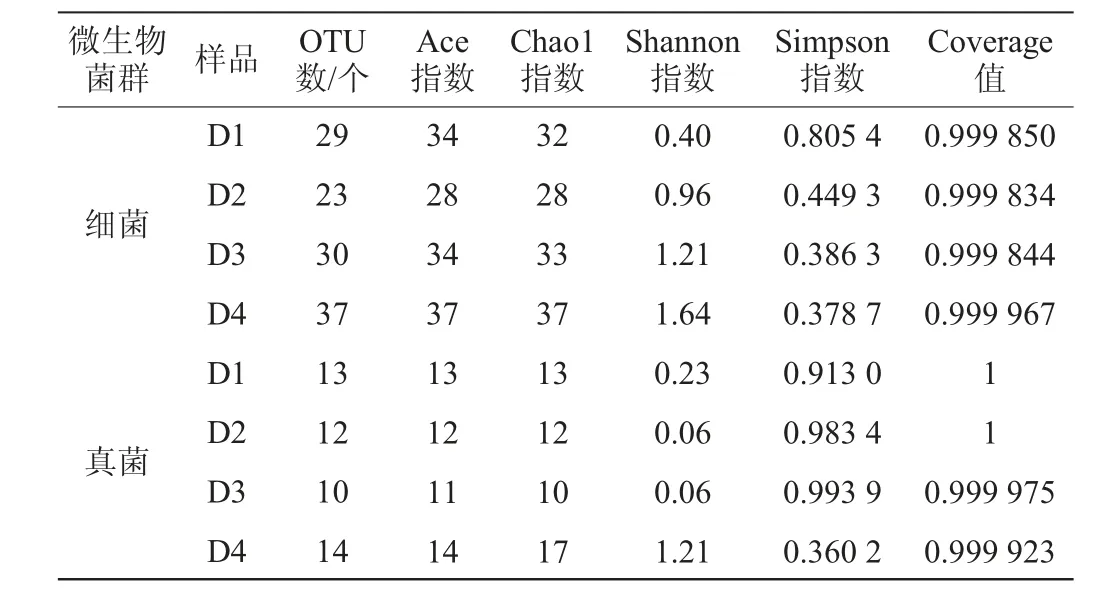
表2 自然发酵酸面团样品微生物菌群的Alpha多样性分析结果Table 2 Alpha diversity analysis results of microflora in spontaneously-fermented sourdough samples
由表2可知,细菌OTU数大于真菌OTU数,说明细菌菌群多样性大于真菌菌群,同时也表明自然发酵酸面团的优势菌群是细菌。Coverage值可反映测序的深度[23],各样品的Coverage值均>0.999,说明数据能真实反映样品中微生物的情况。Ace指数、Chao1指数和Shannon指数值与Alpha多样性呈正相关,而Simpson指数值与Alpha多样性呈负相关[24]。随着发酵时间的延长,真菌和细菌菌群的Chao1指数、Ace指数值均呈先减小后增大的趋势,在发酵后期达最大值,说明在发酵后期,细菌和真菌菌群的丰度最大。细菌和真菌菌群的Shannon指数值均随发酵时间延长而增大,细菌菌群的Simpson指数呈减小趋势,而真菌菌群的Simpson指数在发酵后期显著减小,说明细菌和真菌菌群的多样性随发酵时间延长而升高。
2.2.2 Venn图分析
Venn图通过统计OTU的数量,可以直观显示出不同样本之间的重叠情况及组成的相似性[25]。不同发酵时期自然发酵酸面团中细菌及真菌群落的OTU分布Venn图见图1。由图1a可知,不同发酵时期自然发酵酸面团共有的细菌OTU有12个,分别属于蓝藻门(Cyanobacteria)、厚壁菌门(Firmicutes)及变形菌门(Proteobacteria);由图1b可知,不同发酵时期共有的真菌OTU有9个,均属于子囊菌门(Ascomycota),说明其可能是自然发酵酸面团的优势菌群。整体来看,无论是细菌还是真菌,发酵前期和中期微生物群落组成都较为相似,而发酵后期的微生物多样性增加,说明面团自然发酵过程中,微生物多样性与发酵时间有着密切关系。
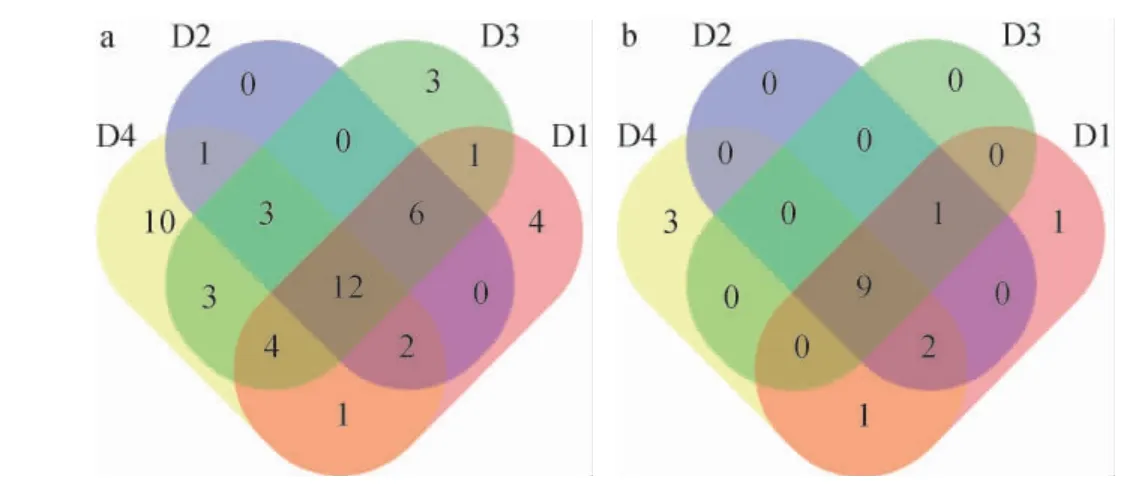
图1 基于操作分类单元自然发酵酸面团样品中细菌(a)及真菌(b)菌群的Venn图Fig.1 Venn diagram of bacteria (a) and fungi (b) community in spontaneously-fermented sourdough samples based on operational taxonomic unit
2.3 微生物菌群结构分析
2.3.1 门分类水平
基于门水平自然发酵酸面团样品中微生物菌群结构见图2。

图2 基于门水平自然发酵酸面团样品中细菌(a)及真菌(b)菌群结构Fig.2 Bacteria (a) and fungi (b) community structure in spontaneously-fermented sourdough samples based on phylum level
由图2a可知,从不同发酵时期自然发酵酸面团样品中共检测到5个细菌门,包括3个共有菌门,分别为蓝藻门(Cyanobacteria)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)。在发酵前期(D1),绝对优势细菌门为蓝藻门(89.28%);在发酵中期(D2~D3),优势细菌门(平均相对丰度>1.00%)为厚壁菌门(53.38%~65.28%);在发酵后期(D4),绝对优势细菌门为变形菌门(77.71%),蓝藻细菌门较少,另外少量拟杆菌门(Bacteroidetes)在发酵后期出现,对面团的发酵过程影响较小。整体来看,蓝藻细菌门波动较大,发酵初期大量存在,发酵后期仅存在少量,说明其可能为原材料中的杂菌。
由图2b可知,从不同发酵时期自然发酵酸面团样品中共检测到13个真菌门,子囊菌门(Ascomycota)在发酵过程中占绝对优势,相对丰度一直>99%,说明子囊菌门对自然发酵酸面团的影响较大。
2.3.2 属分类水平
基于属水平自然发酵酸面团样品中微生物菌群结构见图3。
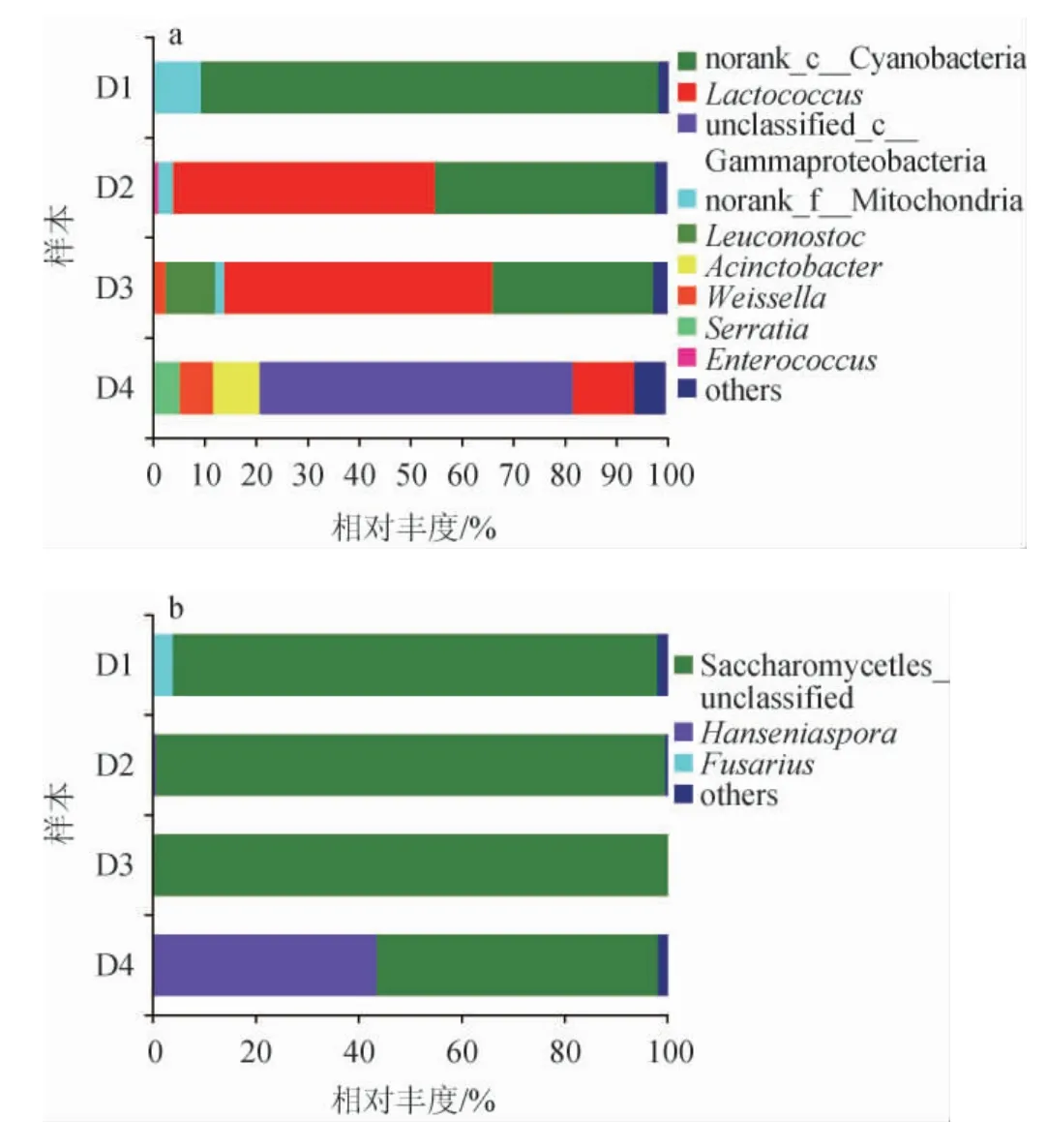
图3 基于属水平自然发酵酸面团样品中细菌(a)及真菌(b)菌群结构Fig.3 Bacteria (a) and fungi (b) community structure in spontaneously-fermented sourdough samples based on genus level
由图3a可知,从不同发酵时期自然发酵酸面团样品中共检测到34个细菌属,包括9个优势细菌属(平均相对丰度>1.00%),分别为未分类的蓝藻门(norank_c__Cyanobacteria)、乳球菌属(Lactococcus)、未分类的γ-变形菌纲(unclassified_c__Gammaproteobacteria)、norank_f__Mitochondria、明串珠菌属(Leuconostoc)、不动杆菌属(Acinetobacter)、魏斯氏菌属(Weissella)、沙雷氏菌属(Serratia)、肠球菌属(Enterococcus)。在发酵前期(D1),绝对优势细菌属为未分类的蓝藻门(89.28%)。在发酵中期(D2~D3),优势细菌属为乳球菌属(50.97%~52.43%),菌群结构较为相似,相对丰度变化幅度不大,说明发酵菌群趋于稳定。酸面团中的乳酸菌可将小分子糖转化为酸类化合物[26],产生较柔和的酸味,发酵代谢中还产生醇,与酸类代谢物反应生成酯,由此使面团具有独特的酯香和醇香[27],另外D3时期还检出了明串珠菌属和乳球菌属。在发酵后期(D4),多种菌属开始被检出,绝对优势细菌属为未分类的γ-变形菌纲。酸面团菌群研究中很少出现未分类的γ-变形菌纲,而在发酵后期大量检出,说明其可能是后期产生的杂菌,另外发酵后期检出的致病菌有沙雷氏菌属(0.22%~5.50%)和肠球菌属(0.04%~1.34%),说明面团发酵时间不宜过长,时间过长会增加致病菌产生的风险。
由图3b可知,从不同发酵时期自然发酵酸面团样品中共检测到12个真菌属,包括3个优势真菌属(平均相对丰度>1%),分别为未分类的酵母目(Saccharomycetales_unclassified)、汉逊酵母属(Hanseniaspora)、镰刀菌属(Fusarium)。各个发酵时期隶属于子囊菌门的未分类的酵母目(95.51%~99.77%)对酸面团的发酵有着重要影响。在发酵后期,优势真菌属汉逊酵母属(22.58%)也被检出。
2.4 PICRUST功能预测
基于OTUs利用PICRUSt预测细菌菌群基因的功能,结果见图4。由图4可知,细菌群落功能多数集中和分布在能量产生和转化、转录、无机离子转运和代谢、碳水化合物转运和代谢、氨基酸转运和代谢等。
3 结论
本研究从辽宁及内蒙古不同地区采集自然发酵酸面团,通过高通量测序对其发酵过程中的微生物多样性分析发现,在门水平上,共检测到5细菌门,不同发酵时期绝对优势菌门不同,发酵前期(D1)为蓝藻门(Cyanobacteria),中期(D2~D3)为厚壁菌门(Firmicutes),后期(D4)为变形菌门(Proteobacteria);共检测到13个真菌门,子囊菌门(Ascomycota)在各个发酵时期均为优势真菌门。属水平上,共检测到34个细菌属,发酵前期绝对优势菌属为未分类的蓝藻细菌门(norank_c__Cyanobacteria),中期为乳球菌属(Lactococcus),后期为未分类的γ-变形菌纲(unclassified_c_Gammaproteobacteria);共检出12种真菌属,未分类的酵母目(Saccharomycetales_unclassified)在各个发酵时期均为绝对优势菌属,后期汉逊酵母属(Hanseniaspora)也被大量检出。通过PICRUSt对细菌群落的基因功能进行预测,结果显示,酸面团在自然发酵过程中细菌群落的基因功能主要有能量物质的产生及转化,转录、无机离子无机离子转运和代谢等。本研究进一步了解了酸面团在不同自然发酵阶段微生物演替模式,为后续研究酸面团中微生物间相互作用提供科学依据,为筛选优良的发酵菌种资源提供数据参考。

