HLA-Ⅰ基因单核苷酸多态性与中国南方汉族女性乳腺癌易感性的相关性研究①
佟 琳 杨学习 李粉霞 孙敏英 刘民锋 董建宇 郭昭泽 叶长生
(南方医科大学南方医院乳腺科,广州510515)
目前,乳腺癌已成为女性最常见的恶性肿瘤。 2008年全球乳腺癌新增病例约138万,占所有新发癌症病例的22.9%(IARC:Cancer Epidemiology Database, GLOBOCAN.2008 [http://www-dep.iarc.fr])。近几年,虽然美国等西方发达国家的乳腺癌发病率和死亡率呈下降趋势;但在中国却逐年增长,2008年中国乳腺癌新发病例(16.9万)比2002年(12.6万)(IARC:Cancer Epidemiology Da-tabase,GLOBOCAN.2002 [http://www-dep.iarc.fr/])增长了34%。
HLA是机体免疫识别,免疫应答及免疫调节的重要系统[1]。它决定机体的组织相容性,并以其多基因性和高度多态性成为人类的重要遗传标记[2]。HLA基因定位于人类染色体6p21.3区域,可分为HLA-Ⅰ、HLA-Ⅱ、HLA-Ⅲ 3 个区域。目前已证明HLA参与肿瘤细胞逃避机体免疫监视过程,与多个系统性疾病存在相关性[2-4]。HLA-Ⅰ类分子广泛分布于人体有核细胞表面,参与内源性抗原肽的加工、处理和提呈[5]。HLA-Ⅰ已被证实与多种肿瘤易感性相关[6-8]。HLA-Ⅰ类分子的表达降低、缺失是肿瘤细胞的一种逃逸方式,主要机制为针对HLA-Ⅰ类分子向T细胞递呈的免疫原性多肽无法被识别,从而无法杀伤肿瘤细胞[9]。此外,HLA-Ⅰ还是预测乳腺癌复发、转移风险的有效指标[10],但HLA-Ⅰ基因单核苷酸多态性(Single nucleotide polymorphism,SNP)与乳腺癌之间的关系尚不清晰。本文选取了HLA-Ⅰ基因编码区的17个单核苷酸多态性位点,采用病例对照研究,通过 Sequenom MassArrayⓇiPLEX GOLD系统,对中国南方汉族乳腺癌患者267例及健康对照组274例进行基因分型分析,探究其多态性与乳腺癌发病风险的相关性。
1 对象与方法
1.1研究对象 2009年5月12日到2010年7月2日,收集广州南方医院、广州军区总医院、江西南昌大学第一附属医院、湛江广东医学院附属第一医院经病理学确诊的乳腺癌患者血液标本267例,平均年龄为(47.8±9.28)岁,中位年龄为47岁。同期收集相应医院体检健康女性274人,要求无乳腺相关病史,无乳腺癌家族史和其它肿瘤病史,平均年龄(51±16.7)岁,中位年龄为49岁。所有对象均为中国南方汉族女性且相互间无血缘关系,获得知情同意后,取外周血1~2 ml于酸性枸橼酸葡萄糖抗凝管中,-80℃保存待用。
1.2方法
1.2.1DNA的提取 取外周静脉血200 μl,用DNA提取试剂盒(北京天根生物技术有限公司,TIANamp Genomic DNA Kit)提取DNA(按说明书操作),DNA保存于-70℃备用。
1.2.2引物的设计 根据HLA-Ⅰ编码区域的目标序列和所选择的多态性位点,利用Sequenom MassArrayⓇAssay Design 3.1软件设计多重PCR特异性扩增引物和单碱基延伸引物,其中针对各位点的PCR特异性扩增引物和单碱基延伸引物见表1。

表1 17个位点的PCR特异性扩增引物和单碱基延伸引物Tab.1 Specific PCR amplification primer and single base extension primer for 17 SNPs
1.2.3基因分型分析 采用 MassArrayⓇ-IPLEX SNP分型技术对所选样本进行基因分型,步骤如下:将提取纯化后的基因组DNA取1.0 μl,按顺序加于特定的384孔板上,再添加PCR扩增体系使反应物的终浓度如下:0.1 U的Taq聚合酶,5 ng基因组DNA,各2.5 pmol的 PCR 引物,2.5 mmol的 dNTP。PCR反应条件为:95℃ 2分钟;95℃ 20秒,56℃ 30秒,72℃ 60秒循环45次;72℃ 5分钟;在上述体系中添加0.3 U的碱性磷酸酶,经37℃ 40分钟,85℃5分钟的反应去除剩余的dNTP;向体系中添加5.4 pmol的各延伸引物,50 μmol的 dNTP/ddNTP 混合物,0.5 U的Thermosequenase DNA聚合酶,反应条件为:94℃ 2分钟;94℃ 5秒,50℃ 5秒,72℃ 5秒循环40次;72℃ 3分钟。反应产物用树脂脱盐15分钟后经自动点样仪点样于SpectroCHIP(Sequenom)芯片,点样结束后的芯片用基质辅助激光解吸附电离飞行时间质谱(SpectroREADER,Sequenom)进行检测。对DNA样品进行质量控制,以保证基因分型成功率为95%以上。
1.2.4数据分析 对所选17个位点的基因型分布频率进行Hardy-Weinberg平衡检验后,以在线分析工具http://bioinfo.iconcologia.net/SNPStats和统计软件SPSS13.0对数据进行病例-对照和受体状态相关性的统计分析,再将病例组样本按免疫组化结果中雌激素受体(Estrogen receptor,ER)、孕激素受体(Progesterone receptor,PR)、人类表皮生长因子受体2(Human epidermal growth factor receptor-2,HER-2)状态分为阳性组和阴性组。分析过程中采用共显性(codominant)、显性(dominant)、隐性(recessive)、超显性(overdominant)四种遗传模型。χ2检验分析不同分组的基因型频率有无差异,非条件Logistic回归计算比数比OR和95%可信区间(95%CI),由此评估所选位点多态性与乳腺癌的相关性。检验水准为双侧α=0.05。
2 结果

表2 不符合Hardy-Weinberg平衡的位点的基因型分布Tab.2 Distribution of SNPs inconsistent with the Hardy-Weinberg equilibrium
本研究所选位点在检测样本中均存在多态性,其中 7 个位点 rs2517749、rs1632902、rs4087726、rs9260475、rs9260489、rs9260571、rs9260619 不符合Hardy-Weinberg平衡(表2),遂在随后的统计分析析中不在包括在内。关联分析发现,rs9260682与乳腺癌的发病风险和HER-2状态存在相关性,rs9260682的AT基因型可增加携带者乳腺癌的患病风险(P=0.04),且与HER-2阴性乳腺癌相关(P=0.04)。rs9260734虽与乳腺癌的发病风险不存在显著相关性,但AA基因型与PR阴性乳腺癌相关(P=0.048),具体结果见表4;其余8个位点rs9404952、 rs9295822、 rs6921314、 rs1632882、rs2517716、rs1632879、rs16896742、rs2571400 基因型分布在病例-对照、ER阳/阴性、PR阳/阴性、HER-2阳/阴性分组中均未显示有明显相关性(表3)。

表3 与乳腺癌患病风险无明显关系的位点的基因型分布Tab.3 Distribution of insignificant SNPs in case-control studies
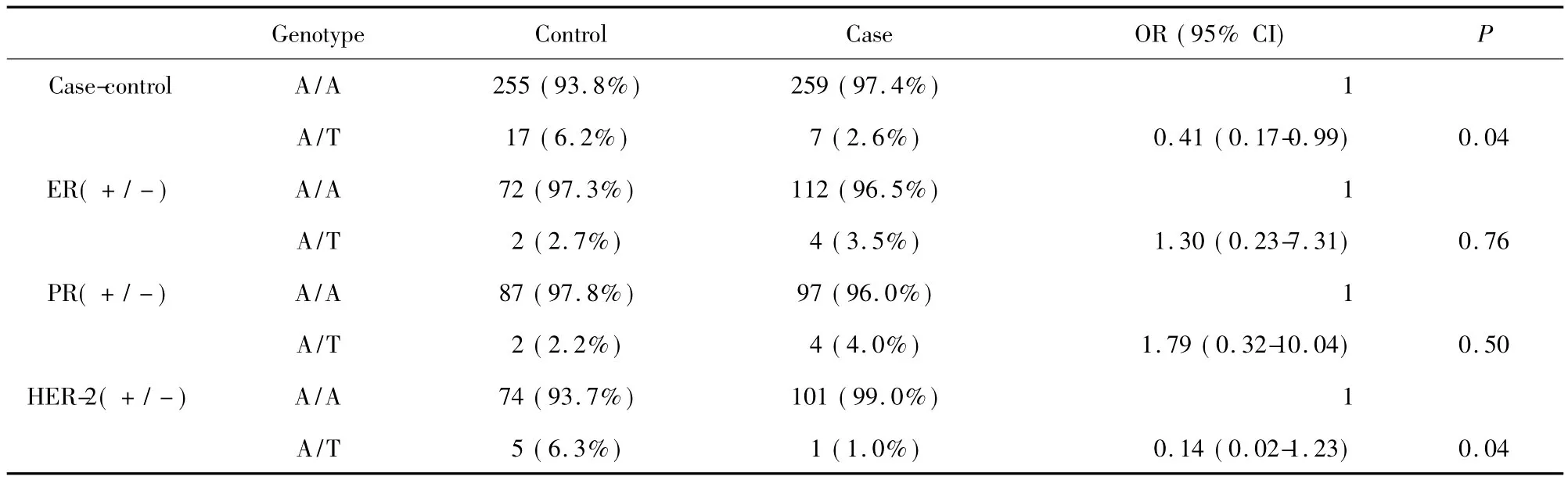
表4 rs9260682位点多态性与乳腺癌患病风险的相关性Tab.4 The association of rs9260682 polymorphisms and breast cancer risk
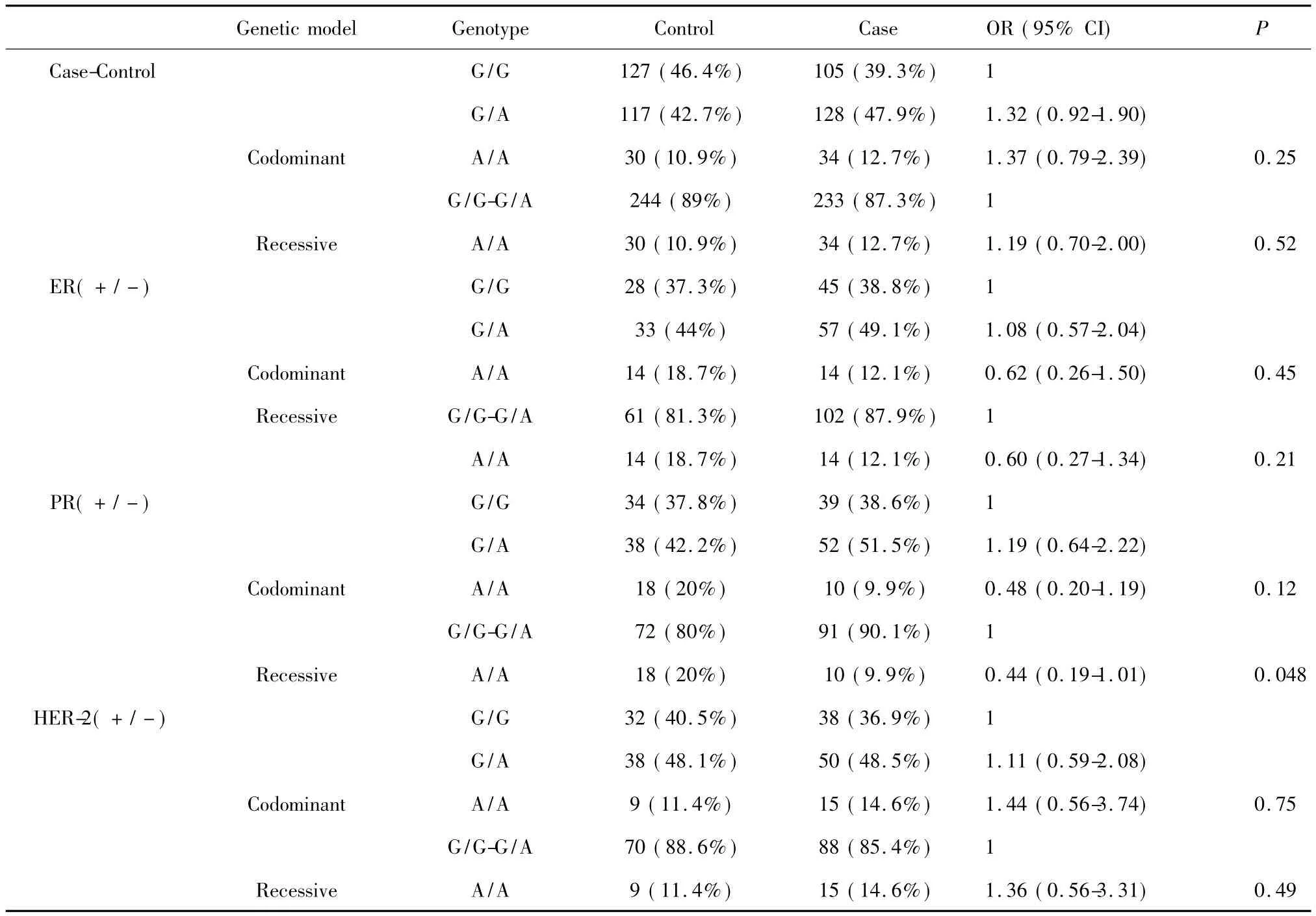
表5 rs9260734位点多态性与乳腺癌患病风险的相关性Tab.5 The association of rs9260734 polymorphisms and breast cancer risk
2.1 位点rs9260682多态性与乳腺癌及ER、PR和HER-2状态的相关性分析 rs9260682在中国南方汉族女性存在AA、AT两种多态性,在总体样本中的分布频率分别为96%、4%,在不同组别中的分别频率如表4。该位点两种基因型分布在病例-对照分组中有显著统计学差异(P=0.04),表明该位点多态性与乳腺癌易感性明显相关;在ER阳/阴性、PR阳/阴性分组比较中发现,基因型分布虽有一定差异,但均无明显统计学意义(P=0.76、P=0.50);HER-2分组比较结果显示,该位点的多态性分布与乳腺癌HER-2状态明显相关(P=0.04),携带杂合型AT者比纯合型AA相比更倾向于HER-2阴性乳腺癌(OR=0.14,95%CI:0.02-1.23)。
2.2 位点rs9260734多态性分析与乳腺癌及ER、PR和HER-2状态的相关性分析 rs9260734存在AA、GA、GG三种多态性,在总体研究样本中的分布频率分别12%、45%、43%。统计分析发现该位点基因型分布在病例-对照、ER阳/阴性和HER-2阳/阴性分组中均无统计学差异(表5);但在PR分组比较中发现rs9260734的多态性分布与PR状态明显相关(P=0.048),AA基因型携带者更倾向于PR阴性乳腺癌(OR=0.44,95%CI:0.19-1.01)。
3 讨论
乳腺癌的发病是多基因的作用结果,通过对乳腺癌易感基因的研究中已经发现,BRCA1和BRCA2是家族性乳腺癌的罪魁祸首。除了寻找易感基因,筛查易感性SNP位点也是乳腺癌基础研究的一个热点。作为第三代遗传标记,SNP更适合作为生物标记在普通人群中进行检测。全基因组关联研究(Genome-wide association studies,GWAS)以大规模群体DNA样本的SNP进行全基因组高密度遗传变异检测,并利用有效统计方法计算进行病例-对照分析,计算出基因分型变异频率的差异,最终达到相关的变异与疾病的关联分析,从而确定特定基因型的个体患病风险,实施个性化预防策略。目前通过GWAS关联分析研究,已发现与乳腺发病相关的数十个基因,如 ABHD8、ANKRD16、CCND1、CDKN2A、CDKN2B、ECHDC1、ESR1、FGFR2、GLG1、GRIK1、KLF4、MAP3K1、MRPS30、TOX3 等的上百个 SNP 位点[11-26],为了解乳腺癌发病的分子机制及易感因素提供了更多的线索。但GWAS研究也存在局限性,对已经发现的肿瘤易感位点需进行大样本,多人群及相应的功能机制分析。
HLA与许多系统疾病,如运动系统、神经系统和感染性疾病等都存在相关性[6-8],尤其是参与免疫调节及应答[27]。此外,HLA还参与肿瘤细胞逃逸免疫监视,在许多肿瘤的形成过程中发挥重要的作用,目前已有研究显示HLA的多态性与肿瘤发生的易感性相关[6,28,29]。根据千人基因组计划(http://www.1000qenomes.orq/),我们在 HLA-Ⅰ选取了17个SNP位点,探讨HLA-Ⅰ基因多态性与乳腺癌的相关性,研究发现有7个位点不符合Hardy-Weinberg平衡。这与HLA基因多态性丰度极高有关,很多关于HLA及疾病的关联研究会面临所获得的数据最终不符合Hardy-Weinberg平衡的问题[30]。
目前有较多证据显示,HLA多态性与肝癌、白血病、卵巢癌、宫颈癌等多种癌症易感性相关[7,8,31,32]。乳腺癌为女性最常见恶性肿瘤,HLA与乳腺癌发生发展密切相关。早在1990年就发现HLA在乳腺癌组织表达显著低于癌旁组织[33]。转移性乳腺癌组织HLA-Ⅰ的表达亦显著下降[34]。此外,有研究显示乳腺癌患者HLA-Ⅰ蛋白表达程度与肿瘤分级明显相关[10,35,36],且发现 HLA-Ⅰ表达越低淋巴结转移越多及无病生存期越短(P<0.05)[10]。Chardhuri[37]首先发现 HLA 区域 DQB3*02017/*0202等位基因与乳腺癌发病风险密切相关。David[38]在对墨西哥散发乳腺癌的研究中也发现HLAI基因上DRB1*1602和DQ*0301位点与乳腺癌的发病风险存在相关性,其他研究同样显示HLA-Ⅰ与乳腺癌明显相关[39,40],但这些数据主要以西方人群为基础,目前尚无中国人群的相关报道。本研究在中国南方汉族女性中首次发现HLA-I基因上的rs9260682与乳腺癌患病风险显著相关。我们实验室已经报道该位点与鼻咽癌易感性同样相关[41],但目前尚无该位点与乳腺癌易感性的相关性研究。
ER、PR、HER-2为乳腺癌指导预后及临床治疗的重要指标,已成为临床上常规检查项目。ER、PR表达阳性的乳腺癌为激素敏感性患者,复发转移风险低,预后较好,且适于内分泌治疗。HER-2蛋白过表达与乳腺癌恶性程度呈正相关,是重要的预后指标,同时HER-2表达情况为临床使用赫赛汀靶向治疗提供依据。因此本研究联合以上指标来分析HLA遗传多态性与其状态的相关性。结果显示rs9260734与PR阴性相关,AA基因型携带者更倾向于PR阴性;rs9260682除与乳腺癌发病风险相关外,其AT基因型比AA基因型更倾向于HER-2阴性乳腺癌。
通过对HLA-I区域17个SNPs位点与乳腺癌易感性及受体状态的相关性研究,发现rs9260682与中国南方汉族女性中乳腺癌易感性及HER-2阴性相关,rs9260734与乳腺癌PR阴性相关。该研究不仅有助于探讨HLA-I在乳腺癌发生、发展中的作用,也可为乳腺癌的早期诊断和个体化医疗提供数据基础。
1 Esteban F,Ruiz-Cabello F,Concha A et al.HLA-DR expression is associated with excellent prognosis in squamous cell carcinoma of the larynx[J].Clin Exp Metastasis,1990;8(4):319-328.
2 Taneja V,David C S.Role of HLA class II genes in susceptibility/resistance to inflammatory arthritis:studies with humanized mice[J].Immunol Rev,2010;233(1):62-78.
3 Ferlini A,Bovolenta M,Neri M et al.Custom CGH array profiling of copy number variations(CNVs)on chromosome 6p21.32(HLA locus)in patients with venous malformations associated with multiple sclerosis[J].BMC Med Genet,2010;11:64-66.
4 Mcmichael A J,Jones E Y.Genetics.First-class control of HIV-1[J].Science,2010;330(6010):1488-1490.
5 吴继周.HLA与人类肿瘤发生的关系研究现状[J].内科,2009;(4):592-594.
6 Tse K P,Su W H,Chang K P et al.Genome-wide association study reveals multiple nasopharyngeal carcinoma-associated loci within the HLA region at chromosome 6p21.3 [J].Am J Hum Genet,2009;85(2):194-203.
7 Gamzatova Z,Villabona L,van der Zanden H et al.Analysis of HLA class I-II haplotype frequency and segregation in a cohort of patients with advanced stage ovarian cancer[J].Tissue Antigens,2007;70(3):205-213.
8 Schiff M A,Apple R J,Lin P et al.HLA alleles and risk of cervical intraepithelial neoplasia among southwestern American Indian women[J].Hum Immunol,2005;66(10):1050-1056.
9 邹 雄,张利宁.分子免疫学与临床[M].济南:山东科学技术出版社,2003:63-80.
10 Kaneko K,Ishigami S,Kijima Y et al.Clinical implication of HLA class I expression in breast cancer[J].BMC Cancer,2011;11:454-457.
11 Sehrawat B,Sridharan M,Ghosh S et al.Potential novel candidate polymorphisms identified in genome-wide association study for breast cancer susceptibility[J].Hum Genet,2011;130(4):529-537.
12 Fletcher O,Johnson N,Orr N et al.Novel breast cancer susceptibility locus at 9q31.2:results of a genome-wide association study[J].J Natl Cancer Inst,2011;103(5):425-435.
13 Li J,Humphreys K,Darabi H et al.A genome-wide association scan on estrogen receptor-negative breast cancer[J].Breast Cancer Res,2010;12(6):R93.
14 Gaudet M M,Kirchhoff T,Green T et al.Common genetic variants and modification of penetrance of BRCA2-associated breast cancer[J].PloS Genet,2010;6(10):e1001183.
15 Li J,Humphreys K,Heikkinen T et al.A combined analysis of genome-wide association studies in breast cancer[J].Breast Cancer Res Treat,2011;126(3):717-727.
16 Antoniou A C,Wang X,Fredericksen Z S et al.A locus on 19p13 modifies risk of breast cancer in BRCA1 mutation carriers and is associated with hormone receptor-negative breast cancer in the general population[J].Nat Genet,2010;42(10):885-892.
17 Long J,Cai Q,Shu X O et al.Identification of a functional genetic variant at 16q12.1 for breast cancer risk:results from the Asian BreastCancer Consortium [J]. Plos Genet, 2010; 6(6):e1001002.
18 Turnbull C,Ahmed S,Morrison J et al.Genomewide association study identifies five new breast cancer susceptibility loci[J].Nat Genet,2010;42(6):504-507.
19 Thomas G,Jacobs K B,Kraft P et al.A multistage genomewide association study in breast cancer identifies two new risk alleles at 1p11.2 and 14q24.1(RAD51L1) [J].Nat Genet,2009;41(5):579-584.
20 Zhang W,Long J R,Gao Y T et al.Genome-wide association study identifies a new breast cancer susceptibility locus at 6q25.1[J].Nat Genet,2009;41(3):324-328.
21 Kibriya M G,Jasmine F,Argos M et al.A pilot genomewide association study of early-onset breast cancer[J].Breast Cancer Res Treat,2009;114(3):463-477.
22 Gold B,Kirchhoff T,Stefanov S et al.Genome-wide association study provides evidence for a breast cancer risk locus at 6q22.33[J].Proc Natl Acad Sci,2008;105(11):4340-4345.
23 Murabito J M,Rosenberg C L,Finger D et al.A genomewide association study of breast and prostate cancer in the NHLBI's Framingham Heart Study[J].BMC Med Genet,2007;8(Suppl 1):S6.
24 Easton D F,Pooley K A,Dunning A M et al.Genome-wide association study identifies novel breast cancer susceptibility loci[J].Nature,2007;447(7148):1087-1093.
25 Hunter D J,Kraft P,Jacobs K B et al.A genome-wide association study identifies alleles in FGFR2 associated with risk of sporadic postmenopausal breast cancer[J].Nat Genet,2007;39(7):870-874.
26 Stacey S N,Manolescu A,Sulem P et al.Common variants on chromosomes 2q35 and 16q12 confer susceptibility to estrogen receptorpositive breast cancer[J].Nat Genet,2007;39(7):865-869.
27 Shiina T,Inoko H,Kulski J K.An update of the HLA genomic region,locus information and disease associations:2004 [J].Tissue Antigens,2004;64(6):631-649.
28 Kubler K,Arndt P F,Wardelmann E et al.HLA-class II haplotype associations with ovarian cancer [J].Int J Cancer,2006;119(12):2980-2985.
29 Kubler K,Arndt P F,Wardelmann E et al.Genetic alterations of HLA-class II in ovarian cancer[J].Int J Cancer,2008;123(6):1350-1356.
30 Yang X X,Pan H Z,Li P Y et al.HLA class II variants in Chinese breast cancer patients[J].Asian Pac J Cancer Prev,2011;12(11):3075-3079.
31 Lopez-Vazquez A,Rodrigo L,Mina-Blanco A et al.Extended human leukocyte antigen haplotype EH18.1 influences progression to hepatocellular carcinoma in patients with hepatitis C virus infection[J].J Infect Dis,2004;189(6):957-963.
32 Khosravi F,Amirzargar A,Sarafnejad A et al.HLA class II allele and haplotype frequencies in Iranian patients with leukemia[J].I-ran J Allergy Asthma Immunol,2007;6(3):137-142.
33 Wintzer H O,Benzing M,von Kleist S.Lacking prognostic significance of beta 2-microglobulin,MHC class I and class II antigen expression in breast carcinomas[J].Br J Cancer,1990;62(2):289-295.
34 Magyarosy E,Martin W J,Chu E W et al.Differential diagnostic significance of the paucity of HLA-I antigens on metastatic breast carcinoma cells in effusions[J].Pathol Oncol Res,1999;5(1):32-35.
35 Klein T,Levin I,Niska A et al.Correlation between tumour and serum beta 2m expression in patients with breast cancer[J].Eur J Immunogenet,1996;23(6):417-423.
36 Zhao S,Yang X,Lu N et al.The amount of surface HLA-I on T lymphocytes decreases in breast infiltrating ductal carcinoma patients[J].J Int Med Res,2011;39(2):508-513.
37 Chaudhuri S,Cariappa A,Tang M et al.Genetic susceptibility to breast cancer:HLA DQB*03032 and HLA DRB1*11 may represent protective alleles[J].Proc Natl Acad Sci USA,2000;97(21):11451-11454.
38 Cantu D L D,Perez-Montiel D,Villavicencio V et al.High resolution human leukocyte antigen(HLA)class I and class II allele typing in Mexican mestizo women with sporadic breast cancer:casecontrol study[J].BMC Cancer,2009;9:48-51.
39 Cabrera T,Angustias F M,Sierra A et al.High frequency of altered HLA class I phenotypes in invasive breast carcinomas[J].Hum Immunol,1996;50(2):127-134.
40 Lavado-Valenzuela R,Benavides M,Carabantes F et al.MHC class I chain-related gene A transmembrane polymorphism in Spanish women with breast cancer[J].Tissue Antigens,2009;74(1):46-49.
41 Zhao M,Cai H,Li X et al.Further evidence for the existence of major susceptibility of nasopharyngeal carcinoma in the region near HLA-A locus in Southern Chinese[J].J Transl Med,2012;10(1):57-60.

