微生物多糖的生物合成及代谢工程研究进展
微生物多糖的生物合成及代谢工程研究进展
曾化伟1,2,郑惠华1,2,陈惠1,2,廖祥儒3,蔡宇杰3
(1.江苏安惠生物科技有限公司, 江苏 南通 226009;
2.中国科学院微生物研究所与江苏安惠生物科技有限公司 药用菌联合实验室, 江苏 南通 226009;
3.江南大学 工业微生物技术教育部重点实验室, 江苏 无锡 214122)
[摘要]综述了微生物多糖的生物合成途径、基因簇结构与功能和代谢工程的研究进展。真菌多糖的合成途径主要包括:前体核苷酸糖(单糖的活化形式)的合成、合成的起始反应、重复单元的延伸、翻转和聚合、多糖的输出。多糖合成过程中涉及大量的基因,大多数基因存在多糖合成基因簇中,随着基因测序技术的发展,大量的多糖合成基因簇被发现和研究。研究表明:通过调控涉及糖核苷酸合成途径的酶水平可提高胞外多糖的产量,而增加多糖合成基因簇基因在微生物中的表达,可产生更高的多糖合成代谢流。
[关键词]微生物多糖;生物合成;基因簇;代谢工程
[文章编号]1673-2944(2015)04-0049-10
[中图分类号]Q936
收稿日期:2015-01-30
基金项目:国家“863”计划项目(2012AA021704);江苏省科技厅企业“博士集聚”计划和江苏省农业科技支撑项目(BE2012341)
作者简介:曾化伟(1981—),男,江西省宁都县人,江苏安惠生物科技有限公司工程师,博士,主要研究方向为药用菌活性成分高效累积代谢工程;[通信作者]郑惠华(1967—),女,江苏省南通市人,江苏安惠生物科技有限公司高级工程师,主要研究方向为食药用菌生物技术。
微生物多糖主要包括细菌、药用真菌在代谢过程中所产的一类由多个单糖或其衍生物聚合而成的大分子物质。根据形态学上的分类,可以分为细胞壁多糖、细胞外多糖和细胞内多糖,细胞壁多糖典型代表为细菌中O-抗原(O-antigen),细胞外多糖包括细菌细胞壁外荚膜多糖(Capsular Polysaccharide,CPS)和胞外多糖(Exopoly Saccharides,EPS)[1]。细菌多糖可作为食品工业中的添加剂(如乳酸菌EPS、海藻酸盐、黄原胶等),或在制造化妆品中作为保湿成分(如透明质酸)[2]。在医药中,病原细菌表面的CPS或O-antigen已被广泛应用于抗感染疾病多糖疫苗的开发,有些多糖疫苗已投入临床使用[3]。近年来,随着人们对保健意识的增强,药用真菌多糖产品(如灵芝多糖)以其良好的抗肿瘤、提高免疫力、抗氧化等生物学活性已受到人们的瞩目[4-5]。因微生物多糖优越的应用价值,科学家们对它们的生物合成途径、途径相关基因、代谢工程领域的研究产生了浓厚的兴趣,并取得了可喜的研究成果,下面将对这些研究成果进行综述。
1微生物多糖生物合成的途径
多糖合成途径的研究主要集中在细菌中,真菌多糖的合成途径研究非常少[2]。现有的研究表明,尽管多糖的结构千差万别,但它的合成途径却相对一致[3]。主要包括4个步骤:(1)前体核苷酸糖(单糖的活化形式)的合成;(2)合成的起始反应;(3)重复单元的延伸、翻转和聚合;(4)多糖的输出。
1.1核苷酸糖的合成
根据多糖所含单糖的种类分为同型多糖和异型多糖[1],异型多糖较同型多糖的核苷酸糖合成更复杂。笔者参考文献[2,6-9],总结了近年发表的异型多糖(如黄原胶、乳酸菌ESP(HePS)、灵芝菌丝体多糖(IPS-1-1)等)的一些主要核苷酸糖合成途径(图1)。以葡萄糖为发酵碳源,经酶1转化为葡萄糖-6-磷酸后,其代谢主要有三条支路。第一条途径为葡萄糖-6-磷酸经酶2转化为葡萄糖-1-磷酸,再合成TDP-葡萄糖、TDP-鼠李糖、UDP-葡萄糖、UDP-半乳糖、UDP-葡糖醛酸,这是基本的合成途径;第二条途径是葡萄糖-6-磷酸可以生成GDP-甘露糖和GDP-岩藻糖;第三条途径是葡萄糖-6-磷酸经酶11作用生成果糖-6-磷酸,再生成UDP-N-乙酰半乳糖胺和GDP-果糖。果糖-6-磷酸还能够进入糖酵解途径产生丙酮酸,丙酮酸经厌氧途径生成乳酸,或通过有氧途径进入柠檬酸循环产生ATP和其它产物,这些ATP为多糖合成提供能量保证。如果以乳糖为发酵碳源,需依次经Leloir途径的酶(12-15)作用生成葡萄糖-1-磷酸,最后进入核苷酸糖的合成;以果糖为碳源直接生成果糖-6-磷酸,然后在相关酶的催化下生成丙酮酸,或生成葡萄糖-1-磷酸,进入核苷酸糖的合成。除此之外,还有大量未标出合成途径,它们能够产生其它的核苷酸糖。如d-TDP-脱氧-太威糖、dTDP-D-3-乙酰-岩藻糖胺、GDP-L-可立糖等[10]。根据发酵碳源和多糖组成的不同,所涉及的合成关键酶并不完全相同,但一般包括酶(2、5、6、11)。
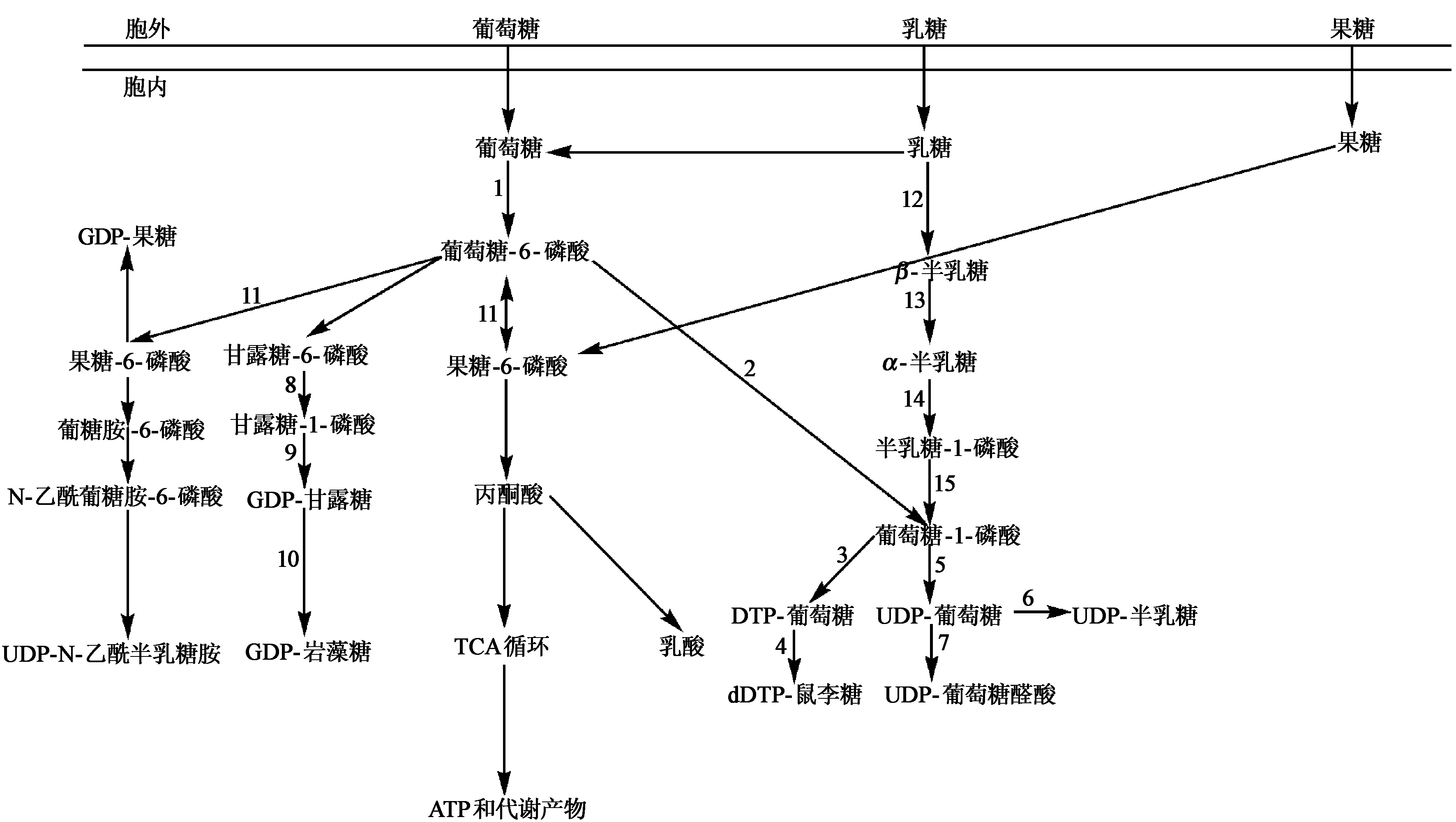
1.葡糖激酶; 2.α-磷酸葡糖变位酶; 3.葡萄糖-1-磷酸胸苷转移酶; 4.鼠李糖合成酶系; 5.UDP-葡萄糖焦磷酸化酶; 6.UDP-半乳糖-4-差向异构酶; 7.NAD连接的脱氢酶; 8.磷酸甘露糖变位酶; 9.GDP-甘露糖焦磷酸化酶; 10.脱水酶和GDP-岩藻糖合成酶; 11.磷酸葡糖异构酶; 12.β-半乳糖苷酶; 13.半乳糖苷变旋酶; 14.半乳糖激酶; 15.半乳糖-1磷酸转移酶 图1 微生物部分核苷酸糖合成途径
1.2合成的起始反应
多糖合成研究主要集中在O-antigen合成中,把它作为一个模型。O-antigen合成的起始反应是在起始糖基转移酶作用下,将第一个核苷酸糖与位于细胞膜上的脂载体十一异戊烯磷酸(Und-P))形成Und-PP-连接的糖复合物,这个过程在不同O-antigen中相对保守[1]。在O-antigen合成中,WecA(PNTP蛋白家族)和WbaP(PHTP蛋白家族)是目前仅知的两个起始转移酶,WecA负责转运N-乙酰氨基葡萄糖(GlcNAc)-/N-乙酰氨基半乳糖(GalNAc),WbaP负责转运乳糖-1-磷酸或葡萄糖-1-磷酸[11]。部分EPS和CPS的合成和这个过程类似,也需要起始糖基转移酶将核苷酸糖加载到脂载体上[12-13]。但乳酸菌同型多糖HoPS不依赖脂载体十一异戊烯磷酸[12]。
1.3重复单元的延伸、翻转和聚合
在O-antigen多糖中,这步是核心过程。根据该过程中组装和翻转机制的不同,可分为三种途径:Wzy-dependent途径、ABC-transporter dependent途径和synthase-dependent途径[1]。
Wzy-dependent途径:起始反应后,基因簇中的糖基转移酶将其余核苷酸糖按照一定顺序转移到单糖-PP-Und上,形成完整的十一异戊二烯二磷酸寡糖重复单元(RU-PP-Und)。RU-PP-Und随后被翻转酶(Wzx)反转到内膜一侧[1]。Wzx如何实现该翻转的清晰机制仍不清楚。但在绿脓杆菌(Pseudomonasaeruginosa)中,开展一系列的巧妙实验,一个更为清晰的机制正在逐渐呈现。Isiam等发现Wzx有12个跨膜螺旋,提出依赖于质子逆向转运机制来转运RU-PP-Und通过内部富含带正电荷的残基空腔[14]。Wzx蛋白被认为与其翻转的RU-PP-Und有不严格底物特异性,但最近发现Wzx与RU-PP-Und的重复单元化学结构的识别也是翻转的部分机制[15]。在周质空间内,聚合酶Wzy将Ru-PP-Und聚合成十一异戊二烯二磷酸多糖(Ps-PP-Und)[16]。推测其聚合方式为PS-PP-Und中的PS转移到Ru-PP-und的非还原端[17]。一般认为Wzy的底物特异性很强,但最新研究表明,某些Wzy即具有比较松弛的底物特异性,它是阐释O-antigen生物合成的关键酶。Wzy的聚合功能及其与Wzz的相互作用在体外试验中得到了证实[18]。然后由Wzz来控制O-antigen的长度。Wzz的作用机理尚不确定。通过研究3个O-antigen链长调节器(WzzB,WzzFepE,WzzECA)的晶体结构,它们分别为五聚体、八聚体和九聚体的哑铃型,具有相似的三维结构,表明其有一个共同调节O-antigen链长的机制,这个观察结构支持了Morona模型[14]。该模型认为Wzz能够以成为伴侣的方式与Wzy和连接酶(WaaL)组成蛋白复合物,并调节Wzy和WaaL不同化学计量比例而影响O-antigen聚合动力学[14]。但也有与该模型不同的结果,Woodward等通过体外重组了O-antigen的合成,发现O-antigen的链长度可单独由Wzy和Wzz决定[14]。
EPS的合成与O-antigen中Wzy-dependent途径相似,在EPS的生物合成系统中,也发现了类似Wzx,Wzy和Wzz的物质[1]。80种大肠杆菌(Escherichia coli)血清型荚膜多糖分4个群,群1和4具有共同的组装系统,与群2和3有根本的不同。群1和4具有与O-antigen相同的Wzy-dependent途径,群1和4的合成依赖ABC-transporter dependent途径[19]。可以看出,Wzy-dependent途径是微生物分布最普遍的合成途径。
ABC-transporter dependent途径:该途径存在于E.coli O8,O9,O52等、Klebsiella.pneumoniaeO1,O2,O12和小肠结肠炎耶尔森菌(Yersiniaenterocolitica)中的O-antigen合成中,也被发现在流感嗜血杆菌(Haemophilusinfluenzae)、E.coli和Neisseriameningitidis荚膜多糖群2和3合成中[1,20-22]。O-antigen链是在内膜细胞质中浅层合成,其通过起始糖基转移酶WecA催化的链反应后,一个特异性的糖基转移酶先在乙酰氨基葡萄糖十一异戊二烯二磷酸(GlcNAc-PP-Und)上添加一个糖残基作为重复单元合成的糖基受体。如在E.coli O9a中,糖基转移酶WbdC转移单个甘露糖残基到GlcNAc中形成糖基受体[1]。在K.pneumoniaeO2a中,双功能半乳糖基转移酶WbbO能转移吡喃半乳糖残基和呋喃半乳糖残基到GlcNAc中形成糖基受体[23]。进一步的链延伸由单功能或多功能糖基转移酶将糖残基依次转移到Und-PP连接的糖基受体的非还原端。在聚合反应完成后,新生糖链由属于ABC转运体(经Wzm编码的2个跨膜结构域(TMDs)和Wzt编码的2个核苷酸结合域(NBDs))转位通过内膜。转运的具体细节和机理依然不明。该途径不用Wzz来调节链的长度,而采用完全不同的方式。O-antigen链的长度可通过终端糖残基的共价修饰来调节,如E.coliO9a的甲基化修饰[24],K.pneumoniaeO2调节与ABC-transporter dependent相关的其它多糖合成组件的化学计量[25]。
荚膜多糖群2、3的合成中,虽然也用ABC-transporter dependent途径,但起始糖基转移酶是将第一糖残基转运到聚2-酮-3-脱氧辛酮糖酸连接体(poly-Kdo linker)中。另外除需要由2个跨膜结构域(KpsM)和2个的核苷酸结合域(KpsT)组成的转运体外,还需要多糖共聚合酶(PCP-3)家族(KpsE)和外膜多糖(OPX)蛋白(KpsD)。这些蛋白组成了蛋白复合物,并以一种类似于药物外排泵的方式将多糖从细胞质转运到细胞表面[26]。
Synthase-dependent pathway途径:直到现在,该途径仅在Salmonellaenterica质粒编码的O54抗原的合成中观察到[26]。该途径是起始糖基转移酶WecA催化反应后,WbbE转移第一个UDP-ManNAc到UndPP-GlcNAc作为糖基受体。WbbF蛋白似乎完成以后所有的步骤。WbbF属于进行性糖基转移酶家族(合酶家族),该家族蛋白涉及合成多种多糖(如透明质酸、软骨素、细菌纤维素等)。WbbF含有两个保守的功能域,一个是负责按顺序依次转移ManNAc来延伸糖链,另一个具有Wzx的功能,即将多糖反转到内膜面向周质空间的一侧[20]。
1.4连接和终止
在细菌内膜向周质空间的一侧,将O-antigen从PS-PP-Und上转移到类脂A核心寡糖上形成完整的脂多糖(Lipopolysaccharides,LPS)[27-28]。该过程在上述3种糖链组装和翻转方式中是相同的,但具体反应机理尚不清楚。仅知道一个WaaL是参与连接的酶类,它的连接功能在外试验已经得到确认[27-28]。近年来,提出该酶为一个不依赖金属的反向寡糖转移酶,能够利用位于周质暴露区域的精氨酸和组氨酸残基转移O-antigen到LPS外核,形成β-自由连锁[34-35]。WaaL对还原端的糖残基具有低的特异性。在一些条件下,甚至能够连接可拉酸(包括非还原末端的葡萄糖)到LPS外核[27-28]。其底物特异性由天然的LPS外核的糖残基受体决定[29]。
2微生物多糖生物合成途径的相关基因
多糖合成过程中涉及大量的基因,大多数基因存在多糖合成基因簇中。随着基因测序技术的发展,大量的多糖合成基因簇被发现和研究。现将不同分类多糖的基因簇进行阐述。
在O-antigen基因簇中,E.coli是研究最为深入的。Plainvert等对血清型O45菌株E.colimeningitis clone:K1:H7的基因簇进行了序列分析,推测该基因簇位于尿苷三磷酸-葡萄糖-1-磷酸尿苷酰转移酶(galF)和6-磷酸葡萄糖酸脱氢酶(gnd)之间。经序列比对推测含有的9个开放阅读框的功能:依次为4个涉及6-脱氧-L-塔罗糖合成途径的酶(rmlABC和Tll)、假蛋白(wbvA)、Wzy、Wzz、糖基转移酶(wbvB)、O-乙酰转移酶(wbvC)。通过突变体的功能分析鉴定了Wzy能有效的对寡糖亚基产生聚合效应[30]。最近,报道了对大量不同血清型O-antigen基因簇的序列比对分析,将使我们更全面的理解该类基因簇中基因的结构与功能。184个血清型O-antigen基因簇均位于galF和gnd之间,galF、gnd和UDP-葡萄糖6磷酸脱氢酶(ugd)是保守基因。一些糖核苷酸合成基因如从dTDP-葡萄糖合成dTDP-L-鼠李糖途径基因(rmlBDAC),广泛分布在56个血清型O-antigen基因簇中。N-乙酰神经氨酸合成前体胞嘧啶核苷酸-N-乙酰神经氨酸仅存在6个血型型O-antigen基因簇。至少有49基因簇中发现没有发现糖核苷酸合成途径基因,暗示这些基因位于基因簇外。每个基因簇中存在2~6个推测的糖基转移酶基因,糖基转移酶1家族(pf00534)和糖基转移酶2家族(pf00535)分布最广。几乎所有基因簇都存在了Wzy-dependent途径基因Wzx/Wzy或ABC-transporter dependent途径基因Wzm/Wzt(血清型O14和O57列外,其可能不存在基因簇)[31]。46个血清型的Salmonella以GlcNAc-/GalNAc起始O-antigens合成基因簇分析表明:大多基因簇也定位于galF和gnd之间,存在Wzx/Wzy基因,4个血清型基因簇中仅存在Wzx或Wzy(除Salmonella O54and O67基因簇外,且其存在Wzm/Wzt基因)。在一些以乳糖起始合成的SalmonellaO抗原基因簇具有盒子结构,即基因簇中间为血清特异性基因,两侧为一些高度同源的基因。但在这46个GlcNAc-/GalNAc起始血清型SalmonellaO抗原基因簇的基因高度多样性,没有这种盒子结构[20]。在所有肠杆菌中O-antigen合成中,起始糖基转移酶WecA不位于基因簇中[20]。
在ESP基因簇中,研究最为深入的是黄原胶和乳酸菌ESP。LactococcuslactisNIZO B40 ESP基因簇在乳酸菌中研究最为深入。Van Kranenburg等对该基因簇(EspRXABCDEFGHIJKL)的序列比对分析表明:EspR是调节的基因。在聚合和输出过程,EspA-B参与决定多糖链长度。EspDEFGH为糖基转移酶功能。EspI仅涉及到聚合功能,EspK仅为输出功能。对糖基转移酶进行同源和异源表达,确认EpsD是起始葡萄糖基转移酶,EpsE和EpsF是将葡萄糖核苷酸转移连接到脂载体上,EpsG负责将半乳糖连接到与脂载体结合纤维二糖,EpsJ涉及到半乳糖磷酸转移酶功能或者是从脂载体产生骨架寡糖,EspB通过磷酸化来控制多糖的合成[32-35]。在比对5个L.lactis菌株的ESP基因簇序列,EpsD是保守的,EpsE序列具有菌株特异性[36]。最近,大量序列比对分析也应用在ESP合成基因簇中,50条Oenococcusoeni基因组序列分析,分布了两个基因簇(eps1和eps2)。eps1基因簇序列分析,将其分为模式A、B、C三种。超过50%的基因高度保守,ugd和吡喃半乳糖酶(glf)是最保守。模式A糖基转移酶基因组成最简单(Woa A、B、C、D、E)。与模式A不同,模式B、C出现其它的糖基转移酶基因或不存一些糖基转移酶基因。无论哪种模式,基因簇中都包括起始糖基转移酶基因woaA。所有的模式均存在Wzz、Wzy、Wzx基因。eps2基因簇序列分析表明,其较eps基因簇复杂。可将其分为15个模式,模式中包括有以下推测的功能基因。5′端高度保守的调节蛋白(AraC,Wzd,Wze)、13个不同聚合酶(Wzy)、9个折叠酶家族、3个序列差异的起始糖基转移酶WobA、其它各种类转移酶、多糖前体合成酶等[37]。
荚膜多糖基因簇研究最多的在Escherichiacoli中,它可为革兰氏阴性菌的模式基因簇。80个E.coli血清型分为4个群。K30为群1的模式血清型,其还存在于K.pneumoniae、Erwinia、创伤弧菌(Vibriovulnificus)等。K1、K5与K10、K54分别为群2、群3的模式血清型,它们还存在于N.meningitidis、Haemophilusinfluenzae等。K40和O111为群4的模式血清型,据我们所知仅存在于Escherichiacoli[19,38]。其它菌属荚膜基因簇与大肠杆菌基因簇的结构具有显著的相似性,可归为相应荚膜多糖群的基因簇。EscherichiacoliK30荚膜多糖群1基因簇定位于galF和gnd基因之间,有12个阅读框。依次有组装新生CPS功能的Wzi、输出聚合物到表面的Wza、起去磷酸化的基因Wzb和Wzc、4个糖基转移酶(起始糖基转移酶WbaP、WcaN、WcaO和WbaZ)、Wzx、Wzy、功能不清楚的orfY、orfZ。其中,起表达表面多糖作用的Wza、Wzb和Wzc基因在1群是保守的[39]。在Vibriovulnificus荚膜多糖群1基因簇也具有保守基因(wza、wzb和wzc),并与E.coli有较高的同源性,但第一个开发阅读框序列在Genbank中没有同源性,不同Vibriovulnificus菌株之间多糖合成基因存在多样性[38]。群4基因簇也出现了与群1有一样的保守基因(Wza、Wzb和Wzc)[40]。K5群2基因簇分为3个独立依赖温度调节的功能区。功能区1包括基因(kpsFEDUCS),kpsFED具有输出或转运功能,kpsUCS与磷脂酰-3-脱氧-D-甘露-辛酮糖的合成有关。功能区2为血清型特异基因(kfiA,B,C and D),kfiA为起始糖基转移酶、KfiC为糖基转移酶,kfiD为UDP-葡萄糖脱氢酶。功能区3包括涉及ABC-transporter dependent途径的kpsM和kpsT。两端的KpsC、KpsD、KpsE、KpsM、KpsS和KpsT为保守区域[41]。流感嗜血杆菌b型荚膜多糖基因簇与K5群2基因簇相似,即中间是与多糖生物合成相关的血清型特异的区域2,两端是在所有流感嗜血杆菌中都保守的区域1和3[42]。群3也出现与群2基因簇具有一样的保守基因(kpsM和kpsT)[40]。
在革兰氏阳性菌中,Streptococcus荚膜多糖基因簇研究最多。35个血清型Streptococcussuis荚膜多糖基因簇与S.suisP1/7全基因组比较,22个血清型的CPS基因簇序列定位于染色体基因orfZ到roA之间,其它13个位于其它基因的两侧。其与乳糖起始合成的SalmonellaO抗原基因簇一样具有盒子结构,即基因簇中5′具有4个高度保守的基因(cpsABCD)。cpsA具有多糖合成调节作用,缺失cpsB不产生荚膜,cpsC和cpsD可能与蛋白的输出有关。3′端相对保守序列。中间区域基因功能包括起始糖基转移酶、多种其它糖基转移酶、多种糖苷前体合成酶、翻转和聚合酶等,各血清型基因簇间(除血清型4)在这个区域展示多样性(存在一个以上特异血清型基因)。所有血清型荚膜多糖合成模式均为Wzx/Wzy pathway途径[43]。在90个血清型S.pneumoniaecapsular基因簇中也存在盒子结构,上游位置也存在高度保守的4个合成调节基因(cpsABCD),基因簇内包含多种糖基转移酶、多糖合成酶、乙酰基转移酶和翻转酶等[44]。其它革兰氏阳性菌S.aureus血清5型荚膜多糖基因簇中,存在16个基因紧密连接的基因,中间区域的基因也存在血清型特异性[45]。
3微生物多糖的代谢工程
随着科学家取得了大量关于微生物多糖生物合成途径和相关基因的研究成果,多糖合成的代谢工程方面的研究逐渐展开。代谢工程是重组DNA技术通过改变细胞内有关的酶活、酶量和输送体系、调节功能,改变细胞的遗传特性以改进微生物某些代谢活性,最大限度的提高目的产物产率的一门技术[46]。微生物多糖合成的代谢工程的研究可以分为两个目标:一个是提高多糖的产量,另外一个是改变多糖的结构。
提高微生物多糖的产量有可能能带来生产效益的提高,降低生产成本。为此近年来国内外研究人员在这方面进行了大量的代谢工程研究。归纳起来可以通过以下策略实现:(1)通过调控涉及糖核苷酸合成途径的酶水平来提高胞外多糖的产量。S.thermophilusLY03在以乳糖为碳源生长时,过表达催化6-磷酸葡萄糖转化葡萄糖-1-磷酸的葡萄糖磷酸变位酶和涉及合成UDP-葡萄糖的UDP-葡萄糖焦磷酸化酶,提高Leloir途径的酶活,提高Leloir途径的酶或和过表达UDP-葡萄糖焦磷酸化酶均能提高胞外多糖产量。通过敲除葡萄糖磷酸变位酶基因和提高Leloir途径的酶活也能提高胞外多糖产量[47]。Leloir途径的UDP半乳糖4-差向异构酶被认为是合成lactic acid bacteria胞外多糖的关键酶[48],Welman AD等通过提高该酶的活力达到提高胞外多糖的产量[46]。但也有一些提高糖核苷酸合成途径的酶活力不能提高多糖产量的报道。在菌株L.lactisUDP-葡萄糖焦磷酸化酶,能够大幅度提高UDP-葡萄糖和UDP-半乳糖的合成能力,胞外多糖产量却没有提高[49]。在其他的乳酸菌和药用菌灵芝中过量表达UDP-葡萄糖焦磷酸化酶,又能提高胞外多糖产量[46,50]。上述研究表明,通过调控涉及糖核苷酸合成途径的酶水平来提高多糖产量的方式具有较大的灵活性。(2)增加多糖合成基因簇基因在微生物中的表达,产生更高的多糖合成代谢流。用高拷贝载体pIL253进行L.lactisNIZO B40 eps基因簇的拷贝,结果发现虽然细胞生长速率由此而降低,EPS的产量则提高了4倍[51]。将多个糖基转移酶基因转入菌株Sphingomonas,增强了糖基转移酶活力,使胞外多糖产量提高了20%[52]。其它策略:一些研究暗示能量和辅酶水平可能提高多糖合成。调节辅酶平衡(NADH/NAD+)能够减少碳代谢流向乳酸,而更多的流向其它代谢物,减少乳酸对乳酸菌生长的胁迫[7,53]。最近通过在LactobacilluscaseiLC2W过表达NADH氧化酶,能使多糖产量提高46%[53]。但在多糖中的透明质酸的研究中,在Streptococcus过表达NADH氧化酶,能量的合成能力提高了30%,生物量提高了15%,透明质酸的产量却没有提高[50];在透明质酸代谢工程研究中,科研人员试图通过敲除透明质酸降解酶,来提高其产量[54]。在菌株S.zooepidemicus共表达有增强细胞摄氧的作用的vgb基因和透明质酸合成hasABC基因,能使透明质酸产量提高了30%[55];在海藻糖代谢工程研究中,通过在P.aeruginosa过表达海藻糖盐合成途径间接调控因子CupB5蛋白和ClpXP proteases基因,海藻糖的产量也能获得提高[56-57]。值得注意的是,近年来Woodward等首先通过化学合成方法制备了多糖合成的中间物质(N-乙酰-d-半乳胺),然后采用依次将酶糖基化的方法,构建了Eoli86寡糖重复单元,再成功的表达出聚合酶Wzy,最终实现了体外合成O-antigen多糖[58]。这项技术虽然超出了代谢工程的范畴,但是其为多糖的产量的提高提供更为广阔的研究空间。
通过代谢工程还能改变多糖的结构,这将有可能改变多糖的物理、化学和生物活性等,为多糖的应用展现更为优越性能。菌株L.delbrueckiisubsp.bulgaricus培养在以果糖为碳源的培养基中,胞外多糖的葡萄糖和半乳糖的比列为1∶2.4。培养在果糖和葡萄糖的混合物中,胞外多糖的葡萄糖、半乳糖和鼠李糖比列为1∶7.0∶0.8[59]。菌株L.rhamnosusE/N在分别以葡萄糖、半乳糖、蔗糖、乳糖、麦芽糖为唯一碳源发酵产的胞外多糖。经凝胶色谱研究表明,半乳糖、蔗糖和乳糖发酵产的多糖具有高、低分子量分布,麦芽糖和葡萄糖发酵产的多糖仅有高分子量分布。原子力显微镜分析表明多糖的分子量比例和聚合链长度不同,且与多糖溶液粘度相关[60]。这些研究结果暗示了通过代谢工程调整碳的代谢流,可以改变多糖的组分、多糖分子量分布、空间构象等结构特征。就在最近,利用代谢工程改变碳代谢进行结构修饰已有成功的实例。Yi等[18]利用含有岩藻糖的E.coliO86 O抗原多糖为研究模式,将Bacterioidesfragilis的GDP-岩藻糖补救代谢途径(能够高效利用培养基中岩藻糖类似物合成GDP-岩藻糖类似物)取代E.coliO86自身的GDP-岩藻糖从头合成代谢途径,将该工程菌生长在岩藻糖类似物中,发现O抗原多糖被该岩藻糖类似物进行了结构同源修饰。将新的多糖合成元件(包括基因簇和糖基转移酶)导入到微生物体内将产生新的多糖合成途径,将有可能改变多糖的单糖组分。将含有S.thermophilusSfi6的多糖合成基因簇的质粒转入不产胞外多糖的L.lactisMG1363菌株中,使L.lactisMG1363能够合成胞外多糖,该多糖重复单元的乙酰半乳糖胺变成了半乳糖,而且糖基侧链缺失。这可能是由于产乙酰半乳糖胺能力的丢失,导致骨架上半乳糖而不是乙酰半乳糖胺[61]。
4灵芝药用模式真菌多糖合成和代谢工程的研究
虽然一些精细的微生物多糖合成途径、相关基因的结构与功能的研究上还不是很清楚,但在该研究领域(尤其在细菌中)已经取得了前所未有的成果。通过代谢工程技术已也大幅度的提高了透明质酸、海藻酸钠和乳酸菌ESP的产量,并在多糖结构改性中展现了应用前景。然而这些研究在药用模式真菌——灵芝(属于真菌界、真菌门、担子菌亚门层菌纲、非褶菌目、灵芝科、灵芝属真菌)中几乎还是空白。近10多年来灵芝多糖的药理作用和结构特征已基本明确,然而其发酵多糖产量和多糖结构急待通过代谢工程技术得以改善。代谢工程技术应用的基础是充分明晰其生物合成和相关基因簇,灵芝多糖合成和代谢工程研究仅局限于对IPS-1-1合成途径进行了初步推测[8],迫切要求我们这一代科研人员进行更为深入的研究。近期,随着中国药用植物研究所采用二代测序技术及光学图谱完成绘制整染色体级的灵芝基因组精细图[62],为灵芝多糖代谢工程研究提供了一定的生物学信息。国家“863”课题中又对企业科研机构与高校共同承担的灵芝多糖合成方面的研究给予大力支持,目前课题组已展开了灵芝多糖生物合成途径分析和基因簇的研究工作,并最终要实现大幅度提高多糖产量和改善多糖生物活性功能。
[参考文献]
[1]WANG Peng,GUO Hong-jie,YI Wen,et al.Current understanding on biosynthesis of microbial polysaccharides[J].Current topics in medicinal chemistry,2008,8(2):141-151.
[2]FREITAS F,ALVES V D,REIS M A.Advances in bacterial exopolysaccharides:from production to biotechnological applications[J].Trends in Biotechnology,2011,29(8):388-398.
[3]陈蕾蕾,王未名,祝清俊,等.细菌多糖的生物合成机制[J].微生物学报,2010,50(12):1583-1589.
[4]WASSER S.Medicinal mushrooms as a source of antitumor and immunomodulating polysaccharides[J].Applied microbiology and biotechnology,2002,60(3):258-274.
[5]NIE Shao-ping,ZHANG Hui,LI Wen-juan,et al.Current development of polysaccharides fromGanoderma:Isolation,structure and bioactivities[J].Bioactive Carbohydrates and Dietary Fibre,2013,1(1):10-20.
[6]VANDAMME E J,BAETS de S,STEINBüCHEL A.Biopolymers.Polysaccharides II:Polysaccharides from Eukaryotes[M].Berlin:Wiley-VCH,2002.
[7]WELMAN A D,MADDOX I S.Exopolysaccharides from lactic acid bacteria:perspectives and challenges[J].Trends in biotechnology,2003,21(6):269-274.
[8]刘高强,赵艳,王晓玲,等.灵芝多糖的生物合成和发酵调控[J].菌物学报.2011,30(2):198-205.
[9]RUAS-MADIEDO P,HUGENHOLTZ J,ZOON P.An overview of the functionality of exopolysaccharides produced by lactic acid bacteria[J].International Dairy Journal,2002,12(2):163-171.
[10]SAMUEL G,REEVES P.Biosynthesis of O-antigens:genes and pathways involved in nucleotide sugar precursor synthesis and O-antigen assembly[J].Carbohydrate research,2003,338(23):2503-2519.
[11]KALYNYCH S,MORONA R,CYGLER M.Progress in understanding the assembly process of bacterial O-antigen[J].FEMS microbiology reviews,2014,38(23):1048-1065.
[12]赵时玮,任静,王荫榆,等.乳酸菌胞外多糖生物合成与遗传调控研究进展[J].山西农业科学,2010,38(12):94-99.
[13]WHITFIELD C,PAIMENT A.Biosynthesis and assembly of Group 1 capsular polysaccharides inEscherichiacoliand related extracellular polysaccharides in other bacteria[J].Carbohydrate research,2003,338(23):2491-2502.
[14]KALYNYCH S,MORONA R,CYGLER M.Progress in understanding the assembly process of bacterial O-antigen[J].FEMS microbiology reviews,2014,38(5):1048-1065.
[15]HONG Y,CUNNEEN M M,REEVES P R.The Wzx translocases forSalmonellaentericaO-antigen processing have unexpected serotype specificity[J].Molecular microbiology,2012,84(4):620-630.
[16]DANIELS C,VINDURAMPULLE C,MORONA R.Overexpression and topology of theShigellaflexneriO-antigen polymerase(Rfc/Wzy)[J].Molecular microbiology,1998,28(6):1211-1222.
[17]PERLSTEIN D L,ZHANG Yi,WANG Tsung-shing,et al.The direction of glycan chain elongation by peptidoglycan glycosyltransferases[J].Journal of the American Chemical Society,2007,129(42):12674-12675.
[18]YI Wang,LIU Xian-wei,LI Yan-hong,et al.Remodeling bacterial polysaccharides by metabolic pathway engineering[J].Proceedings of the National Academy of Sciences,2009,106(11):4207-4212.
[19]WHITFIELD C.Biosynthesis and assembly of capsular polysaccharides inEscherichiacoli[J].Annu.Rev.Biochem.,2006(75):39-68.
[20]LIU Bin,KNIREL Y A,FENG Lu,et al.Structural diversity in Salmonella O antigens and its genetic basis[J].FEMS microbiology reviews,2014,38(1):56-89.
[21]WILLIS L M,WHITFIELD C.Structure,biosynthesis,and function of bacterial capsular polysaccharides synthesized by ABC transporter-dependent pathways[J].Carbohydrate research,2013(378):35-44.
[22]KIDO N,TORGOV V I,SUGIYAMA T,et al.Expression of the O9 polysaccharide ofEscherichiacoli:sequencing of the E.coli O9 rfb gene cluster,characterization of mannosyl transferases,and evidence for an ATP-binding cassette transport system[J].Journal of bacteriology,1995,177(8):2178-2187.
[23]CLARKE B R,BRONNER D,KEENLEYSIDE W J,et al.Role of Rfe and RfbF in the initiation of biosynthesis of D-galactan I,the lipopolysaccharide O antigen fromKlebsiellapneumoniaeserotype O1[J].Journal of bacteriology,1995,177(19):5411-5418.
[24]CLARKE B R,CUTHBERTSON L,WHITFIELD C.Nonreducing terminal modifications determine the chain length of polymannose O antigens ofEscherichiacoliand couple chain termination to polymer export via an ATP-binding cassette transporter[J].Journal of Biological Chemistry,2004,279(34):35709-35718.
[25]PFOESTL A,HOFINGER A,KOSMA P,et al.Biosynthesis of dTDP-3-acetamido-3,6-dideoxy-α-D-galactose inAneurinibacillusthermoaerophilusL420-91T[J].Journal of Biological Chemistry,2003,278(29):26410-26417.
[26]WILLIS L M,WHITFIELD C.Structure,biosynthesis,and function of bacterial capsular polysaccharides synthesized by ABC transporter-dependent pathways[J]. Carbohydrate research,2013,378:35-44.
[27]HAN Wei-qing,WU Bao-lin,LI Lei,et al. Defining function of lipopolysaccharide O-antigen ligase WaaL using chemoenzymatically synthesized substrates[J].Journal of Biological Chemistry,2012,287(8):5357-5365.
[28]RUAN X,LOYOLA D E,MAROLDA C L,et al.The WaaL O-antigen lipopolysaccharide ligase has features in common with metal ion-independent inverting glycosyltransferases[J].Glycobiology,2012,22(2):288-299.
[29]SCHILD S,LAMPRECHT A K,REIDL J.Molecular and functional characterization of O antigen transfer inVibriocholerae[J].Journal of Biological Chemistry,2005,280(27):25936-25947.
[30]PLAINVERT C,BIDET P,PEIGNE C,et al.A new O-antigen gene cluster has a key role in the virulence of theEscherichiacolimeningitis clone O45:K1:H7[J].Journal of bacteriology,2007,189(23):8528-8536.
[31]IGUCHI A,IYODA S,KIKUCHI T,et al.A complete view of the genetic diversity of theEscherichiacoliO-antigen biosynthesis gene cluster[J].DNA Research,2015,22(1):101-107.
[32]KRANENBURG van R,SWAM van I I,MARUGG J D,et al.Exopolysaccharide biosynthesis inLactococcuslactisNIZO B40:functional analysis of the glycosyltransferase genes involved in synthesis of the polysaccharide backbone[J].Journal of bacteriology,1999,181(1):338-340.
[33]V KRANENBURG van R,VOS H R,SWAM van I I,et al.Functional analysis of glycosyltransferase genes fromLactococcuslactisand other gram-positive cocci:complementation,expression,and diversity[J].Journal of bacteriology,1999,181(20):6347-6353.
[34]LAWS A,GU Y,MARSHALL V.Biosynthesis,characterisation,and design of bacterial exopolysaccharides from lactic acid bacteria[J].Biotechnology advances,2001,19(8):597-625.
[35]GROOT N,KLEEREBEZEM M.Mutational analysis of theLactococcuslactisNIZO B40 exopolysaccharide(EPS) gene cluster:EPS biosynthesis correlates with unphosphorylated EpsB[J].Journal of applied microbiology,2007,103(6):2645-2656.
[36]SUZUKI C,KOBAYASHI M,KIMOTO-NIRA H.Novel exopolysaccharides produced byLactococcuslactissubsp.lactis,and the diversity of epsE genes in the exopolysaccharide biosynthesis gene clusters[J].Bioscience Biotechnology Biochemistry,2013,77(10):2013-2018.
[37]DIMOPOULOU M,VUILLEMIN M,CAMPBELL-SILLS H,et al.Exopolysaccharide(EPS) synthesis byOenococcusoeni:from genes to phenotypes[J].PloS one,2014,9(6):e98898.
[38]CHATZIDAKI-LIVANIS M,JONES M K,WRIGHT A C.Genetic variation in theVibriovulnificusgroup 1 capsular polysaccharide operon[J].Journal of bacteriology,2006,188(5):1987-1998.
[39]RAHN A,WHITFIELD C.Transcriptional organization and regulation of theEscherichiacoliK30 group 1 capsule biosynthesis(cps) gene cluster[J].Molecular microbiology,2003,47(4):1045-1060.
[40]CHEN Yuan-sha,DAI Jian-li,MORRIS J G,et al.Genetic analysis of the capsule polysaccharide(K antigen) and exopolysaccharide genes in pandemicVibrioparahaemolyticusO3:K6[J].BMC microbiology,2010,10(1):274.
[41]RIGG G P,BARRETT B.The localization of KpsC,S and T,and KfiA,C and D proteins involved in the biosynthesis of theEscherichiacoliK5 capsular polysaccharide:evidence for a membrane-bound complex[J].Microbiology,1998,144(10):2905-2914.
[42]KROLL J S,LOYNDS B,BROPHY L N,et al.The bex locus in encapsulatedHaemophilusinfluenzae:a chromosomal region involved in capsule polysaccharide export[J].Molecular microbiology,1990,4(11):1853-1862.
[43]OKURA M,TAKAMATSU D,MARUYAMA F,et al.Genetic analysis of capsular polysaccharide synthesis gene clusters from all serotypes ofStreptococcussuis:potential mechanisms for generation of capsular variation[J].Applied and environmental microbiology,2013,79(8):2796-2806.
[44]MAVROIDI A,AANENSEN D M,GODOY D,et al.Genetic relatedness of theStreptococcuspneumoniaecapsular biosynthetic loci[J].Journal of bacteriology,2007,189(21):7841-7855.
[45]O’RIORDAN K,LEE J C.Staphylococcusaureuscapsular polysaccharides[J].Clinical microbiology reviews,2004,17(1):218-234.
[46]WELMAN A D,MADDOX I S,ARCHER R H.Metabolism associated with raised metabolic flux to sugar nucleotide precursors of exopolysaccharides inLactobacillusdelbrueckiisubsp. bulgaricus[J].Journal of Industrial Microbiology and Biotechnology,2006,33(5):391-400.
[47]LEVANDER F,SVENSSON M,Rådström P.Enhanced exopolysaccharide production by metabolic engineering ofStreptococcusthermophilus[J].Applied and environmental microbiology,2002,68(2):784-790.
[48]MOZZI F,SAVOY de G G,FONT de V G.UDP-galactose 4-epimerase:a key enzyme in exopolysaccharide formation byLactobacilluscaseiCRL 87 in controlled pH batch cultures[J].Journal of applied microbiology,2003,94(2):175-183.
[49]BOELS I C,RAMOS A,KLEEREBEZEM M,et al.Functional analysis of theLactococcuslactisgalU and galE genes and their impact on sugar nucleotide and exopolysaccharide biosynthesis[J].Applied and environmental microbiology,2001,67(7):3033-3040.
[50]张帆,钟威,穆虹,等.过量表达OsUgp2基因提高紫芝多糖含量[J].菌物学报,2011,30(3):442-452.
[51]BOELS I C,KRANENBURG van R,KANNING M W,et al.Increased exopolysaccharide production inLactococcuslactisdue to increased levels of expression of the NIZO B40 eps gene cluster[J].Applied and environmental microbiology,2003,69(8):5029-5031.
[52]RUFFING A,CHEN R R.Metabolic engineering of microbes for oligosaccharide and polysaccharide synthesis[J].Microbial cell factories,2006,5(1):25.
[53]LI Nan,WANG Yuan-long,ZHU Ping,et al.Improvement of exopolysaccharide production inLactobacilluscaseiLC2W by overexpression of NADH oxidase gene[J].Microbiological Research,2015(171):73-77.
[54]崔亚娜,苏旭东,王羽,等.兽疫链球菌透明质酸分解酶基因的敲除[J].中国生物工程杂志,2009,29(12):94-99.
[55]郝宁,张晋宇,陈国强.在兽疫链球菌中表达vgb基因和HA合成基因提高透明质酸产量[J].中国生物工程杂志,2005,25(6):56-60.
[56]QIU D,EISINGER V M,HEAD N E,et al.ClpXP proteases positively regulate alginate overexpression and mucoid conversion inPseudomonasaeruginosa[J].Microbiology,2008,154(7):2119-2130.
[57]REGT A K,YIN Y,WITHERS T R,et al.Overexpression of CupB5 activates alginate overproduction inPseudomonasaeruginosaby a novel Alg Wdependent mechanism[J].Molecular microbiology,2014,93(3):415-425.
[58]WOODWARD R,YI Wen,LI Lei,et al.In vitro bacterial polysaccharide biosynthesis:defining the functions of Wzy and Wzz[J].Nature chemical biology,2010,6(6):418-423.
[59]GROBBEN G J,SMITH M R,SIKKEMA J,et al.Influence of fructose and glucose on the production of exopolysaccharides and the activities of enzymes involved in the sugar metabolism and the synthesis of sugar nucleotides inLactobacillusdelbrueckiisubsp.bulgaricus NCFB 2772[J].Applied microbiology and biotechnology,1996,46(3):279-284.
[60]POLAK-BERECKA M,CHOMA A,WAKO A,et al.Physicochemical characterization of exopolysaccharides produced byLactobacillusrhamnosuson various carbon sources[J].Carbohydrate polymers,2015(117):501-509.
[61]STINGELE F,VINCENT S J F,FABER E J,et al.Introduction of the exopolysaccharide gene cluster fromStreptococcusthermophilusSfi6 intoLactococcuslactisMG1363:production and characterization of an altered polysaccharide[J].Molecular microbiology,1999,32(6):1287-1295.
[62]CHEN Shi-lin,XU Jiang,LIU Chang,et al.Genome sequence of the model medicinal mushroomGanodermalucidum[J].Nature communications,2012(3):913.
[责任编辑:谢 平]
Progress in research on biosynthesis and metabolic engineering
of microbial polysaccharides
ZENG Hua-wei1,2,ZHENG Hui-hua1,2,CHEN Hui1,2,LIAO Xiang-ru3,CAI Yu-jie3
(1.Jiangsu Alphay Biotechnology Co.Ltd, Nantong 226009, China;
2.Institute of Microbiology Chinese Academy of Sciences-Jiangsu Alphay Biotechnology
Co. Ltd Joint Laboratory of Medicinal Fungi, Nantong 226009, China;
3.Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University,
Wuxi 214122, China)
Abstract:This paper reviewed progress in research on biosynthetic pathways of microbial polysaccharide, gene cluster structure and function, and metabolic engineering. Biosynthetic pathways of Fungi polysaccharide include: the synthesis of precursor nucleotide sugars (the activated form of monosaccharide), the starting reaction of the synthesis, the extension, flip and aggregation of the repeating unit, and polysaccharides output. Polysaccharide synthesis process involves a large number of genes, most of which are present in the polysaccharide synthesis gene cluster. With the development of genetic sequencing technology, a large number of polysaccharide synthesis gene clusters have been found and researched. Studies have shown that regulating enzymes involved in sugar nucleotide synthesis pathway can increase the production of extracellular polysaccharide. The increase of polysaccharide gene cluster expression in microorganisms can produce higher polysaccharide anabolic flow.
Key words:microbial polysaccharides;biosynthesis;gene clusters;metabolic engineering

