SP-B基因内含子4区(CA)n模体上SRp20蛋白结合位点的初步研究*
倪 岚, 王桂荣, 杨 炯△
1武汉大学中南医院呼吸内科,武汉 4300712美国纽约州立大学上州医学院外科,纽约州 13210
SP-B基因内含子4区(CA)n模体上SRp20蛋白结合位点的初步研究*
倪 岚1, 王桂荣2, 杨 炯1△
1武汉大学中南医院呼吸内科,武汉 4300712美国纽约州立大学上州医学院外科,纽约州 13210
目的 探讨SP-B基因内含子4区第8号(CA)n模体上可能存在的剪切蛋白结合位点,为进一步研究第8号(CA)n模体影响SP-B mRNA剪切的机制提供实验基础。方法 软件分析特定(CA)n模体的潜在RNA结合蛋白,并合成含有特定蛋白潜在结合位点的模体RNA序列的探针及突变序列探针,用凝胶阻滞迁移(EMSA)技术研究相关(CA)n模体的RNA序列与其潜在结合蛋白的相互作用。结果 软件分析预测第8号(CA)n模体上可能存在特定剪切蛋白SRp20的结合位点;EMSA实验中当抗体或突变探针竞争时特异性结合带(迁移带)均消失,显示第8号(CA)n模体RNA序列上的“CAUC”能够与SRp20特异性结合。结论 SP-B内含子4区第8号(CA)n模体的保守序列上存在SRp20的特异性结合位点,从而可能影响SP-B mRNA剪切。
SP-B基因; RNA结合蛋白; SRp20
精确的剪切过程有赖于剪切位点的正确识别以及各种剪切蛋白和RNA之间的相互作用。剪切调控蛋白可结合到称为外显子/内含子剪切增强子(ESE/ISE,exonic/intronic splicing enhancer)或者外显子/内含子剪切沉默子(ESS/ISS,exonic/intronic splicing silencer)的特殊RNA序列,前者增强附近的剪切位点的剪切,后者正好相反。人类SP-B基因内含子4区的前半部分存在11个以(CA)n为特征的模体(motif),每个模体由一段20个碱基左右的保守序列和CA重复序列(2到17个)构成(图1)。我们的前期研究显示SP-B基因内含子4区第8号(CA)n模体对SP-B mRNA剪切产生明显的增强作用[1],第8号模体可能为剪切增强子,但其具体作用方式并不清楚。我们利用在线软件分析了内含子4区相关(CA)n模体的特定RNA序列的潜在RNA结合蛋白,并利用凝胶阻滞迁移技术(EMSA)研究第8号模体保守序列上的RNA序列与其潜在结合蛋白SRp20的相互作用,探讨第8号(CA)n模体上可能存在的剪切蛋白结合位点,为进一步研究第8号(CA)n模体影响SP-B mRNA剪切的机制提供实验基础。
1 材料与方法
1.1 实验材料和仪器
CHO-K1细胞(CCL-61)购于ATCC中心;SRSF3(NM_003017)Human cDNA ORF克隆、TurboFectin 8.0转染试剂盒购于Origene公司;pEGM-11Z载体连接试剂盒购于Promega公司;Quick change lightning site-directed mutagenesi试剂盒购于Stratagene公司;质粒(大)小规模抽提试剂盒、凝胶纯化试剂盒购于Qiagen公司;RNA抽提柱购于Zymo Research公司;AmpliScribe T7-Flash Biotin-RNA转录试剂盒购于Epicentre公司;NE-PER核-胞浆蛋白制备试剂盒、BCA蛋白分析试剂盒、化学发光RNA EMSA试剂盒、化学发光核酸检测试剂盒购于Thermo Scientific公司;SRp20抗体1(抗SFRS3抗体)ab73891、SRp20抗体2 ab80291、抗beta-actin抗体(BA3R)ab125248、二抗山羊抗兔IgG(H+L)-HRP购于Abcam公司;PVDF膜购于Millipore公司;正极尼龙膜购于Roche公司;RNA-bee购于TEL-TEST公司。台式低速离心机、高速低温离心机购于Eppendorf公司;分光光度计、恒温摇床购于Fisher公司;垂直电泳槽、电转移槽购于Bio-rad公司;XL-1000UV交联仪购于Spectronics公司。
1.2 (CA)n模体区的增强子和沉默子分析及结合蛋白预测
利用可变性剪切数据库(Alternative Splicing Database,ASD)项目研究SP-B基因内含子4区的前一部分包含有11个模体加上第11号模体后的46 bp序列,分析增强子或沉默子潜在的结合位点及潜在的剪切蛋白。
1.3 相关(CA)n模体的RNA序列与其潜在结合蛋白SRp20的相互作用
1.3.1 转染及核蛋白抽提 为得到大量SRp20用于RNA凝胶阻滞实验,选取可过表达SRp20的载体SRSF3(NM_003017)Human cDNA ORF克隆转染CHO-K1细胞(中国仓鼠卵巢细胞,培养条件见参考文献[1])使后者大量表达SRp20,然后抽取核蛋白。当CHO-K1细胞汇合度为60%时,每盘(10 cm)转染6 μg载体克隆DNA。转染试剂为TurboFectin 8.0。
用NE-PER核-胞浆蛋白制备试剂盒进行细胞核蛋白抽提:冷冻PBS液洗涤细胞培养皿2次,在培养皿加入1 mL PBS,收取细胞至EP管;4℃下3 000 r/min离心5 min弃上清;将细胞悬浮于1 mL的PBS中,4℃下2 000 r/min离心10 min弃上清;目测沉淀物体积约为50 μL,将细胞悬浮于10倍体积(500 μL)的胞浆蛋白裂解液CER Ⅰ,剧烈振荡15 s使沉淀充分溶解,冰上孵育10 min;加27.5 μL胞浆蛋白裂解液CERⅡ(冰上),剧烈振荡5 s后冰上孵育1 min;剧烈振荡5 s,4℃下14 000 r/min离心5 min,上清液快速转移至EP管储存;将沉淀悬浮于250 μL核蛋白裂解液NER(冰上),剧烈振荡15 s后置于冰上孵育10 min,反复每10 min冰上孵育后振荡15 s,总共40 min;4℃下14 000 r/min离心10 min,快速转移上清(核蛋白抽提物)至EP管并于-70℃保存。
BCA法测定蛋白质浓度:按25体积BCA试剂A+24体积BCA试剂B +1体积BCA试剂C配制BCA工作液。将蛋白样品加于上述溶液中,37℃孵育60 min,移至比色皿,分别测定待测样品孔A570nm值,软件计算待测样品的蛋白含量。
1.3.2 Western blot检测 配制聚丙烯酰胺凝胶,取4 μg蛋白行SDS-PAGE电泳。预电泳电压60 V 30 min后,蛋白上样电压调为100 V电泳至溴酚蓝进入凝胶底部停止电泳。在海绵上放1张滤纸,依次放凝胶、PVDF膜、滤纸和海绵,于冰浴垂直电转槽40 V转印2 h。电转结束后取FVDF膜置于10 mL TBST内,加封闭液10 mL室温孵育1 h。4℃下加一抗,8 h后TBST洗膜。室温下加对应HRP二抗(1∶3 000)孵育1 h。洗膜后将PVDF膜于ECL试剂中孵育1 min,暗室中置于胶片曝光。凝胶成像分析系统密度扫描并测量条带的灰度值,以内参灰度值为基础,测定各组的相对比值。检测完毕后,将PVDF膜TBST洗膜10 min×3次。加入抗体洗脱液20 mL,50℃ 40 min。重新洗膜、封闭并加入抗体检测。
1.3.3 凝胶阻滞探针制备 ①pGEM-11Z M8-D(CA)及pGEM-11Z M8-G4-D(CA)质粒构建:合成含有第8号模体保守序列的质粒pGEM-11Z M8-D(CA)(即M8,pGEM-11Z为载体)以及此段序列的突变质粒pGEM-11Z M8-G4-D(CA)(即G4,用“GGGG”代替“CATC”)。M8合成引物为2053/2054,2053:5′-CCATCCAGACACATACCGCGG-CCGCATGCATAAGC-3′;2054:5′-GCTTATGCATGCGGCCGCGGTATGTGTCTGGATGG-3′。G4合成引物为2061/2062,2061:5′-GATTCGC-ACTCGGGGCAGACACATACCGCGGCCGC-3′,2062:5′-GCGGCCGCGGTATGTGTCTGCCCCG-AGTGCGAATC-3′。M8和G4以合成pcDNA3.1 minigene M8[1]过程中的pGEM-11Z M8为模板合成,片段两端已有酶切位点EcoR Ⅰ和NotⅠ。合成利用Quick change lightning site-directed mutagenesis合成试剂盒,反应系统:5 μL 10×反应缓冲液,0.5 μL模板DNA,2种引物各1 μL,1 μL dNTP混合物,1.5 μL Quick solution试剂,加40 μL ddH2O使反应总体积达50 μL。系统中加入1 μL Quick change lightning酶后行PCR。PCR过程:95℃ 2 min;95℃ 20 s,60℃ 10 s,68℃ 90 s,共18个循环;68℃ 5 min。加入2 μLDpnⅠ酶至反应体系,混合后直接放入37℃水浴5 min,转化至XL-10超级大肠埃希菌感受态细胞,扩增后进行克隆选择。
②探针合成标记:测序正确的pGEM-11Z M8-D(CA)及pGEM-11Z M8-G4-D(CA)质粒在NotⅠ酶切2 h后线性化成为模板,RNA探针体外转录合成利用AmpliScribe T7-Flash Biotin-RNA转录试剂盒。反应体系:110 ng线性化模板DNA,2 μL反应缓冲液,8 μL NTP/Biotin-UTP预混合物,2 μL 100 mmol/L DTT,0.5 μL RiboGuard RNase抑制剂,2 μL聚合酶液,加入去RNase水至总体积20 μL。反应体系在37℃水浴1 h,加入1 μL去RNase的DNase Ⅰ后继续于37℃15 min以去除DNA模板。同时用non-Biotinilated NTPs制备未标记探针作为对照。RNA转录物加入RNA-bee,再用RNA MiniPrep columns进行纯化。分别得到RNA探针M8和G4。M8:CGAAUUGGCCAAGACGGCCGAGCUCGAAUUCGCACUCCAUCCAGAC-ACAUACCGCGGC;G4:CGAAUUGGCCAAGA-CGGCCGAGCUCGAAUUCGCACUCGGGGCAG-ACACAUACCGCGGC。
1.3.4 凝胶阻滞迁移实验 ①探针/蛋白浓度实验:选取25、50、100、200 ng Biotin标记M8探针进行单独反应,检测探针质量和浓度。选取不同量(0.5、1、2、4 μg)核蛋白抽提物与不同浓度Biotin标记M8探针进行反应,以观察蛋白与探针产生的结合条带。综合以上结果选取最佳探针浓度与蛋白量进行反应。凝胶阻滞迁移实验过程如下:探针和核蛋白抽提物混合,室温孵育20 min,再置于80℃水浴10 min后转移至冰上。反应体系:2 μL 10×REMSA binding buffer,1%(v/v)Glycerol,2 μg tRNA,40 U RNAse OUT,10 nmol/L HEPES pH7.3,20 nmol/L KCl,1 nmol/L MgCl2,1 nmol/L DTT,并和探针、核蛋白抽提物以及Nuclease-free water共同构成20 μL反应体积。室温下静置20 min后于4℃下进行非变性6% PAGE胶电泳(100 V,4.5 h)。电泳后将一块同胶等大的正极尼龙膜在转膜缓冲液(0.5×TBE)中平衡5 min后贴于胶上,将4层平衡后的滤纸铺于尼龙膜上,胶另一面贴4层已平衡滤纸。置整个体系于转膜设备中,恒电流400 mA下转膜30 min。紫外灯(254 nm,120 mJ/cm2)交联45 s,使标记探针固定在膜上。37~50℃下将膜于洗涤缓冲液中漂洗2 min并于10 mL封闭液中振荡孵育30 min,室温下20 mL抗体孵育液中振荡孵育30 min,室温下10 mL洗涤缓冲液漂洗2次,每次15 min,10 mL缓冲液中平衡2~5 min。加1 mL显影液室温孵育5 min,暗室中胶片曝光1 h。②未标记(冷)探针竞争实验:Biotin标记M8探针50 ng与核内蛋白抽提物4 μg进行反应,同时未标记冷探针10、20、40 μg(相当于标记探针的200×、400×、800×)与核内蛋白及标记探针50 ng共同孵育,观察冷探针竞争情况,反应过程同①所述。冷探针竞争实验是为证明结合条带为探针与蛋白抽提物产生,而非其他非探针产生的伪影。③抗体竞争实验:Biotin标记M8探针50 ng与核内蛋白抽提物4 μg进行反应,同时Biotin标记M8探针50 ng、核内蛋白抽提物4 μg与抗体anti-SFRS3混合体系进行反应。抗体anti-SFRS3浓度分别为1 μg、2 μg。抗体如能使结合条带减弱或消失,说明与M8探针上序列结合的蛋白确实为SRp20。反应过程同①所述。④突变探针竞争实验:Biotin标记M8探针50 ng,核内蛋白抽提物浓度选取4 μg,同时标记突变探针G4 10 μg、20 μg(相当于标记探针M8的200×、400×)及未标记冷探针M8 10、20、40 μg(相当于标记探针200×、400×、800×)分别与标记探针M8竞争反应,对比观察特异性结合带浓度。突变探针G4是在M8探针基础上使“CAUC”突变为“GGGG”,如果突变探针使特异性结合带减弱消失,说明SRp20蛋白与M8上保守序列的“CAUC”特异性结合的可靠性。反应过程同①所述。
2 结果
2.1 SP-B(CA)n模体区的增强子和沉默子分析及结合蛋白预测
可变性剪切数据库分析SPB内含子4区(CA)n模体上所有潜在增强子(沉默子)及可能结合的潜在蛋白,依据出现频率进行整理作图,我们发现第8号模体存在单个特有基序CATC(RNA序列为CAUC)可能是特定蛋白SRp20的结合位点(图1)。

图1 SP-B内含子4区(CA)n模体上可能存在的RNA结合蛋白Fig.1 Potential binding proteins on the (CA)n motifs within the intron 4 region of SP-B
2.2 SRp20蛋白表达
SRp20抗体1 ab73891为家兔多克隆抗体,免疫原来源于人类SFRS3固有区,SRp20抗体2 ab80291亦为家兔抗体,免疫原来源于人类SFRS3的N端的35~84个氨基酸区域。选择2个抗体是为更好地明确SRp20蛋白表达。Western blot检测结果显示:SRp20抗体1(1 mg/mL,稀释1∶500,5 μg)与核内蛋白抽提物4 μg反应,转染后12 h无表达,36 h表达量较强,较24 h时表达强;SRp20抗体2(1 mg/mL,稀释1∶500,5 μg)与核内蛋白抽提物4 μg反应,12 h无表达,36 h及48 h表达量相当,均较24 h表达强。故选择36 h为转染后收取细胞时间。Endpoint测定转染后24、36、48 h核蛋白抽提物浓度分别为476.94、725.25、640.79 μg/mL。
2.3 相关(CA)n模体的RNA序列与其潜在结合蛋白SRp20的相互作用
2.3.1 Biotin标记的RNA探针(M8)与核蛋白的反应(探针/蛋白浓度实验) 单独检测Biotin标记M8探针(25、50、100、200 ng),浓度为50 ng和100 ng时游离探针条带清晰,故选取这2个探针浓度分别与核内蛋白抽提物(浓度选取0.5、1、2、4 μg)反应。M8探针浓度为50 ng、核蛋白为4 μg时,结合条带清晰,游离探针少(图2)。故选取此探针及核蛋白浓度为标准反应浓度以便与竞争探针对照。
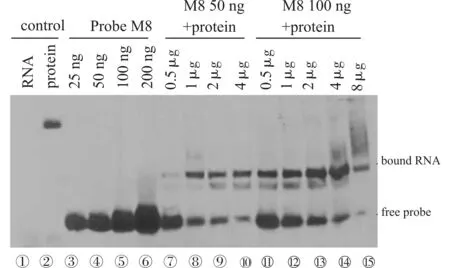
M8探针浓度为50 ng和100 ng时游离探针条带清晰(④⑤),M8探针浓度为50 ng、核蛋白为4 μg时,结合条带(bound RNA)清晰(⑩)图2 Biotin标记的RNA探针(M8)与核蛋白的反应Fig.2 The reaction between Biotin labeled RNA(M8)and nuclear extract
2.3.2 标记/未标记(冷)RNA探针(M8)与核蛋白的反应(未标记冷探针竞争实验) Biotin标记M8探针50 ng,核内蛋白抽提物浓度选取4 μg时,可见清晰结合条带;未标记冷探针10、20、40 μg(相当于标记探针的200×、400×、800×)与核内蛋白4 μg及标记探针50 ng共同孵育,可见结合条带浓度逐渐降低,显示标记探针被竞争(图3),说明结合条带确为探针M8与核蛋白结合引起而非其他非探针产生的伪影。

M8探针50 ng及核蛋白4 μg时结合条带清晰(②),随着未标记冷探针浓度逐渐升高,M8探针与核蛋白结合条带浓度逐渐减弱(③④⑤)图3 标记/未标记(冷)RNA探针(M8)与核蛋白的反应Fig.3 The reaction between Biotin labeled/unlabeled(cold)RNA(M8)and nuclear extract
2.3.3 标记RNA探针(M8)/抗体与核蛋白的反应(抗体竞争实验) Biotin标记M8探针50 ng,核内蛋白抽提物浓度选取4 μg,可见清晰结合条带,抗体anti-SFRS3浓度为2 μg时超级迁移带(super shift)明显,此为抗体与核蛋白结合后电泳移动速度下降所致;抗体为1 μg和2 μg时,结合带中下方条带均消失,说明减弱消失的条带中的结合蛋白为SRp20,显示由于抗体结合SRp20蛋白,引起探针无法和SRp20特异性结合,故被竞争消失的条带为探针M8与SRp20的特异性结合带(specific shift)(图4)。
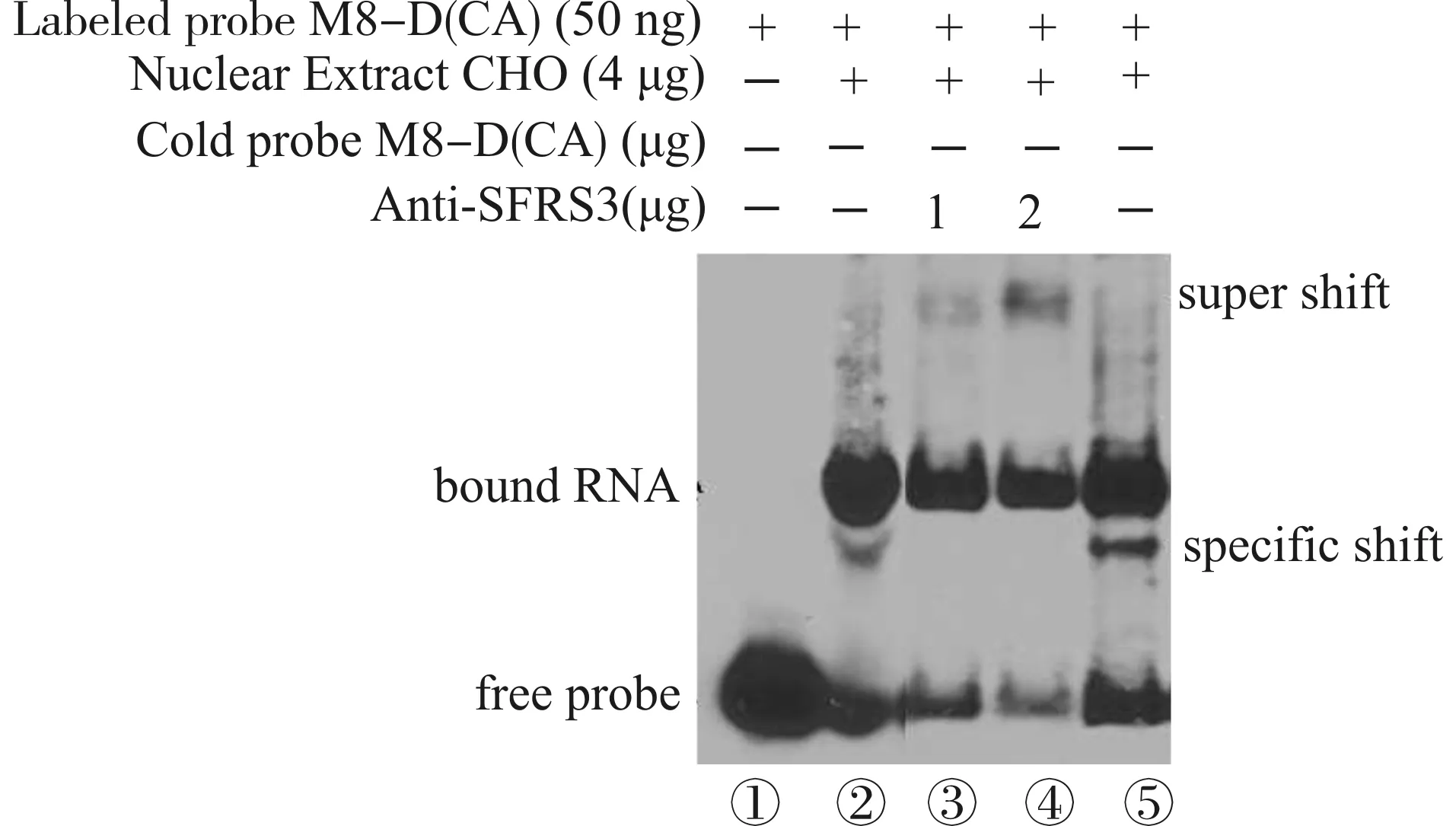
抗体浓度为1 μg和2 μg时,探针M8与核蛋白结合条带中下方条带消失(③④),同时产生超级迁移带(super shift)(④)图4 标记RNA探针(M8)/抗体与核蛋白的反应Fig.4 The reaction between Biotin labeled RNA(M8)/antibody and nuclear extract
2.3.4 标记突变RNA探针(G4)/未标记(冷)RNA探针(M8)与核蛋白的反应(突变探针竞争实验) 标记的突变探针G4(10、20 μg)与未标记冷探针M8(10、20、40 μg)对比,未标记冷探针M8使得所有结合带减弱消失,而随着突变探针G4浓度逐渐增高,只有特异性结合带减弱消失(图5),显示突变探针G4不能与SRp20蛋白相结合,说明被突变掉的特异性序列“CAUC”确实为SRp20蛋白的结合位点。
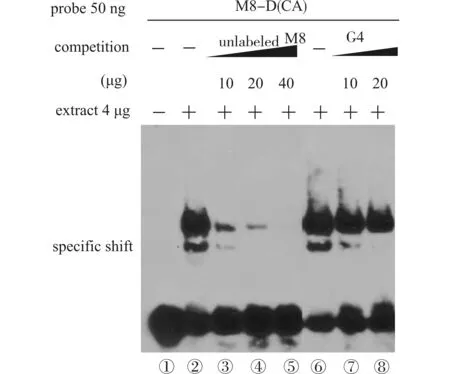
与未标记冷探针M8使所有结合带减弱消失(③④⑤)相比,随着突变探针G4浓度逐渐增高,只有特异性结合带(specific shift)减弱消失(⑦⑧)图5 标记突变RNA探针(G4)/未标记(冷)RNA探针(M8)与核蛋白的反应Fig.5 The reaction between Biotin labeled mutant RNA(G4)/unlabeled (cold) RNA (M8) and nuclear extract
3 讨论
SR蛋白家族(Ser-Arg rich protein family)作为一个在哺乳动物细胞中参与组成和调节pre-mRNA剪切过程的RNA结合蛋白家族而被广泛研究[2]。它们的氨基酸序列特征为包含一个富含精氨酸和丝氨酸重复序列的RS蛋白结构域。SR蛋白的所有9个成员SF2/ASF、SC35、SRp20、SRp40、SRp55、SRp75、SRp30c、9G8以及SRp54都有共同的结构,包含1个或者2个氨基(N)端RNA结合结构域和羧基(C)端长度可变的RS结构域[3-5]。RNA结合结构域是SR蛋白特异性识别并结合pre-mRNA上的RNA序列的功能单位,又称为RNA识别基序(RNA recognition motif,RRM)[4-6]。含有2个RRM的SR蛋白需要2个RRM相配合以完成剪切任务。
可变剪切的经典例子已经显示,pre-mRNA上的顺式作用元件如剪切增强子能通过提供剪切位点来募集如U1、U2 snRNP等剪切因子构成的剪切小体以及一些关联蛋白如U2辅助因子(U2AF),从而使外显子拼接并释放内含子[7]。目前研究较多、较深入的是外显子剪切增强子(ESE),剪切增强子通常识别至少1个SR蛋白将剪切小体募集至内含子毗连区[8-10]。在参与的几步剪切过程中[11-12],SR蛋白需要磷酸化以保证有效的剪切位点识别,同时需要去磷酸化催化剪切反应[13-14]。增强子结合的SR蛋白的RS结构域直接和其他具有RS结构域的剪切因子互相作用以利于剪切小体各因子募集到达功能剪切位点,如使U1 snRNP到达5′端剪切位点或者使U2 AF65到达3′端剪切位点。在功能性剪切小体中可观察到RS结构域同pre-mRNA相接触,提示剪切小体募集的可变模式。除了RS结构域对剪切的活化作用,动力学分析亦显示增强子结合的SR蛋白的相对活性决定了剪切被促进的程度;这个活性取决于SR蛋白组装在增强子上的量以及内含子和外显子之间的距离。有研究已经表明,剪切的活化与结合的SR蛋白的RS结构域上的精氨酸和丝氨酸重复序列的量呈正比,所以精氨酸和丝氨酸重复序列的量说明SR蛋白的活化能力[15]。
SRp20被证实在剪切过程中起着促进作用[16-18],关于内含子区SR蛋白的研究较少,通常认为这些供SR蛋白的内含子区结合位点功能性不强,也有报道证实确有SR蛋白的结合位点[19]。Hargous等[20]于2006年构建了9GB和SRp20的RNA识别基序(RRM)的自由液态结构,发现SRp20可以特异性识别“CAUC”序列,但其识别方式是部分的,序列的所有4个核苷酸都与RRM接触,但是只有5′端的C是主要的特异性识别,显示一种不同寻常的RNA结合模式。
我们的实验发现,SP-B基因内含子4区第8号模体保守区RNA序列上的“CAUC”序列确实可以和剪切调节因子SR家族的SRp20蛋白特异性结合,显示其可能作为一个SRp20剪切蛋白的结合位点而发挥对剪切的影响作用。我们之前研究发现第8号模体对SP-B mRNA剪切的增强作用可能与此结合位点相关,具体机制有待进一步证实。
[1] Yang W,Ni L,Silveyra P,et al.Motifs within the CA-repeat-rich region of Surfactant Protein B(SFTPB)intron 4 differentially affect mRNA splicing[J].J Mol Biochem,2013,2(1):40-55.
[2] Howard J M,Sanford J R.The RNAissance family:SR proteins as multifaceted regulatorsof gene expression[J].Wiley Interdiscip Rev RNA,2015,6(1):93-110.
[3] Mathews D H,Sabina J,Zuker M,et al.Expanded sequen-cedependence of thermodynamic parameters improves prediction of RNA secondary structure[J].J Mol Biol,1999,288(5):911-940.
[4] Ge H,Manley J L.A protein factor,ASF,controls cell-specific alternative splicing of SV40 early pre-mRNAinvitro[J].Cell,1990,62(1):25-34.
[5] Krainer A R,Conway G C,Kozak D.Purification and characterization of pre-mRNA splicing factor SF2 from HeLa cells[J].Genes Dev,1990,4(7):1158-1171.
[6] Fu X D,Maniatis T.The 35-kDa mammalian splicing factor SC35 mediates specific interactions between U1 and U2 small nuclear ribonucle oprotein particles at the 3’splice site[J].Proc Natl Acad Sci USA,1992,89(5):1725-1729.
[7] Hui J.Regulation of mammalian pre-mRNA splicing[J].Sci China C Life Sci,2009,52(3):253-260.
[8] Graveley B R.Sorting out the complexity of SR protein functions[J].RNA,2000,6(9):1197-1211.
[9] Blencowe B J.Exonic splicing enhancers:mechanism of action,diversity and role in human genetic diseases[J].Trends Biochem Sci,2000,25(3):106-110.
[10] Black D L.Mechanisms of alternative pre-messenger RNA splicing[J].Annu Rev Biochem,2003,72(2):291-336.
[11] Fu X D.Specific commitment of different pre-mRNAs to splicing by single SR proteins[J].Nature,1993,365(6441):82-85.
[12] Roscigno R F,Garcia-Blanco M A.SR proteins escort the U4/U6.U5 tri-snRNP to the spliceosome[J].RNA,1995,1(7):692-706.
[13] Mermoud J E,Cohen P,Lamond A I.Ser/Thr-specific protein phosphatases are required for both catalytic steps of pre-mRNA splicing[J].Nucleic Acids Res,1992,20(20):5263-5269.
[14] Mermoud J E,Cohen P T,Lamond A I.Regulation of mammalian spliceosome assembly by a protein phosphorylation mechanism[J].EMBO J,1994,13(23):5679-5688.
[15] Graveley B R,Hertel K J,Maniatis T.A systematic analysis of the factors that determine the strength of pre-mRNA splicing enhancers[J].EMBO J,1998,17(22):6747-6756.
[16] Jumaa H,Wei G,Nielsen P J.Blastocyst formation is blocked in mouse embryos lacking thesplicing factor SRp20[J].Curr Biol,1999,9(16):899-902.
[17] He X,Ee P L,Coon J S,et al.Alternative splicing of the multidrug resistance protein 1/ATP binding cassette transporter subfamily gene in ovarian cancer creates functional splice variants and is associated with increased expression of the splicing factors PTB and SRp20[J].Clin Cancer Res,2004,10(14):4652-4660.
[18] Verma D,Bais S,Gaillard M,et al.Epstein-Barr virus SM protein utilizes cellular splicing factor SRp20 to mediate alternative splicing[J].J Virol,2010,84(22):11781-11789.
[19] Ibrahim E C,Schaal T D,Hertel K J,et al.Serine/arginine-rich protein-dependent suppression of exon skipping by exonic splicing enhancers[J].Proc Natl Acad Sci USA,2005,102(14):5002-5007.
[20] Hargous Y,Hautbergue G M,Tintaru A M,et al.Molecular basis of RNA recognition and TAP binding by the SR proteins SRp20 and 9G8[J].EMBO J,2006,25(21):5126-5137.
(2016-09-28 收稿)
A Preliminary Study of Binding Site of SRp20 in the (CA)nMotif of Intron 4 of Surfactant Protein B Gene
Ni Lan1,Wang Guirong2,Yang Jiong1△
1DepartmentofRespiratoryMedicine,ZhongnanHospital,WuhanUniversity,Wuhan430071,China2DepartmentofSurgery,SUNYUpstateMedicalUniversity,SyracuseNY13210,USA
Objective To investigate the potential protein binding site in the motif 8 of intron 4 of surfactant protein B (SP-B)gene and provide a basis for further investigation on mechanism by which motif 8 affects SP-B mRNA splicing.Methods Specific (CA)nmotifs that potentially bind RNA-binding proteins were analyzed by online software.RNA probes that contained potential binding site of specific protein as well as mutated-sequence probe were synthesized.RNA mobility shift assay was used to study the correlation and interactions between specific (CA)nmotif and potential binding protein.Results The Prediction by software analysis showed potential binding site of SRp20 in the motif 8.The RNA:protein shift was lost upon antibody of SRp20 and mutation of “CAUC” in motif 8 in EMSA,which revealed the specific binding resulted from a Motif 8 sequence that contains a CAUCcis-element and SRp20.Conclusion A specific RNA-binding site of SRp20 exists in the conserved sequence of the motif 8,which may have a role in RNA splicing.
surfactant protein B gene; RNA binding protein; SRp20
*美国国立卫生研究院基金资助项目(No.NIH/R37HL-34788)
R341
10.3870/j.issn.1672-0741.2016.06.007
倪 岚,女,1978年生,主治医师,医学博士,E-mail:lanni_1020@aliyun.com
△通讯作者,Corresponding author,E-mail:jiongyangj@163.com

