川崎病相关甲基化基因及其作用
(湖北省天门市第一人民医院1 新生儿科,2 麻醉科,天门市 431700,电子邮箱:sunting0203@163.com)
川崎病又称为皮肤黏膜淋巴结综合征,是一种全身性、免疫性的血管炎性反应疾病,主要见于5岁以下儿童。该病主要累及中小动脉,尤以冠状动脉损害最为严重。随着发病率的增高,川崎病成为儿童后天获得性心脏病的首要原因,同时也是成年后发生缺血性心脏病的潜在危险因素[1]。尽管发现川崎病已近50年,但其发病机制仍不明确,而部分观点认为其可能与感染及异常的免疫激活有关[2]。因此,有必要进一步探索川崎病可能的发病机制,从而为临床早期诊断及治疗提供依据。
DNA甲基化是主要的表观遗传修饰方式之一,是调节基因功能的重要途径。一般来说,DNA高甲基化与基因沉默相关,低甲基化则与基因活化有关。DNA甲基化与生长、发育以及肿瘤等疾病的形成密切相关,例如抑癌基因的高甲基化已被证实在癌症的发生发展中扮演重要角色[3]。然而,目前仅有少数研究关注川崎病相关甲基化致病基因,且均集中在个别基因[4-5],整个基因组的甲基化水平及其功能尚未明确。近年来,基因微阵列芯片技术的发展为分析基因组甲基化水平提供了可能。本研究通过挖掘基因表达综合数据库(Gene Expression Omnibus,GEO)中DNA甲基化的芯片数据,并联合基因表达芯片数据进行分析,探讨川崎病相关甲基化基因及其在发病机制中的作用。
1 资料与方法
1.1 数据收集 登录GEO数据库(https://www.ncbi.nlm.nih.gov/geo),下载川崎病DNA甲基化数据集GSE84624和基因表达数据集GSE68004。GSE84624共包含24例川崎病患儿和24例健康儿童的外周血单核细胞样本,采用Illumina Human Methylation450 BeadChip芯片进行检测,平台号为GPL13534。GSE68004共收集76例完全型川崎病患儿和37例健康儿童的外周全血样本,采用Illumina Human HT-12 V4.0 Expression BeadChip芯片进行检测,平台号为GPL10558。
1.2 数据预处理 提取数据集GSE84624的甲基化β值矩阵和信号矩阵,β值反映单个基因位点的甲基化程度,即β=甲基化信号/(甲基化信号+非甲基化信号+100)。采用R软件的IMA包对β值矩阵进行过滤,具体如下:(1)过滤含缺失值的探针;(2)过滤在75%以上样本中探测P值均大于0.05的探针;(3)过滤单核苷酸多态性相关探针;(4)过滤XY染色体上的探针;(5)若某一样本75%以上探针的探测P值均大于1×10-5,则过滤掉该样本。最终的β值矩阵包含样本48例,甲基化探针368 067个。此外,提取数据集GSE68004的基因表达矩阵,过滤在5%以上样本中表达量均为负值的探针后进行log2转换,最终得到探针16 195个。根据GPL10558注释文件对探针进行注释,多个探针对应1个基因则取均值作为该基因的表达量。
1.3 差异基因筛选 采用R软件的Limma包筛选差异甲基化位点和差异表达基因(differentially expressed gene,DEG)。差异甲基化位点的阈值条件为|△β|>0.2及P<0.05,根据GPL13534注释文件将差异甲基化位点匹配到相应的基因,即差异甲基化基因(differentially methylated gene,DMG)。DEG的阈值条件为|log2 FC|>1及P<0.05,其中FC表示基因的差异表达倍数。DMG中高甲基化和低甲基化者再分别与DEG中低表达和高表达者取交集,得到最终的甲基化差异表达基因(methylated-differentially expressed gene,mDEG)。
1.4 基因富集分析 利用生物信息注释数据库DAVID(https://david.ncifcrf.gov/)对mDEG进行基因本体论(Gene Ontology,GO)注释,重点分析其相关的生物学过程。该数据库收录了大量基因及蛋白的生物功能注释信息,能筛选出具有富集显著性的生物学注释[6]。同样采用DAVID数据库分析mDEG所富集的京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)信号通路。以P<0.05表示富集具有显著性。
1.5 蛋白质-蛋白质互作网络的构建 采用STRING在线工具(https://string-db.org/)构建mDEG的蛋白质-蛋白质互作(protein-protein interaction,PPI)网络,以互作评分≥0.4作为阈值条件。采用CytoScape 3.6.0软件对网络进行可视化并计算各个节点所对应的连接度,选取连接度相对较高(≥15)的基因作为核心基因。同时,使用MCODE插件筛选网络中具有连接显著性的子模块,筛选条件为:连接度阈值=2、节点得分阈值=0.2、K核=2、最大深度=100,并要求模块得分≥5分。再对子模块中的mDEG进行KEGG通路分析。
2 结 果
2.1 川崎病相关DMG分析 在DNA甲基化数据集GSE84624中,共有2 402个位点存在差异甲基化,而其中97.5%(2 343/2 402)的位点在川崎病患儿中表现为低甲基化(见图1)。通过平台注释文件进行匹配分析后发现,这些位点共对应1 393个DMG,包括高甲基化者35个,低甲基化者1 358个。
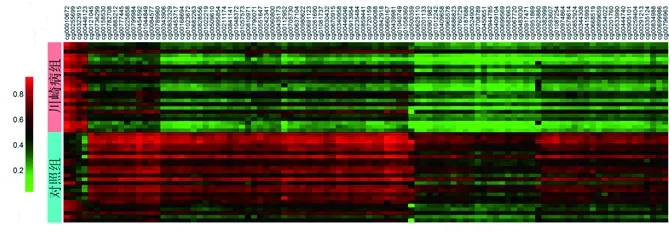
图1 川崎病相关甲基化差异位点的β值热图(前100个差异位点)
2.2 川崎病相关DEG分析 在基因表达数据集GSE68004中,通过Limma包分析共得到1 362个DEG,其中高表达者943个,低表达者419个(见图2)。将DEG与DMG取交集,发现4个基因表现为高甲基化低表达,而187个基因表现为低甲基化高表达。因此,共得到191个mDEG。
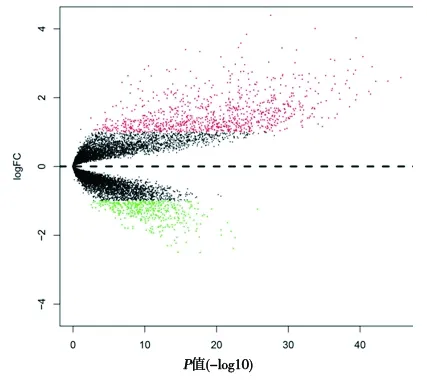
图2 川崎病相关差异表达基因
2.3 功能富集分析 通过DAVID在线工具进行GO功能富集分析,发现上述mDEG主要涉及炎症反应、凝血作用、固有免疫应答、脂多糖刺激反应等生物学过程,见表1。KEGG通路分析表明,这些mDEG主要富集的通路包括血小板活化、破骨细胞分化、趋化因子信号途径等,见表2。
2.4 PPI网络分析 将上述mDEG导入STRING数据库构建PPI网络,该网络呈无尺度分布形式(R2=0.81),共包含95个mDEG和170对相互作用关系(见图3)。其中,MAPK14和PHLPP1为网络中的核心基因,其连接度分别为18和15。利用MCODE插件筛选出一个具有连接显著性的子模块,该模块由ADCY3、CXCR2、ADCY4、GNAI3和CCR7构成。这5个mDEG全部富集在趋化因子信号通路上。
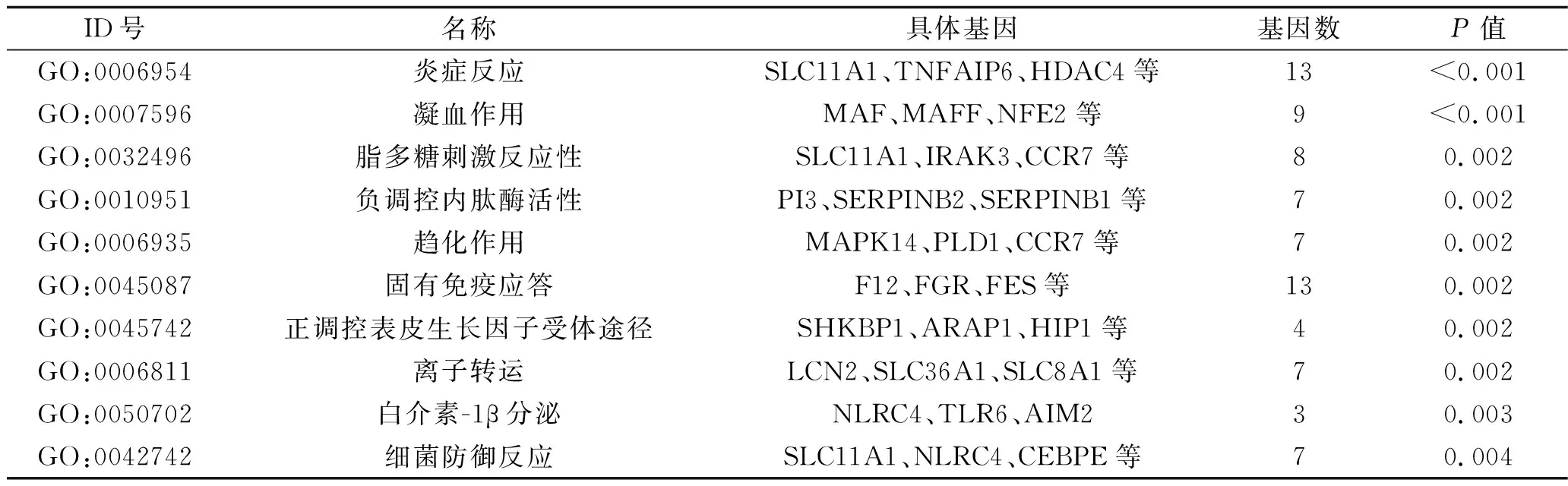
表1 mDEG的GO功能富集分析(前10)
注:SLC为溶质转运蛋白家族;TNFAIP6为人肿瘤坏死因子诱导基因6蛋白;HDAC4为组蛋白去乙酰化酶4;MAF为肌肉腱膜纤维肉瘤癌基因同源物;NFE2为核因子红细胞2;IRAK3为白细胞介素-1受体相关激酶3;CCR7为趋化因子受体7;PI3为磷脂酰基醇3-激酶;SERPINB为丝氨酸蛋白激酶抑制物B;MAPK14为丝裂原活化蛋白激酶14;PLD1为磷脂酶D1;ARAP1为血管紧张素Ⅱ1型受体相关蛋白1;HIP1为人舞蹈病蛋白相互作用蛋白1;LCN2为脂质运载蛋白2;TLR6为Toll样受体6;AIM2为黑色素瘤缺乏因子2;CEBPE为CCAAT/增强子结合蛋白。
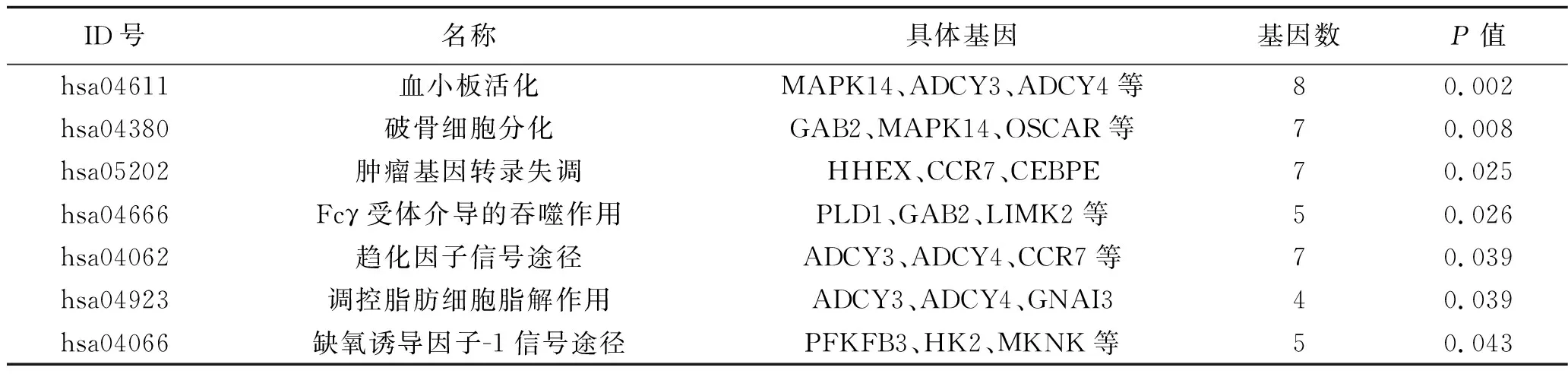
表2 mDEG的通路富集分析
注:ADCY为腺苷酸环化酶;HHEX为造血表达同源异型盒;LIMK2为 LIM结构域蛋白激酶2;GNAI3为鸟嘌呤核苷酸结合蛋白G3;PFKFB3为6-磷酸果糖激酶-2/果糖双磷酸酶-2同工酶3;HK2为己糖激酶2;MKNK为MAP激酶相互作用丝/苏氨酸激酶1。
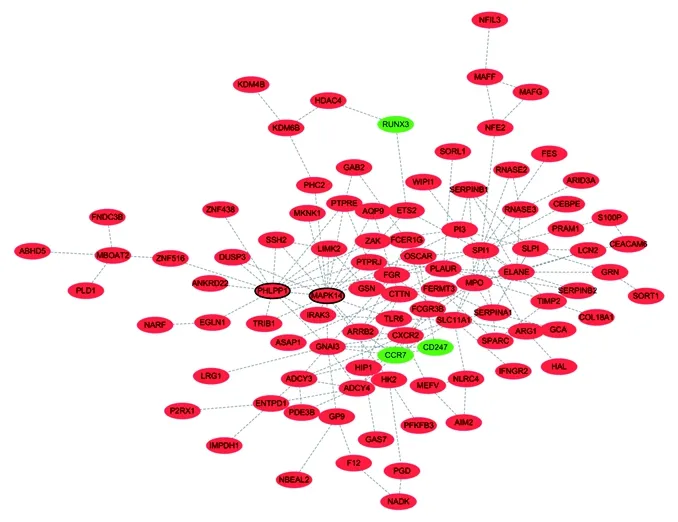
图3 川崎病mDEG的蛋白互作网络图
注:绿色为高甲基化低表达基因,红色为低甲基化高表达基因,边框加粗为网络核心基因。
3 讨 论
流行病学研究表明,川崎病的发病具有遗传易感性,并受外界感染、致敏原和季节变化等环境因素影响[7]。而DNA甲基化是沟通环境因素与遗传因素的桥梁,因此其可能在川崎病的发病过程中发挥重要作用。本研究首次通过整合生物信息学的方法分析DNA甲基化芯片和基因表达芯片数据,发现川崎病相关的甲基化基因多呈低甲基化状态。
免疫失调及其导致的多系统炎症性损伤是川崎病的主要特点。在川崎病的急性期和亚急性期,异常活化的T淋巴细胞和单核细胞释放肿瘤坏死因子α等炎症介质,造成血管内皮损伤[8]。同时,CD4+/CD8+T淋巴细胞比例倒置,自然杀伤细胞数量减少,使其对B淋巴细胞的抑制作用减弱,导致B细胞功能亢进[9]。而在本研究中,在免疫学致病机制方面DNA甲基化主要参与调控川崎病的固有免疫反应。近期观点认为,固有免疫相关的病原体相关分子模式(包括脂多糖、肽聚糖等)可引起血管炎,并且该作用独立于获得性免疫[10]。DNA甲基化是调节固有免疫及其相关炎症反应的重要因素[11]。Huang等[12]的研究更表明,固有免疫所识别的Toll样受体在川崎病中呈低甲基化和高表达状态。本研究提示川崎病相关的甲基化基因也涉及脂多糖刺激反应性、趋化作用及Fcγ受体介导的吞噬作用等生物学功能,而这些功能均与固有免疫应答有关。此外,血小板活化和血液高凝状态是川崎病的重要特征,而这可能与炎症导致的血管内皮损害有关。通过GO和KEGG富集分析,我们发现DNA甲基化也参与调节凝血作用和血小板的活化。这些结果表明DNA甲基化也可能与川崎病患儿的冠状动脉损害有关。
在PPI网络中,MAPK14和PHLPP1是处于核心调控地位的甲基化修饰基因。MAPK14基因(又称作p38)编码一种丝裂素原活化蛋白激酶,位于6号染色体p21.31位置,含有22个外显子。MAPK14由多种环境应激因素和促炎症因子激活,参与细胞的增殖、分化和转录调控等过程[13]。MAPK14也在自身免疫性血管炎中扮演重要角色,如抗中性粒细胞胞浆抗体相关性血管炎[14]。而梁顺姬等[15]认为,抗内皮细胞受体抗体可通过p38-MAPK信号途径诱导细胞内吸附分子表达,从而参与川崎病的发病过程。本研究GO富集分析结果提示MAPK14主要参与调节趋化作用,而PPI显著性模块分析表明趋化因子信号途径在整个网络中处于重要地位。PHLPP1基因编码一种丝氨酸/苏氨酸磷酸酶,位于18号染色体的q21.33位置,含有19个外显子。PHLPP1通过去磷酸化导致Akt失活从而促进细胞凋亡,因此被视作一种抑癌基因[16]。在川崎病患者的内皮细胞中,去磷酸化的Akt比例增加[17],可通过抑制Akt-eNOS信号通路加重血管内皮细胞损害。因此,低甲基化的MAPK14和PHLPP1导致其相应的基因表达上调,而二者通过以上可能的机制参与川崎病的发生发展。
综上所述,川崎病相关的甲基化基因大多呈低甲基化状态,主要参与炎症反应、固有免疫反应、凝血作用和趋化因子信号途径,其中MAPK14和PHLPP1是重要的甲基化修饰基因,这或可为川崎病的表观遗传研究提供新的思路。

