基于生物信息学分析的hsa-miR-206靶基因预测
康治理,武振方,刘晓伟,许 斌
0 引 言
MicroRNAs(miRNAs)是一种长度约为19~24个碱基的单链小分子的非编码RNA家族,它是由一段具有发卡结构的70~80个碱基大小的单链RNA前体经过Dicer酶的作用剪切生成,通过并入到RNA诱导的沉默复合物(RISC)与靶mRNA的5’及3’UTR端(3' untranslated regions,3'-UTR)特异性碱基配对,在转录水平抑制靶mRNA翻译或者诱导靶mRNA降解,从而调控靶基因的表达[1]。其靶向mRNA的主要方式包括翻译抑制、mRNA切割和mRNA稳定性改变。早期研究发现,miRNA对生物学过程的调控至关重要,如发育时间、细胞死亡、细胞增殖、免疫、神经系统模式等[2-3]。越来越多的数据表明,miRNA是许多重要的生命过程的主要调节者,这些生命过程包括细胞增殖、细胞凋亡、病毒感染和细胞癌变等[4-5]。
miR-206是目前最具研究潜力及特性的microRNAs之一,最初在骨骼肌中发现并被认为是骨骼肌特异性miRNA[6]。参与了包括肿瘤在内的诸多疾病的发病机制,目前研究表明miR-206在肿瘤等多种疾病中异常表达并在增殖、分化、凋亡、侵袭和转移中发挥重要作用[7]。miR-206定位在人类第6号染色体上,是骨骼肌特异性表达的“肌特异性miRNA(myomiR)”家族成员之一[8]。miR-206通过与其靶mRNA的3'端非翻译区碱基不完全互补配对,抑制mRNA翻译或直接使其降解而发挥生物学特性,调控细胞的增殖、分化、转移等生物学行为[9]。靶基因与miR-206结合位点越多则受其调控程度越大,由此形成一个巨大分子调控网络,在个体成长发育、生理过程及疾病发展过程中发挥重要作用。然而其通过作用于哪些靶基因,发挥何种生物学效应在许多实验研究中都尚未阐释清楚,有关hsa-miR-206的生物信息学研究也较少。本研究拟通过生物信息学的方法探索对hsa-miR-206进行定位和序列保守性研究、靶基因预测、功能富集分析(GO分析)、信号通路分析(KEGG)、蛋白互作分析(protein protein interaction,PPI)等分析,为今后进一步探索hsa-miR-206在肿瘤发生、发展及分子机制等方面提供线索和理论依据。
1 材料与方法
1.1 miR-206的序列保守性分析应用miRbase(http://www.mirbase.org/)在线查找各物种已被明确的成熟碱基序列,并对比分析miR-206序列在各物种之间的保守性,用美国国家医学图书馆国家生物技术信息中心(National Center for Biotechnology Information, NCBI)开发的生物大分子序列比对搜索工具(Basic Local Alignment Search Tool,BLAST)(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进一步分析其物种保守性。
1.2miR-206的靶基因预测运用在线靶基因预测软件,miRDB(http://www.mirdb.org/),TargetScan V7.2(http://www.targetscan.org/)和DIANA-microT(http://diana.imis.athenainnovation.gr)等3个数据库对has-miR-206可能存在的靶基因进行预测,为得到更加准确的结果,用Venn Diagrams在线作图工具画韦恩图得到3个数据库预测结果的交集来降低假阳性率。
1.3miR-206靶基因的GO功能注释及KEGG通路富集分析运用DAVID6.8(database for annotation, visualization and integrated discovery)(http://David.abcc.ncifcrf.gov/)对靶基因集合进行GO分析以及基于KEGG数据库(Kyoto Encyclopedia of Genes and Genomes)(http://www.genome.jp/)的Pathway分析,选择人类全基因组作为背景基因,用"Functional Annotation Chart"分析工具计算P值,以P<0.05为显著性阈值,分别得到基因集合相对于背景具有统计学意义的GO分析与Pathway分析结果。
1.4关键靶基因筛选利用在线工具STRING(https://string-db.org)[10]联合Cytoscape软件[11]构建miR-206靶基因蛋白互作网络(PPI),并运用Mcode插件[12]筛选出关键基因(hub gene)。
2 结 果
2.1 miR-206的染色体定位及序列保守性分析检索在线数据库miRBase,发现人has-miR-206基因序列号为MIMAT0000462,其成熟体碱基序列为“53-UGGAAUGUAAGGAAGUGUGUGG-74”。通过NCBI BLAST对小鼠(mmu)、大鼠(rno)、野猪(ssc)等10个物种的miR-206序列对比分析,发现miR-206的成熟序列在各物种间高度保守,见表1。
2.2hsa-miR-206的靶基因预测结果使用TargetScan、miRDB、DIANA-microT 3个在线预测软件分别得到hsa-miR-206的靶基因分别为897个、944个、1148个,再进行交集得到预测靶基因420个,见图1。
2.3hsa-miR-206靶基因GO功能注释和KEGG通路富集分析将3个经典数据库预测到的靶基因取最终交集420个作GO分析,发现miR-206的靶基因主要富集在高尔基体、内膜、囊泡等细胞组件(P<0.05),参与DNA结合、蛋白结合、转录调节活性等分子功能(P<0.05),富集于生物合成调节、有机物代谢过程调节、转录调节等生物学过程(P<0.05)。见表2。KEGG信号通路分析显示主要富集在Wnt信号通路、T细胞受体信号通路、癌症通路、调节肌动蛋白细胞骨架等(P<0.05)。见表3。
表1 部分物种成熟的miR-206保守序列

序列号(miRBase)物种名称序列(5'→3')MIMAT0000462人hsa-miR-20653-UGGAAUGUAAGGAAGUGUGUGG-74MIMAT0000239小鼠mmu-miR-206-3p46-ACAUGCUUCUUUAUAUCCUCAUA-67MIMAT0017004小鼠mmu-miR-206-5p8-ACAUGCUUCUUUAUAUCCUCAUA-30MIMAT0017154褐家鼠rno-miR-206-5p13-ACAUGCUUCUUUAUAUCCUCAU-34MIMAT0000879褐家鼠rno-miR-206-3p51-UGGAAUGUAAGGAAGUGUGUGG-72MIMAT0006606家犬cfa-miR-20639-UGGAAUGUAAGGAAGUGUGUGG-60MIMAT0036052山羊chi-miR-20653-UGGAAUGUAAGGAAGUGUGUGGU-75MIMAT0023846黑线仓鼠cgr-miR-20650-UGGAAUGUAAGGAAGUGUGUGG-71MIMAT0002318猩猩ppy-miR-20653-UGGAAUGUAAGGAAGUGUGUGG-74MIMAT0013864野猪ssc-miR-20650-UGGAAUGUAAGGAAGUGUGUGA-71
表2 miR-206靶基因的GO功能分析
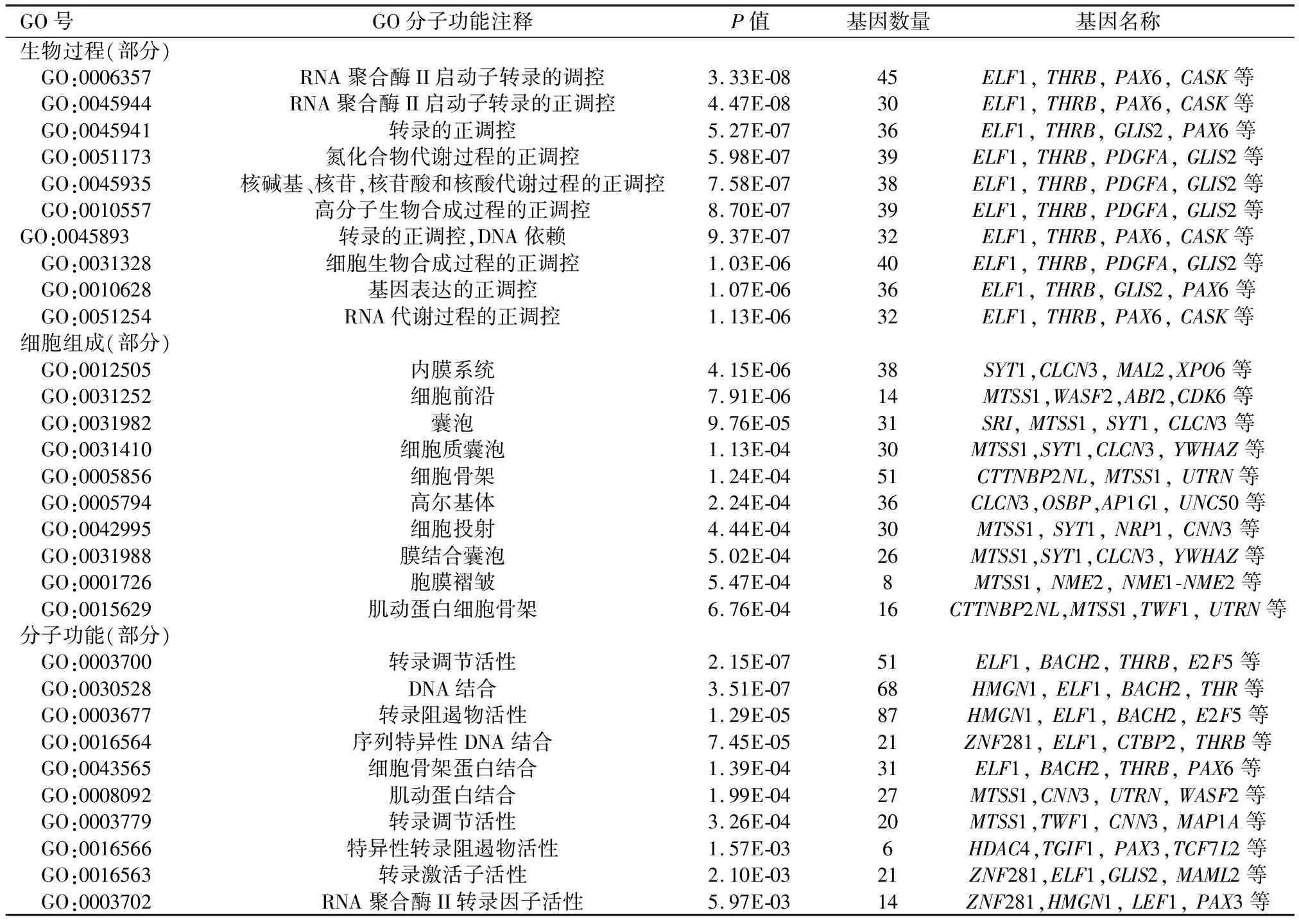
GO号GO分子功能注释P值基因数量基因名称生物过程(部分) GO:0006357RNA聚合酶II启动子转录的调控3.33E-0845ELF1,THRB,PAX6,CASK等 GO:0045944RNA聚合酶II启动子转录的正调控4.47E-0830ELF1,THRB,PAX6,CASK等 GO:0045941转录的正调控5.27E-0736ELF1,THRB,GLIS2,PAX6等 GO:0051173氮化合物代谢过程的正调控5.98E-0739ELF1,THRB,PDGFA,GLIS2等 GO:0045935核碱基、核苷,核苷酸和核酸代谢过程的正调控7.58E-0738ELF1,THRB,PDGFA,GLIS2等 GO:0010557高分子生物合成过程的正调控8.70E-0739ELF1,THRB,PDGFA,GLIS2等GO:0045893转录的正调控,DNA依赖9.37E-0732ELF1,THRB,PAX6,CASK等 GO:0031328细胞生物合成过程的正调控1.03E-0640ELF1,THRB,PDGFA,GLIS2等 GO:0010628基因表达的正调控1.07E-0636ELF1,THRB,GLIS2,PAX6等 GO:0051254RNA代谢过程的正调控1.13E-0632ELF1,THRB,PAX6,CASK等细胞组成(部分) GO:0012505内膜系统4.15E-0638SYT1,CLCN3,MAL2,XPO6等 GO:0031252细胞前沿7.91E-0614MTSS1,WASF2,ABI2,CDK6等 GO:0031982囊泡9.76E-0531SRI,MTSS1,SYT1,CLCN3等 GO:0031410细胞质囊泡1.13E-0430MTSS1,SYT1,CLCN3,YWHAZ等 GO:0005856细胞骨架1.24E-0451CTTNBP2NL,MTSS1,UTRN等 GO:0005794高尔基体2.24E-0436CLCN3,OSBP,AP1G1,UNC50等 GO:0042995细胞投射4.44E-0430MTSS1,SYT1,NRP1,CNN3等 GO:0031988膜结合囊泡5.02E-0426MTSS1,SYT1,CLCN3,YWHAZ等 GO:0001726胞膜褶皱5.47E-048MTSS1,NME2,NME1-NME2等 GO:0015629肌动蛋白细胞骨架6.76E-0416CTTNBP2NL,MTSS1,TWF1,UTRN等分子功能(部分) GO:0003700转录调节活性2.15E-0751ELF1,BACH2,THRB,E2F5等 GO:0030528DNA结合3.51E-0768HMGN1,ELF1,BACH2,THR等 GO:0003677转录阻遏物活性1.29E-0587HMGN1,ELF1,BACH2,E2F5等 GO:0016564序列特异性DNA结合7.45E-0521ZNF281,ELF1,CTBP2,THRB等 GO:0043565细胞骨架蛋白结合1.39E-0431ELF1,BACH2,THRB,PAX6等 GO:0008092肌动蛋白结合1.99E-0427MTSS1,CNN3,UTRN,WASF2等 GO:0003779转录调节活性3.26E-0420MTSS1,TWF1,CNN3,MAP1A等 GO:0016566特异性转录阻遏物活性1.57E-036HDAC4,TGIF1,PAX3,TCF7L2等 GO:0016563转录激活子活性2.10E-0321ZNF281,ELF1,GLIS2,MAML2等 GO:0003702RNA聚合酶II转录因子活性5.97E-0314ZNF281,HMGN1,LEF1,PAX3等
表3 miR-206靶基因KEGG通路富集分析结果

IDKEGG通路名称基因数量P值参与基因hsa04360轴突导向131.11E-04NRP1,MET,EFNB2,MAPK1,KRAS,SEMA6D等hsa04520黏附连接101.47E-04ACTB,MAPK1,MET,WASF2,LEF1,WASL等hsa04320背腹轴形成53.59E-03NOTCH3,MAPK1,KRAS,ETS1,CPEB1hsa04310Wnt信号通路115.55E-03CTBP2,SFRP1,CCND2,PPP2R5A,NFAT5等hsa05216甲状腺癌56.23E-03MAPK1,KRAS,LEF1,TCF7L2,TPM3hsa05211肾细胞癌79.19E-03MAPK1,KRAS,ETS1,PAK3,MET,VEGFA等hsa04810肌动蛋白细胞骨架调节139.50E-03GIT1,ACTB,MAPK1,PFN2,KRAS,ARPC3等hsa05130致病性大肠杆菌感染61.55E-02ACTB,YWHAZ,ARPC3,YWHAQ,WASL,NCLhsa04660T细胞受体82.15E-02MAPK1,KRAS,PAK3,CBL,NFAT5,NFATC2等hsa05200癌症通路162.20E-02CTBP2,PDGFA,CBL,MET,IGF1,LEF1等

图1 数据库预测hsa-miR-206的靶基因个数
2.4miR-206预测靶基因所编码蛋白质间的相互作用分析通过STRING在线工具筛选出靶基因蛋白互作网络,导入Cytoscape,利用Mcode插件筛选出关键基因(Hub基因)共27个,见图2。
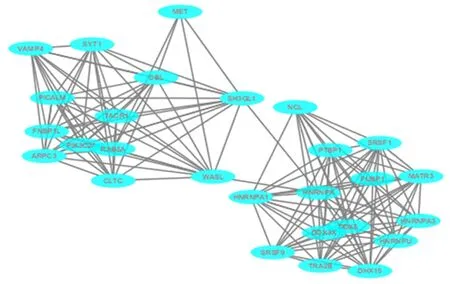
图2 miR-206 预测靶基因所编码蛋白质间的相互作用分析
3 讨 论
miRNA是一类小分子非编码内源性调节RNA,特能结合到靶mRNA的3’非翻译区(Untranslated region,UTR)从而抑制翻译或者直接降解靶mRNA,具有转录后水平的多基因调节功能。miRNA通过碱基互补配对原则特异性结合其靶基因,形成一个巨大的分子调控网络,广泛参与到人类生命活动和疾病的各个方面中去[13]。目前已经在自然界中发现了超过三万种miRNA,参与到细胞的增殖、分化、凋亡及机体的免疫调节和发育等过程,近年来的研究发现miRNA在多种疾病中的表达紊乱[14]。深入研究miRNA与疾病的相关性将有望为疾病的临床诊断及治疗提供新思路和新策略。
随着技术的进步,更多的技术被用来量化临床标本的miRNA,如Northern blotting、基因芯片分析、高通量测序,实时定量PCR等[15-16]。然而,由于通过实验途径一一验证miRNA的靶基因较为困难且繁琐,故而筛选miRNA靶基因,预测其潜在生物学功能,对指导miRNA的实验研究具有重要意义。
考虑到靶基因预测过程中miRNA与mRNA双链特异结合的热稳定性、miRNA与靶基因结合位点的序列匹配、序列的保守性等因素,本研究选用3种经典的miRNA靶基因预测工具Targetscan、miRDB和DIANA-microT,用不同的计算方法预测靶基因,取交集作为靶基因集合。miRbase是由曼彻斯特大学的研究人员开发的一个在线的miRNA数据库,该数据库中收录了来自200多个物种,接近4万个miRNA序列和注释的最全面的miRNA数据库,通过浏览和搜索等方式进行检索,每个条目中包含了该miRNA前体茎环结构的序列,其经过切割后获得的成熟miRNA的序列,以及它在其他物种间的分布情况等的基本信息。本文通过查找各个物种的miR-206与人类miR-206通过NCBI BLAST比对分析发现miR-206的成熟序列在各物种之间高度保守,提示其可能具有重要的生物学功能。利用DAVID在线分析工具进行GO分析和KEGG分析,GO分析从细胞定位、分子功能,生物学途径分析靶基因负富集于那些生物学过程,KEGG分析结果表明靶基因参与哪些细胞代谢通路。通过GO分析我们得知miR-206主要参与生物合成调节、有机物代谢过程调节、转录调节等生物学过程,KEGG分析表明miR-206主要参与Wnt信号通路、T细胞受体信号通路、癌症通路等。这与已有的研究及文献报道的hsa-miR-206在多种疾病中已揭示的一些作用机制相一致,证明各生物信息学软件的预测结果具有一定可靠性,能够为对hsa-miR-206的进一步研究提供指导方向。随后通过String11.0在线数据库和Cytoscapev3.6.1软件对420个差异靶基因进行互作预测分析发现,各靶标蛋白质之间存在复杂的相互作用,共筛选出27个关键基因,如DDX5、FUBP1、DHX15等,因此我们推测这些基因可能在miR-206调控的生物学效应中发挥重要作用。DDX5是在各种恶性肿瘤中过表达的ATP依赖性RNA解旋酶,越来越多的证据表明,DDX5通过促进细胞增殖和转移而参与癌变和癌症进展,研究显示,DDX5通过激活mTOR/S6K1诱导胃癌细胞的生长。特异的mTOR抑制剂依维莫司的治疗显着减弱了DDX5介导的细胞增殖[17]。体外实验表明,FUBP1通过增强糖酵解和ATP产生来促进神经母细胞瘤的细胞增殖并抑制细胞凋亡[18]。DHX15是DEAH-box RNA解旋酶家族的杰出成员,有研究表明[19],DHX15在肝细胞肝癌中显著上调,其高表达与不良预后相关,提示其在肝细胞肝癌进程中的关键作用。
最近的一系列研究证实miR-206在多种人类疾病中具有重要的生物学功能,尤其集中于肿瘤相关疾病,许多研究者发现has-miR-206可在多种肿瘤组织与正常组织间有差异性表达。Zhou等[20]研究发现,与邻近的正常组织相比,miR-206在乳腺癌细胞系和乳腺癌组织中的表达上调,而全长Neurokinin-1的表达与肿瘤淋巴结转移(TNM)阶段和淋巴结转移呈负相关,miR-206结合全长神经激肽-1信使RNA的3'-非翻译区,调节蛋白质表达。与正常邻近组织相比,宫颈癌样品中的miR-206表达明显下调。多变量Cox回归分析显示,miR-206表达降低是整体生存的独立不利预后因素。此外,miR-206模拟物在HeLa细胞中的转染能够减少细胞增殖,促进细胞凋亡并抑制细胞入侵和迁移[21]。Chang等[22]研究指出FOXD2-AS1通过充当miR-206海绵来上调miR-206靶基因膜联蛋白A2(ANXA2)的表达。总之,结论是FOXD2-AS1在HCC中起癌基因的作用,并通过“刺激”miR-206部分上调ANXA2的表达。miR206可以通过调节REST/HDAC4/Sp1/Sp4/BDNF轴来调节MeHg诱导的神经细胞死亡[23]。在宫颈癌中,观察到lncRNA HOTAIR抑制后的迁移和侵袭的表型效应,至少部分是通过抑制Hela细胞miR206表达来调节MKL1[24]。在肺鳞状细胞癌中,miR206通过下调MET和EGFR的mRNA和蛋白水平来抑制EBC-1细胞增殖、迁移和侵袭,另外,ERK1/2和AKT信号转导的磷酸化被癌细胞中的miR-206恢复所抑制[25]。
在动物实验方面,关于miR-206的研究主要集中于miR-206经由TGF-β1/Smad3信号通路影响卫星细胞的增殖和分化来抵抗骨骼肌萎缩[26],miR-206通过靶向Notch3基因来调节骨骼肌细胞的增殖和细胞周期停滞[27],miR-206靶向的AAV有效地下调了miR-206的表达并增加了成熟小鼠肌肉中的内源性治疗基因的表达,且治疗显著改善了小鼠的运动功能和营养不良[28]。而关于miR-206在肿瘤方面的动物模型目前仍较少,这一点可以为将来的研究方向作以参考。
尽管已经有了一些认识,但是我们对miR-206靶基因功能的认识还只是冰山一角,因为miRNAs在不同条件下调控不同细胞的不同靶点发挥不同的作用,这可能是因为同一miRNA能够调控多个靶基因,信号转导通路不同,还可能是因为生物功能的异质性、某种疾病的位置或阶段不同等,从而导致了这种功能上的巨大差异[29-30]。本研究联合采用TargetScan、miRDB、DIANA-microCT3个靶基因预测数据库得到可信度较高的靶基因集合,并对靶基因进行GO功能注释和KEGG信号转导通路富集分析,并进行关键基因筛选,将有助于对miR-206所参与的生物学过程有一个基本的认识,为进一步深入研究其功能奠定了基础。但由于预测靶基因过程中不可避免的存在假阳性率,所以对预测得到的靶基因及其发挥的生物学功能需要进行进一步的实验验证。

